Un virus es un agente infeccioso submicroscópico que se replica solo dentro de las células vivas de un organismo . [1] Los virus infectan todas las formas de vida , desde animales y plantas hasta microorganismos , incluidas bacterias y arqueas . [2] [3] Los virus se encuentran en casi todos los ecosistemas de la Tierra y son el tipo de entidad biológica más numeroso. [4] [5] Desde el artículo de Dmitri Ivanovsky de 1892 que describe un patógeno no bacteriano que infecta las plantas de tabaco y el descubrimiento del virus del mosaico del tabaco por Martinus Beijerinck en 1898, [6] : 4 más de 11.000 de los millones de especies de virus han sido descritas en detalle. [7] [8] El estudio de los virus se conoce como virología , una subespecialidad de la microbiología .
Cuando una célula huésped se infecta, a menudo se ve obligada a producir rápidamente miles de copias del virus original. Cuando no están dentro de una célula infectada o en el proceso de infectar una célula, los virus existen en forma de partículas virales independientes, o viriones , que consisten en (i) material genético , es decir, moléculas largas de ADN o ARN que codifican la estructura de las proteínas mediante las cuales actúa el virus; (ii) una capa proteica , la cápside , que rodea y protege el material genético; y en algunos casos (iii) una envoltura externa de lípidos . Las formas de estas partículas virales varían desde formas helicoidales e icosaédricas simples hasta estructuras más complejas. La mayoría de las especies de virus tienen viriones demasiado pequeños para ser vistos con un microscopio óptico y son una centésima parte del tamaño de la mayoría de las bacterias.
Los orígenes de los virus en la historia evolutiva de la vida aún no están claros. Algunos virus pueden haber evolucionado a partir de plásmidos , que son fragmentos de ADN que pueden moverse entre células. Otros virus pueden haber evolucionado a partir de bacterias. En la evolución, los virus son un medio importante de transferencia horizontal de genes , que aumenta la diversidad genética de una manera análoga a la reproducción sexual . [9] Algunos biólogos consideran que los virus son una forma de vida, porque llevan material genético, se reproducen y evolucionan a través de la selección natural , aunque carecen de algunas características clave, como la estructura celular, que generalmente se consideran criterios necesarios para definir la vida. Debido a que poseen algunas de esas cualidades, pero no todas, los virus han sido descritos como "organismos al borde de la vida" [10] y como replicadores . [11]
Los virus se propagan de muchas maneras. Una vía de transmisión es a través de organismos portadores de enfermedades conocidos como vectores : por ejemplo, los virus a menudo se transmiten de planta a planta por insectos que se alimentan de la savia de las plantas , como los pulgones ; y los virus en animales pueden ser transportados por insectos chupadores de sangre . Muchos virus se propagan en el aire al toser y estornudar, incluidos los virus de la gripe , el SARS-CoV-2 , la varicela , la viruela y el sarampión . El norovirus y el rotavirus , causas comunes de gastroenteritis viral , se transmiten por la vía fecal-oral , a través del contacto mano-boca o en los alimentos o el agua. La dosis infecciosa de norovirus necesaria para producir infección en humanos es inferior a 100 partículas. [12] El VIH es uno de varios virus que se transmiten a través del contacto sexual y por exposición a sangre infectada. La variedad de células hospedadoras que un virus puede infectar se denomina rango de hospedadores : este rango es estrecho para los virus especializados en infectar solo unas pocas especies, o amplio para los virus capaces de infectar a muchas. [13] : 123–124
Las infecciones virales en animales provocan una respuesta inmunitaria que generalmente elimina el virus infectante. Las respuestas inmunitarias también pueden producirse mediante vacunas , que confieren una inmunidad adquirida artificialmente a la infección viral específica. Algunos virus, incluidos los que causan el VIH/SIDA , la infección por VPH y la hepatitis viral , evaden estas respuestas inmunitarias y dan lugar a infecciones crónicas . Se han desarrollado varias clases de medicamentos antivirales .
La palabra inglesa «virus» proviene del latín vīrus , que se refiere a veneno y otros líquidos nocivos. Vīrus proviene de la misma raíz indoeuropea que el sánscrito viṣa , el avéstico vīša y el griego antiguo ἰός ( iós ), que significan «veneno». El primer uso atestiguado de «virus» en inglés apareció en 1398 en la traducción de John Trevisa de De Proprietatibus Rerum de Bartholomeus Anglicus . [14] [15] Virulento , del latín virulentus ('venenoso'), data de alrededor de 1400 . [16] [17] El significado de 'agente que causa enfermedad infecciosa' se registró por primera vez en 1728, [15] mucho antes del descubrimiento de los virus por Dmitri Ivanovsky en 1892. El plural en inglés es viruses (a veces también vira ), [18] mientras que la palabra latina es un sustantivo colectivo , que no tiene un plural atestiguado clásicamente ( vīra se usa en neolatín [19] ). El adjetivo viral data de 1948. [20] El término virión (plural viriones ), que data de 1959, [21] también se usa para referirse a una única partícula viral que se libera de la célula y es capaz de infectar otras células del mismo tipo. [22]
Los virus se encuentran dondequiera que haya vida y probablemente han existido desde que las células vivas evolucionaron por primera vez . [23] El origen de los virus no está claro porque no forman fósiles, por lo que se utilizan técnicas moleculares para inferir cómo surgieron. [24] Además, el material genético viral ocasionalmente se integra en la línea germinal de los organismos hospedadores, por lo que pueden transmitirse verticalmente a la descendencia del hospedador durante muchas generaciones. Esto proporciona una fuente de información invaluable para que los paleovirólogos rastreen virus antiguos que existieron hace millones de años.
Hay tres hipótesis principales que pretenden explicar el origen de los virus: [25]
En el pasado, todas estas hipótesis planteaban problemas: la hipótesis regresiva no explicaba por qué incluso los parásitos celulares más pequeños no se parecen en nada a los virus. La hipótesis del escape no explicaba las complejas cápsides y otras estructuras de las partículas virales. La hipótesis del virus primero contradecía la definición de virus en el sentido de que requieren células hospedadoras. [27] : 24 Ahora se reconoce que los virus son antiguos y que tienen orígenes anteriores a la divergencia de la vida en los tres dominios . [27] : 28 Este descubrimiento ha llevado a los virólogos modernos a reconsiderar y reevaluar estas tres hipótesis clásicas. [27] : 28
La evidencia de un mundo ancestral de células de ARN [27] : 26 y el análisis informático de secuencias de ADN viral y del huésped proporcionan una mejor comprensión de las relaciones evolutivas entre diferentes virus y pueden ayudar a identificar a los ancestros de los virus modernos. Hasta la fecha, dichos análisis no han demostrado cuál de estas hipótesis es correcta. [27] : 26 Parece poco probable que todos los virus conocidos actualmente tengan un ancestro común, y es probable que los virus hayan surgido numerosas veces en el pasado mediante uno o más mecanismos. [31]
La primera evidencia de la existencia de virus provino de experimentos con filtros que tenían poros lo suficientemente pequeños como para retener bacterias. En 1892, Dmitri Ivanovsky utilizó uno de estos filtros para demostrar que la savia de una planta de tabaco enferma seguía siendo infecciosa para las plantas de tabaco sanas a pesar de haber sido filtrada. Martinus Beijerinck llamó a la sustancia infecciosa filtrada un "virus" y este descubrimiento se considera el comienzo de la virología. El posterior descubrimiento y caracterización parcial de los bacteriófagos por Frederick Twort y Félix d'Herelle catalizaron aún más el campo, y a principios del siglo XX se habían descubierto muchos virus. En 1926, Thomas Milton Rivers definió los virus como parásitos obligados. Wendell Meredith Stanley demostró que los virus eran partículas, en lugar de un fluido, y la invención del microscopio electrónico en 1931 permitió visualizar sus complejas estructuras. [32]
Las opiniones científicas difieren sobre si los virus son una forma de vida o estructuras orgánicas que interactúan con organismos vivos. [11] Se les ha descrito como "organismos al borde de la vida", [10] ya que se parecen a los organismos en que poseen genes , evolucionan por selección natural , [33] y se reproducen creando múltiples copias de sí mismos a través del autoensamblaje. Aunque tienen genes, no tienen una estructura celular, que a menudo se considera la unidad básica de la vida. Los virus no tienen su propio metabolismo y requieren una célula huésped para fabricar nuevos productos. Por lo tanto, no pueden reproducirse naturalmente fuera de una célula huésped [34] —aunque algunas bacterias como la rickettsia y la clamidia se consideran organismos vivos a pesar de la misma limitación. [35] [36] Las formas de vida aceptadas utilizan la división celular para reproducirse, mientras que los virus se ensamblan espontáneamente dentro de las células. Se diferencian del crecimiento autónomo de los cristales ya que heredan mutaciones genéticas mientras están sujetos a la selección natural. El autoensamblaje de virus dentro de las células huésped tiene implicaciones para el estudio del origen de la vida , ya que da más credibilidad a la hipótesis de que la vida podría haber comenzado como moléculas orgánicas autoensambladas . [2] El modelo de virocélula propuesto por primera vez por Patrick Forterre considera que la célula infectada es la "forma viva" de los virus y que las partículas virales (viriones) son análogas a las esporas . [37] Aunque el debate sobre lo vivo versus lo no vivo continúa, el modelo de virocélula ha ganado cierta aceptación. [38]
Los virus muestran una amplia diversidad de tamaños y formas, llamadas " morfologías ". En general, los virus son mucho más pequeños que las bacterias y más de mil virus bacteriófagos cabrían dentro de la célula de una bacteria Escherichia coli . [39] : 98 Muchos virus que se han estudiado son esféricos y tienen un diámetro entre 20 y 300 nanómetros . Algunos filovirus , que son filamentos, tienen una longitud total de hasta 1400 nm; sus diámetros son solo de unos 80 nm. [26] : 33–55 La mayoría de los virus no se pueden ver con un microscopio óptico , por lo que se utilizan microscopios electrónicos de barrido y transmisión para visualizarlos. [26] : 33–37 Para aumentar el contraste entre los virus y el fondo, se utilizan "tinciones" electrodensas. Estas son soluciones de sales de metales pesados, como el tungsteno , que dispersan los electrones de las regiones cubiertas con la tinción. Cuando los viriones están recubiertos con una tinción (tinción positiva), los detalles finos se oscurecen. La tinción negativa supera este problema al teñir solo el fondo. [40]
Una partícula viral completa, conocida como virión , consiste en ácido nucleico rodeado por una capa protectora de proteína llamada cápside . Estas se forman a partir de subunidades proteicas llamadas capsómeros . [26] : 40 Los virus pueden tener una "envoltura" lipídica derivada de la membrana de la célula huésped . La cápside está hecha de proteínas codificadas por el genoma viral y su forma sirve como base para la distinción morfológica. [41] [42] Las subunidades proteicas codificadas por el virus se autoensamblan para formar una cápside, lo que en general requiere la presencia del genoma del virus. Los virus complejos codifican proteínas que ayudan en la construcción de su cápside. Las proteínas asociadas con el ácido nucleico se conocen como nucleoproteínas , y la asociación de las proteínas de la cápside viral con el ácido nucleico viral se llama nucleocápside. La cápside y la estructura completa del virus se pueden sondear mecánicamente (físicamente) a través de microscopía de fuerza atómica . [43] [44] En general, hay cinco tipos principales de virus morfológicos:
Los poxvirus son virus grandes y complejos que tienen una morfología inusual. El genoma viral está asociado con proteínas dentro de una estructura de disco central conocida como nucleoide . El nucleoide está rodeado por una membrana y dos cuerpos laterales de función desconocida. El virus tiene una envoltura externa con una capa gruesa de proteína tachonada sobre su superficie. Todo el virión es ligeramente pleomórfico , con una forma que va desde ovoide hasta ladrillo. [50]
El Mimivirus es uno de los virus más grandes que se han caracterizado, con un diámetro de cápside de 400 nm. Los filamentos de proteína que miden 100 nm se proyectan desde la superficie. La cápside parece hexagonal bajo un microscopio electrónico, por lo tanto, la cápside es probablemente icosaédrica. [51] En 2011, los investigadores descubrieron el virus más grande conocido hasta entonces en muestras de agua recolectadas del fondo del océano frente a la costa de Las Cruces, Chile. Nombrado provisionalmente Megavirus chilensis , se puede ver con un microscopio óptico básico. [52] En 2013, se descubrió el género Pandoravirus en Chile y Australia, y tiene genomas aproximadamente el doble de grandes que Megavirus y Mimivirus. [53] Todos los virus gigantes tienen genomas de dsADN y se clasifican en varias familias: Mimiviridae , Pithoviridae, Pandoraviridae , Phycodnaviridae y el género Mollivirus . [54]
Algunos virus que infectan a las arqueas tienen estructuras complejas que no tienen relación con ninguna otra forma de virus, con una amplia variedad de formas inusuales, que van desde estructuras fusiformes hasta virus que se parecen a bastones con forma de gancho, lágrimas o incluso botellas. Otros virus arqueas se parecen a los bacteriófagos con cola y pueden tener múltiples estructuras en la cola. [55]
Se puede observar una enorme variedad de estructuras genómicas entre las especies virales ; como grupo, contienen más diversidad genómica estructural que las plantas, los animales, las arqueas o las bacterias. Hay millones de tipos diferentes de virus, [8] aunque se han descrito en detalle menos de 7000 tipos. [6] : 49 A enero de 2021, la base de datos del genoma del virus del NCBI tiene más de 193 000 secuencias genómicas completas, [56] pero sin duda hay muchas más por descubrir. [57] [58]
Un virus tiene un genoma de ADN o de ARN y se denomina virus de ADN o de ARN , respectivamente. La mayoría de los virus tienen genomas de ARN. Los virus de plantas tienden a tener genomas de ARN monocatenario y los bacteriófagos tienden a tener genomas de ADN bicatenario. [26] : 96–99
Los genomas virales son circulares, como en los poliomavirus , o lineales, como en los adenovirus . El tipo de ácido nucleico es irrelevante para la forma del genoma. Entre los virus de ARN y ciertos virus de ADN, el genoma a menudo se divide en partes separadas, en cuyo caso se llama segmentado. Para los virus de ARN, cada segmento a menudo codifica solo una proteína y generalmente se encuentran juntos en una cápside. No se requiere que todos los segmentos estén en el mismo virión para que el virus sea infeccioso, como lo demuestra el virus del mosaico del bromo y varios otros virus de plantas. [26] : 33–35
Un genoma viral, independientemente del tipo de ácido nucleico, es casi siempre monocatenario (ss) o bicatenario (ds). Los genomas monocatenarios consisten en un ácido nucleico no apareado, análogo a la mitad de una escalera dividida por la mitad. Los genomas bicatenarios consisten en dos ácidos nucleicos complementarios apareados, análogos a una escalera. Las partículas virales de algunas familias de virus, como las que pertenecen a Hepadnaviridae , contienen un genoma que es parcialmente bicatenario y parcialmente monocatenario. [26] : 96–99
En la mayoría de los virus con genomas de ARN y algunos con genomas de ADN monocatenario (ssDNA), se dice que las hebras simples son de sentido positivo (llamadas "hebra positiva") o de sentido negativo (llamadas "hebra negativa"), dependiendo de si son complementarias al ARN mensajero viral (ARNm). El ARN viral de sentido positivo tiene el mismo sentido que el ARNm viral y, por lo tanto, al menos una parte de él puede ser traducido inmediatamente por la célula huésped. El ARN viral de sentido negativo es complementario al ARNm y, por lo tanto, debe convertirse en ARN de sentido positivo mediante una ARN polimerasa dependiente de ARN antes de la traducción. La nomenclatura del ADN para los virus con ssDNA genómico es similar a la nomenclatura del ARN, en que el ssDNA viral de hebra positiva es idéntico en secuencia al ARNm viral y, por lo tanto, es una hebra codificante, mientras que el ssDNA viral de sentido negativo es complementario al ARNm viral y, por lo tanto, es una hebra molde. [26] : 96–99 Varios tipos de virus ssDNA y ssRNA tienen genomas que son ambisentidos en el sentido de que la transcripción puede ocurrir fuera de ambas cadenas en un intermediario replicativo de doble cadena. Los ejemplos incluyen geminivirus , que son virus de plantas ssDNA y arenavirus , que son virus ssRNA de animales. [59]
El tamaño del genoma varía mucho entre especies. Los más pequeños (los circovirus ssDNA, familia Circoviridae ) codifican solo dos proteínas y tienen un tamaño de genoma de solo dos kilobases; [60] los más grandes (los pandoravirus) tienen tamaños de genoma de alrededor de dos megabases que codifican alrededor de 2500 proteínas. [53] Los genes de los virus rara vez tienen intrones y a menudo están dispuestos en el genoma de manera que se superponen . [61]
En general, los virus de ARN tienen genomas más pequeños que los virus de ADN debido a una mayor tasa de error al replicarse, y tienen un límite de tamaño máximo superior. [24] Más allá de esto, los errores al replicarse hacen que el virus sea inútil o no competitivo. Para compensar, los virus de ARN a menudo tienen genomas segmentados (el genoma se divide en moléculas más pequeñas), lo que reduce la posibilidad de que un error en un genoma de un solo componente incapacite a todo el genoma. En contraste, los virus de ADN generalmente tienen genomas más grandes debido a la alta fidelidad de sus enzimas de replicación. [62] Los virus de ADN de cadena sencilla son una excepción a esta regla, ya que las tasas de mutación de estos genomas pueden acercarse al extremo del caso del virus de ARN monocatenario. [63]

Los virus sufren cambios genéticos por varios mecanismos. Estos incluyen un proceso llamado deriva antigénica donde bases individuales en el ADN o ARN mutan a otras bases. La mayoría de estas mutaciones puntuales son "silenciosas" (no cambian la proteína que el gen codifica), pero otras pueden conferir ventajas evolutivas como la resistencia a los medicamentos antivirales . [64] [65] El cambio antigénico ocurre cuando hay un cambio importante en el genoma del virus. Esto puede ser el resultado de la recombinación o reordenamiento . El virus de la influenza A es muy propenso al reordenamiento; ocasionalmente esto ha dado lugar a nuevas cepas que han causado pandemias . [66] Los virus de ARN a menudo existen como cuasiespecies o enjambres de virus de la misma especie pero con secuencias de nucleósidos genómicos ligeramente diferentes. Estas cuasiespecies son un objetivo principal para la selección natural. [67]
Los genomas segmentados confieren ventajas evolutivas; diferentes cepas de un virus con un genoma segmentado pueden mezclar y combinar genes y producir virus descendientes que tienen características únicas. Esto se denomina reordenamiento o "sexo viral". [68]
La recombinación genética es un proceso por el cual una hebra de ADN (o ARN) se rompe y luego se une al extremo de una molécula de ADN (o ARN) diferente. Esto puede ocurrir cuando los virus infectan células simultáneamente y los estudios de evolución viral han demostrado que la recombinación ha sido rampante en las especies estudiadas. [69] La recombinación es común tanto a los virus de ARN como a los de ADN. [70] [71]
Los coronavirus tienen un genoma de ARN monocatenario de sentido positivo . La replicación del genoma está catalizada por una ARN polimerasa dependiente de ARN . El mecanismo de recombinación utilizado por los coronavirus probablemente implica un cambio de plantilla por parte de la polimerasa durante la replicación del genoma. [72] Este proceso parece ser una adaptación para hacer frente al daño del genoma. [73]
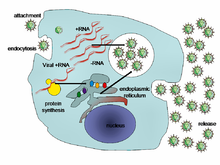
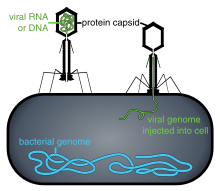
Las poblaciones virales no crecen por división celular, porque son acelulares. En cambio, utilizan la maquinaria y el metabolismo de una célula huésped para producir múltiples copias de sí mismas, y se reúnen en la célula. [74] Cuando se infecta, la célula huésped se ve obligada a producir rápidamente miles de copias del virus original. [75]
Su ciclo de vida difiere mucho entre especies, pero hay seis etapas básicas en su ciclo de vida: [26] : 75–91
La unión es una unión específica entre las proteínas de la cápside viral y receptores específicos en la superficie celular del huésped. Esta especificidad determina el rango de hospedadores y el tipo de célula huésped de un virus. Por ejemplo, el VIH infecta un rango limitado de leucocitos humanos . Esto se debe a que su proteína de superficie, gp120 , interactúa específicamente con la molécula CD4 (un receptor de quimiocina ), que se encuentra más comúnmente en la superficie de las células T CD4+ . Este mecanismo ha evolucionado para favorecer a aquellos virus que infectan solo células en las que son capaces de replicarse. La unión al receptor puede inducir a la proteína de la envoltura viral a sufrir cambios que resultan en la fusión de las membranas virales y celulares, o cambios de las proteínas de superficie del virus sin envoltura que permiten que el virus entre. [76]
La penetración o entrada viral sigue a la unión: los viriones ingresan a la célula huésped a través de endocitosis mediada por receptores o fusión de membranas . La infección de células vegetales y fúngicas es diferente a la de las células animales. Las plantas tienen una pared celular rígida hecha de celulosa y los hongos una de quitina, por lo que la mayoría de los virus pueden ingresar a estas células solo después de un traumatismo en la pared celular. [6] : 70 Casi todos los virus vegetales (como el virus del mosaico del tabaco) también pueden moverse directamente de una célula a otra, en forma de complejos de nucleoproteínas monocatenarias, a través de poros llamados plasmodesmos . [77] Las bacterias, como las plantas, tienen paredes celulares fuertes que un virus debe atravesar para infectar la célula. Dado que las paredes celulares bacterianas son mucho más delgadas que las paredes celulares vegetales debido a su tamaño mucho más pequeño, algunos virus han desarrollado mecanismos que inyectan su genoma en la célula bacteriana a través de la pared celular, mientras que la cápside viral permanece afuera. [6] : 71
La desprotección es un proceso en el cual se elimina la cápside viral: esto puede ser por degradación por enzimas virales o enzimas del huésped o por simple disociación; el resultado final es la liberación del ácido nucleico genómico viral. [78]
La replicación de los virus implica principalmente la multiplicación del genoma. La replicación implica la síntesis del ARN mensajero viral (ARNm) a partir de genes "tempranos" (con excepción de los virus de ARN de sentido positivo), la síntesis de proteínas virales , el posible ensamblaje de proteínas virales y luego la replicación del genoma viral mediada por la expresión de proteínas tempranas o reguladoras. Esto puede ser seguido, para virus complejos con genomas más grandes, por una o más rondas adicionales de síntesis de ARNm: la expresión génica "tardía" es, en general, de proteínas estructurales o virionales. [79]
Ensamblaje – Después del autoensamblaje mediado por la estructura de las partículas virales, a menudo se produce alguna modificación de las proteínas. En virus como el VIH, esta modificación (a veces llamada maduración) ocurre después de que el virus se haya liberado de la célula huésped. [80]
Liberación – Los virus pueden liberarse de la célula huésped por lisis , un proceso que mata la célula al estallar su membrana y pared celular si está presente: esta es una característica de muchos virus bacterianos y algunos animales. Algunos virus experimentan un ciclo lisogénico donde el genoma viral se incorpora por recombinación genética en un lugar específico en el cromosoma del huésped. El genoma viral se conoce entonces como un " provirus " o, en el caso de los bacteriófagos, un " profago ". [13] : 836 Siempre que el huésped se divide, el genoma viral también se replica. El genoma viral es mayormente silencioso dentro del huésped. En algún momento, el provirus o el profago pueden dar lugar al virus activo, que puede lisar las células huésped. [6] : 243–259 Los virus envueltos (por ejemplo, el VIH) generalmente se liberan de la célula huésped por gemación . Durante este proceso, el virus adquiere su envoltura, que es una pieza modificada del plasma del huésped u otra membrana interna. [6] : 185–187
El material genético dentro de las partículas virales y el método mediante el cual se replica dicho material varían considerablemente entre los diferentes tipos de virus.
La gama de efectos estructurales y bioquímicos que los virus tienen sobre la célula huésped es extensa. [26] : 115–146 Estos se llaman ' efectos citopáticos '. [26] : 115 La mayoría de las infecciones virales finalmente resultan en la muerte de la célula huésped. Las causas de muerte incluyen lisis celular, alteraciones en la membrana superficial de la célula y apoptosis . [82] A menudo, la muerte celular es causada por el cese de sus actividades normales debido a la supresión por proteínas específicas del virus, no todas las cuales son componentes de la partícula viral. [83] La distinción entre citopático e inofensivo es gradual. Algunos virus, como el virus de Epstein-Barr , pueden hacer que las células proliferen sin causar malignidad, [84] mientras que otros, como los virus del papiloma , son causas establecidas de cáncer. [85]
Algunos virus no provocan cambios aparentes en la célula infectada. Las células en las que el virus está latente e inactivo muestran pocos signos de infección y suelen funcionar con normalidad. [86] Esto provoca infecciones persistentes y el virus suele permanecer inactivo durante muchos meses o años. Este suele ser el caso de los virus del herpes . [87] [88]
Los virus son, con mucho, las entidades biológicas más abundantes en la Tierra y superan en número a todas las demás juntas. [89] Infectan todos los tipos de vida celular, incluidos animales, plantas, bacterias y hongos . [6] : 49 Los diferentes tipos de virus pueden infectar solo un rango limitado de huéspedes y muchos son específicos de la especie. Algunos, como el virus de la viruela , por ejemplo, pueden infectar solo una especie, en este caso los humanos, [13] : 643 y se dice que tienen un rango estrecho de huéspedes . Otros virus, como el virus de la rabia, pueden infectar diferentes especies de mamíferos y se dice que tienen un rango amplio. [13] : 631 Los virus que infectan plantas son inofensivos para los animales, y la mayoría de los virus que infectan a otros animales son inofensivos para los humanos. [6] : 272 El rango de huéspedes de algunos bacteriófagos está limitado a una sola cepa de bacterias y se pueden usar para rastrear la fuente de brotes de infecciones mediante un método llamado tipificación de fagos . [90] El conjunto completo de virus en un organismo o hábitat se llama viroma ; por ejemplo, todos los virus humanos constituyen el viroma humano . [91]
Un virus nuevo es aquel que no ha sido registrado previamente. Puede ser un virus que se aísla de su reservorio natural o que se aísla como resultado de la propagación a un huésped animal o humano donde el virus no había sido identificado antes. Puede ser un virus emergente , uno que representa un virus nuevo, pero también puede ser un virus existente que no ha sido identificado previamente . [92] El coronavirus SARS-CoV-2 que causó la pandemia de COVID-19 es un ejemplo de un virus nuevo. [93]
La clasificación busca describir la diversidad de virus nombrándolos y agrupándolos sobre la base de similitudes. En 1962, André Lwoff , Robert Horne y Paul Tournier fueron los primeros en desarrollar un medio de clasificación de virus, basado en el sistema jerárquico de Linneo . [94] Este sistema basaba la clasificación en filo , clase , orden , familia , género y especie . Los virus se agrupaban según sus propiedades compartidas (no las de sus huéspedes) y el tipo de ácido nucleico que formaba sus genomas. [95] En 1966, se formó el Comité Internacional de Taxonomía de Virus (ICTV). El sistema propuesto por Lwoff, Horne y Tournier inicialmente no fue aceptado por el ICTV porque el pequeño tamaño del genoma de los virus y su alta tasa de mutación dificultaban determinar su ascendencia más allá del orden. Como tal, el sistema de clasificación de Baltimore ha llegado a utilizarse para complementar la jerarquía más tradicional. [96] A partir de 2018, el ICTV comenzó a reconocer relaciones evolutivas más profundas entre los virus que se han descubierto con el tiempo y adoptó un sistema de clasificación de 15 rangos que van desde el reino hasta la especie. [97] Además, algunas especies dentro del mismo género se agrupan en un genogrupo . [98] [99]
El ICTV desarrolló el sistema de clasificación actual y redactó pautas que dan mayor importancia a ciertas propiedades de los virus para mantener la uniformidad familiar. Se ha establecido una taxonomía unificada (un sistema universal para clasificar los virus). [100] Solo se ha estudiado una pequeña parte de la diversidad total de virus. [101] En 2022, el ICTV ha definido 6 reinos, 10 reinos, 17 filos, 2 subfilos, 40 clases, 72 órdenes, 8 subórdenes, 264 familias, 182 subfamilias , 2818 géneros, 84 subgéneros y 11 273 especies de virus. [7]
A continuación se muestra la estructura taxonómica general de los rangos de taxones y los sufijos utilizados en los nombres taxonómicos. A partir de 2022, los rangos de subreino, subreino y subclase no se utilizan, mientras que todos los demás rangos están en uso. [7]

El biólogo ganador del Premio Nobel David Baltimore ideó el sistema de clasificación de Baltimore . [102] [103] El sistema de clasificación ICTV se utiliza junto con el sistema de clasificación de Baltimore en la clasificación moderna de virus. [104] [105] [106]
La clasificación de Baltimore de los virus se basa en el mecanismo de producción del ARNm . Los virus deben generar ARNm a partir de sus genomas para producir proteínas y replicarse, pero se utilizan diferentes mecanismos para lograrlo en cada familia de virus. Los genomas virales pueden ser monocatenarios (ss) o bicatenarios (ds), de ARN o ADN, y pueden utilizar o no la transcriptasa inversa (RT). Además, los virus ssRNA pueden ser sentido (+) o antisentido (−). Esta clasificación coloca a los virus en siete grupos:

Entre los ejemplos de enfermedades humanas comunes causadas por virus se incluyen el resfriado común , la gripe , la varicela y el herpes labial . Muchas enfermedades graves, como la rabia , la enfermedad del virus del Ébola , el SIDA (VIH) , la gripe aviar y el SARS , son causadas por virus. La capacidad relativa de los virus para causar enfermedades se describe en términos de virulencia . Se están investigando otras enfermedades para descubrir si tienen un virus como agente causal, como la posible conexión entre el herpesvirus humano 6 (HHV6) y enfermedades neurológicas como la esclerosis múltiple y el síndrome de fatiga crónica . [108] Existe controversia sobre si el bornavirus , que anteriormente se creía que causaba enfermedades neurológicas en caballos, podría ser responsable de enfermedades psiquiátricas en humanos. [109]
Los virus tienen diferentes mecanismos por los cuales producen enfermedades en un organismo, lo que depende en gran medida de la especie viral. Los mecanismos a nivel celular incluyen principalmente la lisis celular, la ruptura y posterior muerte de la célula. En los organismos multicelulares , si mueren suficientes células, todo el organismo comenzará a sufrir los efectos. Aunque los virus causan la alteración de la homeostasis saludable , lo que resulta en enfermedad, pueden existir relativamente inofensivamente dentro de un organismo. Un ejemplo incluiría la capacidad del virus del herpes simple , que causa herpes labial, de permanecer en un estado latente dentro del cuerpo humano. Esto se llama latencia [110] y es una característica de los virus del herpes, incluido el virus de Epstein-Barr, que causa fiebre glandular, y el virus de la varicela zóster , que causa varicela y herpes zóster . La mayoría de las personas han sido infectadas con al menos uno de estos tipos de virus del herpes. [111] Estos virus latentes a veces pueden ser beneficiosos, ya que la presencia del virus puede aumentar la inmunidad contra patógenos bacterianos, como Yersinia pestis . [112]
Algunos virus pueden causar infecciones crónicas o de por vida , en las que los virus continúan replicándose en el cuerpo a pesar de los mecanismos de defensa del huésped. [113] Esto es común en las infecciones por el virus de la hepatitis B y el virus de la hepatitis C. Las personas infectadas crónicamente se conocen como portadores, ya que sirven como reservorios del virus infeccioso. [114] En poblaciones con una alta proporción de portadores, se dice que la enfermedad es endémica . [115]
La epidemiología viral es la rama de la ciencia médica que estudia la transmisión y el control de las infecciones virales en los seres humanos. La transmisión de los virus puede ser vertical, es decir, de madre a hijo, u horizontal, es decir, de persona a persona. Entre los ejemplos de transmisión vertical se incluyen el virus de la hepatitis B y el VIH, en los que el bebé nace ya infectado con el virus. [116] Otro ejemplo, más raro, es el virus de la varicela zóster , que, aunque causa infecciones relativamente leves en niños y adultos, puede ser mortal para el feto y el recién nacido. [117]
La transmisión horizontal es el mecanismo más común de propagación de virus en poblaciones. [118] La transmisión horizontal puede ocurrir cuando se intercambian fluidos corporales durante la actividad sexual, por intercambio de saliva o cuando se ingieren alimentos o agua contaminados. También puede ocurrir cuando se inhalan aerosoles que contienen virus o por vectores de insectos , como cuando los mosquitos infectados penetran la piel de un huésped. [118] La mayoría de los tipos de virus se limitan a solo uno o dos de estos mecanismos y se los conoce como "virus respiratorios" o "virus entéricos", etc. La tasa o velocidad de transmisión de infecciones virales depende de factores que incluyen la densidad de población, el número de individuos susceptibles (es decir, aquellos que no son inmunes), [119] la calidad de la atención médica y el clima. [120]
La epidemiología se utiliza para romper la cadena de infección en las poblaciones durante los brotes de enfermedades virales . [13] : 264 Se utilizan medidas de control que se basan en el conocimiento de cómo se transmite el virus. Es importante encontrar la fuente o las fuentes del brote e identificar el virus. Una vez que se ha identificado el virus, la cadena de transmisión a veces se puede romper con vacunas. Cuando las vacunas no están disponibles, el saneamiento y la desinfección pueden ser eficaces. A menudo, las personas infectadas se aíslan del resto de la comunidad, y las que han estado expuestas al virus se ponen en cuarentena . [13] : 894 Para controlar el brote de fiebre aftosa en el ganado en Gran Bretaña en 2001, se sacrificaron miles de cabezas de ganado. [121] La mayoría de las infecciones virales de humanos y otros animales tienen períodos de incubación durante los cuales la infección no causa signos ni síntomas. [13] : 170 Los períodos de incubación de las enfermedades virales varían de unos pocos días a semanas, pero se conocen para la mayoría de las infecciones. [13] : 170–172 Un período de transmisibilidad, que se superpone un poco, pero que se produce principalmente después del período de incubación, es decir, un período en el que un individuo o animal infectado es contagioso y puede infectar a otra persona o animal. [13] : 170–172 Esto también se conoce en el caso de muchas infecciones virales, y el conocimiento de la duración de ambos períodos es importante para controlar los brotes. [13] : 272 Cuando los brotes causan una proporción inusualmente alta de casos en una población, comunidad o región, se denominan epidemias. Si los brotes se propagan por todo el mundo, se denominan pandemias . [13] : 891

Una pandemia es una epidemia mundial . La pandemia de gripe de 1918 , que duró hasta 1919, fue una pandemia de influenza de categoría 5 causada por un virus de influenza A inusualmente grave y mortal. Las víctimas fueron a menudo adultos jóvenes sanos, en contraste con la mayoría de los brotes de influenza, que afectan predominantemente a pacientes jóvenes, ancianos o debilitados de otra manera. [26] : 409–415 Estimaciones más antiguas dicen que mató a 40-50 millones de personas, [122] mientras que investigaciones más recientes sugieren que puede haber matado hasta 100 millones de personas, o el 5% de la población mundial en 1918. [123]
Aunque las pandemias virales son eventos raros, el VIH, que evolucionó a partir de virus encontrados en monos y chimpancés, ha sido una pandemia desde al menos la década de 1980. [124] Durante el siglo XX hubo cuatro pandemias causadas por el virus de la influenza y las que ocurrieron en 1918, 1957 y 1968 fueron graves. [125] La mayoría de los investigadores creen que el VIH se originó en África subsahariana durante el siglo XX; [126] ahora es una pandemia, con un estimado de 37,9 millones de personas viviendo ahora con la enfermedad en todo el mundo. [127] Hubo alrededor de 770.000 muertes por SIDA en 2018. [128] El Programa Conjunto de las Naciones Unidas sobre el VIH/SIDA (ONUSIDA) y la Organización Mundial de la Salud (OMS) estiman que el SIDA ha matado a más de 25 millones de personas desde que fue reconocido por primera vez el 5 de junio de 1981, lo que lo convierte en una de las epidemias más destructivas de la historia registrada. [129] En 2007 hubo 2,7 millones de nuevas infecciones por VIH y 2 millones de muertes relacionadas con el VIH. [130]
Varios patógenos virales altamente letales son miembros de Filoviridae . Los filovirus son virus similares a filamentos que causan fiebre hemorrágica viral , e incluyen ebolavirus y marburgvirus . El virus de Marburgo , descubierto por primera vez en 1967, atrajo la atención generalizada de la prensa en abril de 2005 por un brote en Angola . [131] La enfermedad del virus del Ébola también ha causado brotes intermitentes con altas tasas de mortalidad desde 1976, cuando se identificó por primera vez. El peor y más reciente es la epidemia de África occidental de 2013-2016 . [132]
A excepción de la viruela, la mayoría de las pandemias son causadas por virus de reciente evolución. Estos virus "emergentes" suelen ser mutantes de virus menos dañinos que han circulado previamente en humanos o en otros animales. [133]
El síndrome respiratorio agudo severo ( SARS ) y el síndrome respiratorio de Oriente Medio (MERS) son causados por nuevos tipos de coronavirus . Se sabe que otros coronavirus causan infecciones leves en humanos, [134] por lo que la virulencia y la rápida propagación de las infecciones por SARS (que en julio de 2003 habían causado alrededor de 8000 casos y 800 muertes) fue inesperada y la mayoría de los países no estaban preparados. [135]
Un coronavirus relacionado, el coronavirus 2 del síndrome respiratorio agudo severo (SARS-Cov-2) , que se cree que se originó en murciélagos, surgió en Wuhan , China, en noviembre de 2019 y se propagó rápidamente por todo el mundo. Las infecciones con el virus causaron la pandemia de COVID-19 que comenzó en 2020. [93] [136] [137] Se impusieron restricciones sin precedentes en tiempos de paz a los viajes internacionales, [138] y se impusieron toques de queda en varias ciudades importantes del mundo en respuesta a la pandemia. [139]
Los virus son una causa establecida de cáncer en humanos y otras especies. Los cánceres virales ocurren solo en una minoría de personas infectadas (o animales). Los virus del cáncer provienen de una variedad de familias de virus, incluidos los virus de ARN y ADN, y por lo tanto no hay un solo tipo de " oncovirus " (un término obsoleto utilizado originalmente para retrovirus de transformación aguda). El desarrollo del cáncer está determinado por una variedad de factores como la inmunidad del huésped [140] y mutaciones en el huésped. [141] Los virus aceptados como causantes de cánceres humanos incluyen algunos genotipos del virus del papiloma humano , el virus de la hepatitis B , el virus de la hepatitis C , el virus de Epstein-Barr , el herpesvirus asociado al sarcoma de Kaposi y el virus linfotrópico T humano . El virus del cáncer humano descubierto más recientemente es un poliomavirus ( poliomavirus de células de Merkel ) que causa la mayoría de los casos de una forma rara de cáncer de piel llamado carcinoma de células de Merkel . [142] Los virus de la hepatitis pueden convertirse en una infección viral crónica que conduce al cáncer de hígado . [143] [144] La infección por el virus linfotrópico T humano puede provocar paraparesia espástica tropical y leucemia de células T adultas . [145] Los virus del papiloma humano son una causa establecida de cánceres de cuello uterino , piel, ano y pene . [146] Dentro de Herpesviridae , el herpesvirus asociado al sarcoma de Kaposi causa el sarcoma de Kaposi y el linfoma de cavidad corporal , y el virus de Epstein-Barr causa el linfoma de Burkitt , el linfoma de Hodgkin , el trastorno linfoproliferativo B y el carcinoma nasofaríngeo . [147] El poliomavirus de células de Merkel está estrechamente relacionado con el SV40 y los poliomavirus de ratón que se han utilizado como modelos animales para virus del cáncer durante más de 50 años. [148]
La primera línea de defensa del organismo contra los virus es el sistema inmunitario innato , que está formado por células y otros mecanismos que defienden al huésped de las infecciones de forma no específica. Esto significa que las células del sistema innato reconocen y responden a los patógenos de forma genérica, pero, a diferencia del sistema inmunitario adaptativo , no confieren inmunidad duradera o protectora al huésped. [149]
La interferencia del ARN es una importante defensa innata contra los virus. [150] Muchos virus tienen una estrategia de replicación que implica ARN de doble cadena (dsRNA). Cuando un virus de este tipo infecta una célula, libera su molécula o moléculas de ARN, que se unen inmediatamente a un complejo proteico llamado dicer que corta el ARN en pedazos más pequeños. Se activa una vía bioquímica (el complejo RISC ), que asegura la supervivencia celular al degradar el ARNm viral. Los rotavirus han evolucionado para evitar este mecanismo de defensa al no desenvolverse completamente dentro de la célula y liberar el ARNm recién producido a través de poros en la cápside interna de la partícula. Su dsRNA genómico permanece protegido dentro del núcleo del virión. [151] [152]
Cuando el sistema inmunitario adaptativo de un vertebrado se encuentra con un virus, produce anticuerpos específicos que se unen al virus y, a menudo, lo vuelven no infeccioso. Esto se llama inmunidad humoral . Hay dos tipos de anticuerpos importantes. El primero, llamado IgM , es muy eficaz para neutralizar los virus, pero las células del sistema inmunitario lo producen solo durante unas pocas semanas. El segundo, llamado IgG , se produce indefinidamente. La presencia de IgM en la sangre del huésped se utiliza para detectar una infección aguda, mientras que la IgG indica una infección en algún momento del pasado. [153] El anticuerpo IgG se mide cuando se realizan pruebas de inmunidad . [154]
Los anticuerpos pueden seguir siendo un mecanismo de defensa eficaz incluso después de que los virus hayan logrado entrar en la célula huésped. Una proteína presente en las células, llamada TRIM21 , puede unirse a los anticuerpos en la superficie de la partícula viral. Esto prepara la posterior destrucción del virus por las enzimas del sistema proteosomal de la célula. [155]

Una segunda defensa de los vertebrados contra los virus se denomina inmunidad mediada por células e implica a las células inmunes conocidas como células T. Las células del cuerpo muestran constantemente fragmentos cortos de sus proteínas en la superficie celular y, si una célula T reconoce un fragmento viral sospechoso allí, la célula huésped es destruida por las células "T asesinas" y las células T específicas del virus proliferan. Células como el macrófago son especialistas en esta presentación de antígenos . [156] La producción de interferón es un importante mecanismo de defensa del huésped. Se trata de una hormona producida por el cuerpo cuando hay virus. Su papel en la inmunidad es complejo; finalmente detiene la reproducción de los virus al matar la célula infectada y sus vecinas cercanas. [157]
No todas las infecciones virales producen una respuesta inmunitaria protectora de esta manera. El VIH evade el sistema inmunitario cambiando constantemente la secuencia de aminoácidos de las proteínas en la superficie del virión. Esto se conoce como "mutación de escape", ya que los epítopos virales escapan al reconocimiento por parte de la respuesta inmunitaria del huésped. Estos virus persistentes evaden el control inmunitario mediante secuestro, bloqueo de la presentación de antígenos , resistencia a las citocinas , evasión de las actividades de las células asesinas naturales , escape de la apoptosis y cambio antigénico . [158] Otros virus, llamados " virus neurotrópicos ", se diseminan por propagación neuronal donde el sistema inmunitario puede ser incapaz de alcanzarlos debido al privilegio inmunológico . [159]
Dado que los virus utilizan vías metabólicas vitales dentro de las células huésped para replicarse, es difícil eliminarlos sin el uso de medicamentos que causan efectos tóxicos a las células huésped en general. Los enfoques médicos más eficaces para las enfermedades virales son las vacunas para proporcionar inmunidad a la infección y los medicamentos antivirales que interfieren selectivamente con la replicación viral.
La vacunación es una forma barata y eficaz de prevenir las infecciones por virus. Las vacunas se utilizaban para prevenir las infecciones virales mucho antes del descubrimiento de los virus reales. Su uso ha dado como resultado una disminución drástica de la morbilidad (enfermedad) y la mortalidad (muerte) asociadas con infecciones virales como la polio , el sarampión , las paperas y la rubéola . [160] Las infecciones de viruela han sido erradicadas. [161] Hay vacunas disponibles para prevenir más de trece infecciones virales de humanos, [162] y se utilizan más para prevenir infecciones virales de animales. [163] Las vacunas pueden consistir en virus vivos atenuados o muertos, proteínas virales ( antígenos ) o ARN . [164] [165] Las vacunas vivas contienen formas debilitadas del virus, que no causan la enfermedad pero, no obstante, confieren inmunidad. Estos virus se denominan atenuados. Las vacunas vivas pueden ser peligrosas cuando se administran a personas con una inmunidad débil (que se describen como inmunodeprimidas ), porque en estas personas, el virus debilitado puede causar la enfermedad original. [166] Se utilizan técnicas de biotecnología e ingeniería genética para producir vacunas de subunidades. Estas vacunas utilizan solo las proteínas de la cápside del virus. La vacuna contra la hepatitis B es un ejemplo de este tipo de vacuna. [167] Las vacunas de subunidades son seguras para los pacientes inmunodeprimidos porque no pueden causar la enfermedad. [168] La vacuna contra el virus de la fiebre amarilla , una cepa viva atenuada llamada 17D, es probablemente la vacuna más segura y eficaz jamás generada. [169]
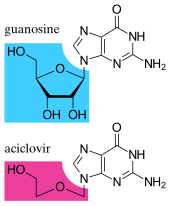
Los fármacos antivirales suelen ser análogos de nucleósidos (bloques de construcción de ADN falsos), que los virus incorporan por error a sus genomas durante la replicación. [170] El ciclo de vida del virus se detiene entonces porque el ADN recién sintetizado está inactivo. Esto se debe a que estos análogos carecen de los grupos hidroxilo , que, junto con los átomos de fósforo , se unen para formar la fuerte "columna vertebral" de la molécula de ADN. Esto se llama terminación de la cadena de ADN . [171] Ejemplos de análogos de nucleósidos son aciclovir para infecciones por el virus del herpes simple y lamivudina para infecciones por VIH y virus de la hepatitis B. El aciclovir es uno de los fármacos antivirales más antiguos y más frecuentemente recetados. [172] Otros fármacos antivirales en uso se dirigen a diferentes etapas del ciclo de vida viral. El VIH depende de una enzima proteolítica llamada proteasa del VIH-1 para que se vuelva completamente infeccioso. Existe una gran clase de fármacos llamados inhibidores de la proteasa que inactivan esta enzima. [173] Existen alrededor de trece clases de medicamentos antivirales, cada uno de los cuales se dirige a diferentes virus o etapas de replicación viral. [170]
La hepatitis C es causada por un virus ARN. En el 80% de las personas infectadas, la enfermedad es crónica y, sin tratamiento, la infección dura el resto de la vida. Existen tratamientos eficaces que utilizan antivirales de acción directa . [174] También se ha desarrollado el tratamiento de los portadores crónicos del virus de la hepatitis B utilizando estrategias similares que incluyen lamivudina y otros fármacos antivirales. [175]
Los virus infectan toda la vida celular y, aunque los virus se encuentran en todo el mundo, cada especie celular tiene su propio rango específico que a menudo infecta sólo a esa especie. [6] : 3 Algunos virus, llamados satélites , pueden replicarse sólo dentro de células que ya han sido infectadas por otro virus. [30]
Los virus son patógenos importantes del ganado. Enfermedades como la fiebre aftosa y la lengua azul son causadas por virus. [176] Los animales de compañía como gatos, perros y caballos, si no están vacunados, son susceptibles a infecciones virales graves. El parvovirus canino es causado por un pequeño virus de ADN y las infecciones suelen ser fatales en las crías. [177] Como todos los invertebrados , la abeja melífera es susceptible a muchas infecciones virales. [178] La mayoría de los virus coexisten sin causar daño en su huésped y no causan signos ni síntomas de enfermedad. [6] : 4

Existen muchos tipos de virus de plantas, pero a menudo sólo causan una pérdida de rendimiento , y no es económicamente viable tratar de controlarlos. Los virus de plantas a menudo se propagan de planta a planta por organismos, conocidos como vectores . Estos suelen ser insectos, pero algunos hongos, gusanos nematodos , organismos unicelulares y plantas parásitas son vectores. [179] Cuando el control de las infecciones por virus de plantas se considera económico, para frutas perennes, por ejemplo, los esfuerzos se concentran en matar a los vectores y eliminar huéspedes alternativos como las malas hierbas. [13] : 802 Los virus de plantas no pueden infectar a los humanos y otros animales porque pueden reproducirse sólo en células vegetales vivas. [13] : 799–807
La papa, originaria de Perú, se ha convertido en un cultivo básico en todo el mundo. [180] El virus Y de la papa causa enfermedades en las papas y especies relacionadas, incluidos los tomates y los pimientos. En la década de 1980, este virus adquirió importancia económica cuando resultó difícil controlarlo en los cultivos de semilla de papa. Este virus, transmitido por pulgones , puede reducir el rendimiento de los cultivos hasta en un 80 por ciento, lo que causa pérdidas significativas en el rendimiento de la papa. [181]
Las plantas tienen mecanismos de defensa elaborados y efectivos contra los virus. Uno de los más efectivos es la presencia de los llamados genes de resistencia (R). Cada gen R confiere resistencia a un virus en particular al desencadenar áreas localizadas de muerte celular alrededor de la célula infectada, que a menudo se pueden ver a simple vista como manchas grandes. Esto impide que la infección se propague. [182] La interferencia del ARN también es una defensa eficaz en las plantas. [13] : 809 > Cuando se infectan, las plantas a menudo producen desinfectantes naturales que matan los virus, como el ácido salicílico , el óxido nítrico y las moléculas reactivas de oxígeno . [183]
Las partículas virales de plantas o partículas similares a virus (VLP) tienen aplicaciones tanto en biotecnología como en nanotecnología . Las cápsides de la mayoría de los virus de plantas son estructuras simples y robustas y pueden producirse en grandes cantidades ya sea por infección de plantas o por expresión en una variedad de sistemas heterólogos. Las partículas virales de plantas pueden modificarse genética y químicamente para encapsular material extraño y pueden incorporarse en estructuras supramoleculares para su uso en biotecnología. [184]

Los bacteriófagos son un grupo común y diverso de virus y son la entidad biológica más abundante en los ambientes acuáticos: hay hasta diez veces más de estos virus en los océanos que bacterias, [185] alcanzando niveles de 250.000.000 de bacteriófagos por mililitro de agua de mar. [186] Estos virus infectan bacterias específicas uniéndose a moléculas receptoras de superficie y luego ingresando a la célula. En un corto período de tiempo, en algunos casos, solo minutos, la polimerasa bacteriana comienza a traducir el ARNm viral en proteínas. Estas proteínas se convierten en nuevos viriones dentro de la célula, proteínas auxiliares, que ayudan al ensamblaje de nuevos viriones, o proteínas involucradas en la lisis celular. Las enzimas virales ayudan en la descomposición de la membrana celular y, en el caso del fago T4 , en poco más de veinte minutos después de la inyección, se podrían liberar más de trescientos fagos. [13] : 834–835
La principal forma en que las bacterias se defienden de los bacteriófagos es mediante la producción de enzimas que destruyen el ADN extraño. Estas enzimas, llamadas endonucleasas de restricción , cortan el ADN viral que los bacteriófagos inyectan en las células bacterianas. [187] Las bacterias también contienen un sistema que utiliza secuencias CRISPR para retener fragmentos de los genomas de los virus con los que las bacterias han estado en contacto previamente, lo que les permite bloquear la replicación del virus a través de una forma de interferencia de ARN . [188] [189] Este sistema genético proporciona a las bacterias inmunidad adquirida a la infección. [190]
Algunos bacteriófagos se denominan " templados " porque causan infecciones latentes y no destruyen inmediatamente sus células huésped. En cambio, su ADN se incorpora al de la célula huésped como profago . Estas infecciones latentes se vuelven productivas cuando el ADN del profago se activa por estímulos como cambios en el medio ambiente. [191] Los intestinos de los animales, incluidos los humanos, contienen bacteriófagos templados, que se activan por diversos estímulos, incluidos los cambios en la dieta y los antibióticos. [192] Aunque se observaron por primera vez en bacteriófagos, se sabe que muchos otros virus forman provirus , incluido el VIH. [191] [193]
Algunos virus se replican dentro de las arqueas : estos son virus de ADN con formas inusuales y a veces únicas. [4] [55] Estos virus han sido estudiados con más detalle en las arqueas termófilas , particularmente en los órdenes Sulfolobales y Thermoproteales . [194] Las defensas contra estos virus involucran la interferencia de ARN de secuencias repetitivas de ADN dentro de los genomas de las arqueas que están relacionadas con los genes de los virus. [195] [196] La mayoría de las arqueas tienen sistemas CRISPR-Cas como defensa adaptativa contra los virus. Estos permiten que las arqueas retengan secciones de ADN viral, que luego se usan para atacar y eliminar infecciones posteriores por el virus usando un proceso similar a la interferencia de ARN. [197]
Los virus son la entidad biológica más abundante en los ambientes acuáticos. [2] Hay alrededor de diez millones de ellos en una cucharadita de agua de mar. [198] La mayoría de estos virus son bacteriófagos que infectan bacterias heterotróficas y cianófagos que infectan cianobacterias y son esenciales para la regulación de los ecosistemas de agua salada y dulce. [199] Los bacteriófagos son inofensivos para las plantas y los animales, y son esenciales para la regulación de los ecosistemas marinos y de agua dulce . [200] son importantes agentes de mortalidad del fitoplancton , la base de la cadena alimentaria en ambientes acuáticos. [201] Infectan y destruyen bacterias en comunidades microbianas acuáticas, y son uno de los mecanismos más importantes de reciclaje del carbono y el ciclo de nutrientes en ambientes marinos. Las moléculas orgánicas liberadas de las células bacterianas muertas estimulan el crecimiento fresco de bacterias y algas, en un proceso conocido como derivación viral . [202] En particular, se ha demostrado que la lisis de bacterias por virus mejora el ciclo del nitrógeno y estimula el crecimiento del fitoplancton. [203] La actividad viral también puede afectar la bomba biológica , el proceso mediante el cual el carbono es secuestrado en las profundidades del océano. [204]
Los microorganismos constituyen más del 90% de la biomasa del mar. Se estima que los virus matan aproximadamente el 20% de esta biomasa cada día y que hay entre 10 y 15 veces más virus en los océanos que bacterias y arqueas. [205] Los virus también son agentes importantes responsables de la destrucción del fitoplancton , incluidas las floraciones de algas nocivas , [206] La cantidad de virus en los océanos disminuye a medida que se alejan de la costa y se adentran más en el agua, donde hay menos organismos hospedadores. [204]
En enero de 2018, los científicos informaron que 800 millones de virus, principalmente de origen marino, se depositan diariamente desde la atmósfera de la Tierra sobre cada metro cuadrado de la superficie del planeta, como resultado de una corriente atmosférica global de virus, que circula por encima del sistema meteorológico pero por debajo de la altitud de los viajes aéreos habituales, distribuyendo virus alrededor del planeta. [207] [208]
Como cualquier organismo, los mamíferos marinos son susceptibles a las infecciones virales. En 1988 y 2002, miles de focas comunes murieron en Europa a causa del virus del moquillo focino . [209] Muchos otros virus, incluidos los calicivirus , los herpesvirus , los adenovirus y los parvovirus , circulan en las poblaciones de mamíferos marinos. [204]
En diciembre de 2022, los científicos informaron sobre la primera observación de virovio a través de un experimento en agua de estanque que contenía clorovirus , que comúnmente infecta a las algas verdes en ambientes de agua dulce. Cuando se eliminaron todas las demás fuentes de alimento microbiano del agua, se observó que el ciliado Halteria había aumentado en número debido al consumo activo de clorovirus como fuente de alimento en lugar de su dieta bacterívora típica . [210] [211]
Los virus son un medio natural importante para transferir genes entre diferentes especies, lo que aumenta la diversidad genética e impulsa la evolución. [9] [212] [213] Se cree que los virus desempeñaron un papel central en la evolución temprana, antes de la diversificación del último ancestro común universal en bacterias, arqueas y eucariotas. [214] Los virus siguen siendo uno de los mayores reservorios de diversidad genética inexplorada en la Tierra. [204]

Los virus son importantes para el estudio de la biología molecular y celular , ya que proporcionan sistemas simples que pueden usarse para manipular e investigar las funciones de las células. [26] : 8 El estudio y uso de virus han proporcionado información valiosa sobre aspectos de la biología celular. [215] Por ejemplo, los virus han sido útiles en el estudio de la genética y han ayudado a nuestra comprensión de los mecanismos básicos de la genética molecular , como la replicación del ADN , la transcripción , el procesamiento del ARN , la traducción , el transporte de proteínas y la inmunología .
Los genetistas suelen utilizar virus como vectores para introducir genes en las células que están estudiando. Esto es útil para hacer que la célula produzca una sustancia extraña o para estudiar el efecto de introducir un nuevo gen en el genoma. De manera similar, la viroterapia utiliza virus como vectores para tratar diversas enfermedades, ya que pueden dirigirse específicamente a las células y al ADN. Muestra un uso prometedor en el tratamiento del cáncer y en la terapia génica . Los científicos de Europa del Este han utilizado la terapia con fagos como una alternativa a los antibióticos durante algún tiempo, y el interés en este enfoque está aumentando, debido al alto nivel de resistencia a los antibióticos que ahora se encuentra en algunas bacterias patógenas. [216] La expresión de proteínas heterólogas por virus es la base de varios procesos de fabricación que se están utilizando actualmente para la producción de varias proteínas, como antígenos de vacunas y anticuerpos. Recientemente se han desarrollado procesos industriales utilizando vectores virales y varias proteínas farmacéuticas se encuentran actualmente en ensayos preclínicos y clínicos. [217]
La viroterapia implica el uso de virus modificados genéticamente para tratar enfermedades. [218] Los científicos han modificado los virus para que se reproduzcan en células cancerosas y las destruyan, pero no infecten las células sanas. Talimogene laherparepvec (T-VEC), por ejemplo, es un virus del herpes simple modificado al que se le ha eliminado un gen, que es necesario para que los virus se repliquen en células sanas, y se lo ha reemplazado por un gen humano ( GM-CSF ) que estimula la inmunidad. Cuando este virus infecta las células cancerosas, las destruye y, al hacerlo, la presencia del gen GM-CSF atrae células dendríticas de los tejidos circundantes del cuerpo. Las células dendríticas procesan las células cancerosas muertas y presentan componentes de ellas a otras células del sistema inmunológico . [219] Tras completar con éxito los ensayos clínicos , el virus obtuvo la aprobación para el tratamiento del melanoma a finales de 2015. [220] Los virus que han sido reprogramados para matar células cancerosas se denominan virus oncolíticos . [221]
Desde el punto de vista de un científico de materiales, los virus pueden considerarse nanopartículas orgánicas . [222] Su superficie lleva herramientas específicas que les permiten cruzar las barreras de sus células anfitrionas. El tamaño y la forma de los virus y el número y la naturaleza de los grupos funcionales en su superficie están definidos con precisión. Como tal, los virus se utilizan comúnmente en la ciencia de los materiales como andamios para modificaciones de superficie enlazadas covalentemente. Una cualidad particular de los virus es que pueden adaptarse mediante evolución dirigida. Las poderosas técnicas desarrolladas por las ciencias de la vida se están convirtiendo en la base de los enfoques de ingeniería hacia los nanomateriales, abriendo una amplia gama de aplicaciones mucho más allá de la biología y la medicina. [223] Debido a su tamaño, forma y estructuras químicas bien definidas, los virus se han utilizado como plantillas para organizar materiales en la nanoescala. Los ejemplos incluyen el trabajo en el Laboratorio de Investigación Naval en Washington, DC, utilizando partículas del virus del mosaico del caupí (CPMV) para amplificar señales en sensores basados en microarrays de ADN . En esta aplicación, las partículas del virus separan los colorantes fluorescentes utilizados para la señalización para evitar la formación de dímeros no fluorescentes que actúan como extintores . [224] Otro ejemplo es el uso de CPMV como una placa de pruebas a nanoescala para la electrónica molecular. [225]
Muchos virus pueden sintetizarse de novo ("desde cero"). El primer virus sintético se creó en 2002. [226] Aunque se trata de una idea un tanto errónea, no es el virus en sí lo que se sintetiza, sino su genoma de ADN (en el caso de un virus de ADN) o una copia de ADNc de su genoma (en el caso de los virus de ARN). Para muchas familias de virus, el ADN o ARN sintético desnudo (una vez convertido enzimáticamente a partir del ADNc sintético) es infeccioso cuando se introduce en una célula. Es decir, contiene toda la información necesaria para producir nuevos virus. Esta tecnología se está utilizando ahora para investigar nuevas estrategias de vacunas. [227] La capacidad de sintetizar virus tiene consecuencias de largo alcance, ya que los virus ya no pueden considerarse extintos, siempre que se conozca la información de su secuencia genómica y haya células permisivas disponibles. En junio de 2021, las secuencias genómicas completas de 11 464 virus diferentes, incluida la viruela, están disponibles públicamente en una base de datos en línea mantenida por los Institutos Nacionales de Salud . [228]
La capacidad de los virus para causar epidemias devastadoras en las sociedades humanas ha generado la preocupación de que los virus pudieran ser utilizados como armas para la guerra biológica . La recreación exitosa del infame virus de la influenza de 1918 en un laboratorio generó más preocupación . [229] El virus de la viruela devastó numerosas sociedades a lo largo de la historia antes de su erradicación. Solo hay dos centros en el mundo autorizados por la OMS para mantener reservas de virus de la viruela: el Centro Estatal de Investigación de Virología y Biotecnología VECTOR en Rusia y los Centros para el Control y la Prevención de Enfermedades en los Estados Unidos. [230] Puede usarse como arma, [230] como la vacuna contra la viruela a veces tenía efectos secundarios graves, ya no se usa rutinariamente en ningún país. Por lo tanto, gran parte de la población humana moderna casi no tiene resistencia establecida a la viruela y sería vulnerable al virus. [230]