
La virología es el estudio científico de los virus biológicos . Es un subcampo de la microbiología que se centra en su detección, estructura, clasificación y evolución, sus métodos de infección y explotación de las células huésped para la reproducción, su interacción con la fisiología e inmunidad del organismo huésped, las enfermedades que causan, las técnicas para aislar y cultivar. ellos y su uso en investigación y terapia.
La identificación del agente causante de la enfermedad del mosaico del tabaco (TMV) como un nuevo patógeno por Martinus Beijerinck (1898) se reconoce ahora como el comienzo oficial del campo de la virología como disciplina distinta de la bacteriología . Se dio cuenta de que la fuente no era una infección bacteriana ni fúngica , sino algo completamente diferente. Beijerinck utilizó la palabra "virus" para describir el misterioso agente en su ' contagium vivum fluidum ' ('fluido vivo contagioso'). Rosalind Franklin propuso la estructura completa del virus del mosaico del tabaco en 1955.
Una motivación principal para el estudio de los virus es que causan muchas enfermedades infecciosas de plantas y animales. [1] El estudio de la manera en que los virus causan enfermedades es la patogénesis viral . El grado en que un virus causa enfermedad es su virulencia . [2] Estos campos de estudio se denominan virología vegetal , virología animal y virología humana o médica . [3]
La virología comenzó cuando no existían métodos para propagar o visualizar virus ni pruebas de laboratorio específicas para infecciones virales. Los métodos para separar los ácidos nucleicos virales ( ARN y ADN ) y las proteínas , que hoy son el pilar de la virología, no existían. Actualmente existen muchos métodos para observar la estructura y funciones de los virus y sus componentes. Actualmente se conocen miles de virus diferentes y los virólogos a menudo se especializan en los virus que infectan a las plantas, a las bacterias y otros microorganismos o a los animales. Los virólogos médicos ahora estudian los virus que infectan a los humanos. La virología es un tema amplio que abarca la biología, la salud, el bienestar animal, la agricultura y la ecología.

Louis Pasteur no pudo encontrar un agente causante de la rabia y especuló sobre un patógeno demasiado pequeño para ser detectado por microscopios. [4] En 1884, el microbiólogo francés Charles Chamberland inventó el filtro Chamberland (o filtro Pasteur-Chamberland) con poros lo suficientemente pequeños como para eliminar todas las bacterias de una solución que pasaba a través de él. [5] En 1892, el biólogo ruso Dmitri Ivanovsky utilizó este filtro para estudiar lo que ahora se conoce como el virus del mosaico del tabaco : los extractos de hojas trituradas de plantas de tabaco infectadas seguían siendo infecciosos incluso después de la filtración para eliminar las bacterias. Ivanovsky sugirió que la infección podría ser causada por una toxina producida por bacterias, pero no siguió adelante con la idea. [6] En ese momento se pensaba que todos los agentes infecciosos podían ser retenidos por filtros y crecer en un medio nutritivo; esto era parte de la teoría de los gérmenes de la enfermedad . [7]
En 1898, el microbiólogo holandés Martinus Beijerinck repitió los experimentos y se convenció de que la solución filtrada contenía una nueva forma de agente infeccioso. [8] Observó que el agente se multiplicaba sólo en células que se estaban dividiendo, pero como sus experimentos no demostraron que estuviera hecho de partículas, lo llamó contagium vivum fluidum (germen vivo soluble) y reintrodujo la palabra virus . Beijerinck sostuvo que los virus eran de naturaleza líquida, teoría posteriormente desacreditada por Wendell Stanley , quien demostró que eran partículas. [6] Ese mismo año, Friedrich Loeffler y Paul Frosch hicieron pasar el primer virus animal, el aftovirus (agente de la fiebre aftosa ), a través de un filtro similar. [9]
A principios del siglo XX, el bacteriólogo inglés Frederick Twort descubrió un grupo de virus que infectan bacterias, ahora llamados bacteriófagos [10] (o comúnmente 'fagos'), y el microbiólogo franco-canadiense Félix d'Herelle describió virus que, cuando se añadían a las bacterias en una placa de agar , produciría áreas de bacterias muertas. Diluyó con precisión una suspensión de estos virus y descubrió que las diluciones más altas (concentraciones de virus más bajas), en lugar de matar todas las bacterias, formaban áreas discretas de organismos muertos. Contar estas áreas y multiplicarlas por el factor de dilución le permitió calcular la cantidad de virus en la suspensión original. [11] Los fagos fueron anunciados como un tratamiento potencial para enfermedades como la fiebre tifoidea y el cólera , pero su promesa fue olvidada con el desarrollo de la penicilina . El desarrollo de resistencia bacteriana a los antibióticos ha renovado el interés en el uso terapéutico de los bacteriófagos. [12]
A finales del siglo XIX, los virus se definían en términos de su infectividad , su capacidad para pasar filtros y su necesidad de huéspedes vivos. Los virus se habían cultivado sólo en plantas y animales. En 1906, Ross Granville Harrison inventó un método para hacer crecer tejido en la linfa , y en 1913 E. Steinhardt, C. Israelí y RA Lambert utilizaron este método para hacer crecer el virus vaccinia en fragmentos de tejido corneal de cobaya. [13] En 1928, HB Maitland y MC Maitland cultivaron virus vaccinia en suspensiones de riñones de gallina picados. Su método no fue ampliamente adoptado hasta la década de 1950, cuando el poliovirus se cultivó a gran escala para la producción de vacunas. [14]
Otro avance se produjo en 1931, cuando el patólogo estadounidense Ernest William Goodpasture y Alice Miles Woodruff cultivaron influenza y varios otros virus en huevos de gallina fertilizados. [15] En 1949, John Franklin Enders , Thomas Weller y Frederick Robbins cultivaron poliovirus en células cultivadas a partir de tejido embrionario humano abortado, [16] el primer virus que se cultivó sin utilizar tejido animal sólido ni óvulos. Este trabajo permitió a Hilary Koprowski , y luego a Jonas Salk , fabricar una vacuna eficaz contra la polio . [17]
Las primeras imágenes de virus se obtuvieron con la invención de la microscopía electrónica en 1931 por los ingenieros alemanes Ernst Ruska y Max Knoll . [18] En 1935, el bioquímico y virólogo estadounidense Wendell Meredith Stanley examinó el virus del mosaico del tabaco y descubrió que estaba compuesto principalmente de proteínas. [19] Poco tiempo después, este virus se separó en partes de proteína y ARN. [20] El virus del mosaico del tabaco fue el primero en cristalizarse y, por lo tanto, su estructura pudo dilucidarse en detalle. Las primeras imágenes de difracción de rayos X del virus cristalizado fueron obtenidas por Bernal y Fankuchen en 1941. Basándose en sus imágenes cristalográficas de rayos X, Rosalind Franklin descubrió la estructura completa del virus en 1955. [21] Ese mismo año, Heinz Fraenkel-Conrat y Robley Williams demostraron que el ARN purificado del virus del mosaico del tabaco y su cubierta proteica pueden ensamblarse por sí mismos para formar virus funcionales, lo que sugiere que este mecanismo simple fue probablemente el medio a través del cual se crearon los virus dentro de sus células huésped. [22]
La segunda mitad del siglo XX fue la época dorada del descubrimiento de virus, y la mayoría de las especies documentadas de virus animales, vegetales y bacterianos se descubrieron durante estos años. [23] En 1957 se descubrieron el arterivirus equino y la causa de la diarrea por virus bovino (un pestivirus ). En 1963, Baruch Blumberg descubrió el virus de la hepatitis B , [24] y en 1965, Howard Temin describió el primer retrovirus . La transcriptasa inversa , la enzima que utilizan los retrovirus para hacer copias de ADN de su ARN, fue descrita por primera vez en 1970 por Temin y David Baltimore de forma independiente. [25] En 1983, el equipo de Luc Montagnier en el Instituto Pasteur de Francia, aisló por primera vez el retrovirus ahora llamado VIH. [26] En 1989, el equipo de Michael Houghton en Chiron Corporation descubrió la hepatitis C. [27] [28]

Existen varios enfoques para detectar virus y estos incluyen la detección de partículas virales (viriones) o sus antígenos o ácidos nucleicos y ensayos de infectividad.

Los virus se observaron por primera vez en la década de 1930, cuando se inventaron los microscopios electrónicos. Estos microscopios utilizan haces de electrones en lugar de luz, que tienen una longitud de onda mucho más corta y pueden detectar objetos que no se pueden ver con los microscopios ópticos. El aumento más alto que se puede obtener con los microscopios electrónicos es de hasta 10.000.000 de veces [29] , mientras que para los microscopios ópticos es de alrededor de 1.500 veces. [30]
Los virólogos suelen utilizar tinciones negativas para ayudar a visualizar los virus. En este procedimiento, los virus se suspenden en una solución de sales metálicas como el acetato de uranio. Los átomos de metal son opacos a los electrones y los virus se ven suspendidos en un fondo oscuro de átomos de metal. [29] Esta técnica se ha utilizado desde la década de 1950. [31] Se descubrieron muchos virus utilizando esta técnica y la microscopía electrónica de tinción negativa sigue siendo un arma valiosa en el arsenal de un virólogo. [32]
La microscopía electrónica tradicional tiene la desventaja de que los virus se dañan al secarse en alto vacío dentro del microscopio electrónico y el propio haz de electrones es destructivo. [29] En la microscopía electrónica criogénica, la estructura de los virus se conserva incrustándolos en un entorno de agua vítrea . [33] Esto permite la determinación de estructuras biomoleculares con una resolución casi atómica, [34] y ha atraído una gran atención al enfoque como alternativa a la cristalografía de rayos X o la espectroscopia de RMN para la determinación de la estructura de los virus. [35]
Los virus son parásitos intracelulares obligados y, debido a que sólo se reproducen dentro de las células vivas de un huésped, estas células son necesarias para cultivarlos en el laboratorio. Para los virus que infectan a animales (generalmente llamados "virus animales") se utilizan células cultivadas en cultivos celulares de laboratorio. En el pasado se utilizaban huevos de gallina fértiles y los virus se cultivaban en las membranas que rodeaban al embrión. Este método todavía se utiliza en la fabricación de algunas vacunas. Para los virus que infectan a las bacterias, los bacteriófagos , se pueden utilizar directamente las bacterias que crecen en los tubos de ensayo. Para los virus de las plantas se pueden utilizar las plantas huéspedes naturales o, especialmente cuando la infección no es evidente, las llamadas plantas indicadoras, que muestran signos de infección más claramente. [36] [37]

Los virus que han crecido en cultivos celulares pueden detectarse indirectamente por el efecto perjudicial que tienen sobre la célula huésped. Estos efectos citopáticos suelen ser característicos del tipo de virus. Por ejemplo, los virus del herpes simple producen un "inflamiento" característico de las células, típicamente fibroblastos humanos . Algunos virus, como el virus de las paperas, hacen que los glóbulos rojos de los pollos se adhieran firmemente a las células infectadas. Esto se llama "hemadsorción" o "hemadsorción". Algunos virus producen "lesiones" localizadas en capas celulares llamadas placas , que son útiles en ensayos de cuantificación y en la identificación de especies de virus mediante ensayos de reducción de placas . [38] [39]
Los virus que crecen en cultivos celulares se utilizan para medir su susceptibilidad a fármacos antivirales novedosos y validados . [40]
Los virus son antígenos que inducen la producción de anticuerpos y estos anticuerpos pueden usarse en laboratorios para estudiar virus. Los virus relacionados a menudo reaccionan con los anticuerpos de otros y algunos virus pueden recibir nombres según los anticuerpos con los que reaccionan. El uso de anticuerpos que alguna vez se derivaron exclusivamente del suero (líquido sanguíneo) de animales se llama serología . [41] Una vez que se ha producido una reacción de anticuerpos en una prueba, se necesitan otros métodos para confirmarla. Los métodos más antiguos incluían pruebas de fijación del complemento , [42] inhibición de la hemaglutinación y neutralización del virus . [43] Los métodos más nuevos utilizan inmunoensayos enzimáticos (EIA). [44]
En los años previos a la invención de la PCR , la inmunofluorescencia se utilizaba para confirmar rápidamente las infecciones virales. Es un ensayo de infectividad que es específico de la especie de virus porque se utilizan anticuerpos. Los anticuerpos se marcan con un tinte luminiscente y, cuando se utiliza un microscopio óptico con una fuente de luz modificada, las células infectadas brillan en la oscuridad. [45]
La PCR es un método fundamental para detectar virus en todas las especies, incluidas plantas y animales. Funciona detectando rastros de ARN o ADN específicos del virus. Es muy sensible y específico, pero puede verse fácilmente comprometido por la contaminación. La mayoría de las pruebas utilizadas en virología veterinaria y virología médica se basan en PCR o métodos similares como la amplificación mediada por transcripción . Cuando surge un nuevo virus, como el coronavirus covid, se puede diseñar rápidamente una prueba específica siempre que se haya secuenciado el genoma viral y se hayan identificado regiones únicas del ADN o ARN viral. [46] La invención de las pruebas de microfluidos permitió automatizar la mayoría de estas pruebas. [47] A pesar de su especificidad y sensibilidad, la PCR tiene la desventaja de que no diferencia virus infecciosos y no infecciosos ni "pruebas de cura". deben retrasarse hasta 21 días para permitir que el ácido nucleico viral residual se elimine del sitio de la infección. [48]
En los laboratorios muchas de las pruebas diagnósticas para detectar virus son métodos de amplificación de ácidos nucleicos como la PCR. Algunas pruebas detectan los virus o sus componentes, como la microscopía electrónica y los inmunoensayos enzimáticos . Los aparatos de prueba llamados "domésticos" o "auto" suelen ser pruebas de flujo lateral , que detectan el virus mediante un anticuerpo monoclonal etiquetado . [49] Estos también se utilizan en la agricultura, la alimentación y las ciencias ambientales. [50]
El recuento de virus (cuantificación) siempre ha tenido un papel importante en virología y se ha vuelto central para el control de algunas infecciones de humanos donde se mide la carga viral . [51] Hay dos métodos básicos: los que cuentan las partículas del virus completamente infectivas, que se denominan ensayos de infectividad, y los que cuentan todas las partículas, incluidas las defectuosas. [29]

Los ensayos de infectividad miden la cantidad (concentración) de virus infecciosos en una muestra de volumen conocido. [52] Para las células huésped se utilizan plantas o cultivos de células bacterianas o animales. También se han utilizado animales de laboratorio como los ratones, especialmente en virología veterinaria. [53] Estos son ensayos cuantitativos donde los resultados están en una escala continua o cuánticos, donde un evento ocurre o no. Los ensayos cuantitativos dan valores absolutos y los ensayos cuánticos dan una probabilidad estadística, como el volumen de la muestra de prueba necesaria para garantizar que el 50% de las células, plantas o animales hospedadores estén infectados. Esto se llama dosis infecciosa mediana o ID 50 . [54] Los bacteriófagos infecciosos se pueden contar sembrándolos en "céspedes" de bacterias en placas de cultivo. Cuando están en bajas concentraciones, los virus forman agujeros en el césped que se pueden contar. El número de virus se expresa entonces como unidades formadoras de placas . Para los bacteriófagos que se reproducen en bacterias que no pueden cultivarse en cultivos, se utilizan ensayos de carga viral. [55]

El ensayo de formación de foco (FFA) es una variación del ensayo de placa, pero en lugar de depender de la lisis celular para detectar la formación de placa, el FFA emplea técnicas de inmunotinción que utilizan anticuerpos marcados con fluorescencia específicos para un antígeno viral para detectar células huésped infectadas y enfermedades infecciosas. partículas de virus antes de que se forme una placa real. La FFA es particularmente útil para cuantificar clases de virus que no lisan las membranas celulares, ya que estos virus no serían susceptibles al ensayo de placa. Al igual que el ensayo de placa, las monocapas de células huésped se infectan con varias diluciones de la muestra de virus y se dejan incubar durante un período de incubación relativamente breve (p. ej., 24 a 72 horas) bajo un medio de recubrimiento semisólido que restringe la propagación del virus infeccioso, creando células localizadas. grupos (focos) de células infectadas. Posteriormente, las placas se sondan con anticuerpos marcados con fluorescencia contra un antígeno viral y se utiliza microscopía de fluorescencia para contar y cuantificar el número de focos. El método FFA generalmente produce resultados en menos tiempo que los ensayos de placa o de dosis infecciosa de cultivo de tejido del cincuenta por ciento (TCID 50 ), pero puede ser más costoso en términos de reactivos y equipos necesarios. El tiempo de finalización del ensayo también depende del tamaño del área que está contando el usuario. Un área más grande requerirá más tiempo pero puede proporcionar una representación más precisa de la muestra. Los resultados de la FFA se expresan como unidades formadoras de focos por mililitro, o FFU/ [56]
Cuando se realiza un ensayo para medir la partícula viral infecciosa (ensayo de placa, ensayo Focus), el título viral a menudo se refiere a la concentración de partículas virales infecciosas, que es diferente de las partículas virales totales. Los ensayos de carga viral generalmente cuentan la cantidad de genomas virales presentes en lugar de la cantidad de partículas y utilizan métodos similares a la PCR . [57] Las pruebas de carga viral son un elemento importante en el control de las infecciones por VIH. [58] Este método versátil se puede utilizar para virus de plantas. [59] [60]
La virología molecular es el estudio de los virus a nivel de ácidos nucleicos y proteínas. Todos los métodos inventados por los biólogos moleculares han demostrado ser útiles en virología. Sus pequeños tamaños y estructuras relativamente simples hacen de los virus un candidato ideal para el estudio mediante estas técnicas.

Para realizar más estudios, los virus cultivados en el laboratorio necesitan purificarse para eliminar los contaminantes de las células huésped. Los métodos utilizados suelen tener la ventaja de concentrar los virus, lo que facilita su investigación.
Las centrífugas se utilizan a menudo para purificar virus. Las centrífugas de baja velocidad, es decir, aquellas con una velocidad máxima de 10.000 revoluciones por minuto (rpm), no son lo suficientemente potentes para concentrar virus, pero las ultracentrífugas con una velocidad máxima de alrededor de 100.000 rpm sí lo son, y esta diferencia se utiliza en un método llamado centrifugación diferencial . En este método, los contaminantes más grandes y pesados se eliminan de una mezcla de virus mediante centrifugación a baja velocidad. Los virus, que son pequeños y livianos y se dejan en suspensión, luego se concentran mediante centrifugación a alta velocidad. [62]
Después de la centrifugación diferencial, las suspensiones de virus a menudo permanecen contaminadas con desechos que tienen el mismo coeficiente de sedimentación y no se eliminan mediante el procedimiento. En estos casos se utiliza una modificación de la centrifugación, denominada centrifugación por densidad flotante . En este método, los virus recuperados de la centrifugación diferencial se centrifugan nuevamente a muy alta velocidad durante varias horas en soluciones densas de azúcares o sales que forman un gradiente de densidad, de menor a mayor, en el tubo durante la centrifugación. En algunos casos, se utilizan gradientes preformados donde se superponen cuidadosamente soluciones de densidad cada vez menor. Como un objeto en el Mar Muerto , a pesar de la fuerza centrífuga, las partículas de virus no pueden hundirse en soluciones que son más densas que ellas y forman capas discretas de virus concentrados, a menudo visibles, en el tubo. El cloruro de cesio se utiliza a menudo para estas soluciones, ya que es relativamente inerte pero autoforma fácilmente un gradiente cuando se centrifuga a alta velocidad en una ultracentrífuga. [61] La centrifugación por densidad flotante también se puede utilizar para purificar los componentes de los virus, como sus ácidos nucleicos o proteínas. [63]
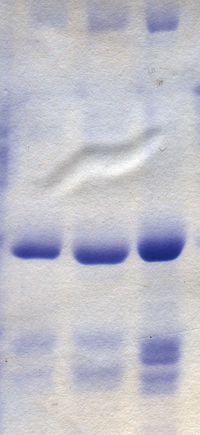
La separación de moléculas en función de su carga eléctrica se llama electroforesis . Los virus y todos sus componentes se pueden separar y purificar mediante este método. Esto generalmente se hace en un medio de soporte como geles de agarosa y poliacrilamida . Las moléculas separadas se revelan mediante tinciones como el azul de coomasie , para las proteínas, o el bromuro de etidio, para los ácidos nucleicos. En algunos casos, los componentes virales se vuelven radiactivos antes de la electroforesis y se revelan utilizando una película fotográfica en un proceso conocido como autorradiografía . [64]
Como la mayoría de los virus son demasiado pequeños para ser vistos con un microscopio óptico, la secuenciación es una de las principales herramientas en virología para identificar y estudiar el virus. La secuenciación de Sanger tradicional y la secuenciación de próxima generación (NGS) se utilizan para secuenciar virus en la investigación básica y clínica, así como para el diagnóstico de infecciones virales emergentes, la epidemiología molecular de patógenos virales y las pruebas de resistencia a los medicamentos. Hay más de 2,3 millones de secuencias virales únicas en GenBank. [65] NGS ha superado al Sanger tradicional como el enfoque más popular para generar genomas virales. [65] La secuenciación del genoma viral se ha convertido en un método central en la epidemiología viral y la clasificación viral .
Los datos de la secuenciación de genomas virales se pueden utilizar para determinar relaciones evolutivas y esto se llama análisis filogenético . [66] Se utiliza software, como PHYLIP , para dibujar árboles filogenéticos . Este análisis también se utiliza para estudiar la propagación de infecciones virales en las comunidades ( epidemiología ). [67]
Cuando se necesitan virus purificados o componentes virales para pruebas de diagnóstico o vacunas, se puede utilizar la clonación en lugar de cultivar los virus. [68] Al comienzo de la pandemia de COVID-19, la disponibilidad de la secuencia de ARN del coronavirus 2 del síndrome respiratorio agudo severo permitió fabricar pruebas rápidamente. [69] Existen varios métodos probados para clonar virus y sus componentes. A menudo se utilizan pequeños trozos de ADN llamados vectores de clonación y los más comunes son plásmidos modificados en laboratorio (pequeñas moléculas circulares de ADN producidas por bacterias). El ácido nucleico viral, o una parte del mismo, se inserta en el plásmido, que las bacterias copian muchas veces. Este ADN recombinante se puede utilizar para producir componentes virales sin necesidad de virus nativos. [70]
Los virus que se reproducen en bacterias, arqueas y hongos se denominan informalmente "fagos" [71] y los que infectan bacterias ( bacteriófagos ) en particular son útiles en virología y biología en general. [72] Los bacteriófagos fueron algunos de los primeros virus que se descubrieron a principios del siglo XX, [73] y debido a que son relativamente fáciles de crecer rápidamente en los laboratorios, gran parte de nuestra comprensión de los virus se originó estudiándolos. [73] Los bacteriófagos, conocidos desde hace mucho tiempo por sus efectos positivos en el medio ambiente, se utilizan en técnicas de presentación de fagos para detectar secuencias de ADN de proteínas. Son una poderosa herramienta en biología molecular. [74]
Todos los virus tienen genes que se estudian mediante genética . [75] Todas las técnicas utilizadas en biología molecular, como la clonación, la creación de mutaciones y el silenciamiento del ARN , se utilizan en genética viral. [76]
La reordenación es el cambio de genes de diferentes padres y es particularmente útil cuando se estudia la genética de virus que tienen genomas segmentados (fragmentados en dos o más moléculas de ácido nucleico), como los virus de la influenza y los rotavirus . De esta forma se pueden identificar los genes que codifican propiedades como el serotipo . [77]
A menudo confundida con recombinación, la recombinación es también la mezcla de genes, pero el mecanismo difiere en que tramos de moléculas de ADN o ARN, a diferencia de las moléculas completas, se unen durante el ciclo de replicación del ARN o ADN. La recombinación no es tan común como la recombinación en la naturaleza, pero es una poderosa herramienta en los laboratorios para estudiar la estructura y funciones de los genes virales. [78]
La genética inversa es un poderoso método de investigación en virología. [79] En este procedimiento se utilizan copias de ADN complementario (ADNc) de genomas de virus llamados "clones infecciosos" para producir virus genéticamente modificados que luego pueden analizarse para detectar cambios en, por ejemplo, virulencia o transmisibilidad. [80]
Una rama importante de la virología es la clasificación de virus . Es artificial porque no se basa en la filogenética evolutiva , sino en propiedades compartidas o distintivas de los virus. [81] [82] Busca describir la diversidad de virus nombrándolos y agrupándolos en función de similitudes. [83] En 1962, André Lwoff , Robert Horne y Paul Tournier fueron los primeros en desarrollar un medio de clasificación de virus, basado en el sistema jerárquico de Linneo . [84] Este sistema basa la clasificación en filo , clase , orden , familia , género y especie . Los virus se agruparon según sus propiedades compartidas (no las de sus huéspedes) y el tipo de ácido nucleico que formaba sus genomas. [85] En 1966, se formó el Comité Internacional sobre Taxonomía de Virus (ICTV). El sistema propuesto por Lwoff, Horne y Tournier no fue aceptado inicialmente por el ICTV porque el pequeño tamaño del genoma de los virus y su alta tasa de mutación hacían difícil determinar su ascendencia fuera de orden. Como tal, el sistema de clasificación de Baltimore ha llegado a utilizarse para complementar la jerarquía más tradicional. [86] A partir de 2018, el ICTV comenzó a reconocer relaciones evolutivas más profundas entre los virus que se han descubierto a lo largo del tiempo y adoptó un sistema de clasificación de 15 rangos que van desde el reino hasta la especie. [87] Además, algunas especies dentro del mismo género se agrupan en un genogrupo . [88] [89]
El ICTV desarrolló el sistema de clasificación actual y redactó pautas que otorgan mayor importancia a ciertas propiedades del virus para mantener la uniformidad familiar. Se ha establecido una taxonomía unificada (un sistema universal para clasificar virus). Sólo se ha estudiado una pequeña parte de la diversidad total de virus. [90] A partir de 2021, 6 reinos, 10 reinos, 17 filos, 2 subfilos, 39 clases, 65 órdenes, 8 subórdenes, 233 familias, 168 subfamilias , 2606 géneros, 84 subgéneros y 10,434 especies de virus han sido definidos por el ICTV. [91]
A continuación se muestran la estructura taxonómica general de los rangos de taxones y los sufijos utilizados en los nombres taxonómicos. A partir de 2021, los rangos de subreino, subreino y subclase no se utilizan, mientras que todos los demás rangos están en uso. [91]

El biólogo David Baltimore, ganador del Premio Nobel, ideó el sistema de clasificación de Baltimore . [92]
La clasificación de virus de Baltimore se basa en el mecanismo de producción de ARNm . Los virus deben generar ARNm a partir de sus genomas para producir proteínas y replicarse, pero se utilizan diferentes mecanismos para lograrlo en cada familia de virus. Los genomas virales pueden ser monocatenarios (ss) o bicatenarios (ds), ARN o ADN, y pueden utilizar o no transcriptasa inversa (RT). Además, los virus ssRNA pueden ser sentido (+) o antisentido (-). Esta clasificación sitúa a los virus en siete grupos:
{{cite book}}: Mantenimiento CS1: falta el editor de la ubicación ( enlace )