
El virus Escherichia T4 es una especie de bacteriófagos que infectan a la bacteria Escherichia coli . Es un virus de ADN bicatenario de la subfamilia Tevenvirinae de la familia Myoviridae . T4 es capaz de experimentar sólo un ciclo de vida lítico y no el ciclo de vida lisogénico . La especie se denominaba anteriormente bacteriófago T-even , nombre que también engloba, entre otras cepas (o aislados), el fago T2 de las enterobacterias , el fago T4 de las enterobacterias y el fago T6 de las enterobacterias .
Los fagos T-incluso, que se remontan a la década de 1940 y continúan en la actualidad, se consideran los organismos modelo mejor estudiados. Generalmente se requiere que los organismos modelo sean simples y tengan tan solo cinco genes . Sin embargo, los fagos T-even se encuentran de hecho entre los virus más grandes y de mayor complejidad , en los que la información genética de estos fagos se compone de alrededor de 300 genes . Coincidiendo con su complejidad, se descubrió que los virus T-incluso tenían la base inusual hidroximetilcitosina (HMC) en lugar de la base del ácido nucleico citosina . [4]
El genoma de ADN bicatenario del virus T4 tiene aproximadamente 169 kpb de largo [5] y codifica 289 proteínas . El genoma T4 es terminalmente redundante . Tras la replicación del ADN, se forman concatémeros de longitud larga y multigenoma, quizás mediante un mecanismo de replicación de círculo rodante. [6] Cuando se empaqueta, el concatémero se corta en posiciones no específicas de la misma longitud, lo que da lugar a varios genomas que representan permutaciones circulares del original. [7] El genoma T4 contiene secuencias de intrones similares a las de los eucariotas .
La secuencia GAGG de Shine-Dalgarno domina en los genes tempranos del virus T4, mientras que la secuencia GGAG es un objetivo para la endonucleasa T4 RegB que inicia la degradación temprana del ARNm. [8]

T4 es un virus relativamente grande, de aproximadamente 90 nm de ancho y 200 nm de largo (la mayoría de los virus varían entre 25 y 200 nm de largo). El genoma del ADN se encuentra contenido en una cabeza icosaédrica , también conocida como cápside . [9] La cola del T4 es hueca para que pueda pasar su ácido nucleico a la célula que está infectando después de la unión. Los fagos Myoviridae como T4 tienen estructuras de cola contráctiles complejas con una gran cantidad de proteínas involucradas en el ensamblaje y función de la cola. [10] Las fibras de la cola también son importantes para reconocer los receptores de la superficie de la célula huésped, por lo que determinan si una bacteria se encuentra dentro del rango de huéspedes del virus. [11]
Recientemente se ha descrito la estructura de la placa base T4 de 6 megadaltones que comprende 127 cadenas polipeptídicas de 13 proteínas diferentes (productos génicos 5, 5.4, 6, 7, 8, 9, 10, 11, 12, 25, 27, 48 y 53). en detalle atómico. También se ha creado un modelo atómico de la región proximal del tubo de cola formada por gp54 y la proteína del tubo principal gp19. La proteína de cinta métrica gp29 está presente en los complejos placa base-tubo de cola, pero no se pudo modelar. [12]
Durante el ensamblaje del virión T4 del bacteriófago (fago) , las proteínas morfogenéticas codificadas por los genes del fago interactúan entre sí en una secuencia característica. Mantener un equilibrio apropiado en las cantidades de cada una de estas proteínas producidas durante la infección viral parece ser fundamental para la morfogénesis normal del fago T4. [13] Las proteínas codificadas por el fago T4 que determinan la estructura del virión incluyen componentes estructurales principales, componentes estructurales menores y proteínas no estructurales que catalizan pasos específicos en la secuencia de morfogénesis. [14] La morfogénesis del fago T4 se divide en tres vías independientes: la cabeza, la cola y las fibras de la cola larga, como lo detallan Yap y Rossman. [15]
El virus T4 inicia una infección por Escherichia coli uniendo proteínas porinas OmpC y lipopolisacáridos (LPS) en la superficie de las células de E. coli con sus fibras de cola larga (LTF). [16] [17] Se envía una señal de reconocimiento a través de los LTF a la placa base. Esto desenreda las fibras cortas de la cola (STF) que se unen irreversiblemente a la superficie de las células de E. coli . La placa base cambia de conformación y la vaina de la cola se contrae, lo que hace que GP5 en el extremo del tubo de la cola perfore la membrana externa de la célula. [18] El dominio lisozima de GP5 se activa y degrada la capa de peptidoglicano periplásmico . La parte restante de la membrana se degrada y luego el ADN de la cabeza del virus puede viajar a través del tubo de la cola y entrar en la célula de E. coli . [ cita necesaria ]
En 1952, Hershey y Chase [19] proporcionaron evidencia clave de que el ADN del fago, a diferencia de las proteínas, ingresa a la célula bacteriana huésped tras la infección y, por lo tanto, es el material genético del fago. Este hallazgo sugirió que el ADN es, en general, el material genético de diferentes organismos. [ cita necesaria ]
El ciclo de vida lítico (desde la entrada en una bacteria hasta su destrucción) dura aproximadamente 30 minutos (a 37 °C). Los bacteriófagos virulentos se multiplican en su huésped bacteriano inmediatamente después de su entrada. Una vez que el número de fagos de la progenie alcanza una cierta cantidad, hacen que el huésped se lise o se descomponga, por lo que se liberarán e infectarán nuevas células huésped. [20] El proceso de lisis y liberación del huésped se denomina ciclo lítico . El ciclo lítico es un ciclo de reproducción viral que implica la destrucción de la célula infectada y su membrana. Este ciclo involucra un virus que supera a la célula huésped y su maquinaria para reproducirse. Por lo tanto, el virus debe pasar por 5 etapas para poder reproducirse e infectar la célula huésped: [ cita necesaria ]
Una vez completado el ciclo de vida, la célula huésped se abre de golpe y expulsa los virus recién construidos al medio ambiente, destruyendo la célula huésped. T4 tiene un tamaño de ráfaga de aproximadamente 100 a 150 partículas virales por huésped infectado. [ cita necesaria ]
Benzer (1955 – 1959) desarrolló un sistema para estudiar la estructura fina del gen utilizando mutantes del bacteriófago T4 defectuosos en los genes rIIA y rIIB . [21] [22] [23] Las técnicas empleadas fueron pruebas de complementación y cruces para detectar recombinación , particularmente entre mutaciones de deleción. Estos experimentos genéticos llevaron al hallazgo de un orden lineal único de sitios mutacionales dentro de los genes. Este resultado proporcionó pruebas sólidas de la idea clave de que el gen tiene una estructura lineal equivalente a una longitud de ADN con muchos sitios que pueden mutar de forma independiente. [ cita necesaria ]
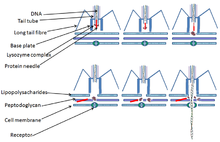
Al igual que todos los demás virus, los fagos T-incluso no se adhieren aleatoriamente a la superficie de su huésped; en lugar de eso, "buscan" y se unen a receptores , estructuras proteicas específicas , que se encuentran en la superficie del huésped. Estos receptores varían con el fago; El ácido teicoico , las proteínas de la pared celular y los lipopolisacáridos , los flagelos y los pili pueden servir como receptores a los que se puede unir el fago. Para que el fago T-incluso infecte a su huésped y comience su ciclo de vida debe entrar en el primer proceso de infección , la adsorción del fago a la célula bacteriana. La adsorción es un valor característico del par fago-huésped y la adsorción del fago en la superficie de la célula huésped se ilustra como un proceso de 2 etapas: reversible e irreversible. Implica la estructura de la cola del fago que comienza cuando las fibras de la cola del fago ayudan a unir el fago al receptor apropiado de su huésped. Este proceso es reversible. Uno o más de los componentes de la placa base median en el proceso irreversible de unión del fago a una bacteria. [ cita necesaria ]
La penetración también es un valor característico de la infección fago-huésped que implica la inyección del material genético del fago dentro de la bacteria . La penetración del ácido nucleico tiene lugar después de la fase de adsorción irreversible. Los mecanismos que implican la penetración del ácido nucleico de los fagos son específicos de cada fago. Este mecanismo de penetración puede involucrar potencial de membrana electroquímico, moléculas de ATP , división enzimática de la capa de peptidoglicano , o los tres factores pueden ser vitales para la penetración del ácido nucleico dentro de la célula bacteriana. Se han realizado estudios sobre el mecanismo de penetración del bacteriófago T2 (fago similar a T4) y se ha demostrado que la cola del fago no penetra dentro de la pared celular bacteriana y la penetración de este fago implica un potencial de membrana electroquímico en la membrana interna. [24] [25]
El genoma del virus T4 se sintetiza dentro de la célula huésped mediante replicación en círculo rodante. [6] El tiempo que tarda la replicación del ADN en una célula viva se midió como la tasa de elongación del ADN del virus T4 en E. coli infectada por virus. [26] Durante el período de aumento exponencial del ADN a 37 °C, la velocidad fue de 749 nucleótidos por segundo. La tasa de mutación por par de bases por replicación durante la síntesis de ADN del virus T4 es de 1,7 por 10 −8 , [27] un mecanismo de copia de ADN muy preciso, con sólo 1 error cada 300 copias. El virus también codifica mecanismos únicos de reparación del ADN . [28] La cabeza del fago T4 se ensambla vacía alrededor de una proteína de andamio, que luego se degrada. En consecuencia, el ADN necesita ingresar a la procabeza a través de un pequeño poro, lo que se logra mediante la interacción primero de un hexámero de gp17 con el ADN, que también sirve como motor y nucleasa. Se ha descubierto que el motor de empaquetado de ADN T4 carga ADN en las cápsides de virus a una velocidad de hasta 2000 pares de bases por segundo. La potencia involucrada, si se ampliara en tamaño, sería equivalente a la de un motor de automóvil promedio. [29]
El paso final en la reproducción y multiplicación viral está determinado por la liberación de viriones de la célula huésped. La liberación de los viriones se produce tras la rotura de la membrana plasmática bacteriana. Los virus sin envoltura lisan la célula huésped, que se caracteriza por proteínas virales que atacan el peptidoglicano o la membrana. La lisis de las bacterias se produce cuando las cápsides del interior de la célula liberan la enzima lisozima que descompone la pared celular. Los bacteriófagos liberados infectan otras células y el ciclo de multiplicación viral se repite dentro de esas células. [ cita necesaria ]

La reactivación de multiplicidad (MR) es el proceso mediante el cual dos o más genomas de virus, cada uno de los cuales contiene daño genómico inactivante, pueden interactuar dentro de una célula infectada para formar un genoma de virus viable. Salvador Luria , mientras estudiaba el virus T4 irradiado con luz ultravioleta en 1946, descubrió la RM y propuso que la reactivación observada del virus dañado se produce mediante un mecanismo de recombinación (ver referencias [30] [31] [32] ). Esto precedió a la confirmación del ADN como el material genético en 1952 en el virus T2 relacionado mediante el experimento de Hershey-Chase . [19]
Como lo recuerda Luria (1984, [33] p. 97), el descubrimiento de la reactivación de virus irradiados (denominada " reactivación de multiplicidad ") inició inmediatamente una oleada de actividad en el estudio de la reparación del daño por radiación dentro del grupo de fagos tempranos ( revisado por Bernstein [28] en 1981). Más tarde resultó que la reparación de virus dañados mediante ayuda mutua que Luria había descubierto era sólo un caso especial de reparación del ADN. Ahora se sabe que las células de todo tipo, no sólo las bacterias y sus virus, sino todos los organismos estudiados, incluidos los humanos, tienen procesos bioquímicos complejos para reparar los daños del ADN (consulte Reparación del ADN ). Ahora también se reconoce que los procesos de reparación del ADN desempeñan funciones críticas en la protección contra el envejecimiento , el cáncer y la infertilidad . [ cita necesaria ]
La RM suele representarse mediante "curvas de supervivencia" en las que la supervivencia de la capacidad de formación de placas de las células infectadas múltiples (multicomplejos) se representa frente a la dosis del agente que daña el genoma. A modo de comparación, la supervivencia de la capacidad de formación de placas virales de células infectadas individualmente (monocomplejos) también se representa gráficamente frente a la dosis del agente que daña el genoma. La figura superior muestra las curvas de supervivencia de los multicomplejos y monocomplejos del virus T4 con una dosis creciente de luz ultravioleta. Dado que la supervivencia se representa en una escala logarítmica, está claro que la supervivencia de los multicomplejos supera a la de los monocomplejos en factores muy importantes (dependiendo de la dosis). La curva de inactivación UV para multicomplejos tiene un hombro inicial. Otros agentes que dañan el ADN del virus T4 con hombros en sus curvas de supervivencia multicomplejas son los rayos X [34] [35] y el metanosulfonato de etilo (EMS). [28] Se ha interpretado que la presencia de un hombro significa que se utilizan dos procesos recombinacionales. [36] El primero repara el ADN con alta eficiencia (en el "hombro"), pero se satura en su capacidad a medida que aumenta el daño; la segunda vía funciona en todos los niveles de daño. Los virus T4 supervivientes liberados de multicomplejos no muestran ningún aumento en la mutación , lo que indica que la RM del virus irradiado con UV es un proceso preciso. [36]
La figura inferior muestra las curvas de supervivencia para la inactivación del virus T4 por el agente que daña el ADN mitomicina C (MMC). En este caso, la curva de supervivencia para multicomplejos no tiene hombro inicial, lo que sugiere que sólo el segundo proceso de reparación recombinacional descrito anteriormente está activo. La eficacia de la reparación mediante este proceso queda indicada por la observación de que una dosis de MMC que permite la supervivencia de sólo 1 en 1.000 monocomplejos permite la supervivencia de aproximadamente el 70% de los multicomplejos. También se obtuvieron curvas de supervivencia multicomplejas similares (sin hombros) para los agentes que dañan el ADN, desintegración P32 , psoraleno más irradiación ultravioleta cercana (PUVA), N-metil-N'-nitro-N-nitrosoguanidina (MNNG), metanosulfonato de metilo (MMS). ) y ácido nitroso . [28]
Varios de los genes que se consideraron necesarios para la RM en el virus T4 resultaron ser ortólogos de genes esenciales para la recombinación en procariotas , eucariotas y arqueas . Esto incluye, por ejemplo, el gen T4 uvsX [37] que especifica una proteína que tiene homología estructural tridimensional con RecA de Escherichia coli y la proteína homóloga RAD51 en eucariotas y RadA en arqueas . Se ha sugerido que la reparación recombinante eficiente y precisa de los daños del ADN durante la RM puede ser análoga al proceso de reparación recombinante que se produce durante la meiosis en eucariotas . [38]
Los bacteriófagos fueron descubiertos por primera vez por el científico inglés Frederick Twort en 1915 y Félix d'Hérelle en 1917. A finales de la década de 1930, T. L. Rakieten propuso a los dos investigadores Milislav una mezcla de aguas residuales sin tratar o un lisado de E. coli infectado con aguas residuales sin tratar. Demerec y Ugo Fano . Estos dos investigadores aislaron T3, T4, T5 y T6 de E. coli . Además, en 1932, el investigador J. Bronfenbrenner había estudiado y trabajado en el fago T2, en el que se aisló el fago T2 del virus. [39] Este aislamiento se hizo a partir de material fecal en lugar de aguas residuales. En cualquier caso, Max Delbrück participó en el descubrimiento de los fagos T-even. Su parte fue nombrar los bacteriófagos en Tipo 1 (T1), Tipo 2 (T2), Tipo 3 (T3), etc. [ cita necesaria ]
El momento y el lugar específicos del aislamiento del virus T4 aún no están claros, aunque probablemente se encontraron en aguas residuales o materia fecal. T4 y virus similares fueron descritos en un artículo de Thomas F. Anderson , Max Delbrück y Milislav Demerec en noviembre de 1944. [40] En 1943, Salvador Luria y Delbrück demostraron que las mutaciones bacterianas para la resistencia a los fagos surgen en ausencia de selección , en lugar de que ser una respuesta a la selección. [33] La sabiduría tradicional entre los bacteriólogos antes de 1943 era que las bacterias no tenían cromosomas ni genes. El experimento de Luria-Delbrück demostró que las bacterias, al igual que otros organismos genéticos modelo establecidos, tienen genes y que éstos pueden mutar espontáneamente para generar mutantes que luego pueden reproducirse para formar linajes clonales. Ese año, también comenzaron a trabajar con Alfred Hershey , otro experimentador de fagos. [41] (Los tres compartirían el Premio Nobel de Fisiología o Medicina de 1969 , "por sus trabajos sobre el mecanismo de replicación y la genética de los virus").
El grupo de fagos era una red informal de biólogos centrada en Max Delbrück que llevó a cabo investigaciones básicas principalmente sobre el bacteriófago T4 e hizo numerosas contribuciones fundamentales a la genética microbiana y los orígenes de la biología molecular a mediados del siglo XX. En 1961, Sydney Brenner , uno de los primeros miembros del grupo de los fagos, colaboró con Francis Crick , Leslie Barnett y Richard Watts-Tobin en el Laboratorio Cavendish de Cambridge para realizar experimentos genéticos que demostraron la naturaleza básica del código genético de las proteínas. [42] Estos experimentos, realizados con mutantes del gen rIIB del fago T4, mostraron que, para un gen que codifica una proteína, tres bases secuenciales del ADN del gen especifican cada aminoácido sucesivo de la proteína. Por tanto, el código genético es un código triplete, donde cada triplete (llamado codón) especifica un aminoácido particular. También obtuvieron pruebas de que los codones no se superponen entre sí en la secuencia de ADN que codifica una proteína y que dicha secuencia se lee desde un punto de partida fijo. [ cita necesaria ]
Durante 1962-1964, los investigadores del fago T4 brindaron la oportunidad de estudiar la función de prácticamente todos los genes que son esenciales para el crecimiento del fago en condiciones de laboratorio. [43] [44] Estos estudios se vieron facilitados por el descubrimiento de dos clases de mutantes letales condicionales . Una clase de tales mutantes se conoce como mutantes ámbar . [45] Otra clase de mutantes letales condicionales se conoce como mutantes sensibles a la temperatura. [46] Los estudios de estas dos clases de mutantes llevaron a una comprensión considerable de numerosos problemas biológicos fundamentales. De este modo se logró comprender las funciones e interacciones de las proteínas empleadas en la maquinaria de replicación , reparación y recombinación del ADN , y cómo se ensamblan los virus a partir de componentes proteicos y de ácidos nucleicos ( morfogénesis molecular ). Además, se aclaró el papel de los codones de terminación de cadena . Un estudio digno de mención utilizó mutantes ámbar defectuosos en el gen que codifica la proteína principal principal del fago T4. [47] Este experimento proporcionó pruebas sólidas de la "hipótesis de la secuencia", ampliamente sostenida, pero antes de 1964 aún no probada, de que la secuencia de aminoácidos de una proteína está especificada por la secuencia de nucleótidos del gen que determina la proteína. Así, este estudio demostró la colinealidad del gen con su proteína codificada.
Varios ganadores del Premio Nobel trabajaron con virus T4 o virus similares a T4, incluidos Max Delbrück , Salvador Luria , Alfred Hershey , James D. Watson y Francis Crick . Otros científicos importantes que trabajaron con el virus T4 incluyen a Michael Rossmann , Seymour Benzer , Bruce Alberts , Gisela Mosig , [48] Richard Lenski y James Bull .
Caudovirales
>
Myoviridae
>
Tevenvirinae
>
T4virus
>
Escherichia virus T4