Una mitocondria ( pl. mitochondria ) es un orgánulo que se encuentra en las células de la mayoría de los eucariotas , como animales , plantas y hongos . Las mitocondrias tienen una estructura de doble membrana y utilizan la respiración aeróbica para generar trifosfato de adenosina (ATP), que se utiliza en toda la célula como fuente de energía química . [2] Fueron descubiertas por Albert von Kölliker en 1857 [3] en los músculos voluntarios de los insectos. El término mitocondria , que significa un gránulo con forma de filamento, fue acuñado por Carl Benda en 1898. La mitocondria es popularmente apodada la "central eléctrica de la célula", una frase popularizada por Philip Siekevitz en un artículo de Scientific American de 1957 del mismo nombre. [4]
Algunas células de algunos organismos multicelulares carecen de mitocondrias (por ejemplo, los glóbulos rojos maduros de los mamíferos ). Se sabe que el animal multicelular Henneguya salminicola ha conservado orgánulos relacionados con las mitocondrias a pesar de una pérdida completa de su genoma mitocondrial. [5] [6] [7] Una gran cantidad de organismos unicelulares , como microsporidia , parabasálidos y diplomonas , han reducido o transformado sus mitocondrias en otras estructuras, [8] por ejemplo, hidrogenosomas y mitosomas . [9] Las oximónadas Monocercomonoides , Streblomastix y Blattamonas han perdido completamente sus mitocondrias. [5] [10]
Las mitocondrias tienen comúnmente entre 0,75 y 3 μm 2 en sección transversal, [11] pero varían considerablemente en tamaño y estructura. A menos que se tiñen específicamente , no son visibles. Además de suministrar energía celular, las mitocondrias están involucradas en otras tareas, como la señalización , la diferenciación celular y la muerte celular , así como en el mantenimiento del control del ciclo celular y el crecimiento celular . [12] La biogénesis mitocondrial está a su vez coordinada temporalmente con estos procesos celulares. [13] [14] Las mitocondrias han sido implicadas en varios trastornos y afecciones humanas, como enfermedades mitocondriales , [15] disfunción cardíaca , [16] insuficiencia cardíaca [17] y autismo . [18]
La cantidad de mitocondrias en una célula puede variar ampliamente según el organismo , el tejido y el tipo de célula. Un glóbulo rojo maduro no tiene mitocondrias, [19] mientras que una célula hepática puede tener más de 2000. [20] [21] La mitocondria está compuesta de compartimentos que realizan funciones especializadas. Estos compartimentos o regiones incluyen la membrana externa, el espacio intermembrana , la membrana interna , las crestas y la matriz .
Aunque la mayor parte del ADN de una célula eucariota está contenido en el núcleo celular , la mitocondria tiene su propio genoma ("mitogenoma") que es sustancialmente similar a los genomas bacterianos . [22] Este hallazgo ha llevado a la aceptación general de la hipótesis endosimbiótica : los ancestros procariotas de vida libre de las mitocondrias modernas se fusionaron permanentemente con células eucariotas en el pasado distante, evolucionando de tal manera que los animales, plantas, hongos y otros eucariotas modernos pueden respirar para generar energía celular . [23]
Las mitocondrias pueden tener distintas formas. [24] Una mitocondria contiene membranas internas y externas compuestas de bicapas de fosfolípidos y proteínas . [20] Las dos membranas tienen propiedades diferentes. Debido a esta organización de doble membrana, hay cinco partes distintas en una mitocondria:
Las mitocondrias se pliegan para aumentar su superficie, lo que a su vez aumenta la producción de ATP (trifosfato de adenosina). Las mitocondrias despojadas de su membrana externa se denominan mitoplastos .
La membrana mitocondrial externa , que encierra todo el orgánulo, tiene un grosor de 60 a 75 angstroms (Å). Tiene una relación proteína-fosfolípido similar a la de la membrana celular (aproximadamente 1:1 en peso). Contiene grandes cantidades de proteínas integrales de membrana llamadas porinas . Una proteína de tráfico importante es el canal aniónico dependiente de voltaje formador de poros (VDAC). El VDAC es el transportador primario de nucleótidos , iones y metabolitos entre el citosol y el espacio intermembrana. [25] [26] Está formado como un barril beta que se extiende por la membrana externa, similar al de la membrana externa bacteriana gramnegativa . [27] Las proteínas más grandes pueden ingresar a la mitocondria si una secuencia de señalización en su extremo N se une a una proteína multisubunidad grande llamada translocasa en la membrana externa , que luego las mueve activamente a través de la membrana. [28] Las proproteínas mitocondriales se importan a través de complejos de translocación especializados.
La membrana externa también contiene enzimas involucradas en actividades tan diversas como la elongación de ácidos grasos , la oxidación de epinefrina y la degradación de triptófano . Estas enzimas incluyen monoaminooxidasa , NADH-citocromo c-reductasa insensible a rotenona , quinurenina hidroxilasa y ácido graso Co-A ligasa . La ruptura de la membrana externa permite que las proteínas en el espacio intermembrana se filtren al citosol, lo que lleva a la muerte celular. [29] La membrana mitocondrial externa puede asociarse con la membrana del retículo endoplasmático (RE), en una estructura llamada MAM (membrana RE asociada a mitocondrias). Esto es importante en la señalización de calcio RE-mitocondria y está involucrado en la transferencia de lípidos entre el RE y las mitocondrias. [30] Fuera de la membrana externa hay partículas pequeñas (diámetro: 60 Å) llamadas subunidades de Parson.
El espacio intermembrana mitocondrial es el espacio entre la membrana externa y la membrana interna. También se conoce como espacio perimitocondrial. Debido a que la membrana externa es libremente permeable a las moléculas pequeñas, las concentraciones de moléculas pequeñas, como iones y azúcares, en el espacio intermembrana son las mismas que en el citosol . [20] Sin embargo, las proteínas grandes deben tener una secuencia de señalización específica para ser transportadas a través de la membrana externa, por lo que la composición proteica de este espacio es diferente de la composición proteica del citosol . Una proteína que se localiza en el espacio intermembrana de esta manera es el citocromo c . [29]
La membrana mitocondrial interna contiene proteínas con tres tipos de funciones: [20]
Contiene más de 151 polipéptidos diferentes y tiene una relación proteína-fosfolípido muy alta (más de 3:1 en peso, que es aproximadamente 1 proteína por 15 fosfolípidos). La membrana interna alberga alrededor de 1/5 de la proteína total en una mitocondria. [31] Además, la membrana interna es rica en un fosfolípido inusual, la cardiolipina . Este fosfolípido se descubrió originalmente en corazones de vaca en 1942 y generalmente es característico de las membranas plasmáticas mitocondriales y bacterianas. [32] La cardiolipina contiene cuatro ácidos grasos en lugar de dos y puede ayudar a hacer que la membrana interna sea impermeable, [20] y su alteración puede conducir a múltiples trastornos clínicos, incluidos trastornos neurológicos y cáncer. [33] A diferencia de la membrana externa, la membrana interna no contiene porinas y es altamente impermeable a todas las moléculas. Casi todos los iones y moléculas requieren transportadores de membrana especiales para ingresar o salir de la matriz. Las proteínas son transportadas a la matriz a través del complejo translocasa de la membrana interna (TIM) o a través de OXA1L . [28] Además, existe un potencial de membrana a través de la membrana interna, formado por la acción de las enzimas de la cadena de transporte de electrones . La fusión de la membrana interna está mediada por la proteína de membrana interna OPA1 . [34]

La membrana mitocondrial interna está compartimentada en numerosos pliegues llamados crestas , que expanden el área de superficie de la membrana mitocondrial interna, mejorando su capacidad para producir ATP. Para las mitocondrias hepáticas típicas, el área de la membrana interna es aproximadamente cinco veces más grande que la de la membrana externa. Esta relación es variable y las mitocondrias de las células que tienen una mayor demanda de ATP, como las células musculares, contienen incluso más crestas. Las mitocondrias dentro de la misma célula pueden tener una densidad de crestas sustancialmente diferente, y las que se requieren para producir más energía tienen una superficie de membrana de cresta mucho mayor. [35] Estos pliegues están tachonados con pequeños cuerpos redondos conocidos como partículas F 1 u oxisomas. [36]
La matriz es el espacio encerrado por la membrana interna. Contiene aproximadamente 2/3 de las proteínas totales en una mitocondria. [20] La matriz es importante en la producción de ATP con la ayuda de la ATP sintasa contenida en la membrana interna. La matriz contiene una mezcla altamente concentrada de cientos de enzimas, ribosomas mitocondriales especiales , ARNt y varias copias del genoma del ADN mitocondrial . De las enzimas, las principales funciones incluyen la oxidación del piruvato y los ácidos grasos , y el ciclo del ácido cítrico . [20] Las moléculas de ADN están empaquetadas en nucleoides por proteínas, una de las cuales es TFAM . [37]
Las funciones más importantes de las mitocondrias son producir la moneda energética de la célula, ATP (es decir, fosforilación de ADP ), a través de la respiración y regular el metabolismo celular . [21] El conjunto central de reacciones involucradas en la producción de ATP se conoce colectivamente como el ciclo del ácido cítrico , o el ciclo de Krebs , y la fosforilación oxidativa . Sin embargo, la mitocondria tiene muchas otras funciones además de la producción de ATP.
Un papel dominante de las mitocondrias es la producción de ATP, como se refleja en la gran cantidad de proteínas en la membrana interna para esta tarea. Esto se hace oxidando los principales productos de la glucosa : piruvato y NADH , que se producen en el citosol. [21] Este tipo de respiración celular , conocida como respiración aeróbica , depende de la presencia de oxígeno . Cuando el oxígeno es limitado, los productos glucolíticos se metabolizarán mediante fermentación anaeróbica , un proceso que es independiente de las mitocondrias. [21] La producción de ATP a partir de glucosa y oxígeno tiene un rendimiento aproximadamente 13 veces mayor durante la respiración aeróbica en comparación con la fermentación. [38] Las mitocondrias de las plantas también pueden producir una cantidad limitada de ATP ya sea rompiendo el azúcar producido durante la fotosíntesis o sin oxígeno utilizando el sustrato alternativo nitrito . [39] El ATP atraviesa la membrana interna con la ayuda de una proteína específica y atraviesa la membrana externa a través de las porinas . [40] Después de la conversión de ATP en ADP por desfosforilación que libera energía, el ADP regresa por la misma ruta.
Las moléculas de piruvato producidas por la glucólisis se transportan activamente a través de la membrana mitocondrial interna y hacia la matriz , donde pueden oxidarse y combinarse con la coenzima A para formar CO2 , acetil -CoA y NADH [21] o pueden carboxilarse (por la piruvato carboxilasa ) para formar oxalacetato. Esta última reacción "completa" la cantidad de oxalacetato en el ciclo del ácido cítrico y, por lo tanto, es una reacción anaplerótica , que aumenta la capacidad del ciclo para metabolizar acetil-CoA cuando las necesidades energéticas del tejido (por ejemplo, en el músculo ) aumentan repentinamente por la actividad. [41]
En el ciclo del ácido cítrico, todos los intermediarios (por ejemplo , citrato , isocitrato , alfa-cetoglutarato , succinato, fumarato , malato y oxaloacetato) se regeneran durante cada vuelta del ciclo. Por lo tanto, agregar más de cualquiera de estos intermediarios a la mitocondria significa que la cantidad adicional se retiene dentro del ciclo, aumentando todos los demás intermediarios a medida que uno se convierte en el otro. Por lo tanto, la adición de cualquiera de ellos al ciclo tiene un efecto anaplerótico , y su eliminación tiene un efecto cataplerótico. Estas reacciones anapleróticas y catapleróticas, durante el curso del ciclo, aumentarán o disminuirán la cantidad de oxaloacetato disponible para combinarse con acetil-CoA para formar ácido cítrico. Esto a su vez aumenta o disminuye la tasa de producción de ATP por la mitocondria y, por lo tanto, la disponibilidad de ATP para la célula. [41]
Por otra parte, el acetil-CoA, derivado de la oxidación del piruvato o de la beta-oxidación de los ácidos grasos , es el único combustible que entra en el ciclo del ácido cítrico. En cada vuelta del ciclo se consume una molécula de acetil-CoA por cada molécula de oxaloacetato presente en la matriz mitocondrial, y nunca se regenera. Es la oxidación de la porción de acetato del acetil-CoA la que produce CO2 y agua, y la energía así liberada se captura en forma de ATP. [41]
En el hígado, la carboxilación del piruvato citosólico en oxaloacetato intramitocondrial es un paso temprano en la vía gluconeogénica , que convierte el lactato y la alanina desaminada en glucosa, [21] [41] bajo la influencia de altos niveles de glucagón y/o epinefrina en la sangre. [41] Aquí, la adición de oxaloacetato a la mitocondria no tiene un efecto anaplerótico neto, ya que otro intermediario del ciclo del ácido cítrico (malato) se elimina inmediatamente de la mitocondria para convertirse en oxaloacetato citosólico y, en última instancia, en glucosa, en un proceso que es casi el inverso de la glucólisis . [41]
Las enzimas del ciclo del ácido cítrico se encuentran en la matriz mitocondrial, con excepción de la succinato deshidrogenasa , que está unida a la membrana mitocondrial interna como parte del Complejo II. [42] El ciclo del ácido cítrico oxida el acetil-CoA a dióxido de carbono y, en el proceso, produce cofactores reducidos (tres moléculas de NADH y una molécula de FADH 2 ) que son una fuente de electrones para la cadena de transporte de electrones , y una molécula de GTP (que se convierte fácilmente en ATP). [21]

Los electrones de NADH y FADH 2 se transfieren al oxígeno (O 2 ) y al hidrógeno (protones) en varios pasos a través de una cadena de transporte de electrones. Las moléculas de NADH y FADH 2 se producen dentro de la matriz a través del ciclo del ácido cítrico y en el citoplasma por glucólisis . Los equivalentes reductores del citoplasma pueden importarse a través del sistema de transporte de malato-aspartato de las proteínas antiportadoras o introducirse en la cadena de transporte de electrones utilizando un transporte de fosfato de glicerol . [21]
Las principales reacciones de liberación de energía [43] [44] que hacen de la mitocondria la "central energética de la célula" ocurren en los complejos proteicos I, III y IV en la membrana mitocondrial interna ( NADH deshidrogenasa (ubiquinona) , citocromo c reductasa y citocromo c oxidasa ). En el complejo IV , el O 2 reacciona con la forma reducida del hierro en el citocromo c :
liberando una gran cantidad de energía libre [44] [43] de los reactivos sin romper los enlaces de un combustible orgánico. La energía libre que se pone en marcha para quitar un electrón del Fe 2+ se libera en el complejo III cuando el Fe 3+ del citocromo c reacciona para oxidar el ubiquinol (QH 2 ):
La ubiquinona (Q) generada reacciona, en el complejo I , con NADH:
Si bien las reacciones están controladas por una cadena de transporte de electrones, los electrones libres no se encuentran entre los reactivos o productos en las tres reacciones mostradas y, por lo tanto, no afectan la energía libre liberada, que se utiliza para bombear protones (H + ) al espacio intermembrana. Este proceso es eficiente, pero un pequeño porcentaje de electrones puede reducir prematuramente el oxígeno, formando especies reactivas de oxígeno como el superóxido . [21] Esto puede causar estrés oxidativo en las mitocondrias y puede contribuir al declive de la función mitocondrial asociado con el envejecimiento. [45]
A medida que aumenta la concentración de protones en el espacio intermembrana, se establece un fuerte gradiente electroquímico a través de la membrana interna. Los protones pueden regresar a la matriz a través del complejo ATP sintasa , y su energía potencial se utiliza para sintetizar ATP a partir de ADP y fosfato inorgánico (P i ). [21] Este proceso se llama quimiosmosis , y fue descrito por primera vez por Peter Mitchell , [46] [47] quien recibió el Premio Nobel de Química en 1978 por su trabajo. Más tarde, parte del Premio Nobel de Química de 1997 fue otorgado a Paul D. Boyer y John E. Walker por su aclaración del mecanismo de funcionamiento de la ATP sintasa. [48]
En determinadas condiciones, los protones pueden volver a entrar en la matriz mitocondrial sin contribuir a la síntesis de ATP. Este proceso se conoce como fuga de protones o desacoplamiento mitocondrial y se debe a la difusión facilitada de protones en la matriz. El proceso da como resultado que la energía potencial no aprovechada del gradiente electroquímico de protones se libere en forma de calor. [21] El proceso está mediado por un canal de protones llamado termogenina o UCP1 . [49] La termogenina se encuentra principalmente en el tejido adiposo pardo o grasa parda y es responsable de la termogénesis sin escalofríos. El tejido adiposo pardo se encuentra en los mamíferos y alcanza sus niveles más altos en los primeros años de vida y en los animales que hibernan. En los seres humanos, el tejido adiposo pardo está presente al nacer y disminuye con la edad. [49]
La síntesis de ácidos grasos mitocondriales (mtFASII) es esencial para la respiración celular y la biogénesis mitocondrial. [50] También se cree que desempeña un papel como mediador en la señalización intracelular debido a su influencia en los niveles de lípidos bioactivos, como los lisofosfolípidos y los esfingolípidos . [51]
Se considera que el octanoil-ACP (C8) es el producto final más importante de mtFASII, que también forma el sustrato inicial de la biosíntesis del ácido lipoico . [52] Dado que el ácido lipoico es el cofactor de importantes complejos enzimáticos mitocondriales, como el complejo de piruvato deshidrogenasa (PDC), el complejo de α-cetoglutarato deshidrogenasa (OGDC), el complejo de α-cetoácido deshidrogenasa de cadena ramificada (BCKDC) y en el sistema de escisión de glicina (GCS), mtFASII tiene una influencia en el metabolismo energético. [53]
Otros productos de mtFASII desempeñan un papel en la regulación de la traducción mitocondrial, la biogénesis del grupo FeS y el ensamblaje de complejos de fosforilación oxidativa. [52]
Además, con la ayuda de mtFASII y ACP acilado, el acetil-CoA regula su consumo en las mitocondrias. [52]

Las concentraciones de calcio libre en la célula pueden regular una serie de reacciones y son importantes para la transducción de señales en la célula. Las mitocondrias pueden almacenar calcio transitoriamente , un proceso que contribuye a la homeostasis del calcio en la célula. [54] [55] Su capacidad de absorber calcio rápidamente para liberarlo más tarde las convierte en buenos "amortiguadores citosólicos" para el calcio. [56] [57] [58] El retículo endoplasmático (RE) es el sitio de almacenamiento de calcio más importante, [59] y existe una interacción significativa entre la mitocondria y el RE con respecto al calcio. [60] El calcio es absorbido por la matriz por el uniportador de calcio mitocondrial en la membrana mitocondrial interna . [61] Está impulsado principalmente por el potencial de membrana mitocondrial . [55] La liberación de este calcio de regreso al interior de la célula puede ocurrir a través de una proteína de intercambio de sodio-calcio o mediante vías de "liberación de calcio inducida por calcio". [61] Esto puede iniciar picos de calcio u ondas de calcio con grandes cambios en el potencial de membrana. Estos pueden activar una serie de proteínas del sistema de segundos mensajeros que pueden coordinar procesos como la liberación de neurotransmisores en las células nerviosas y la liberación de hormonas en las células endocrinas. [62]
Recientemente se ha implicado al influjo de Ca 2+ a la matriz mitocondrial como un mecanismo para regular la bioenergética respiratoria al permitir que el potencial electroquímico a través de la membrana "pulse" transitoriamente desde un estado dominado por ΔΨ a un estado dominado por el pH, lo que facilita una reducción del estrés oxidativo . [63] En las neuronas, los aumentos concomitantes del calcio citosólico y mitocondrial actúan para sincronizar la actividad neuronal con el metabolismo energético mitocondrial. Los niveles de calcio de la matriz mitocondrial pueden alcanzar niveles de decenas de micromolares, lo que es necesario para la activación de la isocitrato deshidrogenasa , una de las enzimas reguladoras clave del ciclo de Krebs . [64]
Se ha investigado la relación entre la proliferación celular y las mitocondrias. Las células tumorales requieren abundante ATP para sintetizar compuestos bioactivos como lípidos , proteínas y nucleótidos para una rápida proliferación. [65] La mayoría del ATP en las células tumorales se genera a través de la vía de fosforilación oxidativa (OxPhos). [66] La interferencia con OxPhos causa la detención del ciclo celular, lo que sugiere que las mitocondrias desempeñan un papel en la proliferación celular. [66] La producción de ATP mitocondrial también es vital para la división celular y la diferenciación en la infección [67] además de las funciones básicas en la célula, incluida la regulación del volumen celular, la concentración de solutos y la arquitectura celular. [68] [69] [70] Los niveles de ATP difieren en varias etapas del ciclo celular, lo que sugiere que existe una relación entre la abundancia de ATP y la capacidad de la célula para ingresar a un nuevo ciclo celular. [71] El papel del ATP en las funciones básicas de la célula hace que el ciclo celular sea sensible a los cambios en la disponibilidad de ATP derivado de las mitocondrias. [71] La variación en los niveles de ATP en diferentes etapas del ciclo celular apoya la hipótesis de que las mitocondrias juegan un papel importante en la regulación del ciclo celular. [71] Aunque los mecanismos específicos entre las mitocondrias y la regulación del ciclo celular no se entienden bien, los estudios han demostrado que los puntos de control del ciclo celular de baja energía monitorean la capacidad energética antes de comprometerse con otra ronda de división celular. [12]
La muerte celular programada (PCD) es crucial para diversas funciones fisiológicas, incluido el desarrollo de órganos y la homeostasis celular. Actúa como un mecanismo intrínseco para prevenir la transformación maligna y desempeña un papel fundamental en la inmunidad al ayudar en la defensa antiviral, la eliminación de patógenos, la inflamación y el reclutamiento de células inmunitarias. [72]
Las mitocondrias han sido reconocidas desde hace mucho tiempo por su papel central en la vía intrínseca de la apoptosis , una forma de PCD. [73] En las últimas décadas, también se las ha identificado como un centro de señalización para gran parte del sistema inmunológico innato . [74] El origen endosimbiótico de las mitocondrias las distingue de otros componentes celulares, y la exposición de elementos mitocondriales al citosol puede desencadenar las mismas vías que los marcadores de infección. Estas vías conducen a la apoptosis , la autofagia o la inducción de genes proinflamatorios. [75] [74]
Las mitocondrias contribuyen a la apoptosis liberando citocromo c , que induce directamente la formación de apoptosomas . Además, son una fuente de varios patrones moleculares asociados a daños (DAMP). Estos DAMP a menudo son reconocidos por los mismos receptores de reconocimiento de patrones (PRR) que responden a los patrones moleculares asociados a patógenos (PAMP) durante las infecciones. [76] Por ejemplo, el ADNmt mitocondrial se parece al ADN bacteriano debido a su falta de metilación de CpG y puede ser detectado por el receptor tipo Toll 9 y cGAS . [77] El ARN bicatenario (dsRNA), producido debido a la transcripción mitocondrial bidireccional, puede activar vías de detección viral a través de receptores tipo RIG-I . [78] Además, la N -formilación de las proteínas mitocondriales , similar a la de las proteínas bacterianas, puede ser reconocida por los receptores de péptidos formilo . [79] [80]
Normalmente, estos componentes mitocondriales son secuestrados del resto de la célula, pero se liberan tras la permeabilización de la membrana mitocondrial durante la apoptosis o de forma pasiva tras un daño mitocondrial. Sin embargo, las mitocondrias también desempeñan un papel activo en la inmunidad innata, liberando ADNmt en respuesta a señales metabólicas. [74] Las mitocondrias también son el sitio de localización de proteínas reguladoras de la inmunidad y la apoptosis, como BAX , MAVS (ubicada en la membrana externa) y NLRX1 (que se encuentra en la matriz). Estas proteínas están moduladas por el estado metabólico mitocondrial y la dinámica mitocondrial. [74] [81] [82]
Las mitocondrias desempeñan un papel central en muchas otras tareas metabólicas , como:
Algunas funciones mitocondriales se realizan solo en tipos específicos de células. Por ejemplo, las mitocondrias de las células hepáticas contienen enzimas que les permiten desintoxicar el amoníaco , un producto de desecho del metabolismo de las proteínas. Una mutación en los genes que regulan cualquiera de estas funciones puede provocar enfermedades mitocondriales .
Las proteínas mitocondriales (proteínas transcritas a partir del ADN mitocondrial) varían según el tejido y la especie. En los seres humanos, se han identificado 615 tipos distintos de proteínas a partir de mitocondrias cardíacas , [92] mientras que en ratas , se han descrito 940 proteínas. [93] Se cree que el proteoma mitocondrial está regulado dinámicamente. [94]

Las mitocondrias (o estructuras relacionadas) se encuentran en todos los eucariotas (excepto en el Oxymonad Monocercomonoides ). [5] Aunque comúnmente se representan como estructuras similares a frijoles, forman una red altamente dinámica en la mayoría de las células donde sufren constantemente fisión y fusión . La población de todas las mitocondrias de una célula dada constituye el condrioma. [95] Las mitocondrias varían en número y ubicación según el tipo de célula. A menudo se encuentra una sola mitocondria en organismos unicelulares, mientras que las células del hígado humano tienen alrededor de 1000 a 2000 mitocondrias por célula, lo que representa 1/5 del volumen celular. [20] El contenido mitocondrial de células similares puede variar sustancialmente en tamaño y potencial de membrana, [96] con diferencias que surgen de fuentes que incluyen una partición desigual en la división celular, lo que lleva a diferencias extrínsecas en los niveles de ATP y los procesos celulares posteriores. [97] Las mitocondrias se pueden encontrar anidadas entre las miofibrillas del músculo o envueltas alrededor del flagelo del espermatozoide . [20] A menudo, forman una red compleja de ramificación 3D dentro de la célula con el citoesqueleto . La asociación con el citoesqueleto determina la forma mitocondrial, que también puede afectar la función: [98] diferentes estructuras de la red mitocondrial pueden proporcionar a la población una variedad de ventajas o desventajas físicas, químicas y de señalización. [99] Las mitocondrias en las células siempre se distribuyen a lo largo de los microtúbulos y la distribución de estos orgánulos también se correlaciona con el retículo endoplásmico . [100] Evidencias recientes sugieren que la vimentina , uno de los componentes del citoesqueleto, también es fundamental para la asociación con el citoesqueleto. [101]
La membrana del RE asociada a las mitocondrias (MAM) es otro elemento estructural que se reconoce cada vez más por su papel crítico en la fisiología celular y la homeostasis . Alguna vez considerado un problema técnico en las técnicas de fraccionamiento celular, los supuestos contaminantes de vesículas del RE que invariablemente aparecían en la fracción mitocondrial han sido reidentificados como estructuras membranosas derivadas de la MAM, la interfaz entre las mitocondrias y el RE. [102] El acoplamiento físico entre estos dos orgánulos se había observado previamente en micrografías electrónicas y más recientemente se ha investigado con microscopía de fluorescencia . [102] Dichos estudios estiman que en la MAM, que puede comprender hasta el 20% de la membrana externa mitocondrial, el RE y las mitocondrias están separados por apenas 10–25 nm y se mantienen unidos por complejos de unión de proteínas. [102] [30] [103]
La MAM purificada del fraccionamiento subcelular está enriquecida con enzimas implicadas en el intercambio de fosfolípidos, además de canales asociados con la señalización de Ca 2+ . [102] [103] Estos indicios de un papel destacado de la MAM en la regulación de los depósitos lipídicos celulares y la transducción de señales se han confirmado, con implicaciones significativas para los fenómenos celulares asociados a las mitocondrias, como se analiza a continuación. La MAM no solo ha proporcionado información sobre la base mecanística subyacente a procesos fisiológicos como la apoptosis intrínseca y la propagación de la señalización del calcio, sino que también favorece una visión más refinada de las mitocondrias. Aunque a menudo se las ve como "centrales eléctricas" estáticas y aisladas secuestradas para el metabolismo celular a través de un antiguo evento endosimbiótico, la evolución de la MAM subraya hasta qué punto las mitocondrias se han integrado en la fisiología celular general, con un acoplamiento físico y funcional íntimo con el sistema de endomembranas.
La MAM está enriquecida con enzimas involucradas en la biosíntesis de lípidos, como la fosfatidilserina sintasa en la cara del RE y la fosfatidilserina descarboxilasa en la cara mitocondrial. [104] [105] Debido a que las mitocondrias son orgánulos dinámicos que experimentan constantemente eventos de fisión y fusión , requieren un suministro constante y bien regulado de fosfolípidos para la integridad de la membrana. [106] [107] Pero las mitocondrias no solo son un destino para los fosfolípidos cuya síntesis finalizan; más bien, este orgánulo también desempeña un papel en el tráfico entre orgánulos de los intermediarios y productos de las vías biosintéticas de fosfolípidos, el metabolismo de ceramidas y colesterol y el anabolismo de los glicoesfingolípidos. [105] [107]
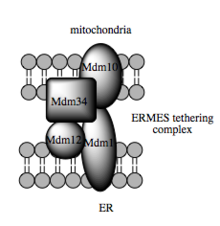
Esta capacidad de tráfico depende del MAM, que se ha demostrado que facilita la transferencia de intermediarios lipídicos entre orgánulos. [104] En contraste con el mecanismo vesicular estándar de transferencia de lípidos, la evidencia indica que la proximidad física del RE y las membranas mitocondriales en el MAM permite la inversión de lípidos entre bicapas opuestas. [107] A pesar de este mecanismo inusual y aparentemente desfavorable desde el punto de vista energético, dicho transporte no requiere ATP. [107] En cambio, en la levadura, se ha demostrado que depende de una estructura de anclaje multiproteica denominada estructura de encuentro RE-mitocondria o ERMES, aunque sigue sin estar claro si esta estructura media directamente la transferencia de lípidos o es necesaria para mantener las membranas en una proximidad suficientemente cercana para reducir la barrera energética para la inversión de lípidos . [107] [108]
La MAM también puede ser parte de la vía secretora, además de su papel en el tráfico intracelular de lípidos. En particular, la MAM parece ser un destino intermedio entre el RE rugoso y el Golgi en la vía que conduce al ensamblaje y secreción de lipoproteínas de muy baja densidad , o VLDL. [105] [109] Por lo tanto, la MAM sirve como un centro metabólico y de tráfico crítico en el metabolismo de los lípidos.
Se reconoció un papel crítico para el RE en la señalización de calcio antes de que se aceptara ampliamente un papel similar para las mitocondrias, en parte porque la baja afinidad de los canales de Ca 2+ localizados en la membrana mitocondrial externa parecía contradecir la supuesta capacidad de respuesta de este orgánulo a los cambios en el flujo intracelular de Ca 2+ . [102] [59] Pero la presencia del MAM resuelve esta aparente contradicción: la estrecha asociación física entre los dos orgánulos da como resultado microdominios de Ca 2+ en los puntos de contacto que facilitan la transmisión eficiente de Ca 2+ desde el RE a las mitocondrias. [102] La transmisión ocurre en respuesta a las llamadas " bocanadas de Ca 2+ " generadas por la agrupación espontánea y la activación de IP3R , un canal de Ca 2+ de la membrana del RE canónico . [102] [30]
El destino de estas bocanadas (en particular, si permanecen restringidas a lugares aislados o integradas en ondas de Ca 2+ para su propagación por toda la célula) está determinado en gran parte por la dinámica del MAM. Aunque la recaptación de Ca 2+ por el RE (concomitante con su liberación) modula la intensidad de las bocanadas, aislando así a las mitocondrias hasta cierto punto de la alta exposición al Ca 2+ , el MAM a menudo sirve como un cortafuegos que esencialmente amortigua las bocanadas de Ca 2+ al actuar como un sumidero en el que se pueden canalizar los iones libres liberados al citosol. [102] [110] [111] Esta tunelización de Ca 2+ ocurre a través del receptor de Ca 2+ de baja afinidad VDAC1 , que recientemente se ha demostrado que está físicamente atado a los grupos de IP3R en la membrana del RE y enriquecido en el MAM. [102] [30] [112] La capacidad de las mitocondrias de servir como sumidero de Ca 2+ es el resultado del gradiente electroquímico generado durante la fosforilación oxidativa, que hace que la tunelización del catión sea un proceso exergónico. [112] La entrada normal y leve de calcio desde el citosol hacia la matriz mitocondrial provoca una despolarización transitoria que se corrige bombeando protones.
Pero la transmisión de Ca 2+ no es unidireccional; más bien, es una calle de doble sentido. [59] Las propiedades de la bomba de Ca 2+ SERCA y el canal IP3R presente en la membrana del RE facilitan la regulación de retroalimentación coordinada por la función de MAM. En particular, la eliminación de Ca 2+ por el MAM permite un patrón espacio-temporal de la señalización de Ca 2+ porque Ca 2+ altera la actividad de IP3R de manera bifásica. [102] SERCA también se ve afectada por la retroalimentación mitocondrial: la captación de Ca 2+ por el MAM estimula la producción de ATP, proporcionando así energía que permite a SERCA recargar el RE con Ca 2+ para el eflujo continuo de Ca 2+ en el MAM. [110] [112] Por lo tanto, el MAM no es un amortiguador pasivo para las bocanadas de Ca 2+ ; más bien, ayuda a modular la señalización de Ca 2+ adicional a través de bucles de retroalimentación que afectan la dinámica del RE.
La regulación de la liberación de Ca 2+ por parte del RE en la MAM es especialmente crítica porque solo una cierta ventana de captación de Ca 2+ mantiene a las mitocondrias, y en consecuencia a la célula, en homeostasis. Se requiere suficiente señalización intraorgánica de Ca 2+ para estimular el metabolismo activando las enzimas deshidrogenasas críticas para el flujo a través del ciclo del ácido cítrico. [113] [114] Sin embargo, una vez que la señalización de Ca 2+ en las mitocondrias pasa un cierto umbral, estimula la vía intrínseca de la apoptosis en parte colapsando el potencial de membrana mitocondrial requerido para el metabolismo. [102] Los estudios que examinan el papel de los factores pro y antiapoptóticos respaldan este modelo; por ejemplo, se ha demostrado que el factor antiapoptótico Bcl-2 interactúa con los IP3R para reducir el llenado de Ca 2+ del RE, lo que conduce a un eflujo reducido en la MAM y previene el colapso del potencial de membrana mitocondrial después de los estímulos apoptóticos. [102] Dada la necesidad de una regulación tan fina de la señalización de Ca 2+ , tal vez no sea sorprendente que el Ca 2+ mitocondrial desregulado haya sido implicado en varias enfermedades neurodegenerativas, mientras que el catálogo de supresores tumorales incluye algunos que están enriquecidos en la MAM. [112]
Los avances recientes en la identificación de los anclajes entre las membranas mitocondriales y del RE sugieren que la función de andamiaje de los elementos moleculares involucrados es secundaria a otras funciones no estructurales. En la levadura, ERMES, un complejo multiproteico de proteínas de membrana residentes en el RE y mitocondriales que interactúan, es necesario para la transferencia de lípidos en la MAM y ejemplifica este principio. Uno de sus componentes, por ejemplo, también es un constituyente del complejo proteico necesario para la inserción de proteínas de barril beta transmembrana en la bicapa lipídica. [107] Sin embargo, aún no se ha identificado un homólogo del complejo ERMES en células de mamíferos. Otras proteínas implicadas en el andamiaje también tienen funciones independientes del anclaje estructural en la MAM; por ejemplo, las mitofusinas residentes en el RE y en las mitocondriales forman heterocomplejos que regulan el número de sitios de contacto entre orgánulos, aunque las mitofusinas se identificaron por primera vez por su papel en los eventos de fisión y fusión entre mitocondrias individuales. [102] La proteína relacionada con la glucosa 75 (grp75) es otra proteína de doble función. Además del conjunto de la matriz de grp75, una porción sirve como chaperona que une físicamente los canales de Ca 2+ mitocondriales y del RE VDAC e IP3R para una transmisión eficiente de Ca 2+ en la MAM. [102] [30] Otro posible vínculo es Sigma-1R , un receptor no opioide cuya estabilización del IP3R residente en el RE puede preservar la comunicación en la MAM durante la respuesta al estrés metabólico. [115] [116]
El MAM es un centro de señalización, metabolismo y tráfico crítico en la célula que permite la integración del RE y la fisiología mitocondrial. El acoplamiento entre estos orgánulos no es simplemente estructural sino también funcional y crítico para la fisiología celular general y la homeostasis . El MAM ofrece así una perspectiva sobre las mitocondrias que diverge de la visión tradicional de este orgánulo como una unidad estática y aislada apropiada por la célula para su capacidad metabólica. [117] En cambio, esta interfaz mitocondrial-RE enfatiza la integración de las mitocondrias, el producto de un evento endosimbiótico, en diversos procesos celulares. Recientemente también se ha demostrado que las mitocondrias y los MAM en las neuronas están anclados a sitios de comunicación intercelular especializados (las llamadas uniones somáticas). Los procesos microgliales monitorean y protegen las funciones neuronales en estos sitios, y se supone que los MAM tienen un papel importante en este tipo de control de calidad celular. [90]
Existen dos hipótesis sobre el origen de las mitocondrias: la endosimbiótica y la autógena . La hipótesis endosimbiótica sugiere que las mitocondrias eran originalmente células procariotas , capaces de implementar mecanismos oxidativos que no eran posibles para las células eucariotas; se convirtieron en endosimbiontes que vivían dentro del eucariota. [23] [118] [119] [120] En la hipótesis autógena, las mitocondrias nacieron al escindirse de una porción de ADN del núcleo de la célula eucariota en el momento de la divergencia con los procariotas; esta porción de ADN habría estado encerrada por membranas, que no podrían ser atravesadas por proteínas. Dado que las mitocondrias tienen muchas características en común con las bacterias , la hipótesis endosimbiótica es la más aceptada de las dos. [120] [121]
Una mitocondria contiene ADN , que está organizado como varias copias de un único cromosoma , generalmente circular . Este cromosoma mitocondrial contiene genes para proteínas redox , como las de la cadena respiratoria. La hipótesis CoRR propone que esta co-ubicación es necesaria para la regulación redox. El genoma mitocondrial codifica algunos ARN de ribosomas y los 22 ARNt necesarios para la traducción de ARNm en proteínas. La estructura circular también se encuentra en procariotas. La proto-mitocondria probablemente estaba estrechamente relacionada con Rickettsia . [122] [123] Sin embargo, la relación exacta del ancestro de las mitocondrias con las alfaproteobacterias y si la mitocondria se formó al mismo tiempo o después del núcleo, sigue siendo controvertido. [124] Por ejemplo, se ha sugerido que el clado SAR11 de bacterias comparte un ancestro común relativamente reciente con las mitocondrias, [125] mientras que los análisis filogenómicos indican que las mitocondrias evolucionaron a partir de un linaje de Pseudomonadota que está estrechamente relacionado con o es miembro de alphaproteobacteria . [126] [127] Algunos artículos describen a las mitocondrias como hermanas de alphaproteobacteriaceae, formando juntas la hermana del grupo marineproteo1, que juntas forman la hermana de Magnetococcidae . [128] [129] [130] [131]
Los ribosomas codificados por el ADN mitocondrial son similares a los de las bacterias en tamaño y estructura. [132] Se parecen mucho al ribosoma 70S bacteriano y no a los ribosomas citoplasmáticos 80S , que están codificados por el ADN nuclear .
La relación endosimbiótica de las mitocondrias con sus células huésped fue popularizada por Lynn Margulis . [133] La hipótesis endosimbiótica sugiere que las mitocondrias descienden de bacterias aeróbicas que de alguna manera sobrevivieron a la endocitosis por otra célula y se incorporaron al citoplasma . La capacidad de estas bacterias para realizar la respiración en células huésped que habían dependido de la glucólisis y la fermentación habría proporcionado una ventaja evolutiva considerable. Esta relación simbiótica probablemente se desarrolló hace entre 1.700 y 2.000 millones de años. [134] [135]

Unos pocos grupos de eucariotas unicelulares tienen solo mitocondrias vestigiales o estructuras derivadas: los microsporidios , las metamónadas y las archamoebas . [136] Estos grupos aparecen como los eucariotas más primitivos en los árboles filogenéticos construidos utilizando información de ARNr , lo que alguna vez sugirió que aparecieron antes del origen de las mitocondrias. Sin embargo, ahora se sabe que esto es un artefacto de atracción de ramas largas : son grupos derivados y retienen genes u orgánulos derivados de las mitocondrias (por ejemplo, mitosomas e hidrogenosomas ). [8] Los hidrogenosomas, mitosomas y orgánulos relacionados que se encuentran en algunos loricíferos (por ejemplo, Spinoloricus ) [137] [138] y mixozoos (por ejemplo, Henneguya zschokkei ) se clasifican juntos como MRO, orgánulos relacionados con las mitocondrias. [7] [139]
Los monocercomonoides y otras oximónadas parecen haber perdido sus mitocondrias por completo y al menos algunas de las funciones mitocondriales parecen ser llevadas a cabo ahora por proteínas citoplasmáticas. [5] [140] [10]

Las mitocondrias contienen su propio genoma. El genoma mitocondrial humano es una molécula de ADN bicatenario circular de aproximadamente 16 kilobases . [141] Codifica 37 genes: 13 para subunidades de complejos respiratorios I, III, IV y V, 22 para ARNt mitocondrial (para los 20 aminoácidos estándar, más un gen extra para leucina y serina), y 2 para ARNr (ARNr 12S y 16S). [141] Una mitocondria puede contener de dos a diez copias de su ADN. [142] Una de las dos hebras de ADN mitocondrial (ADNmt) tiene una proporción desproporcionadamente mayor de los nucleótidos más pesados adenina y guanina, y esto se denomina hebra pesada (o hebra H), mientras que la otra hebra se denomina hebra ligera (o hebra L). La diferencia de peso permite que las dos hebras se separen por centrifugación . El ADNmt tiene un tramo largo no codificante conocido como región no codificante (NCR), que contiene el promotor de la cadena pesada (HSP) y el promotor de la cadena ligera (LSP) para la transcripción del ARN, el origen de replicación de la cadena H (OriH) localizado en la cadena L, tres cajas de secuencia conservadas (CSB 1-3) y una secuencia asociada a la terminación (TAS). El origen de replicación de la cadena L (OriL) está localizado en la cadena H 11.000 pb corriente abajo de OriH, ubicado dentro de un grupo de genes que codifican ARNt. [143]
Al igual que en los procariotas, hay una proporción muy alta de ADN codificante y una ausencia de repeticiones. Los genes mitocondriales se transcriben como transcripciones multigénicas, que se escinden y poliadenilan para producir ARNm maduros . La mayoría de las proteínas necesarias para la función mitocondrial están codificadas por genes en el núcleo celular y las proteínas correspondientes se importan a la mitocondria. [144] El número exacto de genes codificados por el núcleo y el genoma mitocondrial difiere entre especies. La mayoría de los genomas mitocondriales son circulares. [145] En general, el ADN mitocondrial carece de intrones , como es el caso del genoma mitocondrial humano; [144] sin embargo, se han observado intrones en algunos ADN mitocondriales eucariotas, [146] como el de la levadura [147] y los protistas , [148] incluido Dictyostelium discoideum . [149] Entre las regiones codificantes de proteínas, están presentes los ARNt. Los genes de ARNt mitocondriales tienen secuencias diferentes de los ARNt nucleares, pero se han encontrado genes similares a los ARNt mitocondriales en los cromosomas nucleares con una alta similitud de secuencia. [150]
En los animales, el genoma mitocondrial es típicamente un único cromosoma circular que tiene aproximadamente 16 kb de longitud y 37 genes. Los genes, aunque muy conservados, pueden variar en su ubicación. Curiosamente, este patrón no se encuentra en el piojo del cuerpo humano ( Pediculus humanus ). En cambio, este genoma mitocondrial está organizado en 18 cromosomas minicirculares, cada uno de los cuales tiene una longitud de 3-4 kb y tiene de uno a tres genes. [151] Este patrón también se encuentra en otros piojos chupadores , pero no en los piojos masticadores . Se ha demostrado que se produce recombinación entre los minicromosomas.
La casi ausencia de recombinación genética en el ADN mitocondrial lo convierte en una fuente útil de información para estudiar la genética de poblaciones y la biología evolutiva . [152] Debido a que todo el ADN mitocondrial se hereda como una sola unidad, o haplotipo , las relaciones entre el ADN mitocondrial de diferentes individuos se pueden representar como un árbol genético . Los patrones en estos árboles genéticos se pueden utilizar para inferir la historia evolutiva de las poblaciones. El ejemplo clásico de esto es la genética evolutiva humana , donde el reloj molecular se puede utilizar para proporcionar una fecha reciente para la Eva mitocondrial . [153] [154] Esto a menudo se interpreta como un fuerte apoyo a una reciente expansión humana moderna fuera de África . [155] Otro ejemplo humano es la secuenciación del ADN mitocondrial de los huesos neandertales . La distancia evolutiva relativamente grande entre las secuencias de ADN mitocondrial de los neandertales y los humanos vivos se ha interpretado como evidencia de la falta de mestizaje entre los neandertales y los humanos modernos. [156]
Sin embargo, el ADN mitocondrial refleja únicamente la historia de las hembras de una población. Esto se puede solucionar en parte mediante el uso de secuencias genéticas paternas, como la región no recombinante del cromosoma Y. [155]
Mediciones recientes del reloj molecular del ADN mitocondrial [157] informaron un valor de 1 mutación cada 7884 años desde el ancestro común más reciente de humanos y simios, lo que es consistente con las estimaciones de las tasas de mutación del ADN autosómico (10 −8 por base por generación). [158]
Aunque ya se habían previsto ligeras variaciones en el código genético estándar, [159] ninguna se descubrió hasta 1979, cuando los investigadores que estudiaban los genes mitocondriales humanos determinaron que utilizaban un código alternativo. [160] No obstante, las mitocondrias de muchos otros eucariotas, incluidas la mayoría de las plantas, utilizan el código estándar. [161] Desde entonces se han descubierto muchas ligeras variantes, [161] incluidos varios códigos mitocondriales alternativos. [162] Además, los codones AUA, AUC y AUU son todos codones de inicio permitidos.
Algunas de estas diferencias deben considerarse como pseudocambios en el código genético debido al fenómeno de edición de ARN , que es común en las mitocondrias. En las plantas superiores, se pensaba que CGG codificaba para triptófano y no arginina ; sin embargo, se descubrió que el codón en el ARN procesado era el codón UGG, consistente con el código genético estándar para triptófano. [163] Cabe destacar que el código genético mitocondrial de los artrópodos ha experimentado una evolución paralela dentro de un filo, con algunos organismos traduciendo únicamente AGG a lisina. [164]
Las mitocondrias se dividen por fisión mitocondrial , una forma de fisión binaria que también realizan las bacterias [165] aunque el proceso está estrechamente regulado por la célula eucariota huésped e implica comunicación y contacto con varios otros orgánulos. La regulación de esta división difiere entre eucariotas. En muchos eucariotas unicelulares, su crecimiento y división están vinculados al ciclo celular . Por ejemplo, una sola mitocondria puede dividirse sincrónicamente con el núcleo. Este proceso de división y segregación debe controlarse estrictamente para que cada célula hija reciba al menos una mitocondria. En otros eucariotas (en los mamíferos, por ejemplo), las mitocondrias pueden replicar su ADN y dividirse principalmente en respuesta a las necesidades energéticas de la célula, en lugar de en fase con el ciclo celular. Cuando las necesidades energéticas de una célula son altas, las mitocondrias crecen y se dividen. Cuando el uso de energía es bajo, las mitocondrias se destruyen o se vuelven inactivas. En tales ejemplos, las mitocondrias aparentemente se distribuyen aleatoriamente a las células hijas durante la división del citoplasma . La dinámica mitocondrial, el equilibrio entre la fusión y la fisión mitocondrial , es un factor importante en las patologías asociadas a varias enfermedades. [166]
La hipótesis de la fisión binaria mitocondrial se ha basado en la visualización mediante microscopía de fluorescencia y microscopía electrónica de transmisión (MET) convencional. La resolución de la microscopía de fluorescencia (≈200 nm) es insuficiente para distinguir detalles estructurales, como la doble membrana mitocondrial en la división mitocondrial o incluso para distinguir mitocondrias individuales cuando varias están juntas. La MET convencional también tiene algunas limitaciones técnicas [¿ cuáles? ] para verificar la división mitocondrial. Recientemente se utilizó la criotomografía electrónica para visualizar la división mitocondrial en células intactas hidratadas congeladas. Reveló que las mitocondrias se dividen por gemación. [167]
Los genes mitocondriales de un individuo se heredan solo de la madre, con raras excepciones. [168] En los humanos, cuando un óvulo es fertilizado por un espermatozoide, las mitocondrias, y por lo tanto el ADN mitocondrial, generalmente provienen solo del óvulo. Las mitocondrias del espermatozoide ingresan al óvulo, pero no aportan información genética al embrión. [169] En cambio, las mitocondrias paternas se marcan con ubiquitina para seleccionarlas para su posterior destrucción dentro del embrión . [170] El óvulo contiene relativamente pocas mitocondrias, pero estas mitocondrias se dividen para poblar las células del organismo adulto. Este modo se observa en la mayoría de los organismos, incluida la mayoría de los animales. Sin embargo, las mitocondrias en algunas especies a veces pueden heredarse paternalmente. Esta es la norma entre ciertas plantas coníferas , aunque no en pinos y tejos . [171] Para los mitílidos , la herencia paterna solo ocurre dentro de los machos de la especie. [172] [173] [174] Se ha sugerido que se presenta en un nivel muy bajo en humanos. [175]
La herencia uniparental da lugar a pocas oportunidades de recombinación genética entre diferentes linajes de mitocondrias, aunque una sola mitocondria puede contener de 2 a 10 copias de su ADN. [142] La recombinación que se produce mantiene la integridad genética en lugar de mantener la diversidad. Sin embargo, hay estudios que muestran evidencia de recombinación en el ADN mitocondrial. Está claro que las enzimas necesarias para la recombinación están presentes en las células de los mamíferos. [176] Además, la evidencia sugiere que las mitocondrias animales pueden sufrir recombinación. [177] Los datos son más controvertidos en humanos, aunque existe evidencia indirecta de recombinación. [178] [179]
Se puede esperar que las entidades que experimentan herencia uniparental y con poca o ninguna recombinación estén sujetas al trinquete de Muller , la acumulación de mutaciones deletéreas hasta que se pierde la funcionalidad. Las poblaciones animales de mitocondrias evitan esta acumulación a través de un proceso de desarrollo conocido como el cuello de botella del ADNmt . El cuello de botella explota los procesos estocásticos en la célula para aumentar la variabilidad de célula a célula en la carga mutante a medida que un organismo se desarrolla: un solo óvulo con cierta proporción de ADNmt mutante produce un embrión donde diferentes células tienen diferentes cargas mutantes. La selección a nivel celular puede entonces actuar para eliminar aquellas células con más ADNmt mutante, lo que lleva a una estabilización o reducción en la carga mutante entre generaciones. El mecanismo subyacente al cuello de botella es debatido, [180] [181] [182] con un metaestudio matemático y experimental reciente que proporciona evidencia de una combinación de partición aleatoria de ADNmt en divisiones celulares y recambio aleatorio de moléculas de ADNmt dentro de la célula. [183]
Las mitocondrias pueden reparar el daño oxidativo del ADN mediante mecanismos análogos a los que ocurren en el núcleo celular . Las proteínas empleadas en la reparación del ADNmt están codificadas por genes nucleares y se translocan a las mitocondrias. Las vías de reparación del ADN en las mitocondrias de mamíferos incluyen la reparación por escisión de bases , la reparación de rotura de doble cadena, la inversión directa y la reparación de desajustes . [184] [185] Alternativamente, el daño del ADN puede evitarse, en lugar de repararse, mediante la síntesis de translesión.
De los diversos procesos de reparación del ADN en las mitocondrias, la vía de reparación por escisión de bases ha sido la más estudiada. [185] La reparación por escisión de bases se lleva a cabo mediante una secuencia de pasos catalizados por enzimas que incluyen el reconocimiento y la escisión de una base de ADN dañada, la eliminación del sitio abásico resultante, el procesamiento de los extremos, el relleno de los huecos y la ligadura. Un daño común en el ADNmt que se repara mediante la reparación por escisión de bases es la 8-oxoguanina producida por la oxidación de la guanina . [186]
Las roturas de doble cadena se pueden reparar mediante reparación recombinacional homóloga tanto en el ADNmt de mamíferos [187] como en el ADNmt de plantas. [188] Las roturas de doble cadena en el ADNmt también se pueden reparar mediante unión de extremos mediada por microhomología . [189] Aunque hay evidencia de los procesos de reparación de reversión directa y reparación de desajustes en el ADNmt, estos procesos no están bien caracterizados. [185]
Algunos organismos han perdido el ADN mitocondrial por completo. En estos casos, los genes codificados por el ADN mitocondrial se han perdido o transferido al núcleo. [141] Cryptosporidium tiene mitocondrias que carecen de ADN, presumiblemente porque todos sus genes se han perdido o transferido. [190] En Cryptosporidium , las mitocondrias tienen un sistema de generación de ATP alterado que hace que el parásito sea resistente a muchos inhibidores mitocondriales clásicos como el cianuro , la azida y la atovacuona . [190] Se han encontrado mitocondrias que carecen de su propio ADN en un dinoflagelado parásito marino del género Amoebophyra . Este microorganismo, A. cerati , tiene mitocondrias funcionales que carecen de genoma. [191] En especies relacionadas, el genoma mitocondrial todavía tiene tres genes, pero en A. cerati solo se encuentra un único gen mitocondrial, el gen de la citocromo c oxidasa I ( cox1 ), que ha migrado al genoma del núcleo. [192]
El daño y la disfunción posterior en las mitocondrias es un factor importante en una variedad de enfermedades humanas debido a su influencia en el metabolismo celular. Los trastornos mitocondriales a menudo se presentan como trastornos neurológicos, incluido el autismo . [18] También pueden manifestarse como miopatía , diabetes , endocrinopatía múltiple y una variedad de otros trastornos sistémicos. [193] Las enfermedades causadas por mutación en el ADNmt incluyen el síndrome de Kearns-Sayre , el síndrome de MELAS y la neuropatía óptica hereditaria de Leber . [194] En la gran mayoría de los casos, estas enfermedades son transmitidas por una mujer a sus hijos, ya que el cigoto deriva sus mitocondrias y, por lo tanto, su ADNmt del óvulo. Se cree que enfermedades como el síndrome de Kearns-Sayre, el síndrome de Pearson y la oftalmoplejía externa progresiva se deben a reordenamientos del ADNmt a gran escala, mientras que otras enfermedades como el síndrome MELAS, la neuropatía óptica hereditaria de Leber, el síndrome MERRF y otras se deben a mutaciones puntuales en el ADNmt. [193]
También se ha informado que las células cancerosas tolerantes a los fármacos tienen un mayor número y tamaño de mitocondrias, lo que sugiere un aumento en la biogénesis mitocondrial. [195] Un estudio de 2022 en Nature Nanotechnology informó que las células cancerosas pueden secuestrar las mitocondrias de las células inmunes a través de nanotubos de tunelización física. [196]
En otras enfermedades, los defectos en los genes nucleares conducen a la disfunción de las proteínas mitocondriales. Este es el caso de la ataxia de Friedreich , la paraplejía espástica hereditaria y la enfermedad de Wilson . [197] Estas enfermedades se heredan en una relación de dominancia , como se aplica a la mayoría de las otras enfermedades genéticas. Una variedad de trastornos pueden ser causados por mutaciones nucleares de enzimas de fosforilación oxidativa, como la deficiencia de coenzima Q10 y el síndrome de Barth . [193] Las influencias ambientales pueden interactuar con predisposiciones hereditarias y causar enfermedad mitocondrial. Por ejemplo, puede haber un vínculo entre la exposición a pesticidas y la aparición tardía de la enfermedad de Parkinson . [198] [199] Otras patologías con etiología que involucra disfunción mitocondrial incluyen esquizofrenia , trastorno bipolar , demencia , enfermedad de Alzheimer , [200] [201] enfermedad de Parkinson, epilepsia , accidente cerebrovascular , enfermedad cardiovascular , encefalomielitis miálgica/síndrome de fatiga crónica (EM/SFC), retinitis pigmentosa y diabetes mellitus . [202] [203]
El estrés oxidativo mediado por las mitocondrias desempeña un papel en la miocardiopatía en los diabéticos tipo 2. El aumento del suministro de ácidos grasos al corazón aumenta la captación de ácidos grasos por los cardiomiocitos, lo que da como resultado un aumento de la oxidación de ácidos grasos en estas células. Este proceso aumenta los equivalentes reductores disponibles para la cadena de transporte de electrones de las mitocondrias, lo que en última instancia aumenta la producción de especies reactivas de oxígeno (ROS). Las ROS aumentan las proteínas desacopladoras (UCP) y potencian la fuga de protones a través del translocador de nucleótidos de adenina (ANT), cuya combinación desacopla las mitocondrias. El desacoplamiento aumenta entonces el consumo de oxígeno por parte de las mitocondrias, lo que agrava el aumento de la oxidación de ácidos grasos. Esto crea un círculo vicioso de desacoplamiento; además, aunque aumenta el consumo de oxígeno, la síntesis de ATP no aumenta proporcionalmente porque las mitocondrias están desacopladas. Una menor disponibilidad de ATP en última instancia da como resultado un déficit de energía que se presenta como una eficiencia cardíaca reducida y disfunción contráctil. Para agravar el problema, la liberación deficiente de calcio del retículo sarcoplásmico y la recaptación mitocondrial reducida limitan los niveles citosólicos máximos del importante ion de señalización durante la contracción muscular. La concentración reducida de calcio intramitocondrial aumenta la activación de la deshidrogenasa y la síntesis de ATP. Por lo tanto, además de una menor síntesis de ATP debido a la oxidación de ácidos grasos, la síntesis de ATP también se ve afectada por una mala señalización del calcio, lo que causa problemas cardíacos en los diabéticos. [204]
Las mitocondrias también modulan procesos como el desarrollo de células somáticas testiculares, la diferenciación de células madre espermatogoniales, la acidificación luminal, la producción de testosterona en los testículos y más. Por lo tanto, la disfunción de las mitocondrias en los espermatozoides puede ser una causa de infertilidad. [205]
En un esfuerzo por combatir las enfermedades mitocondriales, se ha desarrollado la terapia de reemplazo mitocondrial (TRM). Esta forma de fertilización in vitro utiliza mitocondrias de donantes, lo que evita la transmisión de enfermedades causadas por mutaciones del ADN mitocondrial. [206] Sin embargo, esta terapia aún se está investigando y puede introducir modificaciones genéticas, así como problemas de seguridad. Estas enfermedades son raras, pero pueden ser extremadamente debilitantes y progresivas, lo que plantea cuestiones éticas complejas para las políticas públicas. [207]
Puede haber alguna fuga de los electrones transferidos en la cadena respiratoria para formar especies reactivas de oxígeno . Se pensaba que esto daba como resultado un estrés oxidativo significativo en las mitocondrias con altas tasas de mutación del ADN mitocondrial. [208] Los vínculos hipotéticos entre el envejecimiento y el estrés oxidativo no son nuevos y se propusieron en 1956, [209] que luego se refinó en la teoría de los radicales libres mitocondriales del envejecimiento . [210] Se pensaba que ocurría un círculo vicioso, ya que el estrés oxidativo conduce a mutaciones del ADN mitocondrial, que pueden conducir a anomalías enzimáticas y a un mayor estrés oxidativo.
Durante el proceso de envejecimiento, las mitocondrias pueden sufrir una serie de cambios. [211] Los tejidos de personas mayores muestran una disminución de la actividad enzimática de las proteínas de la cadena respiratoria. [212] Sin embargo, el ADNmt mutado solo se puede encontrar en aproximadamente el 0,2 % de las células muy viejas. [213] Se ha planteado la hipótesis de que las grandes deleciones en el genoma mitocondrial conducen a altos niveles de estrés oxidativo y muerte neuronal en la enfermedad de Parkinson . [214] También se ha demostrado que la disfunción mitocondrial ocurre en la esclerosis lateral amiotrófica . [215] [216]
Dado que las mitocondrias desempeñan un papel fundamental en la función ovárica, al proporcionar el ATP necesario para el desarrollo desde la vesícula germinal hasta el ovocito maduro , una disminución de la función mitocondrial puede provocar inflamación, lo que da lugar a una insuficiencia ovárica prematura y un envejecimiento ovárico acelerado. La disfunción resultante se refleja entonces en daños cuantitativos (como el número de copias de ADNmt y las deleciones de ADNmt), cualitativos (como mutaciones y roturas de cadenas) y oxidativos (como mitocondrias disfuncionales debido a ROS), que no solo son relevantes en el envejecimiento ovárico, sino que perturban la comunicación cruzada ovocito-cúmulo en el ovario, están vinculados a trastornos genéticos (como el síndrome del cromosoma X frágil) y pueden interferir en la selección de embriones. [217]
Las primeras observaciones de estructuras intracelulares que probablemente representaban mitocondrias fueron publicadas en 1857, por el fisiólogo Albert von Kolliker . [218] [219] Richard Altmann , en 1890, las estableció como orgánulos celulares y las llamó "bioblastos". [219] [220] En 1898, Carl Benda acuñó el término "mitocondria" del griego μίτος , mitos , "hilo", y χονδρίον , chondrion , "gránulo". [221] [219] [222] Leonor Michaelis descubrió que el verde Janus puede usarse como una tinción supravital para mitocondrias en 1900. [223] En 1904, Friedrich Meves hizo la primera observación registrada de mitocondrias en plantas en células del nenúfar blanco, Nymphaea alba , [219] [224] y en 1908, junto con Claudius Regaud , sugirió que contienen proteínas y lípidos. Benjamin F. Kingsbury, en 1912, las relacionó por primera vez con la respiración celular, pero casi exclusivamente basándose en observaciones morfológicas. [225] [219] En 1913, Otto Heinrich Warburg relacionó la respiración con partículas que había obtenido de extractos de hígado de cobaya y que llamó "grana". [226] Warburg y Heinrich Otto Wieland , que también habían postulado un mecanismo de partículas similar, no estaban de acuerdo sobre la naturaleza química de la respiración. No fue hasta 1925, cuando David Keilin descubrió los citocromos , que se describió la cadena respiratoria . [219]
En 1939, experimentos con células musculares picadas demostraron que la respiración celular usando una molécula de oxígeno puede formar cuatro moléculas de trifosfato de adenosina (ATP), y en 1941, el concepto de los enlaces de fosfato del ATP como una forma de energía en el metabolismo celular fue desarrollado por Fritz Albert Lipmann . En los años siguientes, el mecanismo detrás de la respiración celular se elaboró más a fondo, aunque su vínculo con las mitocondrias no se conocía. [219] La introducción del fraccionamiento de tejidos por Albert Claude permitió aislar las mitocondrias de otras fracciones celulares y realizar análisis bioquímicos solo en ellas. En 1946, concluyó que la citocromo oxidasa y otras enzimas responsables de la cadena respiratoria estaban aisladas en las mitocondrias. Eugene Kennedy y Albert Lehninger descubrieron en 1948 que las mitocondrias son el sitio de la fosforilación oxidativa en eucariotas. Con el tiempo, el método de fraccionamiento se desarrolló aún más, mejorando la calidad de las mitocondrias aisladas y se determinó que otros elementos de la respiración celular ocurren en las mitocondrias. [219]
Las primeras micrografías electrónicas de alta resolución aparecieron en 1952, reemplazando las tinciones de Janus Green como la forma preferida de visualizar las mitocondrias. [219] Esto condujo a un análisis más detallado de la estructura de las mitocondrias, incluida la confirmación de que estaban rodeadas por una membrana. También mostró una segunda membrana dentro de las mitocondrias que se plegaba en crestas que dividían la cámara interna y que el tamaño y la forma de las mitocondrias variaban de una célula a otra.
El término popular "central eléctrica de la célula" fue acuñado por Philip Siekevitz en 1957. [4] [227]
En 1967, se descubrió que las mitocondrias contenían ribosomas . [228] En 1968, se desarrollaron métodos para mapear los genes mitocondriales, y el mapa genético y físico del ADN mitocondrial de la levadura se completó en 1976. [219]
Por lo tanto, la biogénesis mitocondrial se define como el proceso a través del cual las células aumentan su masa mitocondrial individual [3]. ... La biogénesis mitocondrial ocurre por el crecimiento y la división de orgánulos preexistentes y está coordinada temporalmente con los eventos del ciclo celular [1].
La biogénesis mitocondrial (MB) es el mecanismo esencial por el cual las células controlan el número de mitocondrias.
p. 344: It is a little surprising that organisms with somewhat different codes do not coexist.Further discussion Archived September 3, 2011, at the Wayback Machine.
General