
La recombinación homóloga es un tipo de recombinación genética en el que se intercambia información genética entre dos moléculas similares o idénticas de ácidos nucleicos bicatenarios o monocatenarios (normalmente ADN como en los organismos celulares , pero también puede ser ARN en los virus ).
Las células utilizan ampliamente la recombinación homóloga para reparar con precisión las roturas dañinas del ADN que se producen en ambas hebras de ADN, conocidas como roturas de doble hebra (DSB), en un proceso llamado reparación recombinante homóloga (HRR). [1]
La recombinación homóloga también produce nuevas combinaciones de secuencias de ADN durante la meiosis , el proceso mediante el cual los eucariotas producen células gametas , como los espermatozoides y los óvulos en los animales. Estas nuevas combinaciones de ADN representan una variación genética en la descendencia, lo que a su vez permite que las poblaciones se adapten durante el curso de la evolución . [2]
La recombinación homóloga también se utiliza en la transferencia horizontal de genes para intercambiar material genético entre diferentes cepas y especies de bacterias y virus. La transferencia horizontal de genes es el mecanismo principal para la propagación de la resistencia a los antibióticos en las bacterias.
Aunque la recombinación homóloga varía ampliamente entre diferentes organismos y tipos de células, para el ADN bicatenario ( ADNbc ) la mayoría de las formas implican los mismos pasos básicos. Después de que se produce una rotura de doble hebra, se cortan secciones de ADN alrededor de los extremos 5' de la rotura en un proceso llamado resección . En el paso de invasión de la cadena que sigue, un extremo 3' que sobresale de la molécula de ADN rota "invade" una molécula de ADN similar o idéntica que no está rota. Después de la invasión de la hebra, la secuencia adicional de eventos puede seguir cualquiera de las dos vías principales que se analizan a continuación (ver Modelos); la vía DSBR (reparación de roturas de doble hebra) o la vía SDSA (recocido de hebras dependiente de síntesis). La recombinación homóloga que ocurre durante la reparación del ADN tiende a dar como resultado productos sin entrecruzamiento, restaurando de hecho la molécula de ADN dañada tal como existía antes de la rotura de la doble hebra.
La recombinación homóloga se conserva en los tres dominios de la vida, así como en los virus de ADN y ARN , lo que sugiere que es un mecanismo biológico casi universal. El descubrimiento de genes para la recombinación homóloga en protistas (un grupo diverso de microorganismos eucariotas ) se ha interpretado como evidencia de que la recombinación homóloga surgió temprano en la evolución de los eucariotas. Dado que su disfunción se ha asociado fuertemente con una mayor susceptibilidad a varios tipos de cáncer , las proteínas que facilitan la recombinación homóloga son temas de investigación activa. La recombinación homóloga también se utiliza en la selección de genes , una técnica para introducir cambios genéticos en organismos objetivo. Por el desarrollo de esta técnica, Mario Capecchi , Martin Evans y Oliver Smithies recibieron el Premio Nobel de Fisiología o Medicina en 2007 ; Capecchi [3] y Smithies [4] descubrieron de forma independiente aplicaciones en células madre embrionarias de ratón; sin embargo, los mecanismos altamente conservados que subyacen al modelo de reparación DSB, incluida la integración homóloga uniforme del ADN transformado (terapia génica), se demostraron por primera vez en experimentos con plásmidos por Orr- Weaver, Szostack y Rothstein. [5] [6] [7] La investigación del DSB inducido por plásmidos, utilizando irradiación γ [8] en las décadas de 1970 y 1980, condujo a experimentos posteriores utilizando endonucleasas (por ejemplo, I-SceI) para cortar cromosomas para la ingeniería genética de células de mamíferos. , donde la recombinación no homóloga es más frecuente que en la levadura. [9]

A principios del siglo XX, William Bateson y Reginald Punnett encontraron una excepción a uno de los principios de herencia descritos originalmente por Gregor Mendel en la década de 1860. En contraste con la noción de Mendel de que los rasgos se clasifican independientemente cuando se transmiten de padres a hijos (por ejemplo, que el color del pelo de un gato y la longitud de su cola se heredan independientemente uno del otro), Bateson y Punnett demostraron que ciertos genes asociados con rasgos físicos se pueden heredar juntos. o genéticamente ligados . [10] [11] En 1911, después de observar que los rasgos vinculados en ocasiones podían heredarse por separado, Thomas Hunt Morgan sugirió que pueden ocurrir " cruzamientos " entre genes vinculados, [12] donde uno de los genes vinculados se cruza físicamente con otro diferente. cromosoma . Dos décadas más tarde, Barbara McClintock y Harriet Creighton demostraron que el cruce cromosómico se produce durante la meiosis , [13] [14] el proceso de división celular mediante el cual se producen los espermatozoides y los óvulos . El mismo año del descubrimiento de McClintock, Curt Stern demostró que el entrecruzamiento, más tarde llamado "recombinación", también podría ocurrir en células somáticas como los glóbulos blancos y las células de la piel que se dividen mediante mitosis . [13] [15]
En 1947, el microbiólogo Joshua Lederberg demostró que las bacterias, que se suponía que se reproducían sólo asexualmente mediante fisión binaria , son capaces de realizar recombinación genética, que es más similar a la reproducción sexual. Este trabajo estableció a E. coli como organismo modelo en genética [16] y ayudó a Lederberg a ganar el Premio Nobel de Fisiología o Medicina en 1958 . [17] Basándose en estudios en hongos , en 1964 Robin Holliday propuso un modelo para la recombinación en la meiosis que introdujo detalles clave de cómo puede funcionar el proceso, incluido el intercambio de material entre cromosomas a través de uniones Holliday . [18] En 1983, Jack Szostak y sus colegas presentaron un modelo ahora conocido como vía DSBR, que tenía en cuenta observaciones no explicadas por el modelo de Holliday. [18] [7] Durante la siguiente década, experimentos en Drosophila , levaduras en ciernes y células de mamíferos condujeron a la aparición de otros modelos de recombinación homóloga, llamados vías SDSA, que no siempre dependen de uniones Holliday. [18]
Gran parte del trabajo posterior para identificar proteínas involucradas en el proceso y determinar sus mecanismos ha sido realizado por varios individuos, entre ellos James Haber , Patrick Sung , Stephen Kowalczykowski y otros.
La recombinación homóloga (HR) es esencial para la división celular en eucariotas como plantas, animales, hongos y protistas. La recombinación homóloga repara las roturas de doble cadena del ADN causadas por radiaciones ionizantes o sustancias químicas que dañan el ADN. [19] Si no se reparan, estas roturas de doble cadena pueden provocar una reordenación a gran escala de los cromosomas en las células somáticas , [20] que a su vez puede provocar cáncer. [21]
Además de reparar el ADN, la recombinación homóloga también ayuda a producir diversidad genética cuando las células se dividen en la meiosis para convertirse en gametos especializados : espermatozoides u óvulos en animales, polen u óvulos en plantas y esporas en hongos . Lo hace facilitando el cruce cromosómico , en el que regiones de ADN similares pero no idénticas se intercambian entre cromosomas homólogos . [22] [23] Esto crea nuevas combinaciones de genes, posiblemente beneficiosas, que pueden dar a la descendencia una ventaja evolutiva. [24] El cruce cromosómico a menudo comienza cuando una proteína llamada Spo11 produce una rotura bicatenaria específica en el ADN. [25] Estos sitios no están ubicados al azar en los cromosomas; generalmente en regiones promotoras intergénicas y preferentemente en dominios ricos en GC [26] Estos sitios de rotura de doble cadena a menudo ocurren en puntos críticos de recombinación , regiones en los cromosomas que tienen entre 1000 y 2000 pares de bases de longitud y altas tasas de recombinación. La ausencia de un punto de recombinación entre dos genes en el mismo cromosoma a menudo significa que esos genes serán heredados por generaciones futuras en igual proporción. Esto representa un vínculo entre los dos genes mayor de lo que se esperaría de genes que se distribuyen de forma independiente durante la meiosis. [27]

Las roturas de doble cadena se pueden reparar mediante recombinación homóloga, unión de extremos mediada por polimerasa theta (TMEJ) o mediante unión de extremos no homólogos (NHEJ). [28] NHEJ es un mecanismo de reparación del ADN que, a diferencia de la recombinación homóloga, no requiere una secuencia homóloga larga para guiar la reparación. El uso de recombinación homóloga o NHEJ para reparar roturas de doble hebra está determinado en gran medida por la fase del ciclo celular . La recombinación homóloga repara el ADN antes de que la célula entre en mitosis (fase M). Ocurre durante y poco después de la replicación del ADN , en las fases S y G 2 del ciclo celular, cuando las cromátidas hermanas están más fácilmente disponibles. [29] En comparación con los cromosomas homólogos, que son similares a otro cromosoma pero a menudo tienen alelos diferentes , las cromátidas hermanas son una plantilla ideal para la recombinación homóloga porque son una copia idéntica de un cromosoma determinado. Cuando no hay una plantilla homóloga disponible o cuando no se puede acceder a la plantilla debido a un defecto en la recombinación homóloga, la rotura se repara mediante TMEJ en las fases S y G 2 del ciclo celular. A diferencia de la recombinación homóloga y TMEJ, NHEJ predomina en la fase G 1 del ciclo celular, cuando la célula está creciendo pero aún no está lista para dividirse. Ocurre con menos frecuencia después de la fase G 1 , pero mantiene al menos cierta actividad durante todo el ciclo celular. Los mecanismos que regulan la recombinación homóloga y NHEJ a lo largo del ciclo celular varían ampliamente entre especies. [30]
Las quinasas dependientes de ciclina (CDK), que modifican la actividad de otras proteínas añadiéndoles grupos fosfato (es decir, fosforilándolas ), son reguladores importantes de la recombinación homóloga en eucariotas. [30] Cuando comienza la replicación del ADN en la levadura en ciernes, la quinasa Cdc28 dependiente de ciclina comienza la recombinación homóloga fosforilando la proteína Sae2 . [31] Después de ser activado mediante la adición de un fosfato, Sae2 provoca que se realice un corte limpio cerca de una rotura de doble cadena en el ADN. No está claro si la endonucleasa responsable de este corte es la propia Sae2 u otra proteína, Mre11 . [32] Esto permite que un complejo proteico que incluye Mre11, conocido como complejo MRX , se una al ADN y comienza una serie de reacciones impulsadas por proteínas que intercambian material entre dos moléculas de ADN. [33]
El empaquetamiento del ADN eucariota en cromatina presenta una barrera para todos los procesos basados en el ADN que requieren el reclutamiento de enzimas en sus sitios de acción. Para permitir la reparación del ADN por recombinación homóloga (HR), se debe remodelar la cromatina. En eucariotas, los complejos de remodelación de la cromatina dependientes de ATP y las enzimas modificadoras de histonas son dos factores predominantes empleados para lograr este proceso de remodelación. [34]
La relajación de la cromatina ocurre rápidamente en el sitio de daño del ADN. [35] En uno de los primeros pasos, la proteína quinasa activada por estrés, la quinasa N-terminal c-Jun (JNK) , fosforila SIRT6 en la serina 10 en respuesta a roturas de doble cadena u otros daños en el ADN. [36] Esta modificación postraduccional facilita la movilización de SIRT6 a los sitios de daño del ADN y es necesaria para el reclutamiento eficiente de la poli (ADP-ribosa) polimerasa 1 (PARP1) a los sitios de rotura del ADN y para la reparación eficiente de los DSB. [36] La proteína PARP1 comienza a aparecer en los sitios dañados del ADN en menos de un segundo, con una acumulación máxima de la mitad dentro de 1,6 segundos después de que se produce el daño. [37] A continuación, el remodelador de cromatina Alc1 se une rápidamente al producto de la acción de PARP1, una cadena de ribosa poli-ADP, y Alc1 completa su llegada al daño del ADN dentro de los 10 segundos posteriores a la aparición del daño. [35] Aproximadamente la mitad de la relajación máxima de la cromatina, presumiblemente debido a la acción de Alc1, ocurre en 10 segundos. [35] Esto permite el reclutamiento de la enzima reparadora del ADN MRE11 , para iniciar la reparación del ADN, en 13 segundos. [37]
γH2AX, la forma fosforilada de H2AX , también participa en los primeros pasos que conducen a la descondensación de la cromatina después de que se rompe la doble cadena del ADN. La variante de histona H2AX constituye aproximadamente el 10% de las histonas H2A en la cromatina humana. [38] γH2AX (H2AX fosforilado en serina 139) se puede detectar tan pronto como 20 segundos después de la irradiación de las células (con formación de rotura de la doble cadena del ADN), y la mitad de la acumulación máxima de γH2AX ocurre en un minuto. [38] La extensión de la cromatina con γH2AX fosforilado es de aproximadamente dos millones de pares de bases en el sitio de una rotura de la doble hebra del ADN. [38] γH2AX por sí solo no causa la descondensación de la cromatina, pero dentro de los 30 segundos posteriores a la irradiación, la proteína RNF8 se puede detectar en asociación con γH2AX. [39] RNF8 media la descondensación extensa de la cromatina, a través de su interacción posterior con CHD4 , [40] un componente de la remodelación del nucleosoma y el complejo de desacetilasa NuRD .
Después de sufrir una relajación posterior al daño del ADN, seguida de la reparación del ADN, la cromatina se recupera a un estado de compactación cercano a su nivel previo al daño después de aproximadamente 20 minutos. [35]
En los vertebrados, los lugares en los que se produce la recombinación están determinados por los lugares de unión de PRDM9 , una proteína que reconoce un motivo de secuencia específico por su conjunto de dedos de zinc. [41] En estos sitios, otra proteína, SPO11, cataliza roturas de doble cadena (DSB) que inician la recombinación, un subconjunto de las cuales se repara mediante recombinación con el cromosoma homólogo. PRDM9 deposita marcas de metilación de histonas H3K4me3 y H3K36me3 en los sitios a los que se une, y esta actividad metiltransferasa es esencial para su papel en el posicionamiento de DSB. Después de su formación, los sitios DSB se procesan mediante resección, lo que da como resultado un ADN monocatenario (ADNss) que se adorna con DMC1. Desde la mitad del cigoteno hasta el paquiteno temprano, como parte del proceso de reparación recombinacional, DMC1 se disocia del ssDNA y los recuentos disminuyen hasta que todas las roturas (excepto las de los cromosomas XY) se reparan en el paquiteno tardío. Varias otras proteínas están involucradas en este proceso, incluida ZCWPW1, la [42] primera proteína ubicada directamente junto a las marcas duales de histonas de PRDM9. ZCWPW1 es importante para la reparación de DSB homóloga, no para el posicionamiento.
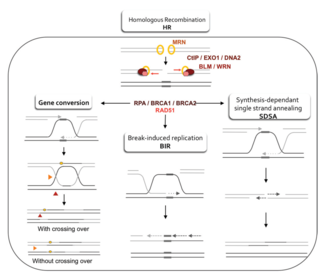
Dos modelos principales de cómo la recombinación homóloga repara las roturas de doble hebra en el ADN son la vía de reparación de roturas de doble hebra (DSBR) (a veces llamada modelo de doble unión Holliday ) y la vía de recocido de hebras dependiente de la síntesis (SDSA). [43] Las dos vías son similares en sus primeros pasos. Después de que se produce una rotura de doble hebra, el complejo MRX ( complejo MRN en humanos) se une al ADN a ambos lados de la rotura. A continuación se lleva a cabo una resección, en la que se corta el ADN alrededor de los extremos 5' de la rotura. Esto sucede en dos pasos distintos: primero, el complejo MRX recluta la proteína Sae2, y estas dos proteínas recortan los extremos 5' a cada lado de la rotura para crear cortos salientes 3' de ADN monocatenario; en el segundo paso, la helicasa Sgs1 y las nucleasas Exo1 y Dna2 continúan la resección 5'→3' . Como helicasa , Sgs1 "descomprime" el ADN de doble hebra, mientras que la actividad nucleasa de Exo1 y Dna2 les permite cortar el ADN monocatenario producido por Sgs1. [31]
La proteína RPA , que tiene una alta afinidad por el ADN monocatenario, se une a los salientes 3'. [44] Con la ayuda de varias otras proteínas que median en el proceso, la proteína Rad51 (y Dmc1 , en la meiosis) forma un filamento de ácido nucleico y proteína en la hebra única de ADN recubierta con RPA. Este filamento de nucleoproteína comienza a buscar secuencias de ADN similares a las del saliente 3'. Después de encontrar dicha secuencia, el filamento de nucleoproteína monocatenaria se mueve hacia (invade) el dúplex de ADN receptor similar o idéntico en un proceso llamado invasión de hebras . En las células que se dividen mediante mitosis, el dúplex de ADN receptor es generalmente una cromátida hermana, que es idéntica a la molécula de ADN dañada y proporciona una plantilla para la reparación. Sin embargo, en la meiosis, el ADN receptor tiende a ser de un cromosoma homólogo similar pero no necesariamente idéntico. [43] Se forma un bucle de desplazamiento ( bucle D ) durante la invasión de la hebra entre la hebra saliente 3' invasora y el cromosoma homólogo. Después de la invasión de la cadena, una ADN polimerasa extiende el extremo de la cadena 3' invasora sintetizando ADN nuevo. Esto cambia el bucle D a una estructura en forma de cruz conocida como cruce Holliday . Después de esto, se produce más síntesis de ADN en la cadena invasora (es decir, uno de los salientes 3' originales), restaurando efectivamente la cadena en el cromosoma homólogo que fue desplazada durante la invasión de la cadena. [43]

Después de las etapas de resección, invasión de hebras y síntesis de ADN, las vías DSBR y SDSA se vuelven distintas. [43] La vía DSBR es única porque el segundo saliente 3' (que no participó en la invasión de la cadena) también forma una unión Holliday con el cromosoma homólogo. Las uniones dobles de Holliday se convierten luego en productos de recombinación mediante mellas endonucleasas , un tipo de endonucleasa de restricción que corta solo una hebra de ADN. La vía DSBR comúnmente da como resultado un cruce, aunque a veces puede dar como resultado productos que no se cruzan; La capacidad de una molécula de ADN rota para recolectar secuencias de loci donantes separados se demostró en levaduras en gemación mitótica utilizando plásmidos o inducción de eventos cromosómicos con endonucleasa. [45] [46] Debido a esta tendencia al cruce cromosómico, la vía DSBR es un modelo probable de cómo se produce la recombinación homóloga cruzada durante la meiosis. [22]
Si la recombinación en la vía DSBR da como resultado un cruce cromosómico está determinado por cómo se corta o "resuelve" la doble unión de Holliday. Se producirá un cruce cromosómico si se corta una unión de Holliday en la hebra que se cruza y la otra unión de Holliday se corta en la hebra que no se cruza (en la Figura 5, a lo largo de las puntas de flecha horizontales de color púrpura en una unión de Holliday y a lo largo de las puntas de flecha verticales de color naranja en la otra ). Alternativamente, si las dos uniones Holliday se cortan en las hebras que se cruzan (a lo largo de las puntas de flecha horizontales de color púrpura en ambas uniones Holliday en la Figura 5), entonces se producirán cromosomas sin cruce. [47]
La recombinación homóloga a través de la vía SDSA ocurre en células que se dividen mediante mitosis y meiosis y da como resultado productos que no se cruzan. En este modelo, la cadena 3' invasora se extiende a lo largo del dúplex de ADN del receptor mediante una ADN polimerasa y se libera a medida que la unión Holliday entre las moléculas de ADN del donante y del receptor se desliza en un proceso llamado migración de rama . El extremo 3' recién sintetizado de la cadena invasora puede entonces hibridarse con el otro extremo 3' del cromosoma dañado mediante apareamiento de bases complementarias. Después de que las hebras se hibridan, a veces puede quedar un pequeño colgajo de ADN. Se eliminan dichos colgajos y la vía SDSA finaliza con el resellado, también conocido como ligadura , de los espacios monocatenarios restantes. [48]
Durante la mitosis, la principal vía de recombinación homóloga para reparar las roturas de la doble hebra del ADN parece ser la vía SDSA (en lugar de la vía DSBR). [49] La vía SDSA produce recombinantes no cruzados (Figura 5). Durante la meiosis, los recombinantes no cruzados también ocurren con frecuencia y parecen surgir también principalmente por la vía SDSA. [49] [50] Los eventos de recombinación sin cruce que ocurren durante la meiosis probablemente reflejan casos de reparación de daños en la doble cadena del ADN u otros tipos de daños en el ADN.

"La vía de recocido de una sola hebra (SSA) de recombinación homóloga repara las roturas de doble hebra entre dos secuencias repetidas ". La vía SSA es única porque no requiere una molécula de ADN separada similar o idéntica, como las vías DSBR o SDSA de recombinación homóloga. En cambio, la vía SSA solo requiere un único dúplex de ADN y utiliza las secuencias repetidas como secuencias idénticas que la recombinación homóloga necesita para la reparación. La vía es relativamente simple en concepto: después de que dos hebras del mismo dúplex de ADN se cortan alrededor del sitio de la rotura de la doble hebra, los dos salientes 3' resultantes se alinean y se hibridan entre sí, restaurando el ADN como un dúplex continuo. . [48] [51]
A medida que se corta el ADN alrededor de la rotura de la doble cadena, los salientes 3' monocatenarios que se producen se recubren con la proteína RPA , lo que evita que los salientes 3' se peguen a sí mismos. [52] Luego, una proteína llamada Rad52 se une a cada una de las secuencias repetidas a cada lado de la rotura y las alinea para permitir que las dos secuencias repetidas complementarias se hibriden. [52] Una vez completado el recocido, las aletas no homólogas sobrantes de los salientes 3' se cortan mediante un conjunto de nucleasas, conocidas como Rad1/Rad10, que son llevadas a las aletas por las proteínas Saw1 y Slx4 . [52] [53] La nueva síntesis de ADN llena cualquier vacío y la ligación restaura el dúplex de ADN como dos hebras continuas. [54] La secuencia de ADN entre las repeticiones siempre se pierde, al igual que una de las dos repeticiones. La vía SSA se considera mutagénica ya que produce este tipo de eliminaciones de material genético. [48]
Durante la replicación del ADN , a veces se pueden encontrar roturas de doble cadena en las bifurcaciones de replicación cuando la ADN helicasa abre la cadena plantilla. Estos defectos se reparan en la vía de replicación inducida por rotura (BIR) de recombinación homóloga. Los mecanismos moleculares precisos de la vía BIR aún no están claros. Tres mecanismos propuestos tienen la invasión de hebras como paso inicial, pero difieren en cómo modelan la migración del bucle D y las fases posteriores de recombinación. [55]
La vía BIR también puede ayudar a mantener la longitud de los telómeros (regiones de ADN al final de los cromosomas eucarióticos) en ausencia de (o en cooperación con) la telomerasa . Sin copias funcionales de la enzima telomerasa, los telómeros normalmente se acortan con cada ciclo de mitosis, lo que eventualmente bloquea la división celular y conduce a la senescencia . En células de levadura en ciernes donde la telomerasa se ha inactivado mediante mutaciones, se ha observado que dos tipos de células "supervivientes" evitan la senescencia durante más tiempo de lo esperado al alargar sus telómeros a través de vías BIR. [55]
Mantener la longitud de los telómeros es fundamental para la inmortalización celular , una característica clave del cáncer. La mayoría de los cánceres mantienen los telómeros mediante la regulación positiva de la telomerasa. Sin embargo, en varios tipos de cáncer humano, una vía similar a BIR ayuda a sostener algunos tumores al actuar como un mecanismo alternativo de mantenimiento de los telómeros. [56] Este hecho ha llevado a los científicos a investigar si dichos mecanismos de mantenimiento de los telómeros basados en la recombinación podrían frustrar los medicamentos contra el cáncer como los inhibidores de la telomerasa . [57]

La recombinación homóloga es un importante proceso de reparación del ADN en las bacterias. También es importante para producir diversidad genética en poblaciones bacterianas, aunque el proceso difiere sustancialmente de la recombinación meiótica , que repara daños en el ADN y genera diversidad en genomas eucariotas . La recombinación homóloga ha sido la más estudiada y mejor comprendida para Escherichia coli . [59] Las roturas del ADN de doble hebra en las bacterias se reparan mediante la vía RecBCD de recombinación homóloga. Se cree que las roturas que se producen en una sola de las dos cadenas de ADN, conocidas como espacios de una sola cadena, se reparan mediante la vía RecF. [60] Tanto la vía RecBCD como la RecF incluyen una serie de reacciones conocidas como migración de ramas , en la que se intercambian hebras individuales de ADN entre dos moléculas cruzadas de ADN dúplex, y resolución , en la que esas dos moléculas cruzadas de ADN se cortan y restauran. a su estado normal de doble cadena.
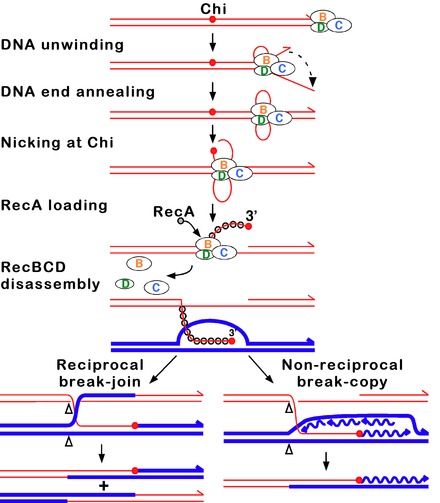

La vía RecBCD es la principal vía de recombinación utilizada en muchas bacterias para reparar roturas de doble hebra en el ADN, y las proteínas se encuentran en una amplia gama de bacterias. [63] [64] [65] Estas roturas de doble cadena pueden ser causadas por la luz ultravioleta y otras radiaciones , así como por mutágenos químicos . Las roturas de doble cadena también pueden surgir por replicación del ADN a través de una muesca o espacio de una sola cadena. Esta situación provoca lo que se conoce como una bifurcación de replicación colapsada y se soluciona mediante varias vías de recombinación homóloga, incluida la vía RecBCD. [66]
En esta vía, un complejo enzimático de tres subunidades llamado RecBCD inicia la recombinación uniéndose a un extremo romo o casi romo de una rotura en el ADN de doble hebra. Después de que RecBCD se une al extremo del ADN, las subunidades RecB y RecD comienzan a descomprimir el dúplex de ADN mediante la actividad de helicasa . La subunidad RecB también tiene un dominio nucleasa , que corta la hebra única de ADN que emerge del proceso de descompresión. Este descomprimido continúa hasta que RecBCD encuentra una secuencia de nucleótidos específica (5'-GCTGGTGG-3') conocida como sitio Chi . [sesenta y cinco]
Al encontrar un sitio Chi, la actividad de la enzima RecBCD cambia drásticamente. [64] [61] [67] El desenrollado del ADN se detiene durante unos segundos y luego se reanuda aproximadamente a la mitad de la velocidad inicial. Esto probablemente se debe a que la helicasa RecB más lenta desenrolla el ADN después de Chi, en lugar de la helicasa RecD más rápida, que desenrolla el ADN antes de Chi. [68] [69] El reconocimiento del sitio Chi también cambia la enzima RecBCD de modo que corta la cadena de ADN con Chi y comienza a cargar múltiples proteínas RecA en el ADN monocatenario con el extremo 3' recién generado. El filamento de nucleoproteína recubierto con RecA resultante busca secuencias similares de ADN en un cromosoma homólogo. El proceso de búsqueda induce el estiramiento del dúplex de ADN, lo que mejora el reconocimiento de homología (un mecanismo denominado corrección conformacional [70] [71] [72] ). Al encontrar dicha secuencia, el filamento de nucleoproteína monocatenaria se mueve hacia el dúplex de ADN del receptor homólogo en un proceso llamado invasión de cadena . [73] El saliente 3' invasor hace que una de las hebras del dúplex de ADN del receptor se desplace y forme un bucle D. Si se corta el bucle D, otro intercambio de hebras forma una estructura en forma de cruz llamada unión Holliday . [65] La resolución de la unión Holliday mediante alguna combinación de RuvABC o RecG puede producir dos moléculas de ADN recombinantes con tipos genéticos recíprocos, si las dos moléculas de ADN que interactúan difieren genéticamente. Alternativamente, el extremo 3' invasor cerca de Chi puede cebar la síntesis de ADN y formar una bifurcación de replicación. Este tipo de resolución produce sólo un tipo de recombinante (no recíproco).
Las bacterias parecen utilizar la vía RecF de recombinación homóloga para reparar huecos monocatenarios en el ADN. Cuando la vía RecBCD se inactiva mediante mutaciones y mutaciones adicionales inactivan las nucleasas SbcCD y ExoI, la vía RecF también puede reparar roturas de doble cadena del ADN. [74] En la vía RecF, la helicasa RecQ desenrolla el ADN y la nucleasa RecJ degrada la hebra con un extremo 5', dejando intacta la hebra con el extremo 3'. La proteína RecA se une a esta cadena y es ayudada por las proteínas RecF, RecO y RecR o estabilizada por ellas. A continuación, el filamento de nucleoproteína RecA busca un ADN homólogo e intercambia lugares con la hebra idéntica o casi idéntica del ADN homólogo.
Aunque las proteínas y los mecanismos específicos involucrados en sus fases iniciales difieren, las dos vías son similares en que ambas requieren ADN monocatenario con un extremo 3' y la proteína RecA para la invasión de la cadena. Las vías también son similares en sus fases de migración de ramas , en las que la unión de Holliday se desliza en una dirección, y de resolución , en la que las enzimas escinden las uniones de Holliday. [75] [76] El tipo de resolución alternativa, no recíproca, también puede ocurrir por cualquiera de las dos vías.
Inmediatamente después de la invasión de la cadena, la unión Holliday se mueve a lo largo del ADN vinculado durante el proceso de migración de la rama. Es en este movimiento de la unión Holliday donde se intercambian los pares de bases entre los dos dúplex de ADN homólogos. Para catalizar la migración de ramas, la proteína RuvA primero reconoce y se une a la unión Holliday y recluta la proteína RuvB para formar el complejo RuvAB. Dos conjuntos de proteínas RuvB, cada uno de los cuales forma una ATPasa en forma de anillo , se cargan en lados opuestos de la unión Holliday, donde actúan como bombas gemelas que proporcionan la fuerza para la migración de las ramas. Entre esos dos anillos de RuvB, dos conjuntos de proteínas RuvA se ensamblan en el centro de la unión Holliday de modo que el ADN en la unión queda intercalado entre cada conjunto de RuvA. Las hebras de ambos dúplex de ADN (el dúplex "donante" y el "receptor") se desenrollan en la superficie de RuvA mientras son guiadas por la proteína de un dúplex al otro. [77] [78]
En la fase de resolución de la recombinación, se cortan todas las uniones Holliday formadas por el proceso de invasión de hebras, restaurando así dos moléculas de ADN separadas. Esta escisión se realiza mediante la interacción del complejo RuvAB con RuvC, que juntos forman el complejo RuvABC . RuvC es una endonucleasa que corta la secuencia degenerada 5'-(A/T)TT(G/C)-3'. La secuencia se encuentra con frecuencia en el ADN, aproximadamente una vez cada 64 nucleótidos. [78] Antes de cortar, RuvC probablemente obtiene acceso a la unión Holliday desplazando uno de los dos tetrámeros de RuvA que cubren el ADN allí. [77] La recombinación da como resultado productos de "empalme" o "parche", dependiendo de cómo RuvC escinde la unión Holliday. [78] Los productos de empalme son productos cruzados, en los que hay una reordenación del material genético alrededor del sitio de recombinación. Los productos de parche, por otro lado, son productos no cruzados en los que no existe tal reordenamiento y sólo hay un "parche" de ADN híbrido en el producto de recombinación. [79]
La recombinación homóloga es un método importante para integrar el ADN del donante en el genoma de un organismo receptor en la transferencia horizontal de genes , el proceso mediante el cual un organismo incorpora ADN extraño de otro organismo sin ser descendiente de ese organismo. La recombinación homóloga requiere que el ADN entrante sea muy similar al genoma receptor, por lo que la transferencia horizontal de genes suele limitarse a bacterias similares. [80] Los estudios en varias especies de bacterias han establecido que existe una disminución log-lineal en la frecuencia de recombinación al aumentar la diferencia en la secuencia entre el ADN del huésped y el receptor. [81] [82] [83]
En la conjugación bacteriana , donde el ADN se transfiere entre bacterias a través del contacto directo de célula a célula, la recombinación homóloga ayuda a integrar el ADN extraño en el genoma del huésped a través de la vía RecBCD. La enzima RecBCD promueve la recombinación después de que el ADN se convierte de ADN monocatenario (en cuya forma ingresa originalmente a la bacteria) a ADN bicatenario durante la replicación. La vía RecBCD también es esencial para la fase final de transducción , un tipo de transferencia genética horizontal en la que un virus transfiere el ADN de una bacteria a otra . El ADN bacteriano extraño a veces se incorpora mal en la cabeza de la cápside de las partículas del virus bacteriófago a medida que el ADN se empaqueta en nuevos bacteriófagos durante la replicación viral. Cuando estos nuevos bacteriófagos infectan a otras bacterias, el ADN de la bacteria huésped anterior se inyecta en la nueva bacteria huésped como ADN de doble hebra. Luego, la enzima RecBCD incorpora este ADN de doble cadena en el genoma del nuevo huésped bacteriano. [sesenta y cinco]
La transformación bacteriana natural implica la transferencia de ADN de una bacteria donante a una bacteria receptora, donde tanto el donante como el receptor son normalmente de la misma especie . La transformación, a diferencia de la conjugación y transducción bacteriana, depende de numerosos productos genéticos bacterianos que interactúan específicamente para realizar este proceso. [84] Por lo tanto, la transformación es claramente una adaptación bacteriana para la transferencia de ADN. Para que una bacteria se una, tome e integre el ADN del donante en su cromosoma residente mediante recombinación homóloga, primero debe entrar en un estado fisiológico especial denominado competencia . La familia de genes RecA / Rad51 / DMC1 desempeña un papel central en la recombinación homóloga durante la transformación bacteriana como lo hace durante la meiosis y la mitosis eucariotas. Por ejemplo, la proteína RecA es esencial para la transformación en Bacillus subtilis y Streptococcus pneumoniae , [85] y la expresión del gen RecA se induce durante el desarrollo de la competencia para la transformación en estos organismos.
Como parte del proceso de transformación, la proteína RecA interactúa con el ADN monocatenario (ssDNA) entrante para formar nucleofilamentos RecA/ssDNA que escanean el cromosoma residente en busca de regiones de homología y llevan el ssDNA entrante a la región correspondiente, donde se intercambian las cadenas y los homólogos. se produce la recombinación. [86] Por lo tanto, el proceso de recombinación homóloga durante la transformación bacteriana tiene similitudes fundamentales con la recombinación homóloga durante la meiosis .
La recombinación homóloga ocurre en varios grupos de virus. En los virus de ADN como el herpesvirus , la recombinación se produce mediante un mecanismo de ruptura y reinserción como en las bacterias y los eucariotas. [87] También hay evidencia de recombinación en algunos virus de ARN , específicamente virus de ARN monocatenario de sentido positivo como retrovirus , picornavirus y coronavirus . Existe controversia sobre si se produce recombinación homóloga en virus ssRNA de sentido negativo como la gripe . [88]
En los virus de ARN, la recombinación homóloga puede ser precisa o imprecisa. En el tipo preciso de recombinación ARN-ARN, no hay diferencia entre las dos secuencias de ARN parentales y la región de ARN cruzada resultante. Debido a esto, a menudo es difícil determinar la ubicación de los eventos de cruce entre dos secuencias de ARN recombinantes. En la recombinación homóloga de ARN imprecisa, la región de cruce tiene alguna diferencia con las secuencias de ARN parentales, causada por adición, eliminación u otra modificación de nucleótidos. El nivel de precisión en el cruce está controlado por el contexto de secuencia de las dos cadenas de ARN recombinantes: las secuencias ricas en adenina y uracilo disminuyen la precisión del cruce. [89] [90]
La recombinación homóloga es importante para facilitar la evolución viral . [89] [91] Por ejemplo, si los genomas de dos virus con diferentes mutaciones desventajosas se someten a recombinación, entonces pueden regenerar un genoma completamente funcional. Alternativamente, si dos virus similares han infectado la misma célula huésped, la recombinación homóloga puede permitir que esos dos virus intercambien genes y así desarrollen variaciones más potentes de sí mismos. [91]
La recombinación homóloga es el mecanismo propuesto mediante el cual el virus de ADN herpesvirus-6 humano se integra en los telómeros humanos. [92]
Cuando dos o más virus, cada uno de los cuales contiene daños genómicos letales, infectan la misma célula huésped, los genomas del virus a menudo pueden emparejarse entre sí y someterse a una reparación recombinante homóloga para producir una progenie viable. Este proceso, conocido como reactivación de la multiplicidad, se ha estudiado en varios bacteriófagos , incluido el fago T4 . [93] Las enzimas empleadas en la reparación recombinante en el fago T4 son funcionalmente homólogas a las enzimas empleadas en la reparación recombinante bacteriana y eucariota. [94] En particular, con respecto a un gen necesario para la reacción de intercambio de cadena, un paso clave en la reparación recombinacional homóloga, existe homología funcional de los virus a los humanos (es decir, uvsX en el fago T4; recA en E. coli y otras bacterias, y rad51 y dmc1 en levaduras y otros eucariotas, incluidos los humanos). [95] La reactivación de la multiplicidad también se ha demostrado en numerosos virus patógenos. [96]
Los coronavirus son capaces de recombinación genética cuando al menos dos genomas virales están presentes en la misma célula infectada. La recombinación de ARN parece ser una fuerza impulsora importante para determinar (1) la variabilidad genética dentro de una especie de CoV, (2) la capacidad de una especie de CoV para saltar de un huésped a otro y (3) con poca frecuencia, la aparición de nuevos CoV. [97] El mecanismo de recombinación en los CoV probablemente implica el cambio de plantilla durante la replicación del genoma. [97] La recombinación en los virus de ARN parece ser una adaptación para hacer frente al daño del genoma. [98]
Todo el motivo de unión al receptor del SARS-CoV-2 pandémico parece haberse introducido mediante recombinación de coronavirus de pangolines . [99] Tal evento de recombinación puede haber sido un paso crítico en la evolución de la capacidad del SARS-CoV-2 para infectar a los humanos. [99] Los eventos de recombinación son probablemente pasos clave en el proceso evolutivo que conduce a la aparición de nuevos coronavirus humanos. [100]
Durante la pandemia de COVID-19 en 2020, muchas secuencias genómicas de aislados australianos de SARS‐CoV‐2 tienen deleciones o mutaciones (29742G>A o 29742G>U; "G19A" o "G19U") en el tallo-loop II-3′ del coronavirus. motivo similar (s2m) , un motivo de ARN en la región 3' no traducida del genoma viral, lo que sugiere que pueden haber ocurrido eventos de recombinación de ARN en s2m del SARS-CoV-2. Basado en el análisis computacional de 1319 secuencias de SARS‐CoV‐2 de Australia utilizando el algoritmo Recco (https://recco.bioinf.mpi-inf.mpg.de/), 29742G("G19"), 29744G("G21") y Se predijo que 29751G ("G28") serían puntos críticos de recombinación. [101]
El brote de SARS-CoV-2 en el crucero Diamond Princess probablemente se originó a partir de una sola persona infectada con una variante del virus idéntica a los aislados WIV04 de Wuhan, o simultáneamente con otro caso primario infectado con un virus que contiene la mutación 11083G > T. El análisis de desequilibrio de ligamiento confirmó que la recombinación de ARN con la mutación 11083G > T también contribuyó al aumento de mutaciones entre la progenie viral. Los hallazgos indican que la mutación 11083G > T del SARS-CoV-2 se propagó durante la cuarentena a bordo y surgió mediante la recombinación de ARN de novo bajo presión de selección positiva. Además, en tres pacientes de este crucero, también se localizaron dos mutaciones 29736G > T y 29751G > T ("G13" y "G28") en el motivo similar al tallo-loop II del coronavirus 3' (s2m) , como "G28". Se predijo que serían puntos críticos de recombinación en mutantes australianos del SARS-CoV-2. Aunque s2m se considera un motivo de ARN altamente conservado entre muchas especies de coronavirus, este resultado también sugiere que s2m del SARS-CoV-2 es más bien un punto crítico de recombinación/mutación de ARN. [102]
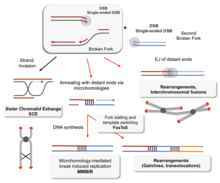
Sin una recombinación homóloga adecuada, los cromosomas a menudo se alinean incorrectamente para la primera fase de la división celular en la meiosis . Esto hace que los cromosomas no logren segregarse adecuadamente en un proceso llamado no disyunción . A su vez, la falta de disyunción puede provocar que los espermatozoides y los óvulos tengan muy pocos o demasiados cromosomas. El síndrome de Down , que es causado por una copia extra del cromosoma 21 , es una de las muchas anomalías que resultan de tal fallo de la recombinación homóloga en la meiosis. [78] [103]
Las deficiencias en la recombinación homóloga se han relacionado fuertemente con la formación de cáncer en humanos. Por ejemplo, cada una de las enfermedades relacionadas con el cáncer, el síndrome de Bloom , el síndrome de Werner y el síndrome de Rothmund-Thomson, son causadas por copias defectuosas de los genes RecQ helicasa implicados en la regulación de la recombinación homóloga: BLM , WRN y RECQL4 , respectivamente. [104] En las células de los pacientes con síndrome de Bloom, que carecen de una copia funcional de la proteína BLM, existe una tasa elevada de recombinación homóloga. [105] Experimentos en ratones con deficiencia de BLM han sugerido que la mutación da lugar al cáncer a través de una pérdida de heterocigosidad causada por una mayor recombinación homóloga. [106] Una pérdida de heterocigosidad se refiere a la pérdida de una de dos versiones (o alelos ) de un gen. Si uno de los alelos perdidos ayuda a suprimir tumores, como por ejemplo el gen de la proteína del retinoblastoma , la pérdida de heterocigosidad puede provocar cáncer. [107] : 1236
La disminución de las tasas de recombinación homóloga provoca una reparación ineficiente del ADN, [107] : 310 , lo que también puede provocar cáncer. [108] Este es el caso de BRCA1 y BRCA2 , dos genes supresores de tumores similares cuyo mal funcionamiento se ha relacionado con un riesgo considerablemente mayor de cáncer de mama y de ovario . Las células a las que les faltan BRCA1 y BRCA2 tienen una tasa reducida de recombinación homóloga y una mayor sensibilidad a la radiación ionizante , lo que sugiere que la disminución de la recombinación homóloga conduce a una mayor susceptibilidad al cáncer. [108] Debido a que la única función conocida de BRCA2 es ayudar a iniciar la recombinación homóloga, los investigadores han especulado que un conocimiento más detallado del papel de BRCA2 en la recombinación homóloga puede ser la clave para comprender las causas del cáncer de mama y de ovario. [108]
Los tumores con una deficiencia de recombinación homóloga (incluidos defectos de BRCA) se describen como HRD positivos. [109]

Si bien las vías pueden variar mecánicamente, la capacidad de los organismos para realizar recombinación homóloga se conserva universalmente en todos los dominios de la vida. [110] Según la similitud de sus secuencias de aminoácidos, se pueden encontrar homólogos de varias proteínas en múltiples dominios de la vida, lo que indica que evolucionaron hace mucho tiempo y que desde entonces se han separado de las proteínas ancestrales comunes. [110]
Los miembros de la familia de recombinasas RecA se encuentran en casi todos los organismos con RecA en bacterias, Rad51 y DMC1 en eucariotas, RadA en arqueas y UvsX en el fago T4 . [111]
En todos los ámbitos de la vida también se encuentran proteínas de unión monocatenarias relacionadas que son importantes para la recombinación homóloga y muchos otros procesos. [112]
Rad54, Mre11 , Rad50 y varias otras proteínas también se encuentran tanto en arqueas como en eucariotas. [110] [111] [113]
Se cree que las proteínas de la familia de proteínas RecA recombinasa descienden de una recombinasa ancestral común. [110] La familia de recombinasas RecA contiene la proteína RecA de bacterias , las proteínas Rad51 y Dmc1 de eucariotas, y RadA de arqueas , y las proteínas parálogas de recombinasa. Los estudios que modelan las relaciones evolutivas entre las proteínas Rad51, Dmc1 y RadA indican que son monofiléticas o que comparten un ancestro molecular común. [110] Dentro de esta familia de proteínas, Rad51 y Dmc1 se agrupan en un clado separado de RadA. Una de las razones para agrupar estas tres proteínas es que todas poseen un motivo hélice-giro-hélice modificado , que ayuda a las proteínas a unirse al ADN, hacia sus extremos N-terminales . [110] Se ha propuesto un antiguo evento de duplicación de un gen eucariota RecA y una mutación posterior como un origen probable de los genes modernos RAD51 y DMC1. [110]
Las proteínas generalmente comparten una región conservada desde hace mucho tiempo conocida como dominio RecA/Rad51. Dentro de este dominio proteico hay dos motivos de secuencia , el motivo Walker A y el motivo Walker B. "Los motivos Walker A y B permiten a los miembros de la familia de proteínas RecA/Rad51 participar en la unión de ATP y la hidrólisis de ATP ". [110] [114]
El descubrimiento de Dmc1 en varias especies de Giardia , uno de los primeros protistas en divergir como eucariota, sugiere que la recombinación homóloga meiótica (y, por tanto, la meiosis misma) surgió muy temprano en la evolución eucariota. [115] Además de la investigación sobre Dmc1, los estudios sobre la proteína Spo11 han proporcionado información sobre los orígenes de la recombinación meiótica. [116] Spo11, una topoisomerasa de tipo II , puede iniciar la recombinación homóloga en la meiosis al realizar roturas bicatenarias específicas en el ADN. [25] Los árboles filogenéticos basados en la secuencia de genes similares a SPO11 en animales, hongos, plantas, protistas y arqueas han llevado a los científicos a creer que la versión Spo11 actualmente en eucariotas surgió en el último ancestro común de eucariotas y arqueas. [116]

Muchos métodos para introducir secuencias de ADN en organismos para crear ADN recombinante y organismos genéticamente modificados utilizan el proceso de recombinación homóloga. [117] También llamado orientación genética , el método es especialmente común en la genética de levaduras y ratones . El método de selección de genes en ratones knockout utiliza células madre embrionarias de ratón para administrar material genético artificial (principalmente de interés terapéutico), que reprime el gen diana del ratón mediante el principio de recombinación homóloga. De este modo, el ratón actúa como modelo de trabajo para comprender los efectos de un gen específico de mamífero. En reconocimiento a su descubrimiento de cómo se puede utilizar la recombinación homóloga para introducir modificaciones genéticas en ratones a través de células madre embrionarias, Mario Capecchi , Martin Evans y Oliver Smithies recibieron el Premio Nobel de Fisiología o Medicina en 2007 . [118]
Los avances en las tecnologías de selección de genes que secuestran la mecánica de recombinación homóloga de las células están conduciendo ahora al desarrollo de una nueva ola de modelos de enfermedades humanas isogénicas más precisas . Se cree que estos modelos de células humanas diseñados reflejan con mayor precisión la genética de las enfermedades humanas que sus predecesores de modelos de ratón. Esto se debe en gran medida a que las mutaciones de interés se introducen en genes endógenos, tal como ocurren en los pacientes reales, y a que se basan en genomas humanos y no en genomas de rata. Además, ciertas tecnologías permiten activar una mutación particular en lugar de simplemente desactivarlas asociadas con tecnologías más antiguas de selección de genes.
La ingeniería de proteínas con recombinación homóloga desarrolla proteínas quiméricas mediante el intercambio de fragmentos entre dos proteínas parentales. Estas técnicas aprovechan el hecho de que la recombinación puede introducir un alto grado de diversidad de secuencias al tiempo que preserva la capacidad de una proteína para plegarse en su estructura terciaria , o forma tridimensional. [119] Esto contrasta con otras técnicas de ingeniería de proteínas, como la mutagénesis puntual aleatoria , en la que la probabilidad de mantener la función de la proteína disminuye exponencialmente con el aumento de las sustituciones de aminoácidos . [120] Las quimeras producidas mediante técnicas de recombinación son capaces de mantener su capacidad de plegarse porque sus fragmentos parentales intercambiados se conservan estructural y evolutivamente. Estos "bloques de construcción" recombinables preservan interacciones estructuralmente importantes, como puntos de contacto físico entre diferentes aminoácidos en la estructura de la proteína. Se pueden utilizar métodos computacionales como SCHEMA y análisis de acoplamiento estadístico para identificar subunidades estructurales adecuadas para la recombinación. [121] [122] [123]
Se han utilizado técnicas que se basan en la recombinación homóloga para diseñar nuevas proteínas. [121] En un estudio publicado en 2007, los investigadores pudieron crear quimeras de dos enzimas involucradas en la biosíntesis de isoprenoides , una clase diversa de compuestos que incluyen hormonas , pigmentos visuales y ciertas feromonas . Las proteínas quiméricas adquirieron la capacidad de catalizar una reacción esencial en la biosíntesis de isoprenoides , una de las vías de biosíntesis más diversas que se encuentran en la naturaleza, que estaba ausente en las proteínas originales. [124] La ingeniería de proteínas mediante recombinación también ha producido enzimas quiméricas con nuevas funciones en miembros de un grupo de proteínas conocido como familia del citocromo P450 , [125] que en los humanos participa en la desintoxicación de compuestos extraños como medicamentos, aditivos alimentarios y conservantes. [22]
Las células cancerosas con capacidad de recombinación homóloga (HRP) son capaces de reparar el daño del ADN causado por quimioterapia como el cisplatino. Por tanto, los cánceres HRP son difíciles de tratar. Los estudios sugieren que la recombinación homóloga puede abordarse mediante la inhibición de c-Abl. [126] [127] Las células cancerosas con mutaciones BRCA tienen deficiencias en la recombinación homóloga y se han desarrollado y utilizado con éxito en ensayos clínicos fármacos para aprovechar esas deficiencias. [128] [129] Olaparib , un inhibidor de PARP1, redujo o detuvo el crecimiento de tumores de cáncer de mama , ovario y próstata causados por mutaciones en los genes BRCA1 o BRCA2 , que son necesarios para la HR. Cuando BRCA1 o BRCA2 están ausentes, otros tipos de mecanismos de reparación del ADN deben compensar la deficiencia de HR, como la reparación por escisión de bases (BER) para horquillas de replicación detenidas o la unión de extremos no homólogos (NHEJ) para roturas de doble cadena. [128] Al inhibir la BER en una célula con deficiencia de recursos humanos, olaparib aplica el concepto de letalidad sintética para atacar específicamente las células cancerosas. Si bien los inhibidores de PARP1 representan un enfoque novedoso para la terapia del cáncer, los investigadores han advertido que pueden resultar insuficientes para tratar los cánceres metastásicos en etapa tardía . [128] Las células cancerosas pueden volverse resistentes a un inhibidor de PARP1 si sufren deleciones de mutaciones en BRCA2, lo que socava la letalidad sintética del fármaco al restaurar la capacidad de las células cancerosas para reparar el ADN mediante HR. [130]