
La proteómica es el estudio a gran escala de las proteínas . [1] [2] Las proteínas son macromoléculas vitales de todos los organismos vivos, con muchas funciones como la formación de fibras estructurales del tejido muscular , la digestión enzimática de los alimentos o la síntesis y replicación del ADN . Además, otros tipos de proteínas incluyen anticuerpos que protegen a un organismo de infecciones y hormonas que envían señales importantes por todo el cuerpo.
El proteoma es el conjunto completo de proteínas producidas o modificadas por un organismo o sistema. La proteómica permite la identificación de un número cada vez mayor de proteínas. Esto varía con el tiempo y los distintos requisitos o tensiones a los que se somete una célula u organismo. [3]
La proteómica es un dominio interdisciplinario que se ha beneficiado enormemente de la información genética de varios proyectos genómicos, incluido el Proyecto Genoma Humano . [4] Cubre la exploración de los proteomas desde el nivel general de composición, estructura y actividad de las proteínas, y es un componente importante de la genómica funcional .
La proteómica generalmente denota el análisis experimental a gran escala de proteínas y proteomas, pero a menudo se refiere específicamente a la purificación de proteínas y la espectrometría de masas . De hecho, la espectrometría de masas es el método más poderoso para el análisis de proteomas, tanto en muestras grandes compuestas por millones de células [5] como en células individuales. [6] [7]
Los primeros estudios de proteínas que podrían considerarse como proteómica comenzaron en 1974, después de la introducción del gel bidimensional y el mapeo de las proteínas de la bacteria Escherichia coli . [8]
Proteoma es una combinación de las palabras "proteína" y "genoma". Fue acuñado en 1994 por el entonces estudiante de doctorado Marc Wilkins en la Universidad Macquarie , [9] que fundó el primer laboratorio dedicado a la proteómica en 1995. [10] [11]
Después de la genómica y la transcriptómica , la proteómica es el siguiente paso en el estudio de los sistemas biológicos. Es más complicada que la genómica porque el genoma de un organismo es más o menos constante, mientras que los proteomas difieren de una célula a otra y de un momento a otro. Los distintos genes se expresan en diferentes tipos de células, lo que significa que incluso el conjunto básico de proteínas producidas en una célula debe identificarse. [12]
En el pasado, este fenómeno se evaluaba mediante análisis de ARN, que no tenía correlación con el contenido de proteínas. [13] [14] Ahora se sabe que el ARNm no siempre se traduce en proteínas, [15] y que la cantidad de proteína producida para una cantidad dada de ARNm depende del gen del que se transcribe y del estado fisiológico de la célula. La proteómica confirma la presencia de la proteína y proporciona una medida directa de su cantidad. [ cita requerida ]
No sólo la traducción del ARNm causa diferencias, sino que muchas proteínas también están sujetas a una amplia variedad de modificaciones químicas después de la traducción. Las modificaciones postraduccionales más comunes y ampliamente estudiadas incluyen la fosforilación y la glicosilación. Muchas de estas modificaciones postraduccionales son fundamentales para la función de la proteína. [ cita requerida ]
Una de estas modificaciones es la fosforilación , que se produce en muchas enzimas y proteínas estructurales durante el proceso de señalización celular . La adición de un fosfato a determinados aminoácidos (más comúnmente serina y treonina [16] mediada por las serina-treonina quinasas , o más raramente tirosina mediada por las tirosina quinasas ) hace que una proteína se convierta en un objetivo para la unión o interacción con un conjunto distinto de otras proteínas que reconocen el dominio fosforilado. [ cita requerida ]
Dado que la fosforilación de proteínas es una de las modificaciones proteicas más estudiadas, muchos estudios "proteómicos" se orientan a determinar el conjunto de proteínas fosforiladas en una célula o tipo de tejido en particular en circunstancias particulares. Esto alerta al científico sobre las vías de señalización que pueden estar activas en ese caso.
La ubiquitina es una proteína pequeña que puede unirse a ciertos sustratos proteicos mediante enzimas llamadas ligasas de ubiquitina E3 . Determinar qué proteínas están poliubiquitinadas ayuda a entender cómo se regulan las vías proteínicas. Por lo tanto, se trata de un estudio "proteómico" legítimo adicional. De manera similar, una vez que un investigador determina qué sustratos están ubiquitinados por cada ligasa, resulta útil determinar el conjunto de ligasas expresadas en un tipo de célula en particular. [ cita requerida ]
Además de la fosforilación y la ubiquitinación , las proteínas pueden sufrir (entre otras) metilación , acetilación , glicosilación , oxidación y nitrosilación . Algunas proteínas sufren todas estas modificaciones, a menudo en combinaciones dependientes del tiempo. Esto ilustra la posible complejidad de estudiar la estructura y la función de las proteínas.
Una célula puede producir diferentes conjuntos de proteínas en diferentes momentos o bajo diferentes condiciones, por ejemplo, durante el desarrollo , la diferenciación celular , el ciclo celular o la carcinogénesis . Para aumentar aún más la complejidad del proteoma, como se mencionó, la mayoría de las proteínas pueden sufrir una amplia gama de modificaciones postraduccionales.
Por lo tanto, un estudio de "proteómica" puede volverse complejo muy rápidamente, incluso si el tema de estudio es restringido. En entornos más ambiciosos, como cuando se busca un biomarcador para un subtipo específico de cáncer, el científico de la proteómica podría optar por estudiar múltiples muestras de suero sanguíneo de múltiples pacientes con cáncer para minimizar los factores de confusión y dar cuenta del ruido experimental. [17] Por lo tanto, a veces son necesarios diseños experimentales complicados para dar cuenta de la complejidad dinámica del proteoma.
La proteómica proporciona un nivel de comprensión diferente al de la genómica por muchas razones:
Reproducibilidad . Un factor importante que afecta la reproducibilidad en los experimentos de proteómica es la elución simultánea de muchos más péptidos de los que pueden medir los espectrómetros de masas. Esto provoca diferencias estocásticas entre experimentos debido a la adquisición de péptidos trípticos dependiente de los datos . Aunque los primeros análisis de proteómica shotgun a gran escala mostraron una variabilidad considerable entre laboratorios, [20] [21] presumiblemente debido en parte a diferencias técnicas y experimentales entre laboratorios, la reproducibilidad ha mejorado en los análisis de espectrometría de masas más recientes, particularmente a nivel de proteínas. [22] En particular, la proteómica dirigida muestra una mayor reproducibilidad y repetibilidad en comparación con los métodos shotgun, aunque a expensas de la densidad de datos y la eficacia. [23]
Calidad de los datos . El análisis proteómico es muy susceptible de automatización y se crean grandes conjuntos de datos que son procesados por algoritmos de software. Se utilizan parámetros de filtro para reducir la cantidad de resultados falsos, pero no se pueden eliminar por completo. Los científicos han expresado la necesidad de concienciar a los científicos de que los experimentos proteómicos deben cumplir con los criterios de la química analítica (suficiente calidad de los datos, verificación de la coherencia, validación). [24] [25] [26] [27]
En proteómica, existen múltiples métodos para estudiar las proteínas. Generalmente, las proteínas se pueden detectar mediante el uso de anticuerpos (inmunoensayos), separación electroforética o espectrometría de masas . Si se analiza una muestra biológica compleja, se debe utilizar un anticuerpo muy específico en un análisis de transferencia de puntos cuantitativo (QDB), o bien se debe utilizar una separación bioquímica antes del paso de detección, ya que hay demasiados analitos en la muestra para realizar una detección y cuantificación precisas.
Los anticuerpos contra proteínas específicas, o sus formas modificadas, se han utilizado en estudios de bioquímica y biología celular . Estas son unas de las herramientas más comunes que utilizan los biólogos moleculares en la actualidad. Existen varias técnicas y protocolos específicos que utilizan anticuerpos para la detección de proteínas. El ensayo inmunoabsorbente ligado a enzimas (ELISA) se ha utilizado durante décadas para detectar y medir cuantitativamente proteínas en muestras. El Western blot se puede utilizar para la detección y cuantificación de proteínas individuales, donde en un paso inicial, una mezcla compleja de proteínas se separa utilizando SDS-PAGE y luego se identifica la proteína de interés utilizando un anticuerpo. [ cita requerida ]
Las proteínas modificadas pueden estudiarse desarrollando un anticuerpo específico para esa modificación. Por ejemplo, algunos anticuerpos solo reconocen ciertas proteínas cuando están fosforiladas en tirosina ; se conocen como anticuerpos fosfoespecíficos. También existen anticuerpos específicos para otras modificaciones. Estos pueden utilizarse para determinar el conjunto de proteínas que han sufrido la modificación de interés. [ cita requerida ]
Los inmunoensayos también pueden llevarse a cabo utilizando derivados de inmunoglobulina generados de forma recombinante o estructuras proteicas diseñadas sintéticamente que se seleccionan por su alta especificidad antigénica. Dichos aglutinantes incluyen fragmentos de anticuerpos de dominio único (Nanobodies), [28] proteínas de repetición de anquirina diseñadas (DARPins) [29] y aptámeros. [30]
La detección de enfermedades a nivel molecular está impulsando la revolución emergente del diagnóstico y el tratamiento tempranos. Un desafío al que se enfrenta este campo es que los biomarcadores proteicos para el diagnóstico temprano pueden estar presentes en cantidades muy bajas. El límite inferior de detección con la tecnología de inmunoensayo convencional es el rango femtomolar superior (10 −13 M). La tecnología de inmunoensayo digital ha mejorado la sensibilidad de detección en tres logaritmos, hasta el rango attomolar (10 −16 M). Esta capacidad tiene el potencial de abrir nuevos avances en el diagnóstico y la terapéutica, pero dichas tecnologías han sido relegadas a procedimientos manuales que no son adecuados para un uso rutinario eficiente. [31]
Aunque la detección de proteínas con anticuerpos sigue siendo muy común en biología molecular, también se han desarrollado otros métodos que no dependen de un anticuerpo. Estos métodos ofrecen varias ventajas; por ejemplo, a menudo pueden determinar la secuencia de una proteína o un péptido, pueden tener un mayor rendimiento que los basados en anticuerpos y, a veces, pueden identificar y cuantificar proteínas para las que no existen anticuerpos.
Uno de los primeros métodos de análisis de proteínas fue la degradación de Edman (introducida en 1967), en la que un único péptido se somete a múltiples pasos de degradación química para resolver su secuencia. Estos primeros métodos han sido reemplazados en su mayoría por tecnologías que ofrecen un mayor rendimiento. [ cita requerida ]
Los métodos implementados más recientemente utilizan técnicas basadas en espectrometría de masas , un desarrollo que fue posible gracias al descubrimiento de métodos de "ionización suave" desarrollados en la década de 1980, como la desorción/ionización láser asistida por matriz (MALDI) y la ionización por electrospray (ESI) . Estos métodos dieron lugar a los flujos de trabajo de proteómica de arriba hacia abajo y de abajo hacia arriba , en los que a menudo se realiza una separación adicional antes del análisis (ver a continuación).
Para el análisis de muestras biológicas complejas, se requiere una reducción de la complejidad de la muestra. Esto se puede realizar fuera de línea mediante una separación unidimensional o bidimensional . Más recientemente, se han desarrollado métodos en línea en los que los péptidos individuales (en enfoques de proteómica de abajo a arriba) se separan mediante cromatografía de fase inversa y luego se ionizan directamente mediante ESI ; el acoplamiento directo de la separación y el análisis explica el término análisis "en línea".
Varias tecnologías híbridas utilizan la purificación basada en anticuerpos de analitos individuales y luego realizan un análisis espectrométrico de masas para su identificación y cuantificación. Ejemplos de estos métodos son el MSIA (inmunoensayo espectrométrico de masas) , desarrollado por Randall Nelson en 1995, [32] y el método SISCAPA (captura de estándar de isótopos estables con anticuerpos antipéptidos), introducido por Leigh Anderson en 2004. [33]
La electroforesis en gel diferencial bidimensional de fluorescencia (2-D DIGE) [34] se puede utilizar para cuantificar la variación en el proceso 2-D DIGE y establecer umbrales estadísticamente válidos para asignar cambios cuantitativos entre muestras. [34]
El análisis proteómico comparativo puede revelar el papel de las proteínas en sistemas biológicos complejos, incluida la reproducción. Por ejemplo, el tratamiento con el insecticida triazofos provoca un aumento en el contenido de proteínas de las glándulas accesorias masculinas (Acps) de la chicharrita marrón ( Nilaparvata lugens (Stål)) que pueden transferirse a las hembras a través del apareamiento, lo que provoca un aumento en la fecundidad (es decir, la tasa de natalidad) de las hembras. [35] Para identificar cambios en los tipos de proteínas de las glándulas accesorias (Acps) y las proteínas reproductivas que las chicharritas hembras apareadas recibieron de los machos, los investigadores llevaron a cabo un análisis proteómico comparativo de hembras apareadas de N. lugens . [36] Los resultados indicaron que estas proteínas participan en el proceso reproductivo de las hembras y los machos adultos de N. lugens . [36]
El análisis del proteoma de los peroxisomas de Arabidopsis [37] se ha establecido como el principal enfoque imparcial para identificar nuevas proteínas peroxisomales a gran escala. [37]
Existen muchos enfoques para caracterizar el proteoma humano, que se estima que contiene entre 20.000 y 25.000 proteínas no redundantes. El número de especies de proteínas únicas probablemente aumentará entre 50.000 y 500.000 debido a los eventos de empalme y proteólisis del ARN, y cuando también se considera la modificación postraduccional, se estima que el número total de proteínas humanas únicas oscila en unos pocos millones. [38] [39]
Además, recientemente se han informado los primeros intentos prometedores de descifrar el proteoma de los tumores animales. [40] Este método se utilizó como método funcional en el perfil de proteínas de Macrobrachium rosenbergii . [41]
La proteómica ha ganado impulso de forma constante durante la última década con la evolución de varios enfoques. Pocos de ellos son nuevos y otros se basan en métodos tradicionales. Los métodos basados en espectrometría de masas, la proteómica de afinidad y las micromatrices son las tecnologías más comunes para el estudio de proteínas a gran escala.
.jpg/440px-Thermo_-_Finnigan_LCQ_Mass_Spectrometer_(15797493459).jpg)
En la actualidad, se utilizan dos métodos basados en espectrometría de masas para la determinación del perfil de proteínas. El método más establecido y extendido utiliza una electroforesis bidimensional de alta resolución para separar las proteínas de diferentes muestras en paralelo, seguida de la selección y tinción de las proteínas expresadas de forma diferencial para su identificación mediante espectrometría de masas. A pesar de los avances en la espectrometría de masas bidimensional y su madurez, también tiene sus límites. La principal preocupación es la incapacidad de resolver todas las proteínas dentro de una muestra, dada su gran variedad de niveles de expresión y sus diferentes propiedades. La combinación del tamaño de los poros y la carga, el tamaño y la forma de las proteínas pueden determinar en gran medida la tasa de migración, lo que conduce a otras complicaciones. [42]
El segundo enfoque cuantitativo utiliza etiquetas de isótopos estables para etiquetar de forma diferencial las proteínas de dos mezclas complejas diferentes. [43] [44] Aquí, las proteínas dentro de una mezcla compleja se etiquetan isotópicamente primero y luego se digieren para producir péptidos marcados. Luego, las mezclas marcadas se combinan, los péptidos se separan mediante cromatografía líquida multidimensional y se analizan mediante espectrometría de masas en tándem. Los reactivos de etiqueta de afinidad codificada por isótopos (ICAT) son las etiquetas de isótopos más utilizadas. En este método, los residuos de cisteína de las proteínas se unen covalentemente al reactivo ICAT, lo que reduce la complejidad de las mezclas y omite los residuos que no son de cisteína.
La proteómica cuantitativa mediante el uso de marcadores isotópicos estables es una herramienta cada vez más útil en el desarrollo moderno. En primer lugar, se han utilizado reacciones químicas para introducir marcadores en sitios específicos o proteínas con el fin de investigar funcionalidades proteicas específicas. El aislamiento de péptidos fosforilados se ha logrado mediante el uso de marcadores isotópicos y químicas selectivas para capturar la fracción de proteína entre la mezcla compleja. En segundo lugar, se utilizó la tecnología ICAT para diferenciar entre complejos macromoleculares parcialmente purificados o purificados, como el complejo de preiniciación de la ARN polimerasa II grande y las proteínas complejadas con el factor de transcripción de levadura. En tercer lugar, el marcado ICAT se combinó recientemente con el aislamiento de cromatina para identificar y cuantificar proteínas asociadas a la cromatina. Finalmente, los reactivos ICAT son útiles para el perfil proteómico de orgánulos celulares y fracciones celulares específicas. [42]
Otro enfoque cuantitativo es el enfoque de etiqueta de masa y tiempo exactos (AMT) desarrollado por Richard D. Smith y colaboradores en el Laboratorio Nacional del Pacífico Noroeste . En este enfoque, se logra un mayor rendimiento y sensibilidad al evitar la necesidad de espectrometría de masas en tándem y hacer uso de información de tiempo de separación determinada con precisión y determinaciones de masa altamente precisas para identificaciones de péptidos y proteínas.
La proteómica de afinidad utiliza anticuerpos u otros reactivos de afinidad (como aptámeros basados en oligonucleótidos) como sondas de detección específicas de proteínas. [45] Actualmente, este método puede analizar varios miles de proteínas, generalmente de biofluidos como plasma, suero o líquido cefalorraquídeo (LCR). Un diferenciador clave de esta tecnología es la capacidad de analizar cientos o miles de muestras en un período de tiempo razonable (en cuestión de días o semanas); los métodos basados en espectrometría de masas no son escalables a este nivel de rendimiento de muestras para análisis proteómicos.
El uso de microarreglos de proteínas es un método que equilibra el uso de espectrómetros de masas en proteómica y en medicina. El objetivo de estos métodos es imprimir miles de características de detección de proteínas para la interrogación de muestras biológicas. Los microarreglos de proteínas son un ejemplo de un conjunto de anticuerpos diferentes que se colocan en un conjunto para detectar sus respectivos antígenos en una muestra de sangre humana. Otro método es el de colocar en un conjunto múltiples tipos de proteínas para estudiar propiedades como las interacciones proteína-ADN, proteína-proteína y proteína-ligando. Lo ideal sería que los microarreglos proteómicos funcionales contuvieran todo el conjunto de proteínas de un organismo determinado. La primera versión de estos microarreglos consistía en 5000 proteínas purificadas de levadura depositadas en portaobjetos de vidrio para microscopio. A pesar del éxito del primer chip, la implementación de los microarreglos de proteínas fue un desafío mayor. Las proteínas son inherentemente mucho más difíciles de trabajar que el ADN. Tienen un amplio rango dinámico, son menos estables que el ADN y su estructura es difícil de preservar en portaobjetos de vidrio, aunque son esenciales para la mayoría de los ensayos. La tecnología global ICAT tiene sorprendentes ventajas sobre las tecnologías de chips de proteínas. [42]
Esta es una aplicación prometedora y nueva de microarrays para el diagnóstico, estudio y tratamiento de enfermedades complejas como el cáncer. La tecnología fusiona la microdisección por captura láser (LCM) con la tecnología de microarrays para producir microarrays de proteínas en fase inversa. En este tipo de microarrays, toda la colección de proteínas se inmoviliza con la intención de capturar varias etapas de la enfermedad dentro de un paciente individual. Cuando se utilizan con LCM, los arreglos de fase inversa pueden monitorear el estado fluctuante del proteoma entre diferentes poblaciones celulares dentro de una pequeña área de tejido humano. Esto es útil para perfilar el estado de las moléculas de señalización celular, entre una sección transversal de tejido que incluye células normales y cancerosas. Este enfoque es útil para monitorear el estado de factores clave en el epitelio de próstata normal y tejidos de cáncer de próstata invasivo. Luego, LCM disecciona estos tejidos y los lisados de proteínas se colocaron en portaobjetos de nitrocelulosa, que se analizaron con anticuerpos específicos. Este método puede rastrear todo tipo de eventos moleculares y puede comparar tejidos enfermos y sanos dentro del mismo paciente, lo que permite el desarrollo de estrategias de tratamiento y diagnóstico. La capacidad de obtener instantáneas proteómicas de poblaciones celulares vecinas, utilizando microarreglos de fase inversa junto con LCM, tiene una serie de aplicaciones más allá del estudio de tumores. El enfoque puede proporcionar información sobre la fisiología y patología normales de todos los tejidos y es invaluable para caracterizar los procesos y anomalías del desarrollo. [42]
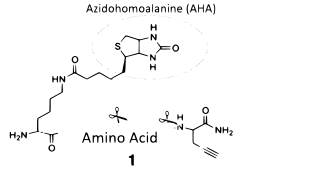
Los recientes avances en química bioortogonal han revelado aplicaciones en el análisis de proteínas. La ampliación del uso de moléculas orgánicas para observar su reacción con proteínas revela métodos extensos para etiquetarlas. Los aminoácidos no naturales y varios grupos funcionales representan nuevas tecnologías en crecimiento en proteómica.
Las biomoléculas específicas que pueden metabolizarse en las células o los tejidos se insertan en proteínas o glicanos. La molécula tendrá una etiqueta de afinidad que modificará la proteína para permitir su detección. La azidohomoalanina (AHA) utiliza esta etiqueta de afinidad mediante la incorporación con la Met-t-ARN sintetasa para incorporarse a las proteínas. Esto ha permitido que la AHA ayude a determinar la identidad de las proteínas recién sintetizadas creadas en respuesta a perturbaciones y a identificar las proteínas secretadas por las células. [46]
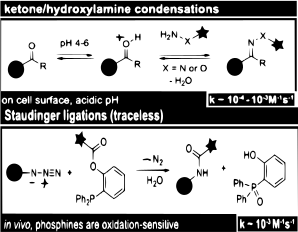
Estudios recientes [47] que utilizan condensaciones de cetonas y aldehídos muestran que son más adecuados para el etiquetado in vitro o de la superficie celular . Sin embargo, el uso de cetonas y aldehídos como reporteros bioortogonales reveló una cinética lenta, lo que indica que, si bien son eficaces para el etiquetado, la concentración debe ser alta.
Algunas proteínas pueden detectarse por su reactividad con los grupos azida . Los aminoácidos no proteinogénicos pueden tener grupos azida que reaccionan con las fosfinas en ligaduras de Staudinger . Esta reacción ya se ha utilizado para marcar otras biomoléculas en células vivas y animales. [48]
El campo de la bioortogénesis se está expandiendo y está impulsando más aplicaciones dentro de la proteómica. Vale la pena señalar las limitaciones y los beneficios. Las reacciones rápidas pueden crear bioconjunciones y generar altas concentraciones con bajas cantidades de reactivos. Por el contrario, las reacciones cinéticas lentas como la condensación de aldehídos y cetonas, si bien son efectivas, requieren una alta concentración, lo que las hace ineficientes en términos de costos.
Un avance importante que ha surgido del estudio de los genes y las proteínas humanas ha sido la identificación de posibles nuevos fármacos para el tratamiento de enfermedades. Esto se basa en la información del genoma y del proteoma para identificar proteínas asociadas con una enfermedad, que luego el software informático puede utilizar como objetivos para nuevos fármacos. Por ejemplo, si una determinada proteína está implicada en una enfermedad, su estructura 3D proporciona la información para diseñar fármacos que interfieran con la acción de la proteína. Una molécula que se adapta al sitio activo de una enzima, pero que no puede ser liberada por la enzima, inactiva la enzima. Esta es la base de las nuevas herramientas de descubrimiento de fármacos, que tienen como objetivo encontrar nuevos fármacos para inactivar las proteínas implicadas en la enfermedad. A medida que se descubran diferencias genéticas entre los individuos, los investigadores esperan utilizar estas técnicas para desarrollar fármacos personalizados que sean más eficaces para el individuo. [49]
La proteómica también se utiliza para revelar interacciones complejas entre plantas e insectos que ayudan a identificar genes candidatos involucrados en la respuesta defensiva de las plantas a la herbivoría. [50] [51] [52]
Una rama de la proteómica llamada quimioproteómica proporciona numerosas herramientas y técnicas para detectar objetivos proteicos de los fármacos. [53]
La proteómica de interacción es el análisis de las interacciones de proteínas desde escalas de interacciones binarias hasta escalas de proteoma o red. La mayoría de las proteínas funcionan a través de interacciones proteína-proteína , y uno de los objetivos de la proteómica de interacción es identificar interacciones proteínicas binarias , complejos proteínicos e interactomas .
Existen varios métodos disponibles para investigar las interacciones proteína-proteína . Si bien el método más tradicional es el análisis de dos híbridos de levadura , un método emergente poderoso es la purificación por afinidad seguida de espectrometría de masas de proteínas utilizando cebos de proteínas marcados . Otros métodos incluyen resonancia de plasmón de superficie (SPR), [54] [55] microarreglos de proteínas , interferometría de polarización dual , termoforesis a microescala , ensayo de exclusión cinética y métodos experimentales como presentación de fagos y métodos computacionales in silico .
El conocimiento de las interacciones proteína-proteína es especialmente útil en relación con las redes biológicas y la biología de sistemas , por ejemplo en las cascadas de señalización celular y las redes reguladoras de genes (GRN, donde el conocimiento de las interacciones proteína-ADN también es informativo). El análisis de las interacciones de proteínas a nivel de proteoma y la integración de estos patrones de interacción en redes biológicas más grandes es crucial para comprender la biología a nivel de sistemas. [56] [57]
La proteómica de expresión incluye el análisis de la expresión de proteínas a una escala mayor. Ayuda a identificar las proteínas principales en una muestra en particular y aquellas proteínas que se expresan de manera diferencial en muestras relacionadas, como tejido enfermo y tejido sano. Si una proteína se encuentra solo en una muestra enferma, puede ser un objetivo farmacológico útil o un marcador de diagnóstico. Las proteínas con perfiles de expresión iguales o similares también pueden estar funcionalmente relacionadas. Existen tecnologías como la electroforesis en gel de poliacrilamida 2D y la espectrometría de masas que se utilizan en la proteómica de expresión. [58]
Los Institutos Nacionales de Salud han definido un biomarcador como "una característica que se mide y evalúa objetivamente como un indicador de procesos biológicos normales, procesos patógenos o respuestas farmacológicas a una intervención terapéutica". [59] [60]
Comprender el proteoma, la estructura y función de cada proteína y las complejidades de las interacciones proteína-proteína son fundamentales para desarrollar las técnicas de diagnóstico y los tratamientos de enfermedades más eficaces en el futuro. Por ejemplo, la proteómica es muy útil para la identificación de biomarcadores candidatos (proteínas en los fluidos corporales que son de valor para el diagnóstico), la identificación de los antígenos bacterianos que son el objetivo de la respuesta inmunitaria y la identificación de posibles marcadores inmunohistoquímicos de enfermedades infecciosas o neoplásicas. [61]
Un uso interesante de la proteómica es el uso de biomarcadores proteicos específicos para diagnosticar enfermedades. Varias técnicas permiten analizar las proteínas producidas durante una enfermedad en particular, lo que ayuda a diagnosticar la enfermedad rápidamente. Las técnicas incluyen el Western blot , la tinción inmunohistoquímica , el ensayo inmunoabsorbente ligado a enzimas (ELISA) o la espectrometría de masas . [40] [62] La secretómica , un subcampo de la proteómica que estudia las proteínas secretadas y las vías de secreción utilizando enfoques proteómicos, ha surgido recientemente como una herramienta importante para el descubrimiento de biomarcadores de enfermedades. [63]
En proteogenómica , se utilizan tecnologías proteómicas como la espectrometría de masas para mejorar las anotaciones genéticas . El análisis paralelo del genoma y del proteoma facilita el descubrimiento de modificaciones postraduccionales y eventos proteolíticos, [64] especialmente cuando se comparan múltiples especies (proteogenómica comparativa). [65]
La proteómica estructural incluye el análisis de las estructuras de las proteínas a gran escala. Compara las estructuras de las proteínas y ayuda a identificar las funciones de los genes recién descubiertos. El análisis estructural también ayuda a comprender dónde se unen los fármacos a las proteínas y también muestra dónde interactúan las proteínas entre sí. Esta comprensión se logra utilizando diferentes tecnologías, como la cristalografía de rayos X y la espectroscopia de RMN. [58]
Muchos datos proteómicos se recopilan con la ayuda de tecnologías de alto rendimiento, como la espectrometría de masas y los microarrays. A menudo, se necesitan semanas o meses para analizar los datos y realizar comparaciones a mano. Por este motivo, los biólogos y químicos están colaborando con científicos informáticos y matemáticos para crear programas y procesos para analizar computacionalmente los datos proteínicos. Mediante el uso de técnicas bioinformáticas , los investigadores pueden realizar análisis y almacenar datos con mayor rapidez. Un buen lugar para encontrar listas de programas y bases de datos actuales es el portal de recursos bioinformáticos ExPASy . Las aplicaciones de la proteómica basada en la bioinformática incluyen la medicina, el diagnóstico de enfermedades, la identificación de biomarcadores y muchas más.
La espectrometría de masas y los microarrays producen información sobre la fragmentación de péptidos, pero no permiten identificar proteínas específicas presentes en la muestra original. Debido a la falta de identificación de proteínas específicas, los investigadores del pasado se vieron obligados a descifrar los fragmentos de péptidos por sí mismos. Sin embargo, actualmente hay programas disponibles para la identificación de proteínas. Estos programas toman las secuencias de péptidos resultantes de la espectrometría de masas y los microarrays y devuelven información sobre proteínas coincidentes o similares. Esto se hace a través de algoritmos implementados por el programa que realizan alineaciones con proteínas de bases de datos conocidas como UniProt [66] y PROSITE [67] para predecir qué proteínas hay en la muestra con un cierto grado de certeza.
La estructura biomolecular forma la configuración 3D de la proteína. Comprender la estructura de la proteína ayuda a identificar las interacciones y la función de la proteína. Antes, la estructura 3D de las proteínas solo se podía determinar mediante cristalografía de rayos X y espectroscopia de RMN . A partir de 2017, la microscopía crioelectrónica es una técnica líder que resuelve las dificultades con la cristalización (en cristalografía de rayos X) y la ambigüedad conformacional (en RMN); la resolución era de 2,2 Å a partir de 2015. Ahora, a través de la bioinformática, existen programas informáticos que en algunos casos pueden predecir y modelar la estructura de las proteínas. Estos programas utilizan las propiedades químicas de los aminoácidos y las propiedades estructurales de las proteínas conocidas para predecir el modelo 3D de las proteínas de muestra. Esto también permite a los científicos modelar las interacciones de las proteínas a mayor escala. Además, los ingenieros biomédicos están desarrollando métodos para tener en cuenta la flexibilidad de las estructuras de las proteínas para hacer comparaciones y predicciones. [68]
La mayoría de los programas disponibles para el análisis de proteínas no están escritos para proteínas que han sufrido modificaciones postraduccionales . [69] Algunos programas aceptarán modificaciones postraduccionales para ayudar en la identificación de proteínas, pero luego ignorarán la modificación durante el análisis posterior de proteínas. Es importante tener en cuenta estas modificaciones, ya que pueden afectar la estructura de la proteína. A su vez, el análisis computacional de las modificaciones postraduccionales ha ganado la atención de la comunidad científica. Los programas de modificación postraduccional actuales son solo predictivos. [70] Los químicos, biólogos y científicos informáticos están trabajando juntos para crear e introducir nuevos procesos que permitan el análisis de modificaciones postraduccionales que se han identificado experimentalmente por su efecto en la estructura y función de la proteína.
Un ejemplo del uso de la bioinformática y el uso de métodos computacionales es el estudio de biomarcadores proteicos. Los modelos predictivos computacionales [71] han demostrado que durante el embarazo se produce un tráfico proteico feto-materno extenso y diverso que se puede detectar fácilmente de forma no invasiva en la sangre materna completa. Este enfoque computacional eludió una limitación importante, la abundancia de proteínas maternas que interfieren con la detección de proteínas fetales , para el análisis proteómico fetal de la sangre materna. Los modelos computacionales pueden utilizar transcripciones de genes fetales previamente identificadas en la sangre materna completa para crear una red proteómica integral del término neonato . Tal trabajo muestra que las proteínas fetales detectadas en la sangre de la mujer embarazada se originan a partir de un grupo diverso de tejidos y órganos del feto en desarrollo. Las redes proteómicas contienen muchos biomarcadores que son indicadores del desarrollo e ilustran la posible aplicación clínica de esta tecnología como una forma de monitorear el desarrollo fetal normal y anormal.
También se ha introducido un marco teórico de la información para el descubrimiento de biomarcadores , integrando información de biofluidos y tejidos. [72] Este nuevo enfoque aprovecha la sinergia funcional entre ciertos biofluidos y tejidos con el potencial de hallazgos clínicamente significativos que no serían posibles si los tejidos y los biofluidos se consideraran individualmente. Al conceptualizar los tejidos y los biofluidos como canales de información, se pueden identificar indicadores significativos de los biofluidos y luego utilizarlos para el desarrollo guiado de diagnósticos clínicos. Luego, se predicen los biomarcadores candidatos en función de los criterios de transferencia de información a través de los canales de tejido y biofluido. Las relaciones significativas entre biofluidos y tejidos se pueden utilizar para priorizar la validación clínica de los biomarcadores. [72]
Varios conceptos emergentes tienen el potencial de mejorar las características actuales de la proteómica. Obtener la cuantificación absoluta de las proteínas y monitorear las modificaciones postraduccionales son las dos tareas que impactan en la comprensión de la función de las proteínas en células sanas y enfermas. Además, el rendimiento y la sensibilidad de los ensayos proteómicos, a menudo medidos como muestras analizadas por día y profundidad de la cobertura del proteoma, respectivamente, han impulsado el desarrollo de instrumentación y metodologías de vanguardia. [73] Para muchos eventos celulares, las concentraciones de proteínas no cambian; más bien, su función es modulada por modificaciones postraduccionales (PTM). Los métodos de monitoreo de PTM son un área subdesarrollada en proteómica. Seleccionar un subconjunto particular de proteínas para el análisis reduce sustancialmente la complejidad de las proteínas, lo que lo hace ventajoso para fines de diagnóstico donde la sangre es el material de partida. Otro aspecto importante de la proteómica, aún no abordado, es que los métodos de proteómica deben centrarse en estudiar las proteínas en el contexto del medio ambiente. El uso cada vez mayor de reticulantes químicos, introducidos en células vivas para fijar interacciones proteína-proteína, proteína-ADN y otras, puede mejorar parcialmente este problema. El desafío es identificar métodos adecuados para preservar las interacciones relevantes. Otro objetivo para el estudio de las proteínas es el desarrollo de métodos más sofisticados para obtener imágenes de proteínas y otras moléculas en células vivas y en tiempo real. [42]
Los avances en proteómica cuantitativa claramente permitirían un análisis más profundo de los sistemas celulares. [56] [57] Otra frontera de investigación es el análisis de células individuales, [74] [75] y la covariación de proteínas entre células individuales [76] que refleja procesos biológicos como la formación de complejos proteicos, funciones inmunes, [77] así como el ciclo celular y la preparación de células cancerosas para la resistencia a fármacos [78] Los sistemas biológicos están sujetos a una variedad de perturbaciones ( ciclo celular , diferenciación celular , carcinogénesis , medio ambiente (biofísico) , etc.). Las respuestas transcripcionales y traduccionales a estas perturbaciones resultan en cambios funcionales en el proteoma implicado en la respuesta al estímulo. Por lo tanto, describir y cuantificar los cambios en la abundancia de proteínas en todo el proteoma es crucial para comprender el fenómeno biológico de manera más holística , a nivel de todo el sistema. De esta manera, la proteómica puede verse como complementaria a la genómica , transcriptómica , epigenómica , metabolómica y otros enfoques -ómicos en análisis integrativos que intentan definir fenotipos biológicos de manera más completa. Como ejemplo, The Cancer Proteome Atlas proporciona datos cuantitativos de expresión de proteínas para ~200 proteínas en más de 4000 muestras de tumores con datos transcriptómicos y genómicos coincidentes de The Cancer Genome Atlas . [79] Conjuntos de datos similares en otros tipos de células, tipos de tejidos y especies, particularmente usando espectrometría de masas de escopeta profunda, serán un recurso inmensamente importante para la investigación en campos como la biología del cáncer , la biología del desarrollo y de células madre , la medicina y la biología evolutiva .
La caracterización del proteoma del plasma humano se ha convertido en un objetivo importante en el campo de la proteómica, pero también es el proteoma más desafiante de todos los tejidos humanos. [80] Contiene inmunoglobulina, citocinas, hormonas proteicas y proteínas secretadas indicativas de infección, además de proteínas hemostáticas residentes. También contiene proteínas de fuga tisular debido a la circulación sanguínea a través de diferentes tejidos del cuerpo. Por lo tanto, la sangre contiene información sobre el estado fisiológico de todos los tejidos y, combinado con su accesibilidad, hace que el proteoma sanguíneo sea invaluable para fines médicos. Se cree que caracterizar el proteoma del plasma sanguíneo es un desafío abrumador.
La profundidad del proteoma plasmático abarca un rango dinámico de más de 10 10 entre la proteína más abundante (albúmina) y la más baja (algunas citocinas) y se cree que es uno de los principales desafíos para la proteómica. [81] La dinámica temporal y espacial complica aún más el estudio del proteoma plasmático humano. El recambio de algunas proteínas es bastante más rápido que el de otras y el contenido proteico de una arteria puede variar sustancialmente del de una vena. Todas estas diferencias hacen que incluso la tarea proteómica más simple de catalogar el proteoma parezca fuera de alcance. Para abordar este problema, es necesario establecer prioridades. Capturar el subconjunto más significativo de proteínas entre todo el proteoma para generar una herramienta de diagnóstico es una de esas prioridades. En segundo lugar, dado que el cáncer está asociado con una mayor glicosilación de las proteínas, los métodos que se centran en esta parte de las proteínas también serán útiles. Nuevamente: el análisis multiparamétrico revela mejor un estado patológico. A medida que estas tecnologías mejoren, los perfiles de la enfermedad deben relacionarse continuamente con los cambios respectivos en la expresión genética. [42] Debido a los problemas mencionados anteriormente, la proteómica plasmática siguió siendo un desafío. Sin embargo, los avances tecnológicos y los desarrollos continuos parecen dar como resultado un resurgimiento de la proteómica plasmática, como se demostró recientemente mediante una tecnología llamada perfilación del proteoma plasmático. [82] Gracias a estas tecnologías, los investigadores pudieron investigar los procesos de inflamación en ratones, la heredabilidad de los proteomas plasmáticos y demostrar el efecto de un cambio de estilo de vida tan común como la pérdida de peso en el proteoma plasmático. [83] [84] [85]
Existen numerosas revistas especializadas en el campo de la proteómica y áreas relacionadas. Cabe señalar que las revistas que tratan sobre proteínas suelen estar más centradas en la estructura y la función, mientras que las revistas de proteómica se centran más en el análisis a gran escala de proteomas completos o, al menos, de grandes conjuntos de proteínas. A continuación se enumeran algunas revistas de proteómica relevantes (con sus editores).
{{cite book}}: |journal=ignorado ( ayuda )