En biología , la palabra gen ( griego : γένος , génos ; [1] generación, [2] o nacimiento, [1] o género ) tiene dos significados. El gen mendeliano es una unidad básica de la herencia . El gen molecular es una secuencia de nucleótidos en el ADN , que se transcribe para producir un ARN funcional . Hay dos tipos de genes moleculares: genes codificadores de proteínas y genes no codificantes. [3] [4] [5] [6]
Durante la expresión genética , el ADN se copia primero en ARN . El ARN puede ser directamente funcional o ser el molde intermedio para la síntesis de una proteína.
La transmisión de genes a la descendencia de un organismo es la base de la herencia de rasgos fenotípicos de una generación a la siguiente. Estos genes forman diferentes secuencias de ADN, llamadas en conjunto un genotipo , que es específico de cada individuo determinado, dentro del acervo genético de una población de una especie determinada . El genotipo, junto con los factores ambientales y de desarrollo, determina en última instancia el fenotipo del individuo. La mayoría de los rasgos biológicos ocurren bajo la influencia combinada de poligenes (un conjunto de genes diferentes) e interacciones entre genes y ambiente . Algunos rasgos genéticos son visibles instantáneamente, como el color de ojos o el número de extremidades, otros no, como el tipo de sangre , el riesgo de enfermedades específicas o los miles de procesos bioquímicos básicos que constituyen la vida .
Un gen puede adquirir mutaciones en su secuencia , dando lugar a diferentes variantes, conocidas como alelos , en la población . Estos alelos codifican versiones ligeramente diferentes de un gen, lo que puede causar rasgos fenotípicos diferentes . [7] Los genes evolucionan debido a la selección natural / supervivencia del más apto y a la deriva genética de los alelos.
El término gen fue introducido por el botánico, fisiólogo vegetal y genetista danés Wilhelm Johannsen en 1909. [8] Está inspirado en el griego antiguo : γόνος, gonos , que significa descendencia y procreación.
Hay muchas formas diferentes de utilizar el término "gen" según diferentes aspectos de su herencia, selección, función biológica o estructura molecular, pero la mayoría de estas definiciones se dividen en dos categorías: gen mendeliano o gen molecular. [3] [9] [10] [11] [12]
El gen mendeliano es el gen clásico de la genética y se refiere a cualquier rasgo hereditario. Este es el gen descrito en "El gen egoísta". [13] Se pueden encontrar discusiones más exhaustivas sobre esta versión de un gen en los artículos sobre Genética y Visión de la evolución centrada en los genes .
La definición de gen molecular se usa más comúnmente en bioquímica, biología molecular y la mayor parte de la genética: el gen que se describe en términos de secuencia de ADN. [3] Hay muchas definiciones diferentes de este gen, algunas de las cuales son engañosas o incorrectas. [9] [14]
Los primeros trabajos en el campo que se convirtió en genética molecular sugirieron el concepto de que un gen produce una proteína (originalmente "un gen, una enzima"). [15] [16] Sin embargo, los genes que producen ARN represores se propusieron en la década de 1950 [17] y en la década de 1960, los libros de texto utilizaban definiciones de genes moleculares que incluían aquellas que especificaban moléculas de ARN funcionales como el ARN ribosómico y el ARNt (genes no codificantes). así como genes codificadores de proteínas. [18]
Esta idea de dos tipos de genes todavía forma parte de la definición de gen en la mayoría de los libros de texto. Por ejemplo,
Las partes importantes de tales definiciones son: (1) que un gen corresponde a una unidad de transcripción; (2) que los genes producen tanto ARNm como ARN no codificantes; y (3) las secuencias reguladoras controlan la expresión genética pero no son parte del gen en sí. Sin embargo, hay otra parte importante de la definición y se enfatiza en el libro de Kostas Kampourakis "Making Sense of Genes".
El énfasis en la función es esencial porque hay tramos de ADN que producen transcripciones no funcionales y no califican como genes. Estos incluyen ejemplos obvios, como los pseudogenes transcritos, así como ejemplos menos obvios, como el ARN basura producido como ruido debido a errores de transcripción. Para calificar como un gen verdadero, según esta definición, uno tiene que demostrar que la transcripción tiene una función biológica. [9]
Las primeras especulaciones sobre el tamaño de un gen típico se basaban en mapas genéticos de alta resolución y en el tamaño de proteínas y moléculas de ARN. Una longitud de 1.500 pares de bases parecía razonable en aquel momento (1965). [18] Esto se basó en la idea de que el gen era el ADN directamente responsable de la producción del producto funcional. El descubrimiento de los intrones en la década de 1970 significó que muchos genes eucariotas eran mucho más grandes de lo que implicaría el tamaño del producto funcional. Los genes típicos que codifican proteínas de mamíferos, por ejemplo, tienen una longitud de aproximadamente 62 000 pares de bases (región transcrita) y, dado que hay alrededor de 20 000, ocupan aproximadamente entre el 35 y el 40 % del genoma de los mamíferos (incluido el genoma humano). [22] [23] [24]
A pesar de que tanto los genes codificadores de proteínas como los no codificantes se conocen desde hace más de 50 años, todavía existen varios libros de texto, sitios web y publicaciones científicas que definen un gen como una secuencia de ADN que especifica una proteína. En otras palabras, la definición se restringe a los genes que codifican proteínas. A continuación se muestra un ejemplo de un artículo reciente de American Scientist.
Esta definición restringida es tan común que ha generado muchos artículos recientes que critican esta "definición estándar" y exigen una nueva definición ampliada que incluya genes no codificantes. [26] [27] [28] Sin embargo, esta llamada "nueva" definición ha existido durante más de medio siglo y no está claro por qué algunos escritores modernos están ignorando los genes no codificantes. [ editorializando ]
Aunque algunas definiciones pueden tener una aplicación más amplia que otras, la complejidad fundamental de la biología significa que ninguna definición de gen puede capturar todos los aspectos a la perfección. No todos los genomas son ADN (por ejemplo, virus de ARN ), [29] los operones bacterianos son múltiples regiones codificantes de proteínas transcritas en ARNm individuales grandes, el empalme alternativo permite que una única región genómica codifique productos de distritos múltiples y el empalme trans concatena ARNm de secuencias codificantes más cortas a lo largo del genoma. [30] [31] [32] Dado que las definiciones moleculares excluyen elementos como intrones, promotores y otras regiones reguladoras , se piensa que estos están "asociados" con el gen y afectan su función.
A veces se utiliza una definición operativa aún más amplia para abarcar la complejidad de estos diversos fenómenos, donde un gen se define como una unión de secuencias genómicas que codifican un conjunto coherente de productos funcionales potencialmente superpuestos. [33] Esta definición clasifica los genes por sus productos funcionales (proteínas o ARN) en lugar de por sus loci de ADN específicos, con elementos reguladores clasificados como regiones asociadas a genes . [33]

Gregor Mendel (1822-1884) sugirió por primera vez la existencia de unidades heredables discretas . [34] De 1857 a 1864, en Brno , Imperio austríaco (hoy República Checa), estudió los patrones de herencia en 8000 plantas de guisantes comestibles comunes , rastreando distintos rasgos de padres a hijos. Las describió matemáticamente como 2 n combinaciones donde n es el número de características diferentes en los guisantes originales. Aunque no utilizó el término gen , explicó sus resultados en términos de unidades hereditarias discretas que dan lugar a características físicas observables. Esta descripción prefiguró la distinción de Wilhelm Johannsen entre genotipo (el material genético de un organismo) y fenotipo (los rasgos observables de ese organismo). Mendel también fue el primero en demostrar el surtido independiente , la distinción entre rasgos dominantes y recesivos , la distinción entre heterocigoto y homocigoto y el fenómeno de la herencia discontinua.
Antes del trabajo de Mendel, la teoría dominante de la herencia era la de la herencia mixta , [35] que sugería que cada padre aportaba fluidos al proceso de fertilización y que los rasgos de los padres se mezclaban y mezclaban para producir la descendencia. Charles Darwin desarrolló una teoría de la herencia que denominó pangénesis , del griego pan ("todo, todo") y génesis ("nacimiento")/genos ("origen"). [36] [37] Darwin usó el término gémula para describir partículas hipotéticas que se mezclarían durante la reproducción.
El trabajo de Mendel pasó prácticamente desapercibido después de su primera publicación en 1866, pero fue redescubierto a finales del siglo XIX por Hugo de Vries , Carl Correns y Erich von Tschermak , quienes (afirmaron haber) llegado a conclusiones similares en sus propias investigaciones. [38] En concreto, en 1889, Hugo de Vries publicó su libro Pangénesis intracelular , [39] en el que postulaba que diferentes caracteres tienen portadores hereditarios individuales y que la herencia de rasgos específicos en los organismos viene en partículas. De Vries llamó a estas unidades "pangenes" ( Pangens en alemán), en honor a la teoría de la pangénesis de Darwin de 1868.
Veinte años más tarde, en 1909, Wilhelm Johannsen introdujo el término 'gen' [8] y en 1906, William Bateson , el de ' genética ' [40] [33] mientras que Eduard Strasburger , entre otros, todavía utilizaba el término 'pangene'. para la unidad física y funcional fundamental de la herencia. [39] : Prefacio del traductor, viii
Los avances en la comprensión de los genes y la herencia continuaron a lo largo del siglo XX. Se demostró que el ácido desoxirribonucleico (ADN) era el depósito molecular de la información genética mediante experimentos realizados entre los años 1940 y 1950. [41] [42] La estructura del ADN fue estudiada por Rosalind Franklin y Maurice Wilkins utilizando cristalografía de rayos X , lo que llevó a James D. Watson y Francis Crick a publicar un modelo de la molécula de ADN bicatenario cuyas bases de nucleótidos emparejadas indicaban una Hipótesis convincente sobre el mecanismo de replicación genética. [43] [44]
A principios de la década de 1950, la opinión predominante era que los genes de un cromosoma actuaban como entidades discretas dispuestas como cuentas en un hilo. Los experimentos de Benzer utilizando mutantes defectuosos en la región rII del bacteriófago T4 (1955-1959) demostraron que los genes individuales tienen una estructura lineal simple y probablemente sean equivalentes a una sección lineal de ADN. [45] [46]
En conjunto, este cuerpo de investigación estableció el dogma central de la biología molecular , que establece que las proteínas se traducen a partir de ARN , que a su vez se transcribe a partir del ADN . Desde entonces se ha demostrado que este dogma tiene excepciones, como la transcripción inversa en los retrovirus . El estudio moderno de la genética a nivel del ADN se conoce como genética molecular .
En 1972, Walter Fiers y su equipo fueron los primeros en determinar la secuencia de un gen: el de la proteína de cubierta del bacteriófago MS2 . [47] El desarrollo posterior de la secuenciación de ADN por terminación de cadena en 1977 por Frederick Sanger mejoró la eficiencia de la secuenciación y la convirtió en una herramienta de laboratorio de rutina. [48] En las primeras fases del Proyecto Genoma Humano se utilizó una versión automatizada del método Sanger . [49]
Las teorías desarrolladas a principios del siglo XX para integrar la genética mendeliana con la evolución darwiniana se denominan síntesis moderna , término introducido por Julian Huxley . [50]
Esta visión de la evolución fue enfatizada por la visión de la evolución centrada en los genes de George C. Williams . Propuso que el gen mendeliano es una unidad de selección natural con la definición: "aquello que se segrega y recombina con frecuencia apreciable". [51] : 24 Richard Dawkins popularizó ideas relacionadas que enfatizaban la centralidad de los genes mendelianos y la importancia de la selección natural en la evolución . [13] [52]
El desarrollo de la teoría neutral de la evolución a finales de la década de 1960 llevó al reconocimiento de que la deriva genética aleatoria es un actor importante en la evolución y que la teoría neutral debería ser la hipótesis nula de la evolución molecular. [53] Esto condujo a la construcción de árboles filogenéticos y al desarrollo del reloj molecular , que es la base de todas las técnicas de datación que utilizan secuencias de ADN. Estas técnicas no se limitan a secuencias de genes moleculares, sino que pueden usarse en todos los segmentos de ADN del genoma.

La gran mayoría de organismos codifican sus genes en largas hebras de ADN (ácido desoxirribonucleico). El ADN consta de una cadena formada por cuatro tipos de subunidades de nucleótidos , cada una compuesta por: un azúcar de cinco carbonos ( 2-desoxirribosa ), un grupo fosfato y una de las cuatro bases adenina , citosina , guanina y timina . [54] : 2.1
Dos cadenas de ADN se retuercen entre sí para formar una doble hélice de ADN con la columna vertebral de fosfato-azúcar girando en espiral alrededor del exterior y las bases apuntando hacia adentro con las bases de adenina emparentadas con timina y guanina con citosina. La especificidad del emparejamiento de bases se produce porque la adenina y la timina se alinean para formar dos enlaces de hidrógeno , mientras que la citosina y la guanina forman tres enlaces de hidrógeno. Por lo tanto, las dos hebras de una doble hélice deben ser complementarias , con su secuencia de bases coincidente de modo que las adeninas de una hebra estén emparejadas con las timinas de la otra hebra, y así sucesivamente. [54] : 4.1
Debido a la composición química de los residuos de pentosas de las bases, las cadenas de ADN tienen direccionalidad. Un extremo de un polímero de ADN contiene un grupo hidroxilo expuesto en la desoxirribosa ; esto se conoce como el extremo 3' de la molécula. El otro extremo contiene un grupo fosfato expuesto; este es el extremo 5' . Las dos hebras de una doble hélice corren en direcciones opuestas. La síntesis de ácidos nucleicos, incluida la replicación y transcripción del ADN , se produce en la dirección 5'→3', porque se agregan nuevos nucleótidos mediante una reacción de deshidratación que utiliza el hidroxilo 3' expuesto como nucleófilo . [55] : 27,2
La expresión de genes codificados en el ADN comienza con la transcripción del gen en ARN , un segundo tipo de ácido nucleico muy similar al ADN, pero cuyos monómeros contienen el azúcar ribosa en lugar de desoxirribosa . El ARN también contiene la base uracilo en lugar de timina . Las moléculas de ARN son menos estables que las de ADN y suelen ser monocatenarias. Los genes que codifican proteínas están compuestos por una serie de secuencias de tres nucleótidos llamadas codones , que sirven como "palabras" en el "lenguaje" genético. El código genético especifica la correspondencia durante la traducción de proteínas entre codones y aminoácidos . El código genético es casi el mismo para todos los organismos conocidos. [54] : 4.1


El complemento total de genes de un organismo o célula se conoce como genoma y puede almacenarse en uno o más cromosomas . Un cromosoma consta de una única hélice de ADN muy larga en la que se codifican miles de genes. [54] : 4.2 La región del cromosoma en la que se encuentra un gen particular se llama locus . Cada locus contiene un alelo de un gen; sin embargo, los miembros de una población pueden tener diferentes alelos en el locus, cada uno con una secuencia genética ligeramente diferente.
La mayoría de los genes eucariotas se almacenan en un conjunto de cromosomas lineales grandes. Los cromosomas están empaquetados dentro del núcleo en complejos con proteínas de almacenamiento llamadas histonas para formar una unidad llamada nucleosoma . El ADN empaquetado y condensado de esta manera se llama cromatina . [54] : 4.2 La manera en que el ADN se almacena en las histonas, así como las modificaciones químicas de la propia histona, regulan si una región particular del ADN es accesible para la expresión genética . Además de los genes, los cromosomas eucarióticos contienen secuencias que garantizan que el ADN se copie sin degradación de las regiones terminales y se clasifique en células hijas durante la división celular: orígenes de replicación , telómeros y centrómero . [54] : 4.2 Los orígenes de replicación son las regiones de secuencia donde se inicia la replicación del ADN para producir dos copias del cromosoma. Los telómeros son largos tramos de secuencias repetitivas que cubren los extremos de los cromosomas lineales y evitan la degradación de las regiones codificantes y reguladoras durante la replicación del ADN . La longitud de los telómeros disminuye cada vez que se replica el genoma y se ha implicado en el proceso de envejecimiento . [57] El centrómero es necesario para unir las fibras del huso para separar las cromátidas hermanas en células hijas durante la división celular . [54] : 18,2
Los procariotas ( bacterias y arqueas ) normalmente almacenan sus genomas en un único cromosoma circular grande . De manera similar, algunos orgánulos eucariotas contienen un cromosoma circular remanente con una pequeña cantidad de genes. [54] : 14.4 Los procariotas a veces complementan sus cromosomas con pequeños círculos adicionales de ADN llamados plásmidos , que normalmente codifican sólo unos pocos genes y son transferibles entre individuos. Por ejemplo, los genes de resistencia a los antibióticos suelen estar codificados en plásmidos bacterianos y pueden transmitirse entre células individuales, incluso entre especies diferentes, mediante transferencia genética horizontal . [58]
Mientras que los cromosomas de los procariotas son relativamente densos en genes, los de los eucariotas a menudo contienen regiones de ADN que no cumplen ninguna función obvia. Los eucariotas unicelulares simples tienen cantidades relativamente pequeñas de dicho ADN, mientras que los genomas de organismos multicelulares complejos , incluidos los humanos, contienen una mayoría absoluta de ADN sin una función identificada. [59] Este ADN a menudo se ha denominado " ADN basura ". Sin embargo, análisis más recientes sugieren que, aunque el ADN que codifica proteínas constituye apenas el 2% del genoma humano , alrededor del 80% de las bases del genoma pueden expresarse, por lo que el término "ADN basura" puede ser un nombre inapropiado. [30]
La estructura de un gen codificante de proteínas consta de muchos elementos de los cuales la secuencia codificante de proteínas real suele ser sólo una pequeña parte. Estos incluyen intrones y regiones no traducidas del ARNm maduro. Los genes no codificantes también pueden contener intrones que se eliminan durante el procesamiento para producir ARN funcional maduro.
Todos los genes están asociados con secuencias reguladoras necesarias para su expresión. Primero, los genes requieren una secuencia promotora . El promotor es reconocido y unido por factores de transcripción que reclutan y ayudan a la ARN polimerasa a unirse a la región para iniciar la transcripción. [54] : 7.1 El reconocimiento normalmente ocurre como una secuencia de consenso como la caja TATA . Un gen puede tener más de un promotor, lo que da como resultado ARN mensajeros ( ARNm ) que difieren en su extensión en el extremo 5'. [61] Los genes altamente transcritos tienen secuencias promotoras "fuertes" que forman fuertes asociaciones con factores de transcripción, iniciando así la transcripción a un ritmo elevado. Otros genes tienen promotores "débiles" que forman asociaciones débiles con factores de transcripción e inician la transcripción con menos frecuencia. [54] : 7.2 Las regiones promotoras eucariotas son mucho más complejas y difíciles de identificar que los promotores procarióticos . [54] : 7,3
Además, los genes pueden tener regiones reguladoras de muchas kilobases aguas arriba o aguas abajo del gen que alteran la expresión. Estos actúan uniéndose a factores de transcripción que luego hacen que el ADN forme un bucle de modo que la secuencia reguladora (y el factor de transcripción unido) se acerquen al sitio de unión de la ARN polimerasa. [62] Por ejemplo, los potenciadores aumentan la transcripción al unirse a una proteína activadora que luego ayuda a reclutar la ARN polimerasa al promotor; por el contrario, los silenciadores se unen a proteínas represoras y hacen que el ADN esté menos disponible para la ARN polimerasa. [63]
El ARN mensajero maduro producido a partir de genes codificadores de proteínas contiene regiones no traducidas en ambos extremos que contienen sitios de unión para ribosomas , proteínas de unión a ARN , miARN , así como terminadores y codones de inicio y parada . [64] Además, la mayoría de los marcos de lectura abiertos de eucariotas contienen intrones no traducidos , que se eliminan, y exones , que se conectan entre sí en un proceso conocido como empalme de ARN . Finalmente, los extremos de las transcripciones de genes están definidos por sitios de escisión y poliadenilación (CPA) , donde se escinde el pre-ARNm recién producido y se agrega una cadena de ~200 monofosfatos de adenosina en el extremo 3'. La cola poli(A) protege el ARNm maduro de la degradación y tiene otras funciones que afectan la traducción, la localización y el transporte del transcrito desde el núcleo. El empalme, seguido de CPA, genera el ARNm maduro final , que codifica la proteína o el producto de ARN. [65] Aunque se conocen los mecanismos generales que definen las ubicaciones de los genes humanos, la identificación de los factores exactos que regulan estos procesos celulares es un área de investigación activa. Por ejemplo, las características de secuencia conocidas en la 3'-UTR sólo pueden explicar la mitad de todos los extremos de los genes humanos. [66]
Muchos genes no codificantes en eucariotas tienen diferentes mecanismos de terminación de la transcripción y no tienen colas pol(A).
Muchos genes procarióticos están organizados en operones , con múltiples secuencias codificadoras de proteínas que se transcriben como una unidad. [67] [68] Los genes de un operón se transcriben como un ARN mensajero continuo , denominado ARNm policistrónico . El término cistrón en este contexto equivale a gen. La transcripción del ARNm de un operón suele estar controlada por un represor que puede ocurrir en un estado activo o inactivo dependiendo de la presencia de metabolitos específicos. [69] Cuando está activo, el represor se une a una secuencia de ADN al comienzo del operón, llamada región operadora , y reprime la transcripción del operón ; cuando el represor está inactivo puede ocurrir la transcripción del operón (ver, por ejemplo, operón Lac ). Los productos de los genes de operones suelen tener funciones relacionadas y están implicados en la misma red reguladora . [54] : 7,3
Aunque muchos genes tienen estructuras simples, como ocurre con gran parte de la biología, otros pueden ser bastante complejos o representar casos extremos inusuales. Los genes eucariotas a menudo tienen intrones que suelen ser mucho más grandes que sus exones, [70] [71] y esos intrones pueden incluso tener otros genes anidados en su interior . [72] Los potenciadores asociados pueden estar a muchas kilobases de distancia, o incluso en cromosomas completamente diferentes que operan a través del contacto físico entre dos cromosomas. [73] [74] Un solo gen puede codificar múltiples productos funcionales diferentes mediante empalme alternativo y, a la inversa, el gen puede dividirse entre cromosomas, pero esas transcripciones se concatenan nuevamente en una secuencia funcional mediante empalme trans . [75] También es posible que genes superpuestos compartan parte de su secuencia de ADN, ya sea en cadenas opuestas o en la misma cadena (en un marco de lectura diferente, o incluso en el mismo marco de lectura). [76]
En todos los organismos, se requieren dos pasos para leer la información codificada en el ADN de un gen y producir la proteína que especifica. Primero, el ADN del gen se transcribe a ARN mensajero ( ARNm ). [54] : 6.1 En segundo lugar, que el ARNm se traduce en proteína. [54] : 6.2 Los genes codificadores de ARN aún deben pasar por el primer paso, pero no se traducen en proteínas. [77] El proceso de producir una molécula biológicamente funcional de ARN o proteína se llama expresión genética , y la molécula resultante se llama producto genético .

La secuencia de nucleótidos del ADN de un gen especifica la secuencia de aminoácidos de una proteína a través del código genético . Conjuntos de tres nucleótidos, conocidos como codones , corresponden cada uno a un aminoácido específico. [54] : 6 El principio de que tres bases secuenciales del ADN codifican para cada aminoácido se demostró en 1961 utilizando mutaciones de desplazamiento del marco de lectura en el gen rIIB del bacteriófago T4 [78] (ver experimento de Crick, Brenner et al. ).
Además, un " codón de inicio " y tres " codones de parada " indican el comienzo y el final de la región codificante de la proteína . Hay 64 codones posibles (cuatro nucleótidos posibles en cada una de las tres posiciones, por lo tanto, 4 3 codones posibles) y sólo 20 aminoácidos estándar; por tanto, el código es redundante y varios codones pueden especificar el mismo aminoácido. La correspondencia entre codones y aminoácidos es casi universal entre todos los organismos vivos conocidos. [79]
La transcripción produce una molécula de ARN monocatenaria conocida como ARN mensajero , cuya secuencia de nucleótidos es complementaria al ADN a partir del cual fue transcrita. [54] : 6.1 El ARNm actúa como intermediario entre el gen del ADN y su producto proteico final. El ADN del gen se utiliza como plantilla para generar un ARNm complementario . El ARNm coincide con la secuencia de la cadena codificante de ADN del gen porque se sintetiza como complemento de la cadena plantilla . La transcripción la realiza una enzima llamada ARN polimerasa , que lee la cadena plantilla en la dirección 3' a 5' y sintetiza el ARN de 5' a 3' . Para iniciar la transcripción, la polimerasa primero reconoce y se une a una región promotora del gen. Por lo tanto, un mecanismo importante de regulación genética es el bloqueo o secuestro de la región promotora, ya sea mediante una unión estrecha mediante moléculas represoras que bloquean físicamente la polimerasa o organizando el ADN de manera que la región promotora no sea accesible. [54] : 7
En los procariotas , la transcripción se produce en el citoplasma ; para transcripciones muy largas, la traducción puede comenzar en el extremo 5' del ARN mientras el extremo 3' todavía se está transcribiendo. En los eucariotas , la transcripción se produce en el núcleo, donde se almacena el ADN de la célula. La molécula de ARN producida por la polimerasa se conoce como transcripción primaria y sufre modificaciones postranscripcionales antes de ser exportada al citoplasma para su traducción. Una de las modificaciones realizadas es el empalme de intrones que son secuencias en la región transcrita que no codifican una proteína. Mecanismos de corte y empalme alternativos pueden dar como resultado transcripciones maduras del mismo gen que tienen secuencias diferentes y, por lo tanto, codifican proteínas diferentes. Esta es una forma importante de regulación en las células eucariotas y también ocurre en algunas procariotas. [54] : 7,5 [80]

La traducción es el proceso mediante el cual una molécula de ARNm madura se utiliza como plantilla para sintetizar una nueva proteína . [54] : 6.2 La traducción la llevan a cabo los ribosomas , grandes complejos de ARN y proteínas responsables de llevar a cabo las reacciones químicas para agregar nuevos aminoácidos a una cadena polipeptídica en crecimiento mediante la formación de enlaces peptídicos . El código genético se lee tres nucleótidos a la vez, en unidades llamadas codones , mediante interacciones con moléculas de ARN especializadas llamadas ARN de transferencia (ARNt). Cada ARNt tiene tres bases desapareadas conocidas como anticodón que son complementarias al codón que lee en el ARNm. El ARNt también está unido covalentemente al aminoácido especificado por el codón complementario. Cuando el ARNt se une a su codón complementario en una cadena de ARNm, el ribosoma une su carga de aminoácidos a la nueva cadena polipeptídica, que se sintetiza desde el extremo amino al extremo carboxilo . Durante y después de la síntesis, la mayoría de las proteínas nuevas deben plegarse a su estructura tridimensional activa antes de que puedan llevar a cabo sus funciones celulares. [54] : 3
Los genes están regulados para que se expresen sólo cuando se necesita el producto, ya que la expresión requiere recursos limitados. [54] : 7 Una célula regula su expresión genética dependiendo de su entorno externo (p. ej. , nutrientes disponibles , temperatura y otras tensiones ), su entorno interno (p. ej. , ciclo de división celular , metabolismo , estado de infección ) y su función específica si se encuentra en una célula multicelular . organismo. La expresión génica se puede regular en cualquier paso: desde el inicio de la transcripción hasta el procesamiento del ARN y la modificación postraduccional de la proteína. La regulación de los genes del metabolismo de la lactosa en E. coli ( operón lac ) fue el primer mecanismo de este tipo que se describió en 1961. [81]
Un gen codificador de proteínas típico se copia primero en ARN como intermediario en la fabricación del producto proteico final. [54] : 6.1 En otros casos, las moléculas de ARN son los productos funcionales reales, como en la síntesis de ARN ribosómico y ARN de transferencia . Algunos ARN conocidos como ribozimas son capaces de realizar funciones enzimáticas , mientras que otros, como los microARN y los riboswitches, tienen funciones reguladoras. Las secuencias de ADN a partir de las cuales se transcriben dichos ARN se conocen como genes de ARN no codificantes . [77]
Algunos virus almacenan su genoma completo en forma de ARN y no contienen ADN alguno. [82] [83] Debido a que utilizan ARN para almacenar genes, sus huéspedes celulares pueden sintetizar sus proteínas tan pronto como se infectan y sin la demora de espera de la transcripción. [84] Por otro lado, los retrovirus de ARN , como el VIH , requieren la transcripción inversa de su genoma de ARN a ADN antes de que sus proteínas puedan sintetizarse.

Los organismos heredan sus genes de sus padres. Los organismos asexuales simplemente heredan una copia completa del genoma de sus padres. Los organismos sexuales tienen dos copias de cada cromosoma porque heredan un juego completo de cada padre. [54] : 1
Según la herencia mendeliana , las variaciones en el fenotipo de un organismo (características físicas y de comportamiento observables) se deben en parte a variaciones en su genotipo (un conjunto particular de genes). Cada gen especifica un rasgo particular con una secuencia diferente de un gen ( alelos ) que da lugar a diferentes fenotipos. La mayoría de los organismos eucariotas (como las plantas de guisantes en las que trabajó Mendel) tienen dos alelos para cada rasgo, uno heredado de cada padre. [54] : 20
Los alelos de un locus pueden ser dominantes o recesivos ; Los alelos dominantes dan lugar a sus fenotipos correspondientes cuando se combinan con cualquier otro alelo para el mismo rasgo, mientras que los alelos recesivos dan lugar a su fenotipo correspondiente sólo cuando se combinan con otra copia del mismo alelo. Si conoces los genotipos de los organismos, podrás determinar qué alelos son dominantes y cuáles son recesivos. Por ejemplo, si el alelo que especifica tallos altos en las plantas de guisantes es dominante sobre el alelo que especifica tallos cortos, entonces las plantas de guisantes que heredan un alelo alto de un padre y un alelo corto del otro padre también tendrán tallos altos. El trabajo de Mendel demostró que los alelos se clasifican de forma independiente en la producción de gametos o células germinales , lo que garantiza la variación en la siguiente generación. Aunque la herencia mendeliana sigue siendo un buen modelo para muchos rasgos determinados por genes individuales (incluidos varios trastornos genéticos bien conocidos ), no incluye los procesos físicos de replicación del ADN y división celular. [85] [86]
El crecimiento, desarrollo y reproducción de los organismos depende de la división celular ; el proceso por el cual una sola célula se divide en dos células hijas generalmente idénticas . Esto requiere primero hacer una copia duplicada de cada gen del genoma en un proceso llamado replicación del ADN . [54] : 5.2 Las copias son realizadas por enzimas especializadas conocidas como ADN polimerasas , que "lee" una hebra del ADN de doble hélice, conocida como hebra plantilla, y sintetiza una nueva hebra complementaria. Debido a que la doble hélice del ADN se mantiene unida mediante el emparejamiento de bases , la secuencia de una hebra especifica completamente la secuencia de su complemento; por lo tanto, la enzima sólo necesita leer una hebra para producir una copia fiel. El proceso de replicación del ADN es semiconservativo ; es decir, la copia del genoma heredada por cada célula hija contiene una hebra de ADN original y otra recién sintetizada. [54] : 5,2
La tasa de replicación del ADN en células vivas se midió por primera vez como la tasa de elongación del ADN del fago T4 en E. coli infectada con fagos y se descubrió que era impresionantemente rápida. [87] Durante el período de aumento exponencial del ADN a 37 °C, la tasa de elongación fue de 749 nucleótidos por segundo.
Una vez completada la replicación del ADN, la célula debe separar físicamente las dos copias del genoma y dividirse en dos células distintas unidas a una membrana. [54] : 18.2 En los procariotas ( bacterias y arqueas ), esto generalmente ocurre mediante un proceso relativamente simple llamado fisión binaria , en el que cada genoma circular se adhiere a la membrana celular y se separa en las células hijas a medida que la membrana se invagina para dividir el citoplasma en dos porciones unidas a membrana. La fisión binaria es extremadamente rápida en comparación con las tasas de división celular de los eucariotas . La división de las células eucariotas es un proceso más complejo conocido como ciclo celular ; La replicación del ADN ocurre durante una fase de este ciclo conocida como fase S , mientras que el proceso de segregación de cromosomas y división del citoplasma ocurre durante la fase M. [54] : 18,1
La duplicación y transmisión de material genético de una generación de células a la siguiente es la base de la herencia molecular y el vínculo entre las imágenes clásica y molecular de los genes. Los organismos heredan las características de sus padres porque las células de la descendencia contienen copias de los genes en las células de sus padres. En los organismos que se reproducen asexualmente , la descendencia será una copia genética o un clon del organismo padre. En los organismos que se reproducen sexualmente , una forma especializada de división celular llamada meiosis produce células llamadas gametos o células germinales que son haploides o contienen solo una copia de cada gen. [54] : 20.2 Los gametos producidos por las hembras se llaman óvulos u óvulos, y los producidos por los machos se llaman espermatozoides . Dos gametos se fusionan para formar un óvulo fertilizado diploide , una única célula que tiene dos conjuntos de genes, con una copia de cada gen de la madre y otra del padre. [54] : 20
Durante el proceso de división celular meiótica, a veces puede ocurrir un evento llamado recombinación o entrecruzamiento genético , en el que una longitud de ADN en una cromátida se intercambia con una longitud de ADN en la cromátida no hermana homóloga correspondiente. Esto puede dar lugar a una reordenación de alelos que de otro modo estarían vinculados. [54] : 5.5 El principio mendeliano de distribución independiente afirma que cada uno de los dos genes de un padre para cada rasgo se clasificará de forma independiente en gametos; qué alelo hereda un organismo para un rasgo no está relacionado con qué alelo hereda para otro rasgo. De hecho, esto sólo es cierto para los genes que no residen en el mismo cromosoma o que están ubicados muy lejos unos de otros en el mismo cromosoma. Cuanto más cerca se encuentren dos genes en el mismo cromosoma, más estrechamente estarán asociados en los gametos y con mayor frecuencia aparecerán juntos (lo que se conoce como ligamiento genético ). [88] Los genes que están muy cerca esencialmente nunca se separan porque es extremadamente improbable que se produzca un punto de cruce entre ellos. [88]
La replicación del ADN es en su mayor parte extremadamente precisa, sin embargo, se producen errores ( mutaciones ). [54] : 7,6 La tasa de error en células eucariotas puede ser tan baja como 10 −8 por nucleótido por replicación, [89] [90] mientras que para algunos virus de ARN puede ser tan alta como 10 −3 . [91] Esto significa que cada generación, cada genoma humano acumula alrededor de 30 nuevas mutaciones. [92] Las pequeñas mutaciones pueden ser causadas por la replicación del ADN y las consecuencias del daño del ADN e incluyen mutaciones puntuales en las que se altera una sola base y mutaciones de cambio de marco en las que se inserta o elimina una sola base. Cualquiera de estas mutaciones puede cambiar el gen por sentido erróneo (cambiar un codón para codificar un aminoácido diferente) o sin sentido (un codón de parada prematuro ). [93] Las mutaciones más grandes pueden ser causadas por errores en la recombinación que causan anomalías cromosómicas, incluida la duplicación , eliminación, reordenamiento o inversión de grandes secciones de un cromosoma. Además, los mecanismos de reparación del ADN pueden introducir errores mutacionales al reparar el daño físico de la molécula. La reparación, incluso en caso de mutación, es más importante para la supervivencia que restaurar una copia exacta, por ejemplo cuando se reparan roturas de doble cadena . [54] : 5,4
Cuando varios alelos diferentes de un gen están presentes en la población de una especie, se denomina polimórfico . La mayoría de los alelos diferentes son funcionalmente equivalentes; sin embargo, algunos alelos pueden dar lugar a rasgos fenotípicos diferentes . El alelo más común de un gen se llama tipo salvaje y los alelos raros se llaman mutantes . La variación genética en las frecuencias relativas de diferentes alelos en una población se debe tanto a la selección natural como a la deriva genética . [94] El alelo de tipo salvaje no es necesariamente el ancestro de alelos menos comunes, ni es necesariamente más apto .
La mayoría de las mutaciones dentro de los genes son neutrales y no tienen ningún efecto sobre el fenotipo del organismo ( mutaciones silenciosas ). Algunas mutaciones no cambian la secuencia de aminoácidos porque múltiples codones codifican el mismo aminoácido ( mutaciones sinónimas ). Otras mutaciones pueden ser neutrales si conducen a cambios en la secuencia de aminoácidos, pero la proteína aún funciona de manera similar al nuevo aminoácido (por ejemplo, mutaciones conservadoras ). Sin embargo, muchas mutaciones son perjudiciales o incluso letales y se eliminan de las poblaciones mediante selección natural. Los trastornos genéticos son el resultado de mutaciones nocivas y pueden deberse a una mutación espontánea en el individuo afectado o pueden heredarse. Finalmente, una pequeña fracción de las mutaciones son beneficiosas , mejoran la aptitud del organismo y son extremadamente importantes para la evolución, ya que su selección direccional conduce a una evolución adaptativa . [54] : 7,6
La relación entre genes se puede medir comparando las secuencias de su ADN. Si el nivel de similitud excede un valor mínimo, se puede concluir que los genes descienden de un ancestro común; son homólogos . [95] [96] Los genes que están relacionados por descendencia directa de un ancestro común son genes ortólogos: generalmente se encuentran en el mismo locus en diferentes especies. Los genes que están relacionados como resultado de un evento de duplicación genética son genes parólogos. [97] [98]
A menudo se supone que las funciones de los genes ortólogos son más similares que las de los genes parálogos, aunque la diferencia es mínima. [99] [100]
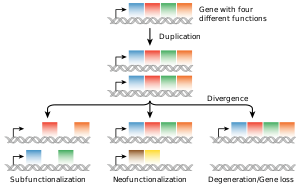
La fuente más común de nuevos genes en linajes eucariotas es la duplicación de genes , que crea una variación en el número de copias de un gen existente en el genoma. [101] [102] Los genes resultantes (parálogos) pueden entonces divergir en secuencia y función. Conjuntos de genes formados de esta manera componen una familia de genes . Las duplicaciones y pérdidas de genes dentro de una familia son comunes y representan una fuente importante de biodiversidad evolutiva . [103] A veces, la duplicación genética puede dar como resultado una copia no funcional de un gen, o una copia funcional puede estar sujeta a mutaciones que resultan en la pérdida de función; Estos genes no funcionales se denominan pseudogenes . [54] : 7,6
Los genes "huérfanos" , cuya secuencia no muestra similitud con genes existentes, son menos comunes que los duplicados de genes. El genoma humano contiene aproximadamente entre 18 [104] y 60 [105] genes sin homólogos identificables fuera de los humanos. Los genes huérfanos surgen principalmente de la aparición de novo de una secuencia previamente no codificante o de la duplicación de genes seguida de un cambio de secuencia tan rápido que la relación original se vuelve indetectable. [106] Los genes de novo suelen ser más cortos y de estructura más simple que la mayoría de los genes eucarióticos, con pocos o ningún intrón. [101] Durante largos períodos de tiempo evolutivos, el nacimiento de genes de novo puede ser responsable de una fracción significativa de familias de genes taxonómicamente restringidas. [107]
La transferencia horizontal de genes se refiere a la transferencia de material genético a través de un mecanismo distinto a la reproducción . Este mecanismo es una fuente común de nuevos genes en los procariotas , y a veces se piensa que contribuye más a la variación genética que a la duplicación de genes. [108] Es un medio común para propagar la resistencia a los antibióticos , la virulencia y las funciones metabólicas adaptativas . [58] [109] Aunque la transferencia horizontal de genes es rara en eucariotas, se han identificado ejemplos probables de genomas de protistas y algas que contienen genes de origen bacteriano. [110] [111]
El genoma es el material genético total de un organismo e incluye tanto los genes como las secuencias no codificantes . [112] Los genes eucariotas se pueden anotar utilizando FINDER. [113]

El tamaño del genoma y la cantidad de genes que codifica varían ampliamente entre organismos. Los genomas más pequeños se encuentran en virus , [122] y viroides (que actúan como un único gen de ARN no codificante). [123] Por el contrario, las plantas pueden tener genomas extremadamente grandes, [124] y el arroz contiene >46.000 genes codificadores de proteínas. [118] Se estima que el número total de genes codificadores de proteínas (el proteoma de la Tierra ) es de 5 millones de secuencias. [125]
Aunque el número de pares de bases del ADN en el genoma humano se conoce desde la década de 1950, el número estimado de genes ha cambiado con el tiempo a medida que se han perfeccionado las definiciones de genes y los métodos para detectarlos. Las predicciones teóricas iniciales sobre el número de genes humanos en las décadas de 1960 y 1970 se basaban en estimaciones de la carga de mutaciones y del número de ARNm, y estas estimaciones tendían a ser de unos 30.000 genes codificadores de proteínas. [126] [127] [128] Durante la década de 1990 hubo estimaciones de hasta 100.000 genes y los primeros datos sobre la detección de ARNm ( etiquetas de secuencia expresadas ) sugirieron más que el valor tradicional de 30.000 genes que se había informado en los libros de texto durante la década de 1990. Década de 1980. [129]
El borrador inicial de secuencias del genoma humano confirmó las predicciones anteriores de alrededor de 30.000 genes codificadores de proteínas; sin embargo, esa estimación se ha reducido a alrededor de 19.000 con el proyecto de anotación GENCODE en curso . [130] El número de genes no codificantes no se conoce con certeza, pero las últimas estimaciones de Ensembl sugieren 26.000 genes no codificantes. [131]
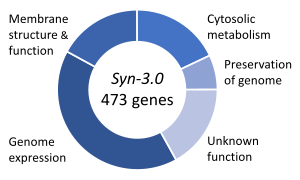
Los genes esenciales son el conjunto de genes que se cree que son críticos para la supervivencia de un organismo. [133] Esta definición supone la disponibilidad abundante de todos los nutrientes relevantes y la ausencia de estrés ambiental. Sólo una pequeña porción de los genes de un organismo son esenciales. En las bacterias, se estima que entre 250 y 400 genes son esenciales para Escherichia coli y Bacillus subtilis , lo que representa menos del 10% de sus genes. [134] [135] [136] La mitad de estos genes son ortólogos en ambos organismos y participan en gran medida en la síntesis de proteínas . [136] En la levadura en ciernes Saccharomyces cerevisiae, el número de genes esenciales es ligeramente mayor, 1000 genes (~20% de sus genes). [137] Aunque el número es más difícil de medir en eucariotas superiores, se estima que los ratones y los humanos tienen alrededor de 2000 genes esenciales (~10% de sus genes). [138] El organismo sintético, Syn 3 , tiene un genoma mínimo de 473 genes esenciales y genes cuasi esenciales (necesarios para un crecimiento rápido), aunque 149 tienen una función desconocida. [132]
Los genes esenciales incluyen genes de mantenimiento (críticos para las funciones celulares básicas) [139] , así como genes que se expresan en diferentes momentos del desarrollo o ciclo de vida del organismo . [140] Los genes de mantenimiento se utilizan como controles experimentales al analizar la expresión genética , ya que se expresan constitutivamente a un nivel relativamente constante.
La nomenclatura genética ha sido establecida por el Comité de Nomenclatura Genética de HUGO (HGNC), un comité de la Organización del Genoma Humano , para cada gen humano conocido en forma de un nombre de gen aprobado y un símbolo ( abreviatura abreviada ), al que se puede acceder a través de una base de datos mantenida por HGNC. Los símbolos se eligen para que sean únicos y cada gen tiene un solo símbolo (aunque los símbolos aprobados a veces cambian). Es preferible que los símbolos se mantengan coherentes con otros miembros de una familia de genes y con homólogos de otras especies, en particular el ratón debido a su papel como organismo modelo común . [141]
La ingeniería genética es la modificación del genoma de un organismo mediante la biotecnología . Desde la década de 1970, se han desarrollado una variedad de técnicas para agregar, eliminar y editar genes específicamente en un organismo. [142] Las técnicas de ingeniería genómica desarrolladas recientemente utilizan enzimas nucleasas diseñadas para crear una reparación específica del ADN en un cromosoma para alterar o editar un gen cuando se repara la rotura. [143] [144] [145] [146] El término relacionado biología sintética se utiliza a veces para referirse a la ingeniería genética extensa de un organismo. [147]
La ingeniería genética es ahora una herramienta de investigación rutinaria con organismos modelo . Por ejemplo, los genes se añaden fácilmente a las bacterias [148] y se utilizan linajes de ratones knockout con la función de un gen específico alterada para investigar la función de ese gen. [149] [150] Muchos organismos han sido modificados genéticamente para aplicaciones en agricultura , biotecnología industrial y medicina .
En el caso de los organismos multicelulares, normalmente se modifica el embrión , que crece hasta convertirse en un organismo adulto genéticamente modificado . [151] Sin embargo, los genomas de las células de un organismo adulto se pueden editar utilizando técnicas de terapia génica para tratar enfermedades genéticas.
… la ciencia misma [es decir, el estudio del mejoramiento e hibridación de plantas] aún no tiene nombre, y sólo podemos describir nuestra búsqueda mediante perífrasis engorrosas y a menudo engañosas.
Para resolver esta dificultad sugiero para la consideración de este Congreso el término
Genética
, que indica suficientemente que nuestros trabajos están dedicados a la elucidación de los fenómenos de la herencia y la variación: en otras palabras, a la fisiología de la Descendencia, con implicación en la problemas teóricos del evolucionista y del sistematista, y aplicación a los problemas prácticos de los criadores, ya sean de animales o de plantas.