
La química computacional es una rama de la química que utiliza simulaciones por computadora para ayudar a resolver problemas químicos. [2] Utiliza métodos de química teórica incorporados en programas informáticos para calcular las estructuras y propiedades de moléculas , grupos de moléculas y sólidos. [3] La importancia de este tema se debe al hecho de que, con la excepción de algunos hallazgos relativamente recientes relacionados con el ion molecular de hidrógeno ( catión dihidrógeno ), no es factible lograr una representación mecánica cuántica precisa de los sistemas químicos de forma analítica o en forma cerrada. [4] La complejidad inherente al problema de muchos cuerpos exacerba el desafío de proporcionar descripciones detalladas de los sistemas mecánicos cuánticos. [5] Si bien los resultados computacionales normalmente complementan la información obtenida por experimentos químicos , ocasionalmente pueden predecir fenómenos químicos no observados . [6]
La química computacional se diferencia de la química teórica , que implica una descripción matemática de la química. Sin embargo, la química computacional implica el uso de programas informáticos y habilidades matemáticas adicionales para modelar con precisión varios problemas químicos. En la química teórica, los químicos, físicos y matemáticos desarrollan algoritmos y programas informáticos para predecir propiedades atómicas y moleculares y vías de reacción para reacciones químicas. Los químicos computacionales, por el contrario, pueden simplemente aplicar programas informáticos y metodologías existentes a cuestiones químicas específicas. [7]
Históricamente, la química computacional ha tenido dos vertientes diferentes:
Estos aspectos, junto con el propósito de la química computacional, han dado lugar a toda una serie de algoritmos.
Basándose en los descubrimientos y teorías fundacionales en la historia de la mecánica cuántica , los primeros cálculos teóricos en química fueron los de Walter Heitler y Fritz London en 1927, utilizando la teoría del enlace de valencia . [9] Los libros que fueron influyentes en el desarrollo temprano de la química cuántica computacional incluyen Introducción a la mecánica cuántica de Linus Pauling y E. Bright Wilson de 1935 , con aplicaciones a la química , [10] Química cuántica de Eyring , Walter y Kimball de 1944 , [11] Mecánica de ondas elemental de Heitler de 1945 , con aplicaciones a la química cuántica , [12] y más tarde el libro de texto Valence de Coulson de 1952 , cada uno de los cuales sirvió como referencias principales para los químicos en las décadas siguientes. [13]
Con el desarrollo de la tecnología informática eficiente en la década de 1940, las soluciones de ecuaciones de onda elaboradas para sistemas atómicos complejos comenzaron a ser un objetivo realizable. A principios de la década de 1950, se realizaron los primeros cálculos orbitales atómicos semiempíricos. Los químicos teóricos se convirtieron en usuarios extensivos de las primeras computadoras digitales. Un avance significativo fue marcado por el artículo de Clemens CJ Roothaan de 1951 en Reviews of Modern Physics. [14] [15] Este artículo se centró principalmente en el enfoque "LCAO MO" (combinación lineal de orbitales atómicos y orbitales moleculares). Durante muchos años, fue el segundo artículo más citado en esa revista. [14] [15] Smith y Sutcliffe ofrecen una descripción muy detallada de dicho uso en el Reino Unido. [16] Los primeros cálculos ab initio del método Hartree-Fock sobre moléculas diatómicas se realizaron en 1956 en el MIT, utilizando un conjunto base de orbitales de Slater . [17] Para las moléculas diatómicas, un estudio sistemático utilizando un conjunto base mínimo y el primer cálculo con un conjunto base más grande fueron publicados por Ransil y Nesbet respectivamente en 1960. [18] Los primeros cálculos poliatómicos utilizando orbitales gaussianos se realizaron a finales de la década de 1950. Los primeros cálculos de interacción de configuración se realizaron en Cambridge en la computadora EDSAC en la década de 1950 utilizando orbitales gaussianos por Boys y colaboradores. [19] Para 1971, cuando se publicó una bibliografía de cálculos ab initio , [20] las moléculas más grandes incluidas eran naftaleno y azuleno . [21] [22] Schaefer ha publicado resúmenes de muchos desarrollos anteriores en la teoría ab initio . [23]
En 1964, los cálculos del método Hückel (utilizando un método de combinación lineal simple de orbitales atómicos (LCAO) para determinar las energías electrónicas de los orbitales moleculares de los electrones π en sistemas de hidrocarburos conjugados) de moléculas, que varían en complejidad desde butadieno y benceno hasta ovaleno , se generaron en computadoras en Berkeley y Oxford. [24] Estos métodos empíricos fueron reemplazados en la década de 1960 por métodos semiempíricos como CNDO . [25]
A principios de la década de 1970, comenzaron a usarse programas informáticos eficientes ab initio como ATMOL, Gaussian , IBMOL y POLYAYTOM, para acelerar los cálculos ab initio de orbitales moleculares. [26] De estos cuatro programas, solo Gaussian, ahora ampliamente expandido, todavía está en uso, pero muchos otros programas ahora están en uso. [26] Al mismo tiempo, los métodos de mecánica molecular , como el campo de fuerza MM2 , fueron desarrollados, principalmente por Norman Allinger . [27]
Una de las primeras menciones del término química computacional se puede encontrar en el libro Computers and Their Role in the Physical Sciences de 1970 de Sidney Fernbach y Abraham Haskell Taub, donde afirman: "Parece, por lo tanto, que la 'química computacional' puede finalmente ser cada vez más una realidad". [28] Durante la década de 1970, se empezaron a considerar métodos muy diferentes como parte de una nueva disciplina emergente de la química computacional . [29] El Journal of Computational Chemistry se publicó por primera vez en 1980.
La química computacional ha aparecido en varios premios Nobel, más notablemente en 1998 y 2013. Walter Kohn , "por su desarrollo de la teoría funcional de la densidad", y John Pople , "por su desarrollo de métodos computacionales en química cuántica", recibieron el Premio Nobel de Química de 1998. [30] Martin Karplus , Michael Levitt y Arieh Warshel recibieron el Premio Nobel de Química de 2013 por "el desarrollo de modelos multiescala para sistemas químicos complejos". [31]
Hay varios campos dentro de la química computacional.
Estos campos pueden dar lugar a varias aplicaciones como se muestra a continuación.

La química computacional es una herramienta para analizar sistemas catalíticos sin hacer experimentos. La teoría de la estructura electrónica moderna y la teoría del funcional de la densidad han permitido a los investigadores descubrir y comprender los catalizadores . [37] Los estudios computacionales aplican la química teórica a la investigación de la catálisis. Los métodos de la teoría del funcional de la densidad calculan las energías y los orbitales de las moléculas para dar modelos de esas estructuras. [38] Usando estos métodos, los investigadores pueden predecir valores como la energía de activación , la reactividad del sitio [39] y otras propiedades termodinámicas. [38]
Los datos que son difíciles de obtener experimentalmente se pueden encontrar utilizando métodos computacionales para modelar los mecanismos de los ciclos catalíticos. [39] Los químicos computacionales expertos proporcionan predicciones que se acercan a los datos experimentales con consideraciones adecuadas de métodos y conjuntos de bases. Con buenos datos computacionales, los investigadores pueden predecir cómo se pueden mejorar los catalizadores para reducir el costo y aumentar la eficiencia de estas reacciones. [38]
La química computacional se utiliza en el desarrollo de fármacos para modelar moléculas de fármacos potencialmente útiles y ayudar a las empresas a ahorrar tiempo y dinero en el desarrollo de fármacos. El proceso de descubrimiento de fármacos implica analizar datos, encontrar formas de mejorar las moléculas actuales, encontrar rutas sintéticas y probar esas moléculas. [36] La química computacional ayuda con este proceso al proporcionar predicciones de qué experimentos sería mejor hacer sin realizar otros experimentos. Los métodos computacionales también pueden encontrar valores que son difíciles de encontrar experimentalmente, como los pKa de los compuestos. [40] Los métodos como la teoría funcional de la densidad se pueden utilizar para modelar moléculas de fármacos y encontrar sus propiedades, como sus energías HOMO y LUMO y orbitales moleculares. Los químicos computacionales también ayudan a las empresas con el desarrollo de la informática, la infraestructura y los diseños de fármacos. [41]
Además de la síntesis de fármacos, los químicos computacionales también investigan los portadores de fármacos para nanomateriales . Esto permite a los investigadores simular entornos para probar la eficacia y la estabilidad de los portadores de fármacos. Comprender cómo interactúa el agua con estos nanomateriales garantiza la estabilidad del material en los cuerpos humanos. Estas simulaciones computacionales ayudan a los investigadores a optimizar el material y encontrar la mejor manera de estructurar estos nanomateriales antes de fabricarlos. [42]
Las bases de datos son útiles tanto para los químicos computacionales como para los no computacionales en la investigación y para verificar la validez de los métodos computacionales. Los datos empíricos se utilizan para analizar el error de los métodos computacionales en comparación con los datos experimentales. Los datos empíricos ayudan a los investigadores con sus métodos y conjuntos de bases para tener una mayor confianza en los resultados de los investigadores. Las bases de datos de química computacional también se utilizan para probar software o hardware para química computacional. [43]
Las bases de datos también pueden utilizar datos puramente calculados. Los datos puramente calculados utilizan valores calculados en lugar de valores experimentales para las bases de datos. Los datos puramente calculados evitan tener que lidiar con estos ajustes para diferentes condiciones experimentales, como la energía del punto cero. Estos cálculos también pueden evitar errores experimentales en el caso de moléculas difíciles de probar. Aunque los datos puramente calculados a menudo no son perfectos, identificar problemas suele ser más fácil para los datos calculados que para los experimentales. [43]
Las bases de datos también brindan acceso público a la información para que la utilicen los investigadores. Contienen datos que otros investigadores han encontrado y cargado en estas bases de datos para que cualquiera pueda buscarlos. Los investigadores utilizan estas bases de datos para encontrar información sobre moléculas de interés y aprender qué se puede hacer con ellas. Algunas bases de datos de química disponibles públicamente incluyen las siguientes. [43]
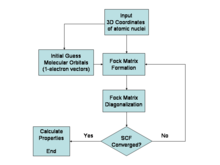
Los programas utilizados en química computacional se basan en muchos métodos químicos cuánticos diferentes que resuelven la ecuación molecular de Schrödinger asociada con el hamiltoniano molecular . [46] Los métodos que no incluyen ningún parámetro empírico o semiempírico en sus ecuaciones (derivados directamente de la teoría, sin incluir datos experimentales) se denominan métodos ab initio . [47] Una aproximación teórica se define rigurosamente en primeros principios y luego se resuelve dentro de un margen de error que se conoce cualitativamente de antemano. Si se deben utilizar métodos iterativos numéricos, el objetivo es iterar hasta obtener la precisión total de la máquina (lo mejor que sea posible con una longitud de palabra finita en la computadora y dentro de las aproximaciones matemáticas y/o físicas realizadas). [48]
Los métodos ab initio necesitan definir un nivel de teoría (el método) y un conjunto de bases. [49] Un conjunto de bases consiste en funciones centradas en los átomos de la molécula. Estos conjuntos se utilizan luego para describir orbitales moleculares a través del método de orbitales moleculares de combinación lineal de orbitales atómicos (LCAO) . [50]
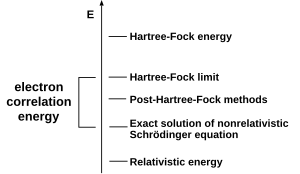
Un tipo común de cálculo de la estructura electrónica ab initio es el método Hartree-Fock (HF), una extensión de la teoría de orbitales moleculares , donde las repulsiones electrón-electrón en la molécula no se tienen en cuenta específicamente; solo se incluye en el cálculo el efecto promedio de los electrones. A medida que aumenta el tamaño del conjunto base, la energía y la función de onda tienden hacia un límite llamado límite Hartree-Fock. [50]
Muchos tipos de cálculos comienzan con un cálculo Hartree-Fock y posteriormente corrigen la repulsión electrón-electrón, también denominada correlación electrónica . [51] Estos tipos de cálculos se denominan métodos post-Hartree-Fock . Al mejorar continuamente estos métodos, los científicos pueden acercarse cada vez más a la predicción perfecta del comportamiento de los sistemas atómicos y moleculares en el marco de la mecánica cuántica, tal como se define en la ecuación de Schrödinger. [52] Para obtener una concordancia exacta con el experimento, es necesario incluir términos específicos, algunos de los cuales son mucho más importantes para los átomos pesados que para los más ligeros. [53]
En la mayoría de los casos, la función de onda de Hartree-Fock ocupa una única configuración o determinante. [54] En algunos casos, en particular para los procesos de ruptura de enlaces, esto es inadecuado y se deben utilizar varias configuraciones . [55]
La energía molecular total puede evaluarse como una función de la geometría molecular ; en otras palabras, la superficie de energía potencial . [56] Una superficie de este tipo puede utilizarse para la dinámica de la reacción. Los puntos estacionarios de la superficie conducen a predicciones de diferentes isómeros y las estructuras de transición para la conversión entre isómeros, pero estas pueden determinarse sin un conocimiento completo de la superficie completa. [53]

Un objetivo particularmente importante, llamado termoquímica computacional , es calcular magnitudes termoquímicas como la entalpía de formación con precisión química. La precisión química es la precisión requerida para hacer predicciones químicas realistas y generalmente se considera que es de 1 kcal/mol o 4 kJ/mol. Para alcanzar esa precisión de manera económica, es necesario utilizar una serie de métodos post-Hartree-Fock y combinar los resultados. Estos métodos se denominan métodos compuestos de química cuántica . [57]
Después de que las variables electrónicas y nucleares se separan dentro de la representación de Born-Oppenheimer), el paquete de ondas correspondiente a los grados de libertad nucleares se propaga a través del operador de evolución temporal (física) asociado a la ecuación de Schrödinger dependiente del tiempo (para el hamiltoniano molecular completo ). [58] En el enfoque complementario dependiente de la energía, la ecuación de Schrödinger independiente del tiempo se resuelve utilizando el formalismo de la teoría de dispersión . El potencial que representa la interacción interatómica está dado por las superficies de energía potencial . En general, las superficies de energía potencial están acopladas a través de los términos de acoplamiento vibrónico . [59]
Los métodos más populares para propagar el paquete de ondas asociado a la geometría molecular son:
La forma en que un método computacional resuelve ecuaciones cuánticas afecta la precisión y la eficiencia del método. La técnica del operador dividido es uno de estos métodos para resolver ecuaciones diferenciales. En química computacional, la técnica del operador dividido reduce los costos computacionales de la simulación de sistemas químicos. Los costos computacionales son aproximadamente el tiempo que les toma a las computadoras calcular estos sistemas químicos, ya que puede llevar días para sistemas más complejos. Los sistemas cuánticos son difíciles y requieren mucho tiempo para ser resueltos por humanos. Los métodos del operador dividido ayudan a las computadoras a calcular estos sistemas rápidamente al resolver los subproblemas en una ecuación diferencial cuántica . El método hace esto al separar la ecuación diferencial en dos ecuaciones diferentes, como cuando hay más de dos operadores. Una vez resueltas, las ecuaciones divididas se combinan nuevamente en una ecuación para dar una solución fácilmente calculable. [62]
Este método se utiliza en muchos campos que requieren la resolución de ecuaciones diferenciales, como la biología . Sin embargo, la técnica conlleva un error de división. Por ejemplo, con la siguiente solución para una ecuación diferencial. [62]
La ecuación se puede descomponer, pero las soluciones no serán exactas, solo similares. Este es un ejemplo de descomposición de primer orden. [62]
Hay formas de reducir este error, que incluyen tomar un promedio de dos ecuaciones divididas. [62]
Otra forma de aumentar la precisión es utilizar la división de orden superior. Por lo general, la división de segundo orden es la más utilizada porque la división de orden superior requiere mucho más tiempo para calcularse y no vale la pena el costo. Los métodos de orden superior se vuelven demasiado difíciles de implementar y no son útiles para resolver ecuaciones diferenciales a pesar de la mayor precisión. [62]
Los químicos computacionales dedican mucho tiempo a hacer que los sistemas calculados con la técnica del operador dividido sean más precisos y, al mismo tiempo, minimicen el costo computacional. Calcular métodos es un desafío enorme para muchos químicos que intentan simular moléculas o entornos químicos. [62]

Los métodos de la teoría funcional de la densidad (DFT) suelen considerarse métodos ab initio para determinar la estructura electrónica molecular, aunque muchos de los funcionales más comunes utilizan parámetros derivados de datos empíricos o de cálculos más complejos. En la DFT, la energía total se expresa en términos de la densidad total de un electrón en lugar de la función de onda. En este tipo de cálculo, existe un hamiltoniano aproximado y una expresión aproximada para la densidad total de electrones. Los métodos DFT pueden ser muy precisos con un bajo costo computacional. Algunos métodos combinan el funcional de intercambio de la densidad con el término de intercambio de Hartree-Fock y se denominan métodos funcionales híbridos . [63]
Los métodos semiempíricos de la química cuántica se basan en el formalismo del método Hartree-Fock , pero realizan muchas aproximaciones y obtienen algunos parámetros a partir de datos empíricos. Fueron muy importantes en la química computacional desde los años 60 hasta los 90, especialmente para tratar moléculas grandes, donde el método Hartree-Fock completo sin las aproximaciones era demasiado costoso. El uso de parámetros empíricos parece permitir cierta inclusión de efectos de correlación en los métodos. [64]
Los métodos semiempíricos primitivos fueron diseñados incluso antes, donde la parte de dos electrones del hamiltoniano no está incluida explícitamente. Para los sistemas de electrones π, este fue el método de Hückel propuesto por Erich Hückel , y para todos los sistemas de electrones de valencia, el método de Hückel extendido propuesto por Roald Hoffmann . A veces, los métodos de Hückel se denominan "completamente empíricos" porque no derivan de un hamiltoniano. [65] Sin embargo, el término "métodos empíricos" o "campos de fuerza empíricos" se usa generalmente para describir la mecánica molecular. [66]

En muchos casos, se pueden modelar con éxito sistemas moleculares grandes evitando por completo los cálculos mecánicos cuánticos. Las simulaciones de mecánica molecular , por ejemplo, utilizan una expresión clásica para la energía de un compuesto, por ejemplo, el oscilador armónico . Todas las constantes que aparecen en las ecuaciones deben obtenerse de antemano a partir de datos experimentales o cálculos ab initio . [64]
La base de datos de compuestos utilizados para la parametrización, es decir, el conjunto resultante de parámetros y funciones se denomina campo de fuerza , y es crucial para el éxito de los cálculos de mecánica molecular. Se esperaría que un campo de fuerza parametrizado contra una clase específica de moléculas, por ejemplo, proteínas, solo tuviera relevancia al describir otras moléculas de la misma clase. [64] Estos métodos se pueden aplicar a proteínas y otras moléculas biológicas grandes, y permiten estudios del enfoque e interacción (acoplamiento) de posibles moléculas de fármacos. [67] [68]
La dinámica molecular (MD) utiliza la mecánica cuántica , la mecánica molecular o una mezcla de ambas para calcular fuerzas que luego se utilizan para resolver las leyes de movimiento de Newton y examinar el comportamiento dependiente del tiempo de los sistemas. El resultado de una simulación de dinámica molecular es una trayectoria que describe cómo la posición y la velocidad de las partículas varían con el tiempo. El punto de fase de un sistema descrito por las posiciones y los momentos de todas sus partículas en un punto de tiempo anterior determinará el siguiente punto de fase en el tiempo mediante la integración sobre las leyes de movimiento de Newton. [69]
El método Monte Carlo (MC) genera configuraciones de un sistema haciendo cambios aleatorios en las posiciones de sus partículas, junto con sus orientaciones y conformaciones cuando sea apropiado. [70] Es un método de muestreo aleatorio, que hace uso del llamado muestreo de importancia . Los métodos de muestreo de importancia son capaces de generar estados de baja energía, ya que esto permite calcular las propiedades con precisión. La energía potencial de cada configuración del sistema se puede calcular, junto con los valores de otras propiedades, a partir de las posiciones de los átomos. [71] [72]
QM/MM es un método híbrido que intenta combinar la precisión de la mecánica cuántica con la velocidad de la mecánica molecular. Es útil para simular moléculas muy grandes como las enzimas . [73]
La química computacional cuántica tiene como objetivo explotar la computación cuántica para simular sistemas químicos, lo que la distingue del enfoque QM/MM (mecánica cuántica/mecánica molecular). [74] Mientras que QM/MM utiliza un enfoque híbrido, combinando la mecánica cuántica para una parte del sistema con la mecánica clásica para el resto, la química computacional cuántica utiliza exclusivamente métodos de computación cuántica para representar y procesar información, como los operadores hamiltonianos. [75]
Los métodos convencionales de química computacional suelen tener dificultades para resolver las complejas ecuaciones de la mecánica cuántica, en particular debido al crecimiento exponencial de la función de onda de un sistema cuántico. La química computacional cuántica aborda estos desafíos utilizando métodos de computación cuántica , como la qubitización y la estimación de fase cuántica , que se cree que ofrecen soluciones escalables. [76]
La qubitización implica adaptar el operador hamiltoniano para un procesamiento más eficiente en computadoras cuánticas, mejorando la eficiencia de la simulación. La estimación de fase cuántica, por otro lado, ayuda a determinar con precisión los estados propios de energía, que son fundamentales para comprender el comportamiento del sistema cuántico. [77]
Si bien estas técnicas han hecho avanzar el campo de la química computacional, especialmente en la simulación de sistemas químicos, su aplicación práctica actualmente se limita principalmente a sistemas más pequeños debido a limitaciones tecnológicas. No obstante, estos avances pueden conducir a un progreso significativo hacia la consecución de simulaciones de química cuántica más precisas y eficientes en el uso de recursos. [76]
El costo computacional y la complejidad algorítmica en química se utilizan para ayudar a comprender y predecir fenómenos químicos. Ayudan a determinar qué algoritmos o métodos computacionales utilizar al resolver problemas químicos. Esta sección se centra en la escala de la complejidad computacional con el tamaño de la molécula y detalla los algoritmos que se utilizan comúnmente en ambos dominios. [78]
En química cuántica, en particular, la complejidad puede crecer exponencialmente con el número de electrones involucrados en el sistema. Este crecimiento exponencial es una barrera importante para simular sistemas grandes o complejos con precisión. [79]
Los algoritmos avanzados en ambos campos buscan equilibrar la precisión con la eficiencia computacional. Por ejemplo, en la química cuántica, se emplean métodos como la integración de Verlet o el algoritmo de Beeman por su eficiencia computacional. En la química cuántica, se utilizan cada vez más métodos híbridos que combinan diferentes enfoques computacionales (como QM/MM) para abordar grandes sistemas biomoleculares. [80]
La siguiente lista ilustra el impacto de la complejidad computacional en los algoritmos utilizados en los cálculos químicos. Es importante señalar que, si bien esta lista proporciona ejemplos clave, no es exhaustiva y sirve como guía para comprender cómo las demandas computacionales influyen en la selección de métodos computacionales específicos en química.
Resuelve las ecuaciones de movimiento de Newton para átomos y moléculas. [81]
El cálculo estándar de interacción por pares en MD genera complejidad para las partículas. Esto se debe a que cada partícula interactúa con todas las demás partículas, lo que da como resultado interacciones. [82] Los algoritmos avanzados, como la suma de Ewald o el método multipolar rápido, reducen esto a o incluso agrupando partículas distantes y tratándolas como una sola entidad o utilizando aproximaciones matemáticas inteligentes. [83] [84]
Combina cálculos mecánicos cuánticos para una región pequeña con mecánica molecular para un entorno más amplio. [85]
La complejidad de los métodos QM/MM depende tanto del tamaño de la región cuántica como del método utilizado para los cálculos cuánticos. Por ejemplo, si se utiliza un método Hartree-Fock para la parte cuántica, la complejidad se puede aproximar como , donde es el número de funciones base en la región cuántica. Esta complejidad surge de la necesidad de resolver un conjunto de ecuaciones acopladas de forma iterativa hasta que se logre la autoconsistencia. [86]
Encuentra un único estado de Fock que minimiza la energía. [87]
NP-hard o NP-complete como se demuestra al incorporar instancias del modelo de Ising en los cálculos de Hartree-Fock. El método de Hartree-Fock implica la resolución de las ecuaciones de Roothaan-Hall, que escalan en función de la implementación, siendo el número de funciones base. El costo computacional proviene principalmente de la evaluación y transformación de las integrales de dos electrones. Esta prueba de NP-hardness o NP-completeness proviene de la incorporación de problemas como el modelo de Ising en el formalismo de Hartree-Fock. [87]
Investiga la estructura electrónica o estructura nuclear de sistemas de muchos cuerpos como átomos, moléculas y fases condensadas . [89]
Las implementaciones tradicionales de DFT suelen escalar como , principalmente debido a la necesidad de diagonalizar la matriz de Kohn-Sham . [90] El paso de diagonalización, que encuentra los valores propios y los vectores propios de la matriz, contribuye más a este escalamiento. Los avances recientes en DFT apuntan a reducir esta complejidad a través de varias aproximaciones y mejoras algorítmicas. [91]
Los métodos CCSD y CCSD(T) son técnicas avanzadas de estructura electrónica que implican excitaciones simples, dobles y, en el caso de CCSD(T), triples perturbativas para calcular los efectos de correlación electrónica. [92]
Escalas como donde es el número de funciones base. Esta intensa demanda computacional surge de la inclusión de excitaciones simples y dobles en el cálculo de correlación electrónica. [92]
Con la adición de triples perturbativos, la complejidad aumenta a . Esta complejidad elevada restringe el uso práctico a sistemas más pequeños, típicamente hasta 20-25 átomos en implementaciones convencionales. [92]

Una adaptación del método estándar CCSD(T) que utiliza orbitales naturales locales (NO) para reducir significativamente la carga computacional y permitir la aplicación a sistemas más grandes. [92]
Se logra una escala lineal con el tamaño del sistema, una mejora importante con respecto a la escala tradicional de quinta potencia del CCSD. Este avance permite aplicaciones prácticas para moléculas de hasta 100 átomos con conjuntos de bases razonables, lo que marca un avance significativo en la capacidad de la química computacional para manejar sistemas más grandes con alta precisión. [92]
La demostración de las clases de complejidad de los algoritmos implica una combinación de pruebas matemáticas y experimentos computacionales. Por ejemplo, en el caso del método Hartree-Fock, la demostración de la NP-dureza es un resultado teórico derivado de la teoría de la complejidad, específicamente a través de reducciones de problemas NP-duros conocidos. [93]
En el caso de otros métodos, como MD o DFT, la complejidad computacional suele observarse empíricamente y respaldarse mediante análisis de algoritmos. En estos casos, la prueba de corrección tiene menos que ver con pruebas matemáticas formales y más con la observación consistente del comportamiento computacional en varios sistemas e implementaciones. [93]
La química computacional no es una descripción exacta de la química de la vida real, ya que los modelos matemáticos y físicos de la naturaleza sólo pueden proporcionar una aproximación. Sin embargo, la mayoría de los fenómenos químicos pueden describirse hasta cierto punto en un esquema computacional cualitativo o cuantitativo aproximado. [94]
Las moléculas están formadas por núcleos y electrones, por lo que se aplican los métodos de la mecánica cuántica . Los químicos computacionales a menudo intentan resolver la ecuación de Schrödinger no relativista , con correcciones relativistas añadidas, aunque se han logrado algunos avances en la resolución de la ecuación de Dirac totalmente relativista . En principio, es posible resolver la ecuación de Schrödinger en su forma dependiente o independiente del tiempo, según sea apropiado para el problema en cuestión; en la práctica, esto no es posible excepto para sistemas muy pequeños. Por lo tanto, una gran cantidad de métodos aproximados intentan lograr el mejor equilibrio entre precisión y costo computacional. [95]
La precisión siempre se puede mejorar con un mayor costo computacional. Pueden presentarse errores significativos en modelos ab initio que comprenden muchos electrones, debido al costo computacional de los métodos que incluyen la relatividad total. [92] Esto complica el estudio de moléculas que interactúan con átomos de unidades de masa atómica alta, como los metales de transición y sus propiedades catalíticas. Los algoritmos actuales en química computacional pueden calcular rutinariamente las propiedades de moléculas pequeñas que contienen hasta aproximadamente 40 electrones con errores para energías menores a unos pocos kJ/mol. Para las geometrías, las longitudes de enlace se pueden predecir con una precisión de unos pocos picómetros y los ángulos de enlace con una precisión de 0,5 grados. El tratamiento de moléculas más grandes que contienen unas pocas docenas de átomos es computacionalmente manejable con métodos más aproximados como la teoría funcional de la densidad (DFT). [96]
Existe cierta controversia en el campo sobre si estos últimos métodos son suficientes o no para describir reacciones químicas complejas, como las de la bioquímica. Las moléculas grandes se pueden estudiar mediante métodos aproximados semiempíricos. Incluso las moléculas más grandes se tratan mediante métodos de mecánica clásica que utilizan lo que se denomina mecánica molecular (MM). En los métodos QM-MM, pequeñas partes de grandes complejos se tratan de forma mecánica cuántica (QM) y el resto se trata de forma aproximada (MM). [97]
Existen muchos paquetes de software de química computacional autosuficientes . Algunos incluyen muchos métodos que cubren un amplio espectro, mientras que otros se concentran en un rango muy específico o incluso en un solo método. Los detalles de la mayoría de ellos se pueden encontrar en: