La edición genética CRISPR (CRISPR, pronunciado /ˈkrɪspər/ "crisper", se refiere a "repeticiones palindrómicas cortas agrupadas regularmente interespaciadas " ) es una técnica de ingeniería genética en biología molecular mediante la cual se pueden modificar los genomas de organismos vivos . Se basa en una versión simplificada del sistema de defensa antiviral bacteriano CRISPR-Cas9. Al introducir la nucleasa Cas9 en un complejo con un ARN guía sintético (gRNA) en una célula, se puede cortar el genoma de la célula en la ubicación deseada, lo que permite eliminar genes existentes y/o agregar otros nuevos in vivo . [1]
La técnica se considera de gran importancia en biotecnología y medicina , ya que permite editar genomas in vivo de forma muy precisa, barata y sencilla. Se puede utilizar en la creación de nuevos medicamentos, productos agrícolas y organismos modificados genéticamente , o como medio para controlar patógenos y plagas . También tiene posibilidades en el tratamiento de enfermedades genéticas hereditarias , así como enfermedades derivadas de mutaciones somáticas como el cáncer. Sin embargo, su uso en la modificación genética de la línea germinal humana es muy controvertido. El desarrollo de la técnica le valió a Jennifer Doudna y Emmanuelle Charpentier el Premio Nobel de Química en 2020. [2] [3] El tercer grupo de investigadores que compartió el Premio Kavli por el mismo descubrimiento, [4] dirigido por Virginijus Šikšnys , no recibió el premio Nobel. [5] [6] [7]
La nucleasa Cas9, que funciona como una tijera genética, abre ambas hebras de la secuencia de ADN objetivo para introducir la modificación mediante uno de dos métodos. Las mutaciones knock-in, facilitadas mediante la reparación dirigida por homología (HDR), son la vía tradicional de los enfoques de edición genómica dirigida. [1] Esto permite la introducción de daños y reparaciones del ADN dirigidas . La HDR emplea el uso de secuencias de ADN similares para impulsar la reparación de la rotura mediante la incorporación de ADN exógeno para que funcione como plantilla de reparación. [1] Este método se basa en la aparición periódica y aislada de daños en el ADN en el sitio objetivo para que comience la reparación. Las mutaciones knock-out causadas por CRISPR-Cas9 son el resultado de la reparación de la rotura de doble cadena mediante unión de extremos no homólogos (NHEJ) o unión de extremos mediada por POLQ/polimerasa theta (TMEJ). Estas vías de unión de extremos a menudo pueden dar lugar a deleciones o inserciones aleatorias en el sitio de reparación, lo que puede interrumpir o alterar la funcionalidad del gen. Por lo tanto, la ingeniería genómica mediante CRISPR-Cas9 brinda a los investigadores la capacidad de generar disrupciones genéticas aleatorias y dirigidas.
Si bien la edición genómica en células eucariotas ha sido posible mediante diversos métodos desde la década de 1980, los métodos empleados habían demostrado ser ineficientes y poco prácticos para implementar a gran escala. Con el descubrimiento de CRISPR y, específicamente, la molécula de nucleasa Cas9, se hizo posible una edición eficiente y altamente selectiva. Cas9 derivada de la especie bacteriana Streptococcus pyogenes ha facilitado la modificación genómica dirigida en células eucariotas al permitir un método confiable para crear una ruptura dirigida en una ubicación específica designada por las cadenas guía crRNA y tracrRNA. [8] La facilidad con la que los investigadores pueden insertar Cas9 y ARN molde para silenciar o causar mutaciones puntuales en loci específicos ha demostrado ser invaluable para el mapeo rápido y eficiente de modelos genómicos y procesos biológicos asociados con varios genes en una variedad de eucariotas. Se han desarrollado variantes recientemente diseñadas de la nucleasa Cas9 que reducen significativamente la actividad fuera del objetivo. [9]
Las técnicas de edición genómica CRISPR-Cas9 tienen muchas aplicaciones potenciales. El uso del complejo CRISPR-Cas9-gRNA para la edición genómica [10] fue elegido por la AAAS como el Avance del Año en 2015. [11] Se han planteado muchas preocupaciones bioéticas sobre la posibilidad de utilizar CRISPR para la edición de la línea germinal , especialmente en embriones humanos . [12] En 2023, el primer fármaco que utiliza la edición genética CRISPR, Casgevy , fue aprobado para su uso en el Reino Unido para curar la anemia de células falciformes y la beta talasemia . [13] [14] Casgevy fue aprobado para su uso en los Estados Unidos el 8 de diciembre de 2023 por la Administración de Alimentos y Medicamentos . [15]
A principios de la década de 2000, los investigadores alemanes comenzaron a desarrollar nucleasas de dedo de zinc (ZFN), proteínas sintéticas cuyos dominios de unión al ADN les permiten crear roturas de doble cadena en el ADN en puntos específicos. Las ZFN tienen una mayor precisión y la ventaja de ser más pequeñas que Cas9, pero las ZFN no se utilizan tan comúnmente como los métodos basados en CRISPR. En 2010, las nucleasas sintéticas llamadas nucleasas efectoras similares a activadores de la transcripción (TALEN) proporcionaron una forma más fácil de dirigir una rotura de doble cadena a una ubicación específica en la cadena de ADN. Tanto las nucleasas de dedo de zinc como las TALEN requieren el diseño y la creación de una proteína personalizada para cada secuencia de ADN objetivo, que es un proceso mucho más difícil y que requiere más tiempo que el de diseñar ARN guía. Los CRISPR son mucho más fáciles de diseñar porque el proceso requiere sintetizar solo una secuencia corta de ARN, un procedimiento que ya se usa ampliamente para muchas otras técnicas de biología molecular (por ejemplo, la creación de cebadores de oligonucleótidos ). [16]
Mientras que métodos como la interferencia de ARN (RNAi) no suprimen por completo la función de los genes, CRISPR, ZFN y TALEN proporcionan una eliminación completa e irreversible de los genes . [17] CRISPR también puede dirigirse a varios sitios de ADN simultáneamente simplemente introduciendo diferentes gRNA. Además, los costos de emplear CRISPR son relativamente bajos. [17] [18] [19]
En 2005, Alexander Bolotin del Instituto Nacional de Investigación Agrícola de Francia (INRA) descubrió un locus CRISPR que contenía nuevos genes Cas, especialmente uno que codificaba una proteína grande conocida como Cas9. [20]
En 2006, Eugene Koonin del Centro Nacional de Información Biotecnológica de Estados Unidos (NIH) propuso una explicación de cómo CRISPR actúa como un sistema inmunológico bacteriano. [20]
En 2007, Philippe Horvath de Danisco France SAS demostró experimentalmente cómo los sistemas CRISPR son un sistema inmunológico adaptativo e integran nuevo ADN de fago en la matriz CRISPR, que es la forma en que combaten la siguiente ola de fagos atacantes. [20]
En 2012, el equipo de investigación dirigido por la Universidad de California, Berkeley, la profesora Jennifer Doudna y la profesora de la Universidad de Umea Emmanuelle Charpentier, fueron las primeras personas en identificar, divulgar y presentar una solicitud de patente para el sistema CRISPR-Cas9 necesario para editar ADN. [20] También publicaron su hallazgo de que CRISPR- Cas9 podría programarse con ARN para editar ADN genómico, ahora considerado uno de los descubrimientos más significativos en la historia de la biología .
En noviembre de 2013 [update], SAGE Labs (parte del grupo Horizon Discovery ) tenía derechos exclusivos de una de esas empresas para producir y vender ratas modificadas genéticamente y derechos no exclusivos para modelos de ratón y conejo. [21] En 2015 [update], Thermo Fisher Scientific había obtenido la licencia de propiedad intelectual de ToolGen para desarrollar kits de reactivos CRISPR. [22]
En diciembre de 2014 [update], los derechos de patente de CRISPR fueron impugnados. Se formaron varias empresas para desarrollar medicamentos y herramientas de investigación relacionados. [23] A medida que las empresas aumentaron la financiación, surgieron dudas sobre si CRISPR podría monetizarse rápidamente. [24] En 2014, Feng Zhang del Broad Institute del MIT y Harvard y otras nueve personas recibieron la patente estadounidense número 8.697.359 [25] sobre el uso de la edición genética CRISPR-Cas9 en eucariotas. Aunque a Charpentier y Doudna (conocidas como CVC) se les atribuyó la concepción de CRISPR, el Broad Institute fue el primero en lograr una "reducción a la práctica" según los jueces de patentes Sally Gardner Lane, James T. Moore y Deborah Katz. [26]
El primer conjunto de patentes fue otorgado al equipo Broad en 2015, lo que llevó a los abogados del grupo CVC a solicitar el primer procedimiento de interferencia. [27] En febrero de 2017, la Oficina de Patentes de los EE. UU. dictó sentencia sobre un caso de interferencia de patentes presentado por la Universidad de California con respecto a las patentes emitidas al Instituto Broad , y encontró que las patentes de Broad, con reclamos que cubren la aplicación de CRISPR-Cas9 en células eucariotas, eran distintas de las invenciones reclamadas por la Universidad de California. [28] [29] [30]
Poco después, la Universidad de California presentó una apelación contra esta sentencia. [31] [32] En 2019 se abrió la segunda disputa por interferencia. Esto fue en respuesta a las solicitudes de patente presentadas por CVC que requerían que la junta de apelaciones determinara el inventor original de la tecnología. La USPTO falló en marzo de 2022 en contra de la UC, declarando que el Instituto Broad fue el primero en presentar la solicitud. La decisión afectó a muchos de los acuerdos de licencia para la tecnología de edición CRISPR que fue licenciada por UC Berkeley. UC manifestó su intención de apelar la decisión de la USPTO. [33]
En marzo de 2017, la Oficina Europea de Patentes (OEP) anunció su intención de permitir reivindicaciones para la edición de todo tipo de células al Instituto Max-Planck de Berlín, la Universidad de California y la Universidad de Viena, [34] [35] y en agosto de 2017, la OEP anunció su intención de permitir reivindicaciones CRISPR en una solicitud de patente que MilliporeSigma había presentado. [34] En agosto de 2017, [update]la situación de las patentes en Europa era compleja, con MilliporeSigma, ToolGen, la Universidad de Vilnius y Harvard compitiendo por reivindicaciones, junto con la Universidad de California y Broad. [36]
En julio de 2018, el TJUE dictaminó que la edición genética de plantas era una subcategoría de los alimentos transgénicos y, por lo tanto, la técnica CRISPR en adelante estaría regulada en la Unión Europea por sus normas y reglamentos para los OGM . [37]
En febrero de 2020, un ensayo realizado en Estados Unidos demostró que la edición genética mediante CRISPR es segura en tres pacientes con cáncer. [38]
En octubre de 2020, las investigadoras Emmanuelle Charpentier y Jennifer Doudna recibieron el Premio Nobel de Química por su trabajo en este campo. [39] [40] Hicieron historia al ser las primeras dos mujeres en compartir este premio sin un colaborador masculino. [41] [5]
En junio de 2021, el primer ensayo clínico a pequeña escala de edición genética CRISPR intravenosa en humanos concluye con resultados prometedores. [42] [43]
En septiembre de 2021, el primer alimento editado con CRISPR salió a la venta en Japón. Los tomates fueron modificados genéticamente para obtener una cantidad aproximadamente cinco veces mayor de la normal de GABA , un posible calmante [44] . [45] CRISPR se aplicó por primera vez en tomates en 2014. [46]
En diciembre de 2021, se informó que el primer animal marino/ marisco editado genéticamente con CRISPR y el segundo conjunto de alimentos editados con CRISPR salieron a la venta al público en Japón: dos peces, de los cuales una especie crece hasta el doble del tamaño de los especímenes naturales debido a la alteración de la leptina , que controla el apetito, y la otra crece hasta 1,2 del tamaño medio natural con la misma cantidad de alimento debido a la miostatina desactivada , que inhibe el crecimiento muscular . [47] [48] [49]
Un estudio de 2022 ha descubierto que saber más sobre los tomates CRISPR tuvo un fuerte efecto en las preferencias de los participantes. "Casi la mitad de los 32 participantes de Alemania que son científicos demostraron elecciones constantes, mientras que la mayoría mostró una mayor disposición a comprar tomates CRISPR, en su mayoría no científicos". [50] [51]
La Universidad de California en Berkeley anunció en mayo de 2021 su intención de subastar tokens no fungibles tanto de la patente de edición genética CRISPR como de la inmunoterapia contra el cáncer . Sin embargo, en este caso, la universidad conservaría la propiedad de las patentes. [52] [53] El 85 % de los fondos obtenidos mediante la venta de la colección denominada The Fourth Pillar se utilizarían para financiar la investigación. [54] [55] Se vendió en junio de 2022 por 22 Ether, que en ese momento eran unos 54.000 dólares estadounidenses . [56]
En noviembre de 2023, la Agencia Reguladora de Medicamentos y Productos Sanitarios (MHRA) del Reino Unido se convirtió en la primera del mundo en aprobar el uso del primer fármaco basado en la edición genética CRISPR, Casgevy, para tratar la anemia falciforme y la beta talasemia . Casgevy, o exagamglogene autotemcel , actúa directamente sobre los genes de las células madre del interior de los huesos del paciente, haciendo que produzcan glóbulos rojos sanos. Este tratamiento evita así la necesidad de realizar transfusiones de sangre periódicas y costosas. [13] [14]
En diciembre de 2023, la FDA aprobó la primera terapia génica en los EE. UU. para tratar a pacientes con enfermedad de células falciformes (ECF). La FDA aprobó dos tratamientos emblemáticos, Casgevy y Lyfgenia, que representan las primeras terapias génicas basadas en células para el tratamiento de la ECF. [57]

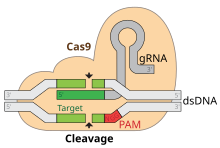
La edición genómica mediante CRISPR-Cas9 se lleva a cabo con un sistema CRISPR de tipo II . Cuando se utiliza para la edición genómica, este sistema incluye una ribonucleoproteína (RNP), que consta de Cas9 , crRNA y tracrRNA, junto con una plantilla de reparación de ADN opcional.

CRISPR-Cas9 a menudo emplea plásmidos , que codifican los componentes de RNP, para transfectar las células objetivo, o el RNP se ensambla antes de la adición a las células a través de la nucleofección. [58] Los componentes principales de este plásmido se muestran en la imagen y se enumeran en la tabla. El crRNA está diseñado exclusivamente para cada aplicación, ya que esta es la secuencia que Cas9 usa para identificar y unirse directamente a secuencias específicas dentro del ADN de la célula huésped. El crRNA debe unirse solo donde se desea la edición. La plantilla de reparación también está diseñada exclusivamente para cada aplicación, ya que debe complementar hasta cierto punto las secuencias de ADN a cada lado del corte y también contener cualquier secuencia que se desee para la inserción en el genoma huésped.
Se pueden empaquetar varios ARNcr y el ARNtracr juntos para formar un ARN guía único (ARNsg). [59] Este ARNsg se puede incluir junto con el gen que codifica la proteína Cas9 y transformarse en un plásmido para ser transfectado en células. Hay muchas herramientas en línea disponibles para ayudar a diseñar secuencias de ARNsg efectivas. [60] [61]

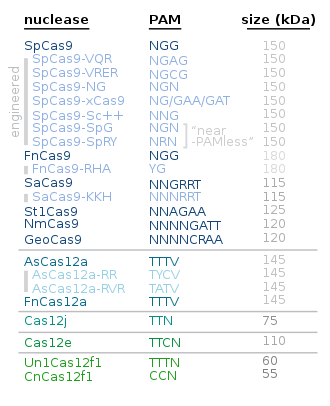
Las proteínas alternativas a Cas9 incluyen las siguientes:
CRISPR-Cas9 ofrece un alto grado de fidelidad y una construcción relativamente simple. Depende de dos factores para su especificidad: la secuencia objetivo y la secuencia del motivo adyacente al protoespaciador (PAM). La secuencia objetivo tiene 20 bases de longitud como parte de cada locus CRISPR en la matriz de crRNA. [58] Una matriz de crRNA típica tiene múltiples secuencias objetivo únicas. Las proteínas Cas9 seleccionan la ubicación correcta en el genoma del huésped utilizando la secuencia para unirse con pares de bases en el ADN del huésped. La secuencia no es parte de la proteína Cas9 y, como resultado, es personalizable y se puede sintetizar de forma independiente . [75] [76]
La secuencia PAM del genoma del huésped es reconocida por Cas9. Cas9 no se puede modificar fácilmente para reconocer una secuencia PAM diferente. Sin embargo, esto no es en última instancia demasiado limitante, ya que normalmente es una secuencia muy corta y no específica que aparece con frecuencia en muchos lugares a lo largo del genoma (por ejemplo, la secuencia PAM de SpCas9 es 5'-NGG-3' y en el genoma humano aparece aproximadamente cada 8 a 12 pares de bases). [58]
Una vez que estas secuencias se han ensamblado en un plásmido y se han transfectado en las células, la proteína Cas9 con la ayuda del crRNA encuentra la secuencia correcta en el ADN de la célula huésped y, dependiendo de la variante de Cas9, crea una ruptura mono o bicatenaria en la ubicación apropiada en el ADN. [77]
Las roturas monocatenarias adecuadamente espaciadas en el ADN del huésped pueden desencadenar una reparación dirigida por homología , que es menos propensa a errores que la unión de extremos no homólogos que normalmente sigue a una rotura de doble cadena. Proporcionar una plantilla de reparación del ADN permite la inserción de una secuencia de ADN específica en una ubicación exacta dentro del genoma. La plantilla de reparación debe extenderse de 40 a 90 pares de bases más allá de la rotura del ADN inducida por Cas9. [58] El objetivo es que el proceso HDR nativo de la célula utilice la plantilla de reparación proporcionada y, por lo tanto, incorpore la nueva secuencia al genoma. Una vez incorporada, esta nueva secuencia ahora es parte del material genético de la célula y pasa a sus células hijas. La inhibición transitoria combinada de NHEJ y TMEJ por una molécula pequeña y ARNi puede aumentar la eficiencia de HDR hasta un 93% y, al mismo tiempo, prevenir la edición fuera del objetivo. [78]
La administración de Cas9, sgRNA y complejos asociados a las células puede ocurrir a través de sistemas virales y no virales. La electroporación de ADN, ARN o ribonucleocomplejos es una técnica común, aunque puede producir efectos nocivos en las células diana. [79] También se han utilizado técnicas de transfección química que utilizan lípidos y péptidos para introducir sgRNA en complejos con Cas9 en las células. [80] [81] También se ha utilizado la administración basada en nanopartículas para la transfección. [82] Los tipos de células que son más difíciles de transfectar (por ejemplo, células madre, neuronas y células hematopoyéticas) requieren sistemas de administración más eficientes, como los basados en lentivirus (LV), adenovirus (AdV) y virus adenoasociados (AAV). [83] [84] [85]
Se ha descubierto que la eficiencia de CRISPR-Cas9 aumenta considerablemente cuando varios componentes del sistema, incluida la estructura completa de CRISPR/Cas9 hasta los complejos Cas9-gRNA, se entregan en forma ensamblada en lugar de usar transgénicos. [86] [87] Esto ha encontrado un valor particular en cultivos genéticamente modificados para comercialización masiva. [88] [89] Dado que la maquinaria de replicación del huésped no es necesaria para producir estas proteínas, la posibilidad de que la secuencia de reconocimiento del sgRNA sea casi nula, lo que disminuye la posibilidad de efectos fuera del objetivo. [82]
Las mejoras y variantes posteriores del sistema CRISPR-Cas9 se han centrado en introducir un mayor control en su uso. En concreto, la investigación destinada a mejorar este sistema incluye la mejora de su especificidad, su eficiencia y la granularidad de su capacidad de edición. Las técnicas se pueden dividir y clasificar además según el componente del sistema que modifican. Entre ellas se incluyen el uso de variantes diferentes o creaciones novedosas de la proteína Cas, el uso de una proteína efectora totalmente diferente, la modificación del ARNsg o el uso de un enfoque algorítmico para identificar las soluciones óptimas existentes.
La especificidad es un aspecto importante para mejorar el sistema CRISPR-Cas9 porque los efectos fuera del objetivo que genera tienen graves consecuencias para el genoma de la célula e invocan precaución para su uso. Minimizar los efectos fuera del objetivo es, por tanto, maximizar la seguridad del sistema. Las nuevas variaciones de las proteínas Cas9 que aumentan la especificidad incluyen proteínas efectoras con una eficiencia y especificidad comparables a las del SpCas9 original que pueden dirigirse a las secuencias previamente inalcanzables y una variante que prácticamente no tiene mutaciones fuera del objetivo. [90] [91] También se han realizado investigaciones en la ingeniería de nuevas proteínas Cas9, incluidas algunas que reemplazan parcialmente los nucleótidos de ARN en crRNA con ADN y un procedimiento de generación de mutantes Cas9 guiado por la estructura que habían reducido los efectos fuera del objetivo. [92] [93] Se ha demostrado que los sgRNA truncados iterativamente y los gRNA altamente estabilizados también reducen los efectos fuera del objetivo. [94] [95] Se han utilizado métodos computacionales, incluido el aprendizaje automático, para predecir la afinidad y crear secuencias únicas para el sistema a fin de maximizar la especificidad para objetivos determinados. [96] [97]
Varias variantes de CRISPR-Cas9 permiten la activación genética o la edición del genoma con un desencadenante externo, como la luz o moléculas pequeñas. [98] [99] [100] Estos incluyen sistemas CRISPR fotoactivables desarrollados mediante la fusión de proteínas sensibles a la luz asociadas con un dominio activador y un dCas9 para la activación genética, [101] [102] o mediante la fusión de dominios similares sensibles a la luz con dos construcciones de Cas9 dividido, [103] [104] o mediante la incorporación de aminoácidos no naturales enjaulados en Cas9, [105] o mediante la modificación de los ARN guía con complementos fotoescindibles para la edición del genoma. [106]
Los métodos para controlar la edición del genoma con moléculas pequeñas incluyen un Cas9 alostérico, sin edición de fondo detectable, que activará la unión y la escisión tras la adición de 4-hidroxitamoxifeno (4-HT), [98] Cas9 ligado a inteína sensible a 4-HT , [107] o un Cas9 que responde a 4-HT cuando se fusiona a cuatro dominios ERT2. [108] El Cas9 dividido inducible por inteína permite la dimerización de fragmentos de Cas9 [109] y el sistema de Cas9 dividido inducible por rapamicina se desarrolló fusionando dos construcciones de Cas9 dividido con fragmentos de FRB y FKBP . [110] Otros estudios han podido inducir la transcripción de Cas9 con una molécula pequeña, la doxiciclina . [111] [112] Las moléculas pequeñas también se pueden utilizar para mejorar la reparación dirigida por homología, [113] a menudo inhibiendo la vía de unión de extremos no homólogos y/o la vía de unión de extremos mediada por theta. [114] [115] Se creó un sistema con la proteína efectora Cpf1 que es inducida por las moléculas pequeñas VE-822 y AZD-7762. [116] Estos sistemas permiten el control condicional de la actividad CRISPR para mejorar la precisión, la eficiencia y el control espaciotemporal. El control espaciotemporal es una forma de eliminar los efectos no deseados: solo es posible que sea necesario modificar ciertas células o partes del organismo y, por lo tanto, se pueden utilizar moléculas ligeras o pequeñas como una forma de llevar a cabo esto. La eficiencia del sistema CRISPR-Cas9 también aumenta en gran medida mediante la entrega adecuada de las instrucciones de ADN para crear las proteínas y los reactivos necesarios. [116]
CRISPR también utiliza proteínas de edición de un solo par de bases para crear ediciones específicas en una o dos bases en la secuencia objetivo. CRISPR/Cas9 se fusionó con enzimas específicas que inicialmente solo podían cambiar las mutaciones de C a T y de G a A y su reverso. Esto se logró finalmente sin requerir ninguna escisión del ADN. [117] [118] [119] Con la fusión de otra enzima, el sistema de edición de bases CRISPR-Cas9 también puede editar C a G y su reverso. [120]
El sistema de repeticiones de palíndromo corto agrupadas y regularmente interespaciadas (CRISPR)/Cas9 es una tecnología de edición genética que puede inducir roturas de doble cadena (DSB) en cualquier lugar donde los ácidos ribonucleicos guía ( gRNA ) puedan unirse con la secuencia del motivo adyacente al protoespaciador (PAM). [121] Las muescas de cadena simple también pueden ser inducidas por mutantes del sitio activo de Cas9, [122] también conocidos como nickasas de Cas9. [123] Simplemente cambiando la secuencia de gRNA, la endonucleasa Cas9 puede ser entregada a un gen de interés e inducir DSB. [124] La eficiencia de la endonucleasa Cas9 y la facilidad con la que los genes pueden ser seleccionados llevaron al desarrollo de bibliotecas CRISPR-knockout (KO) tanto para células de ratón como humanas, que pueden cubrir conjuntos de genes específicos de interés o el genoma completo. [125] [126] El cribado CRISPR ayuda a los científicos a crear una perturbación genética sistemática y de alto rendimiento dentro de organismos modelo vivos. Esta perturbación genética es necesaria para comprender plenamente la función de los genes y la regulación epigenética. [127] La ventaja de las bibliotecas CRISPR agrupadas es que se pueden seleccionar más genes a la vez.
Las bibliotecas knock-out se crean de manera de lograr una representación y un rendimiento iguales en todos los gRNA expresados y llevan un marcador de selección antibiótico o fluorescente que se puede usar para recuperar células transducidas. [121] Hay dos sistemas de plásmidos en las bibliotecas CRISPR/Cas9. Primero, es un plásmido todo en uno, donde el sgRNA y Cas9 se producen simultáneamente en una célula transfectada. Segundo, es un sistema de dos vectores: los plásmidos sgRNA y Cas9 se entregan por separado. [127] Es importante entregar miles de vectores únicos que contienen sgRNA a un solo recipiente de células por transducción viral a baja multiplicidad de infección (MOI, típicamente a 0,1-0,6), previene la probabilidad de que un clon celular individual obtenga más de un tipo de sgRNA de lo contrario puede conducir a una asignación incorrecta de genotipo a fenotipo . [125]
Una vez preparada la biblioteca agrupada, es necesario llevar a cabo una secuenciación profunda (NGS, secuenciación de próxima generación) del ADN plasmídico amplificado por PCR para revelar la abundancia de sgRNAs. Las células de interés pueden ser infectadas por la biblioteca y luego seleccionadas según el fenotipo. Hay dos tipos de selección: negativa y positiva. Mediante la selección negativa, se detectan de manera eficiente las células muertas o de crecimiento lento. Puede identificar genes esenciales para la supervivencia, que pueden servir además como candidatos para fármacos dirigidos molecularmente. Por otro lado, la selección positiva proporciona una colección de poblaciones adquiridas con ventaja de crecimiento mediante mutagénesis aleatoria. [121] Después de la selección, se recolecta y secuencia el ADN genómico mediante NGS. Se detecta la disminución o el enriquecimiento de sgRNAs y se compara con la biblioteca de sgRNA original, anotada con el gen objetivo al que corresponde el sgRNA. Luego, el análisis estadístico identifica los genes que es significativamente probable que sean relevantes para el fenotipo de interés. [125]
Además de la eliminación, también existen bibliotecas de eliminación (CRISPRi) y activación (CRISPRa), que utilizan la capacidad de las proteínas de fusión Cas9 desactivadas proteolíticamente (dCas9) para unirse al ADN objetivo, lo que significa que el gen de interés no se corta, sino que se sobreexpresa o reprime. Esto hizo que el sistema CRISPR/Cas9 fuera aún más interesante en la edición genética. La proteína dCas9 inactiva modula la expresión genética al dirigir los represores o activadores de dCas9 hacia el promotor o los sitios de inicio de la transcripción de los genes objetivo. Para reprimir genes, Cas9 se puede fusionar al dominio efector KRAB que forma un complejo con el ARNm, mientras que CRISPRa utiliza dCas9 fusionado a diferentes dominios de activación transcripcional, que luego son dirigidos por el ARNm a las regiones promotoras para regular positivamente la expresión. [129] [130] [131]
La modificación genómica de Cas9 ha permitido la generación rápida y eficiente de modelos transgénicos dentro del campo de la genética. Cas9 se puede introducir fácilmente en las células diana junto con sgRNA a través de la transfección de plásmidos para modelar la propagación de enfermedades y la respuesta y defensa de la célula frente a las infecciones. [132] La capacidad de Cas9 para introducirse in vivo permite la creación de modelos más precisos de la función genética y los efectos de las mutaciones, todo ello evitando las mutaciones fuera del objetivo que se observan típicamente con los métodos más antiguos de ingeniería genética.
La revolución de CRISPR y Cas9 en el modelado genómico no se extiende solo a los mamíferos. Los modelos genómicos tradicionales como Drosophila melanogaster , uno de los primeros organismos modelo, han visto un mayor refinamiento en su resolución con el uso de Cas9. [132] Cas9 utiliza promotores específicos de células que permiten un uso controlado de Cas9. Cas9 es un método preciso para tratar enfermedades debido a que la enzima Cas9 se dirige a ciertos tipos de células. Las células que se someten a la terapia Cas9 también se pueden eliminar y reintroducir para proporcionar efectos amplificados de la terapia. [133]
La técnica CRISPR-Cas9 se puede utilizar para editar el ADN de organismos in vivo y eliminar genes individuales o incluso cromosomas enteros de un organismo en cualquier momento de su desarrollo. Entre los cromosomas que se han eliminado con éxito in vivo mediante técnicas CRISPR se encuentran el cromosoma Y y el cromosoma X de ratones de laboratorio adultos y los cromosomas humanos 14 y 21, en líneas de células madre embrionarias y ratones aneuploides respectivamente. Este método podría ser útil para tratar trastornos genéticos causados por un número anormal de cromosomas, como el síndrome de Down y los trastornos intersexuales . [134]
Se ha demostrado que la edición genómica in vivo exitosa usando CRISPR-Cas9 se ha realizado en numerosos organismos modelo, incluidos Escherichia coli , [135] Saccharomyces cerevisiae , [136] [137] Candida albicans , Methanosarcina acetivorans , [138] [139] Caenorhabditis elegans , [140] Arabidopsis spp., [141] Danio rerio , [142] y Mus musculus . [143] [144] Se han logrado éxitos en el estudio de la biología básica, en la creación de modelos de enfermedades, [140] [145] y en el tratamiento experimental de modelos de enfermedades. [146]
Se han planteado inquietudes sobre la posibilidad de que los efectos no deseados (edición de genes distintos a los previstos) puedan confundir los resultados de un experimento de edición genética con CRISPR (es decir, el cambio fenotípico observado puede no deberse a la modificación del gen objetivo, sino a algún otro gen). Se han realizado modificaciones a CRISPR para minimizar la posibilidad de efectos no deseados. A menudo se recomiendan experimentos con CRISPR ortogonales para confirmar los resultados de un experimento de edición genética. [147] [148]
CRISPR simplifica la creación de organismos modificados genéticamente para la investigación que imitan enfermedades o muestran lo que sucede cuando un gen es eliminado o mutado. CRISPR puede usarse a nivel de la línea germinal para crear organismos en los que el gen objetivo se modifica en todas partes (es decir, en todas las células/tejidos/órganos de un organismo multicelular), o puede usarse en células que no pertenecen a la línea germinal para crear cambios locales que solo afecten a ciertas poblaciones de células dentro del organismo. [149] [150] [151]
CRISPR se puede utilizar para crear modelos celulares humanos de enfermedades. [152] Por ejemplo, cuando se aplica a células madre pluripotentes humanas , CRISPR se ha utilizado para introducir mutaciones dirigidas en genes relevantes para la enfermedad renal poliquística (PKD) y la glomeruloesclerosis focal y segmentaria (FSGS). [153] Estas células madre pluripotentes modificadas con CRISPR se cultivaron posteriormente en organoides renales humanos que exhibieron fenotipos específicos de la enfermedad. Los organoides renales de células madre con mutaciones PKD formaron estructuras quísticas grandes y translúcidas a partir de túbulos renales. Los quistes fueron capaces de alcanzar dimensiones macroscópicas, hasta un centímetro de diámetro. [154] Los organoides renales con mutaciones en un gen vinculado a FSGS desarrollaron defectos de unión entre podocitos , las células filtrantes afectadas en esa enfermedad. Esto se atribuyó a la incapacidad de los podocitos para formar microvellosidades entre células adyacentes. [155] Es importante destacar que estos fenotipos de enfermedad no estaban presentes en los organoides de control con antecedentes genéticos idénticos, pero que carecían de las modificaciones CRISPR. [153]
Se adoptó un enfoque similar para modelar el síndrome de QT largo en cardiomiocitos derivados de células madre pluripotentes. [156] Estos modelos celulares generados mediante CRISPR, con controles isogénicos, proporcionan una nueva forma de estudiar enfermedades humanas y probar medicamentos.
La tecnología CRISPR-Cas se ha propuesto como un tratamiento para múltiples enfermedades humanas, especialmente aquellas con una causa genética. [157] Su capacidad para modificar secuencias de ADN específicas la convierte en una herramienta con potencial para corregir mutaciones causantes de enfermedades. Las primeras investigaciones en modelos animales sugieren que las terapias basadas en la tecnología CRISPR tienen potencial para tratar una amplia gama de enfermedades, [158] incluyendo cáncer, [159] progeria , [160] beta-talasemia, [161] [162] [163] anemia de células falciformes , [163] [164] hemofilia , [165] fibrosis quística , [166] distrofia muscular de Duchenne , [167] enfermedad de Huntington , [168] [169] amiloidosis por transtiretina [43] y enfermedad cardíaca. [170] CRISPR también se ha utilizado para curar la malaria en mosquitos, lo que podría eliminar el vector y la enfermedad en humanos. [171] CRISPR también puede tener aplicaciones en ingeniería de tejidos y medicina regenerativa, por ejemplo mediante la creación de vasos sanguíneos humanos que carecen de expresión de proteínas MHC de clase II , que a menudo causan rechazo de trasplantes. [172]
Además, los ensayos clínicos para curar la beta talasemia y la enfermedad de células falciformes en pacientes humanos utilizando la tecnología CRISPR-Cas9 han mostrado resultados prometedores. [173] [174]
Sin embargo, aún quedan algunas limitaciones en el uso de la tecnología en la terapia génica : la frecuencia relativamente alta de efectos fuera del objetivo , el requisito de una secuencia PAM cerca del sitio objetivo, la apoptosis mediada por p53 por roturas de doble cadena inducidas por CRISPR y la toxicidad inmunogénica debido al sistema de administración típicamente por virus . [175]
CRISPR también ha encontrado muchas aplicaciones en el desarrollo de inmunoterapias basadas en células. [176] El primer ensayo clínico que involucró CRISPR comenzó en 2016. Implicó tomar células inmunes de personas con cáncer de pulmón, usar CRISPR para editar el gen PD-1 expresado y luego administrar las células alteradas nuevamente a la misma persona. Otros 20 ensayos estaban en marcha o casi listos, principalmente en China, en 2017. [update][ 159]
En 2016, la Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) aprobó un ensayo clínico en el que se utilizaría CRISPR para alterar células T extraídas de personas con diferentes tipos de cáncer y luego administrar esas células T modificadas a las mismas personas. [177]
En noviembre de 2020, en modelos animales de ratón, se utilizó CRISPR de manera eficaz para tratar el glioblastoma (tumor cerebral de rápido crecimiento) y el cáncer de ovario metastásico , ya que se trata de dos cánceres con uno de los peores pronósticos en el mejor de los casos y que suelen diagnosticarse durante sus últimas etapas. Los tratamientos han dado como resultado una inhibición del crecimiento tumoral y un aumento de la supervivencia en un 80 % en el caso del cáncer de ovario metastásico y la apoptosis de células tumorales , una inhibición del crecimiento tumoral en un 50 % y una mejora de la supervivencia en un 30 % en el caso del glioblastoma. [178]
En octubre de 2021, CRISPR Therapeutics anunció los resultados de su ensayo de fase 1 en curso en EE. UU. para una terapia con células T alogénicas. Estas células provienen de donantes sanos y se editan para atacar las células cancerosas y evitar que el sistema inmunológico del receptor las considere una amenaza, y luego se multiplican en lotes enormes que se pueden administrar a un gran número de receptores. [179]
En diciembre de 2022, una niña británica de 13 años a la que se le había diagnosticado leucemia linfoblástica aguda de células T incurable fue curada por médicos del Great Ormond Street Hospital , en el primer uso documentado de la edición genética terapéutica para este propósito, después de someterse a seis meses de un tratamiento experimental, donde los intentos anteriores de otros tratamientos fracasaron. El procedimiento incluyó la reprogramación de una célula T sana para destruir las células T cancerosas para primero librarla de la leucemia y luego reconstruir su sistema inmunológico desde cero utilizando células inmunes sanas. [180] El equipo utilizó la edición BASE y previamente había tratado un caso de leucemia linfoblástica aguda en 2015 utilizando TALEN . [181]
La diabetes tipo 1 es un trastorno endocrino que resulta de la falta de células beta pancreáticas que produzcan insulina, un compuesto vital para transportar el azúcar en sangre a las células para producir energía. Los investigadores han estado tratando de trasplantar células beta sanas. Se utiliza CRISPR para editar las células con el fin de reducir la posibilidad de que el cuerpo del paciente rechace el trasplante.
El 17 de noviembre de 2021, CRISPR therapeutics y ViaCyte anunciaron que la agencia médica canadiense había aprobado su solicitud de ensayo clínico para VCTX210, una terapia con células madre editadas mediante CRISPR diseñada para tratar la diabetes tipo 1. Esto fue significativo porque fue la primera terapia editada genéticamente para la diabetes que se acercó a las clínicas. Las mismas empresas también desarrollaron un tratamiento novedoso para la diabetes tipo 1 para producir insulina a través de un pequeño implante médico que utiliza millones de células pancreáticas derivadas de células madre editadas genéticamente mediante CRISPR. [182]
En febrero de 2022, se llevó a cabo un ensayo de fase 1 en el que un paciente voluntario recibió el tratamiento. [179] [183]
El virus de inmunodeficiencia humana o VIH es un virus que ataca el sistema inmunológico del cuerpo. Si bien existen tratamientos efectivos que pueden permitir que los pacientes vivan vidas saludables, el VIH es retroactivo, lo que significa que incorpora una versión inactiva de sí mismo en el genoma humano. CRISPR se puede utilizar para eliminar selectivamente el virus del genoma mediante el diseño de ARN guía para dirigirse al genoma retroactivo del VIH. Un problema con este enfoque es que requiere la eliminación del genoma del VIH de casi todas las células, lo que puede ser difícil de lograr de manera realista. [179]
Los resultados iniciales en el tratamiento y la cura del VIH han sido bastante exitosos: en 2021, 9 de 23 ratones humanizados fueron tratados con una combinación de antirretrovirales y CRISPR/Cas-9 que hicieron que el virus se volviera indetectable, incluso después del período de rebote habitual. Ninguno de los dos tratamientos por sí solo tuvo tal efecto. [184] Los ensayos clínicos en humanos de una terapia basada en CRISPR-Cas9, EBT-101, comenzaron en 2022. [185] [186] En octubre de 2023, un estudio en etapa temprana en 3 personas de EBT-101 informó que el tratamiento parecía ser seguro y no tenía efectos secundarios importantes, pero no se revelaron datos sobre su efectividad. [187] En marzo de 2024, otra terapia CRISPR de investigadores de la Universidad de Ámsterdam informó sobre la eliminación del VIH en cultivos celulares. [188] [189]
Las "nucleasas guiadas por ARN" basadas en CRISPR-Cas se pueden utilizar para atacar factores de virulencia , genes que codifican resistencia a antibióticos y otras secuencias de interés médicamente relevantes. Por lo tanto, esta tecnología representa una nueva forma de terapia antimicrobiana y una estrategia para manipular poblaciones bacterianas. [190] [191] Estudios recientes sugieren una correlación entre la interferencia del locus CRISPR-Cas y la adquisición de resistencia a antibióticos. [192] Este sistema proporciona protección a las bacterias contra la invasión de ADN extraño, como transposones , bacteriófagos y plásmidos. Se demostró que este sistema es una fuerte presión selectiva para la adquisición de resistencia a antibióticos y factor de virulencia en patógenos bacterianos. [192]
Las terapias basadas en la tecnología de edición genética CRISPR-Cas3 administrada por bacteriófagos diseñados podrían usarse para destruir ADN específico en patógenos. [193] Cas3 es más destructivo que el más conocido Cas9. [194] [195]
Las investigaciones sugieren que la CRISPR es una forma eficaz de limitar la replicación de múltiples virus del herpes . Fue capaz de erradicar el ADN viral en el caso del virus de Epstein-Barr (VEB). Las CRISPR anti-herpesvirus tienen aplicaciones prometedoras, como la eliminación del VEB que causa cáncer de las células tumorales, ayudar a eliminar los invasores virales de los órganos donados para pacientes inmunodeprimidos o prevenir los brotes de herpes labial y las infecciones oculares recurrentes al bloquear la reactivación del VHS-1 . En agosto de 2016 [update], estas técnicas estaban pendientes de ser probadas. [196]
La tecnología CRISPR puede revivir el concepto de trasplantar órganos animales a personas. Los retrovirus presentes en los genomas animales podrían dañar a los receptores de trasplantes. En 2015, un equipo eliminó 62 copias de una secuencia particular de ADN retroviral del genoma de cerdo en una célula epitelial de riñón. [197] Recientemente, los investigadores demostraron la capacidad de generar especímenes de cerdo vivos después de eliminar estos retrovirus de su genoma utilizando CRISPR por primera vez. [198]
CRISPR se puede utilizar para suprimir mutaciones que causan ganancia de función, y también para reparar mutaciones que causan pérdida de función en trastornos neurológicos. [199] La herramienta de edición genética se ha convertido en una aplicación in vivo de referencia para la asimilación de vías moleculares.
CRISPR es único en el desarrollo de soluciones para enfermedades neurológicas por varias razones. Por ejemplo, CRISPR permite a los investigadores generar rápidamente modelos de células animales y humanas. Esto les permite estudiar cómo funcionan los genes en un sistema nervioso. Al introducir mutaciones relacionadas con varias enfermedades dentro de estas células, los investigadores pueden estudiar los efectos de los cambios en el desarrollo, la función y el comportamiento del sistema nervioso. [200] Pueden descubrir los mecanismos moleculares que contribuyen a estos trastornos, lo que es esencial para desarrollar tratamientos efectivos. Esto es particularmente útil para modelar y tratar trastornos neurológicos complejos como el Alzheimer , el Parkinson y la epilepsia , entre otros.
La enfermedad de Alzheimer (EA) es una enfermedad neurodegenerativa caracterizada por la pérdida de neuronas y la acumulación de ovillos neurofibrilares intracelulares y placas amiloides extracelulares en el cerebro. [201] Se han identificado tres genes patógenos que causan la aparición temprana de EA en humanos, específicamente la proteína precursora amiloide (APP), la presenilina 1 (PSEN1) y la presenilina 2 (PSEN2). [201] Se han detectado más de 300 mutaciones en estos genes, lo que resulta en un aumento del β-amiloide total (Aβ), la relación Aβ42/40 y/o la polimerización de Aβ.
En el caso de la distrofia muscular de Duchenne , la mutación responsable de la enfermedad se produce en el gen de la distrofina. [202] Se ha utilizado CRISPR para corregir esto. De manera similar, para el síndrome de Dravet , un trastorno de epilepsia, se ha utilizado CRISPR para corregir la mutación del gen SCN1A . [203] A pesar del progreso que se ha logrado, aún existen desafíos con el uso de CRISPR, uno de los cuales es la transferencia de componentes CRISPR a través de la barrera hematoencefálica . Sin embargo, los avances recientes en sistemas de administración de nanopartículas y vectores virales han demostrado ser prometedores para superar este obstáculo [ cita requerida ] . De cara al futuro, se espera que el uso de CRISPR en neurociencia aumente a medida que evolucione la tecnología.
Las enfermedades oculares más comunes en todo el mundo son las cataratas y la retinitis pigmentosa (RP). Estas son causadas por una mutación sin sentido en la cadena alfa que conduce a la ceguera permanente. Un desafío para el uso de CRISPR en enfermedades oculares es que el tejido retiniano del ojo está libre de la respuesta inmune del cuerpo. El enfoque de los investigadores para usar CRISPR es embolsar el gen que codifica la proteína retiniana y editar el genoma. [204]
El tratamiento CRISPR para LCA10 (la variante más común de la amaurosis congénita de Leber , que es la principal causa de ceguera infantil hereditaria) modifica el gen fotorreceptor defectuoso del paciente.
En marzo de 2020, el primer paciente voluntario de este estudio con sede en EE. UU., patrocinado por Editas Medicine , recibió una dosis baja del tratamiento para probar su seguridad. En junio de 2021, comenzó la inscripción para una cohorte de adultos y pediátricos de dosis alta de 4 pacientes voluntarios cada uno. Se espera que la dosificación de las nuevas cohortes se complete en julio de 2022. [179] En noviembre de 2022, Editas informó que el 20% de los pacientes tratados tuvieron mejoras significativas, pero también anunció que la población objetivo resultante era demasiado pequeña para sustentar un desarrollo independiente continuo. [205]
Se ha demostrado que la tecnología CRISPR funciona de manera eficaz en el tratamiento de enfermedades cardíacas. En el caso de la hipercolesterolemia familiar (HF), la deposición de colesterol en las paredes de las arterias provoca el bloqueo del flujo sanguíneo. Esto es causado por una mutación en los receptores de colesterol de lipoproteínas de baja densidad (LDLC) , que da como resultado una liberación excesiva de colesterol en la sangre. Esto se puede tratar mediante la eliminación de un par de bases en el exón 4 del receptor LDLC. Esta es una mutación sin sentido . [ cita requerida ]
Esta enfermedad se enmarca dentro de los trastornos genéticos que son causados por mutaciones que ocurren en la estructura de la hemoglobina o debido a la sustitución de diferentes aminoácidos en las cadenas de globina. Debido a esto, los glóbulos rojos (RBC) causan una serie de obstáculos como insuficiencia cardíaca, obstrucción de los vasos sanguíneos, defectos en el crecimiento y problemas ópticos. [206] Para rehabilitar las β-hemoglobinopatías, las células multipotentes del paciente se transfieren a un modelo de ratones para estudiar la tasa de terapia génica ex vivo que da como resultado la expresión del ARNm y la rectificación del gen. Curiosamente, la vida media de los glóbulos rojos también aumentó.
La hemofilia es una pérdida de función en la sangre en la que los factores de coagulación no funcionan correctamente. Mediante el uso de CRISPR-Cas9, se inserta un vector en las bacterias. [207] El vector utilizado es un vector adenoviral que ayuda a corregir los genes.
La aplicación de CRISPR en plantas se logró con éxito en el año 2013. CRISPR Cas9 se ha convertido en una herramienta influyente en la edición de genomas en cultivos. Ha dejado una marca en los sistemas de mejoramiento actuales, [208]
Para aumentar el rendimiento de los cultivos de cereales, se modifica el equilibrio de citoquinina. La citoquinina oxidasa/deshidrogenasa (CKX) es una enzima que inhibe el crecimiento de brotes en el arroz, [209] por lo que se eliminó el gen que codifica esta enzima para aumentar el rendimiento.
Los cereales tienen una gran cantidad de polisacárido amilosa. Para disminuir el contenido de amilosa, se utiliza CRISPR para alterar los aminoácidos y reducir la producción de sacáridos. Además, el trigo contiene la proteína gluten , a la que algunas personas son intolerantes y causa la enfermedad celíaca . La herramienta de edición genética se dirige a los genes que codifican el gluten, lo que da como resultado un trigo con una menor producción de gluten. [210]
El estrés biótico de las plantas se puede reducir mediante el uso de CRISPR. Las infecciones bacterianas en el arroz provocan la activación de la transcripción de genes, cuyos productos son susceptibles a la enfermedad. Mediante el uso de CRISPR, los científicos pudieron generar resistencia hereditaria al oídio. [211]
En la actualidad se conocen unos 6000 trastornos genéticos, la mayoría de los cuales no tienen tratamiento. El papel de CRISPR en la terapia génica es sustituir el ADN exógeno por genes defectuosos. [212] La terapia génica ha tenido un enorme impacto y ha abierto muchas nuevas posibilidades en la biotecnología médica.
Existen dos tipos de ediciones base:
El editor de bases de citidina es una nueva terapia en la que la citidina (C) cambia a timidina (T).
Editor de bases de adenina (ABE), [213] en este hay un cambio en los complementos de bases de adenina (A) a guanina (G).
Las mutaciones se instalaron directamente en el ADN celular, por lo que no se requiere la plantilla donante. Las ediciones de base solo pueden editar mutaciones puntuales. Además, solo pueden corregir hasta cuatro mutaciones puntuales. [214] Para abordar este problema, el sistema CRISPR introdujo una nueva técnica conocida como fusión Cas9 para aumentar la escala de genes que se pueden editar.
Además, la proteína CRISPR Cas9 puede modular genes ya sea activándolos o silenciándolos en función de los genes de interés. [215] Existe una nucleasa llamada dCas9 (endonucleasa) que se utiliza para silenciar o activar la expresión de genes.
Los investigadores se enfrentan a muchos desafíos en la edición genética. [216] Los principales obstáculos que se presentan en las aplicaciones clínicas son las cuestiones éticas y el sistema de transporte al sitio objetivo. Como las unidades del sistema CRISPR se toman de las bacterias, cuando se transfieren a las células huésped, se produce una respuesta inmune contra ellas. Se utilizan vectores físicos, químicos y virales como vehículos para entregar el complejo al huésped. [ cita requerida ] Debido a esto, surgen muchas complicaciones, como el daño celular que conduce a la muerte celular. En el caso de los vectores virales, la capacidad del virus es pequeña y la proteína Cas9 es grande. Entonces, para superarlos, se desarrollaron nuevos métodos en los que se toman cepas más pequeñas de Cas9 de las bacterias. Finalmente, todavía se necesita una gran cantidad de trabajo para mejorar el sistema.


Las nucleasas asociadas a CRISPR han demostrado ser útiles como herramienta para pruebas moleculares debido a su capacidad para apuntar específicamente a secuencias de ácidos nucleicos en un alto fondo de secuencias no objetivo. [219] En 2016, la nucleasa Cas9 se utilizó para agotar secuencias de nucleótidos no deseadas en bibliotecas de secuenciación de próxima generación mientras que solo requería 250 picogramos de entrada de ARN inicial. [220] A partir de 2017, las nucleasas asociadas a CRISPR también se utilizaron para pruebas de diagnóstico directo de ácidos nucleicos, hasta la sensibilidad de una sola molécula. [221] [222] La diversidad de CRISPR se utiliza como un objetivo de análisis para discernir la filogenia y la diversidad en bacterias, como en xantomonas por Martins et al. , 2019. [223] : 552 Las detecciones tempranas de patógenos vegetales mediante la tipificación molecular de los CRISPR del patógeno se pueden utilizar en la agricultura como lo demostraron Shen et al. , 2020. [223] : 553
Al acoplar los diagnósticos basados en CRISPR a procesos enzimáticos adicionales, es posible la detección de moléculas más allá de los ácidos nucleicos. Un ejemplo de una tecnología acoplada es el perfil de transcripción IN vitro basado en SHERLOCK (SPRINT). SPRINT se puede utilizar para detectar una variedad de sustancias, como metabolitos en muestras de pacientes o contaminantes en muestras ambientales, con alto rendimiento o con dispositivos portátiles de punto de atención. [224] También se están explorando plataformas CRISPR-Cas para la detección [225] [226] [ 218] [227] [228] e inactivación del SARS-CoV-2 , el virus que causa COVID-19 . [229] Se han identificado dos pruebas de diagnóstico integrales diferentes, AIOD-CRISPR y la prueba SHERLOCK para el SARS-CoV-2. [230] La prueba SHERLOCK se basa en un ARN reportero de prensa marcado con fluorescencia que tiene la capacidad de identificar 10 copias por microlitro. [231] La AIOD-CRISPR ayuda a la detección visual robusta y altamente sensible del ácido nucleico viral. [232]
CRISPR-Cas9 se puede utilizar para investigar e identificar las diferencias genéticas de los humanos con respecto a otros simios, especialmente en el cerebro . Por ejemplo, al reintroducir variantes genéticas arcaicas en organoides cerebrales para mostrar un impacto en la neurogénesis, [233] la longitud de la metafase de los progenitores apicales del neocórtex en desarrollo, [234] o al inactivar un gen en células madre embrionarias para identificar un regulador genético que, a través de la transición temprana de la forma celular, impulsa la expansión evolutiva del prosencéfalo humano . [235] [236] Un estudio describió un impacto importante de una variante genética arcaica en el desarrollo neurológico [237] [238] que puede ser un artefacto de un efecto secundario de CRISPR, [239] [240] ya que no se pudo replicar en un estudio posterior. [78]
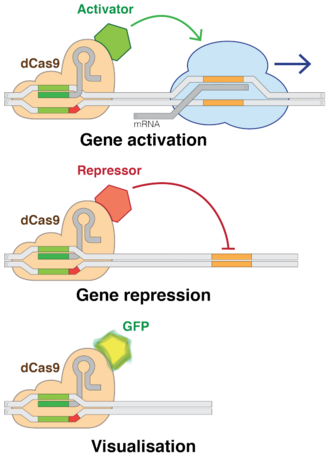
El uso de versiones "muertas" de Cas9 ( dCas9 ) elimina la capacidad de corte de ADN de CRISPR, al tiempo que preserva su capacidad de dirigirse a secuencias deseables. Múltiples grupos agregaron varios factores reguladores a dCas9s, lo que les permitió activar o desactivar casi cualquier gen o ajustar su nivel de actividad. [197] Al igual que RNAi, la interferencia CRISPR (CRISPRi) desactiva genes de manera reversible al dirigirse, pero no cortar un sitio. El sitio dirigido se metila, modificando epigenéticamente el gen. Esta modificación inhibe la transcripción. Estas modificaciones colocadas con precisión pueden luego usarse para regular los efectos sobre las expresiones genéticas y la dinámica del ADN después de la inhibición de ciertas secuencias genómicas dentro del ADN. En los últimos años, las marcas epigenéticas en diferentes células humanas se han investigado de cerca y se ha descubierto que ciertos patrones dentro de las marcas se correlacionan con todo, desde el crecimiento tumoral hasta la actividad cerebral. [10] Por el contrario, la activación mediada por CRISPR (CRISPRa) promueve la transcripción genética. [241] Cas9 es una forma eficaz de atacar y silenciar genes específicos a nivel de ADN. [242] En las bacterias, la presencia de Cas9 por sí sola es suficiente para bloquear la transcripción. Para aplicaciones en mamíferos, se añade una sección de proteína. Su ARN guía ataca secuencias de ADN reguladoras llamadas promotores que preceden inmediatamente al gen diana. [243]
Se utilizó Cas9 para transportar factores de transcripción sintéticos que activaban genes humanos específicos. La técnica logró un efecto potente al dirigir múltiples construcciones CRISPR a lugares ligeramente diferentes en el promotor del gen. [243]
En 2016, los investigadores demostraron que el CRISPR de una bacteria bucal común podía usarse para editar el ARN . Los investigadores buscaron en bases de datos que contenían cientos de millones de secuencias genéticas aquellas que se parecían a los genes CRISPR. Consideraron la fusobacteria Leptotrichia shahii . Tenía un grupo de genes que se parecían a los genes CRISPR, pero con diferencias importantes. Cuando los investigadores equiparon otras bacterias con estos genes, a los que llamaron C2c2, descubrieron que los organismos adquirían una defensa novedosa. [244] C2c2 fue posteriormente renombrado como Cas13a para adaptarse a la nomenclatura estándar de los genes Cas. [245]
Muchos virus codifican su información genética en ARN en lugar de ADN, que luego reutilizan para crear nuevos virus. El VIH y el poliovirus son algunos de ellos. Las bacterias con Cas13 crean moléculas que pueden desmembrar el ARN y destruir el virus. La adaptación de estos genes abrió cualquier molécula de ARN a la edición. [244]
Los sistemas CRISPR-Cas también se pueden emplear para editar genes de micro-ARN y ARN largo no codificante en plantas. [246]
La edición dirigida para corregir secuencias mutadas se propuso y demostró por primera vez en 1995. [247] Este trabajo inicial utilizó oligonucleótidos antisentido de ARN sintético complementarios a una mutación prematura del codón de terminación en una secuencia de distrofina para activar la edición de A a I del codón de terminación a un codón de lectura directa en un sistema de células modelo de xenopus. [247] Si bien esto también condujo a transiciones inadvertidas cercanas de A a I, las transiciones de A a I (leídas como G) pueden corregir los tres codones de terminación, pero no pueden crear un codón de terminación. Por lo tanto, los cambios condujeron a una corrección de >25% del codón de terminación objetivo con lectura directa a una secuencia reportera de luciferasa corriente abajo. El trabajo posterior de Rosenthal logró la edición de la secuencia de ARNm mutada en un cultivo de células de mamíferos al dirigir un oligonucleótido unido a una citidina desaminasa para corregir una secuencia de fibrosis quística mutada. [248] Más recientemente, se ha empleado CRISPR-Cas13 fusionado a desaminasas para dirigir la edición del ARNm. [249]
En 2022, se informó sobre la edición terapéutica del ARN para Cas7-11. [250] [251] Permite cortes suficientemente específicos y una versión temprana del mismo se utilizó para la edición in vitro en 2021. [252]Las unidades genéticas pueden proporcionar una herramienta poderosa para restablecer el equilibrio de los ecosistemas mediante la eliminación de especies invasoras. Se han planteado inquietudes sobre la eficacia y las consecuencias no deseadas en las especies objetivo, así como en las especies no objetivo, en particular en relación con la posibilidad de liberación accidental de los laboratorios a la naturaleza. Los científicos han propuesto varias salvaguardas para asegurar la contención de las unidades genéticas experimentales, incluidas las moleculares, reproductivas y ecológicas. [254] Muchos recomiendan que se desarrollen unidades de inmunización y de reversión en conjunto con las unidades genéticas para sobrescribir sus efectos si es necesario. [255] Sigue habiendo consenso en que los efectos a largo plazo deben estudiarse más a fondo, en particular en relación con el potencial de alteración ecológica que no se puede corregir con unidades de reversión. [256]
Las bibliotecas de secuenciación no enriquecidas suelen contener abundantes secuencias no deseadas. Cas9 puede eliminar específicamente las secuencias no deseadas con rotura de doble cadena con una eficacia de hasta el 99 % y sin efectos significativos fuera del objetivo , como se observa con las enzimas de restricción . El tratamiento con Cas9 puede eliminar abundante ARNr al tiempo que aumenta la sensibilidad a los patógenos en las bibliotecas de secuenciación de ARN. [257]

La edición o ingeniería del epigenoma es un tipo de ingeniería genética en la que el epigenoma se modifica en sitios específicos utilizando moléculas diseñadas específicamente para esos sitios (en lugar de modificaciones del genoma completo). Mientras que la edición genética implica cambiar la secuencia de ADN en sí, la edición epigenética implica modificar y presentar secuencias de ADN a proteínas y otros factores de unión al ADN que influyen en la función del ADN. Al "editar" características epigenómicas de esta manera, los investigadores pueden determinar el papel biológico exacto de una modificación epigenética en el sitio en cuestión.
Las proteínas diseñadas que se utilizan para la edición del epigenoma se componen de un dominio de unión al ADN que se dirige a secuencias específicas y un dominio efector que modifica las características epigenómicas. En la actualidad, se han utilizado predominantemente tres grupos principales de proteínas de unión al ADN para la edición del epigenoma: proteínas con dedos de zinc , efectores similares a activadores de la transcripción (TALE) y fusiones Cas9 deficientes en nucleasas ( CRISPR ).La regulación dirigida de genes relacionados con enfermedades puede permitir nuevas terapias para muchas enfermedades, especialmente en casos en los que aún no se han desarrollado terapias genéticas adecuadas o son inadecuadas. [258] Si bien las consecuencias transgeneracionales y a nivel poblacional no se comprenden por completo, puede convertirse en una herramienta importante para la genómica funcional aplicada y la medicina personalizada . [259] Al igual que con la edición de ARN , no implica cambios genéticos y sus riesgos acompañantes. [258] En 2021 se describió un ejemplo de un posible uso funcional de la edición del epigenoma: la represión de la expresión del gen Na v 1.7 a través de CRISPR-dCas9 , que mostró potencial terapéutico en tres modelos de ratón de dolor crónico. [260] [261]
En 2022, una investigación evaluó su utilidad para reducir los niveles de proteína tau , regular una proteína involucrada en la enfermedad de Huntington , atacar una forma hereditaria de obesidad y el síndrome de Dravet . [262]La combinación de CRISPR-Cas9 con integrasas permitió una técnica para realizar ediciones a gran escala sin problemas de rotura de doble cadena, como se demostró con PASTE en 2022. Los investigadores informaron que podría usarse para administrar genes de hasta 36 000 pares de bases de ADN a varios tipos de células humanas y, por lo tanto, potencialmente para tratar enfermedades causadas por una gran cantidad de mutaciones. [263] [264]
La edición primaria [265] (o edición de base) es un refinamiento de CRISPR para insertar o eliminar con precisión secciones de ADN. Las ediciones CRISPR no siempre son perfectas y los cortes pueden terminar en el lugar equivocado. Ambos problemas son un problema para el uso de la tecnología en medicina. [266] La edición primaria no corta el ADN de doble cadena, sino que utiliza el aparato de orientación CRISPR para transportar una enzima adicional a una secuencia deseada, donde convierte un solo nucleótido en otro. [267] La nueva guía, llamada pegRNA, contiene una plantilla de ARN para una nueva secuencia de ADN que se agregará al genoma en la ubicación objetivo. Eso requiere una segunda proteína, unida a Cas9: una enzima transcriptasa inversa, que puede crear una nueva cadena de ADN a partir de la plantilla de ARN e insertarla en el sitio mellado. [268] Cada uno de esos tres eventos de emparejamiento independientes brinda una oportunidad para prevenir secuencias fuera del objetivo, lo que aumenta significativamente la flexibilidad de la orientación y la precisión de la edición. [267] La edición primaria fue desarrollada por investigadores del Instituto Broad del MIT y Harvard en Massachusetts. [269] Se necesita más trabajo para optimizar los métodos. [269] [268]
En marzo de 2015, varios grupos habían anunciado investigaciones en curso con la intención de sentar las bases para aplicar CRISPR a embriones humanos para la ingeniería de la línea germinal humana , incluidos laboratorios en los EE. UU., China y el Reino Unido, así como la empresa de biotecnología estadounidense OvaScience . [270] Los científicos, incluido un codescubridor de CRISPR, instaron a una moratoria mundial sobre la aplicación de CRISPR a la línea germinal humana, especialmente para uso clínico. Dijeron que "los científicos deberían evitar incluso intentar, en jurisdicciones laxas, la modificación del genoma de la línea germinal para la aplicación clínica en humanos" hasta que las implicaciones completas "se discutan entre las organizaciones científicas y gubernamentales". [271] [272] Estos científicos apoyan una mayor investigación de bajo nivel sobre CRISPR y no ven que CRISPR esté lo suficientemente desarrollado para cualquier uso clínico en la realización de cambios hereditarios en humanos. [273]
En abril de 2015, científicos chinos informaron los resultados de un intento de alterar el ADN de embriones humanos no viables utilizando CRISPR para corregir una mutación que causa beta talasemia , un trastorno hereditario letal. [274] [275] El estudio había sido rechazado anteriormente tanto por Nature como por Science en parte debido a preocupaciones éticas. [276] Los experimentos dieron como resultado cambiar con éxito solo algunos de los genes previstos y tuvieron efectos no deseados en otros genes. Los investigadores afirmaron que CRISPR no está listo para su aplicación clínica en medicina reproductiva . [276] En abril de 2016, se informó que científicos chinos habían hecho un segundo intento fallido de alterar el ADN de embriones humanos no viables utilizando CRISPR, esta vez para alterar el gen CCR5 para hacer que el embrión sea resistente a la infección por VIH . [277]
En diciembre de 2015, se celebró en Washington una Cumbre Internacional sobre Edición Genética Humana bajo la dirección de David Baltimore . Miembros de las academias científicas nacionales de los EE. UU., el Reino Unido y China discutieron la ética de la modificación de la línea germinal. Acordaron apoyar la investigación básica y clínica bajo ciertas pautas legales y éticas. Se hizo una distinción específica entre las células somáticas , donde los efectos de las ediciones se limitan a un solo individuo, y las células de la línea germinal, donde los cambios del genoma pueden ser heredados por los descendientes. Las modificaciones hereditarias podrían tener consecuencias no deseadas y de largo alcance para la evolución humana, genéticamente (por ejemplo, interacciones gen-ambiente) y culturalmente (por ejemplo, darwinismo social ). La alteración de gametocitos y embriones para generar cambios hereditarios en humanos se definió como irresponsable. El grupo acordó iniciar un foro internacional para abordar tales preocupaciones y armonizar las regulaciones en todos los países. [278]
En febrero de 2017, el Comité de Edición Genética Humana de las Academias Nacionales de Ciencias, Ingeniería y Medicina ( NASEM ) de los Estados Unidos publicó un informe en el que se analizaban las cuestiones éticas, jurídicas y científicas de la tecnología de ingeniería genómica. La conclusión del informe afirmaba que la edición genómica hereditaria no está permitida en la actualidad, pero que podría justificarse en el caso de determinadas enfermedades; sin embargo, no justificaba el uso de CRISPR para la mejora genética. [279]
En noviembre de 2018, Jiankui He anunció que había editado dos embriones humanos para intentar desactivar el gen CCR5 , que codifica un receptor que el VIH usa para ingresar a las células. Dijo que las niñas gemelas, Lulu y Nana , habían nacido unas semanas antes. Dijo que las niñas todavía portaban copias funcionales de CCR5 junto con CCR5 desactivado ( mosaicismo ) y aún eran vulnerables al VIH. El trabajo fue ampliamente condenado como poco ético, peligroso y prematuro. [280] Un grupo internacional de científicos pidió una moratoria global sobre la edición genética de embriones humanos. [281]
Bebés de diseño
La aparición de la tecnología de edición genética CRISPR-Cas9 ha abierto la posibilidad de crear "bebés de diseño". Esta tecnología tiene la posibilidad de eliminar ciertas enfermedades genéticas o mejorar la salud al potenciar ciertos rasgos genéticos.
Las normativas sobre el sistema CRISPR-Cas9 varían en todo el mundo. En febrero de 2016, los organismos reguladores dieron permiso a científicos británicos para modificar genéticamente embriones humanos mediante el uso de CRISPR-Cas9 y técnicas relacionadas. Sin embargo, se les prohibió a los investigadores implantar los embriones y estos debían ser destruidos después de siete días. [282]
Estados Unidos cuenta con un elaborado sistema regulatorio interdepartamental para evaluar nuevos alimentos y cultivos modificados genéticamente. Por ejemplo, la Ley de Protección de Riesgos Agrícolas de 2000 otorga al Departamento de Agricultura de los Estados Unidos la autoridad para supervisar la detección, control, erradicación, supresión, prevención o retraso de la propagación de plagas de plantas o malezas nocivas para proteger la agricultura, el medio ambiente y la economía de los Estados Unidos. La ley regula cualquier organismo modificado genéticamente que utilice el genoma de una "plaga de plantas" predefinida o cualquier planta no categorizada previamente. [283] En 2015, Yinong Yang desactivó con éxito 16 genes específicos en el champiñón blanco para que no se oscurecieran. Dado que no había agregado ADN de especies extrañas ( transgénico ) a su organismo, el hongo no podía ser regulado por el USDA según la Sección 340.2. [284] El champiñón blanco de Yang fue el primer organismo modificado genéticamente con el sistema de proteína CRISPR-Cas9 en pasar la regulación estadounidense. [285]
En 2016, el USDA patrocinó un comité para considerar la política regulatoria futura para las próximas técnicas de modificación genética. Con la ayuda de las Academias Nacionales de Ciencias, Ingeniería y Medicina de EE. UU ., grupos de intereses especiales se reunieron el 15 de abril para contemplar los posibles avances en ingeniería genética dentro de los próximos cinco años y cualquier nueva regulación que pudiera ser necesaria como resultado. [286] En 2017, la Administración de Alimentos y Medicamentos propuso una norma que clasificaría las modificaciones de ingeniería genética a los animales como "medicamentos animales", sometiéndolos a una estricta regulación si se ofrecen para la venta y reduciendo la capacidad de los individuos y las pequeñas empresas para rentabilizarlos. [287] [288]
En China, donde las condiciones sociales contrastan marcadamente con las de Occidente, las enfermedades genéticas conllevan un fuerte estigma. [289] Esto deja a China con menos barreras políticas para el uso de esta tecnología. [290] [291]
En 2012 y 2013, CRISPR fue finalista en el premio Breakthrough of the Year de la revista Science . En 2015, fue el ganador de ese premio. [197] CRISPR fue nombrado como una de las 10 tecnologías innovadoras de MIT Technology Review en 2014 y 2016. [292] [293] En 2016, Jennifer Doudna y Emmanuelle Charpentier , junto con Rudolph Barrangou, Philippe Horvath y Feng Zhang ganaron el premio Gairdner International. En 2017, Doudna y Charpentier recibieron el Premio Japón en Tokio, Japón por su revolucionaria invención de CRISPR-Cas9. En 2016, Charpentier, Doudna y Zhang ganaron el Premio Tang en Ciencia Biofarmacéutica. [294] En 2020, Charpentier y Doudna recibieron el Premio Nobel de Química , el primer premio de este tipo para un equipo formado exclusivamente por mujeres, "por el desarrollo de un método de edición del genoma". [295]
<...> Bueno, fuimos nosotros los que enviamos el artículo primero, pero no tuvimos mucha suerte.
{{cite book}}: CS1 maint: location missing publisher (link){{cite web}}: CS1 maint: numeric names: authors list (link){{cite journal}}: CS1 maint: DOI inactive as of March 2024 (link)The biologists writing in Science support continuing laboratory research with the technique, and few if any scientists believe it is ready for clinical use.