
Las enzimas ( / ˈɛnzaɪmz / ) son proteínas que actúan como catalizadores biológicos al acelerar las reacciones químicas . Las moléculas sobre las que pueden actuar las enzimas se denominan sustratos , y la enzima convierte los sustratos en diferentes moléculas conocidas como productos . Casi todos los procesos metabólicos en la célula necesitan catálisis enzimática para ocurrir a velocidades lo suficientemente rápidas para sostener la vida. [1] : 8.1 Las vías metabólicas dependen de las enzimas para catalizar los pasos individuales. El estudio de las enzimas se llama enzimología y el campo del análisis de pseudoenzimas reconoce que durante la evolución, algunas enzimas han perdido la capacidad de llevar a cabo catálisis biológica, lo que a menudo se refleja en sus secuencias de aminoácidos y propiedades "pseudocatalíticas" inusuales. [2] [3]
Se sabe que las enzimas catalizan más de 5.000 tipos de reacciones bioquímicas. [4]
Otros biocatalizadores son las moléculas catalíticas de ARN , también llamadas ribozimas . A veces se las describe como un tipo de enzima en lugar de como una enzima, pero incluso en las décadas transcurridas desde el descubrimiento de las ribozimas en 1980-1982, la palabra enzima sola a menudo se refiere específicamente al tipo de proteína (como se usa en este artículo).
La especificidad de una enzima proviene de su estructura tridimensional única .
Como todos los catalizadores, las enzimas aumentan la velocidad de reacción al disminuir su energía de activación . Algunas enzimas pueden hacer que su conversión de sustrato a producto ocurra muchos millones de veces más rápido. Un ejemplo extremo es la orotidina 5'-fosfato descarboxilasa , que permite que una reacción que de otro modo tardaría millones de años ocurra en milisegundos. [5] [6] Químicamente, las enzimas son como cualquier catalizador y no se consumen en reacciones químicas, ni alteran el equilibrio de una reacción. Las enzimas se diferencian de la mayoría de los demás catalizadores por ser mucho más específicas. La actividad enzimática puede verse afectada por otras moléculas: los inhibidores son moléculas que disminuyen la actividad enzimática y los activadores son moléculas que aumentan la actividad. Muchos fármacos terapéuticos y venenos son inhibidores de enzimas. La actividad de una enzima disminuye notablemente fuera de su temperatura y pH óptimos , y muchas enzimas se desnaturalizan (permanentemente) cuando se exponen a un calor excesivo, perdiendo su estructura y propiedades catalíticas.
Algunas enzimas se utilizan comercialmente, por ejemplo, en la síntesis de antibióticos . Algunos productos domésticos utilizan enzimas para acelerar las reacciones químicas: las enzimas de los detergentes biológicos descomponen las manchas de proteínas, almidón o grasa en la ropa, y las enzimas del ablandador de carne descomponen las proteínas en moléculas más pequeñas, lo que hace que la carne sea más fácil de masticar.

A finales del siglo XVII y principios del XVIII, se conocía la digestión de la carne mediante secreciones estomacales [7] y la conversión del almidón en azúcares mediante extractos de plantas y saliva , pero no se habían identificado los mecanismos por los cuales esto ocurría. [8]
El químico francés Anselme Payen fue el primero en descubrir una enzima, la diastasa , en 1833. [9] Unas décadas más tarde, al estudiar la fermentación del azúcar en alcohol por la levadura , Louis Pasteur concluyó que esta fermentación era causada por una fuerza vital contenida dentro de las células de levadura llamadas "fermentos", que se pensaba que funcionaban solo dentro de los organismos vivos. Escribió que "la fermentación alcohólica es un acto correlacionado con la vida y la organización de las células de levadura, no con la muerte o putrefacción de las células". [10]
En 1877, el fisiólogo alemán Wilhelm Kühne (1837-1900) utilizó por primera vez el término enzima , que proviene del griego antiguo ἔνζυμον (énzymon) ' leudado , en levadura', para describir este proceso. [11] La palabra enzima se utilizó más tarde para referirse a sustancias no vivas como la pepsina , y la palabra fermento se utilizó para referirse a la actividad química producida por organismos vivos. [12]
Eduard Buchner presentó su primer artículo sobre el estudio de extractos de levadura en 1897. En una serie de experimentos en la Universidad de Berlín , descubrió que el azúcar era fermentado por extractos de levadura incluso cuando no había células de levadura vivas en la mezcla. [13] Llamó a la enzima que provocaba la fermentación de la sacarosa " zimasa ". [14] En 1907, recibió el Premio Nobel de Química por "su descubrimiento de la fermentación sin células". Siguiendo el ejemplo de Buchner, las enzimas suelen nombrarse según la reacción que llevan a cabo: el sufijo -asa se combina con el nombre del sustrato (p. ej., la lactasa es la enzima que escinde la lactosa ) o con el tipo de reacción (p. ej., la ADN polimerasa forma polímeros de ADN). [15]
La identidad bioquímica de las enzimas era todavía desconocida a principios del siglo XX. Muchos científicos observaron que la actividad enzimática estaba asociada con las proteínas, pero otros (como el premio Nobel Richard Willstätter ) argumentaron que las proteínas eran simplemente portadoras de las enzimas verdaderas y que las proteínas per se eran incapaces de catálisis. [16] En 1926, James B. Sumner demostró que la enzima ureasa era una proteína pura y la cristalizó; hizo lo mismo con la enzima catalasa en 1937. La conclusión de que las proteínas puras pueden ser enzimas fue demostrada definitivamente por John Howard Northrop y Wendell Meredith Stanley , quienes trabajaron en las enzimas digestivas pepsina (1930), tripsina y quimotripsina . Estos tres científicos fueron galardonados con el Premio Nobel de Química de 1946. [17]
El descubrimiento de que las enzimas podían cristalizarse permitió finalmente que sus estructuras se resolvieran mediante cristalografía de rayos X. Esto se hizo por primera vez con la lisozima , una enzima que se encuentra en las lágrimas, la saliva y las claras de huevo y que digiere el recubrimiento de algunas bacterias; la estructura fue resuelta por un grupo dirigido por David Chilton Phillips y publicada en 1965. [18] Esta estructura de alta resolución de la lisozima marcó el comienzo del campo de la biología estructural y el esfuerzo por comprender cómo funcionan las enzimas a un nivel atómico de detalle. [19]
Las enzimas se pueden clasificar según dos criterios principales: similitud de secuencia de aminoácidos (y, por tanto, relación evolutiva) o actividad enzimática.
Actividad enzimática . El nombre de una enzima suele derivar de su sustrato o de la reacción química que cataliza, y la palabra termina en -asa . [1] : 8.1.3 Algunos ejemplos son la lactasa , la alcohol deshidrogenasa y la ADN polimerasa . Las diferentes enzimas que catalizan la misma reacción química se denominan isoenzimas . [1] : 10.3
La Unión Internacional de Bioquímica y Biología Molecular ha desarrollado una nomenclatura para las enzimas, los números EC (por "Enzyme Commission") . Cada enzima se describe con "EC" seguido de una secuencia de cuatro números que representan la jerarquía de la actividad enzimática (de muy general a muy específica). Es decir, el primer número clasifica ampliamente la enzima en función de su mecanismo, mientras que los demás dígitos añaden más y más especificidad. [20]
La clasificación de nivel superior es:
Estas secciones se subdividen según otras características, como el sustrato, los productos y el mecanismo químico . Una enzima se especifica completamente mediante cuatro designaciones numéricas. Por ejemplo, la hexoquinasa (EC 2.7.1.1) es una transferasa (EC 2) que añade un grupo fosfato (EC 2.7) a un azúcar hexosa, una molécula que contiene un grupo alcohol (EC 2.7.1). [21]
Similitud de secuencia . Las categorías de EC no reflejan la similitud de secuencia. Por ejemplo, dos ligasas del mismo número de EC que catalizan exactamente la misma reacción pueden tener secuencias completamente diferentes. Independientemente de su función, las enzimas, como cualquier otra proteína, se han clasificado por su similitud de secuencia en numerosas familias. Estas familias se han documentado en docenas de bases de datos de proteínas y familias de proteínas diferentes, como Pfam . [22]
Enzimas isofuncionales no homólogas . Las enzimas no relacionadas que tienen la misma actividad enzimática se han denominado enzimas isofuncionales no homólogas . [23] La transferencia horizontal de genes puede propagar estos genes a especies no relacionadas, especialmente bacterias, donde pueden reemplazar genes endógenos de la misma función, lo que conduce al desplazamiento de genes homólogos.

Las enzimas son generalmente proteínas globulares , que actúan solas o en complejos más grandes . La secuencia de los aminoácidos especifica la estructura que a su vez determina la actividad catalítica de la enzima. [24] Aunque la estructura determina la función, una nueva actividad enzimática aún no se puede predecir solo a partir de la estructura. [25] Las estructuras enzimáticas se despliegan ( se desnaturalizan ) cuando se calientan o se exponen a desnaturalizantes químicos y esta alteración de la estructura generalmente causa una pérdida de actividad. [26] La desnaturalización enzimática normalmente está vinculada a temperaturas superiores al nivel normal de una especie; como resultado, las enzimas de las bacterias que viven en entornos volcánicos como las aguas termales son apreciadas por los usuarios industriales por su capacidad de funcionar a altas temperaturas, lo que permite que las reacciones catalizadas por enzimas se realicen a una velocidad muy alta.
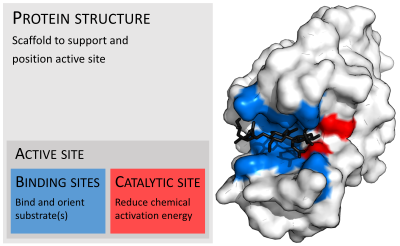
Las enzimas suelen ser mucho más grandes que sus sustratos. Los tamaños varían desde solo 62 residuos de aminoácidos, para el monómero de la 4-oxalocrotonato tautomerasa , [27] hasta más de 2500 residuos en la sintasa de ácidos grasos animales . [28] Solo una pequeña parte de su estructura (alrededor de 2-4 aminoácidos) está directamente involucrada en la catálisis: el sitio catalítico. [29] Este sitio catalítico está ubicado junto a uno o más sitios de unión donde los residuos orientan los sustratos. El sitio catalítico y el sitio de unión juntos componen el sitio activo de la enzima . La mayoría restante de la estructura de la enzima sirve para mantener la orientación y dinámica precisas del sitio activo. [30]
En algunas enzimas, ningún aminoácido está directamente involucrado en la catálisis; en cambio, la enzima contiene sitios para unir y orientar cofactores catalíticos . [30] Las estructuras enzimáticas también pueden contener sitios alostéricos donde la unión de una molécula pequeña causa un cambio conformacional que aumenta o disminuye la actividad. [31]
Existe una pequeña cantidad de catalizadores biológicos basados en ARN , llamados ribozimas , que pueden actuar solos o en combinación con proteínas. El más común de ellos es el ribosoma , que es un complejo de proteínas y componentes catalíticos de ARN. [1] : 2.2
Las enzimas deben unirse a sus sustratos antes de poder catalizar cualquier reacción química. Las enzimas suelen ser muy específicas en cuanto a los sustratos a los que se unen y luego la reacción química que catalizan. La especificidad se logra mediante la unión de bolsas con forma, carga y características hidrofílicas / hidrofóbicas complementarias a los sustratos. Por lo tanto, las enzimas pueden distinguir entre moléculas de sustrato muy similares para ser quimioselectivas , regioselectivas y estereoespecíficas . [32]
Algunas de las enzimas que muestran la mayor especificidad y precisión están involucradas en la copia y expresión del genoma . Algunas de estas enzimas tienen mecanismos de " corrección de pruebas ". Aquí, una enzima como la ADN polimerasa cataliza una reacción en un primer paso y luego verifica que el producto sea correcto en un segundo paso. [33] Este proceso de dos pasos da como resultado tasas de error promedio de menos de 1 error en 100 millones de reacciones en polimerasas de mamíferos de alta fidelidad. [1] : 5.3.1 También se encuentran mecanismos de corrección de pruebas similares en la ARN polimerasa , [34] las aminoacil ARNt sintetasas [35] y los ribosomas . [36]
Por el contrario, algunas enzimas muestran promiscuidad enzimática , tienen una amplia especificidad y actúan sobre una variedad de sustratos fisiológicamente relevantes. Muchas enzimas poseen pequeñas actividades secundarias que surgieron de manera fortuita (es decir, de manera neutral ), lo que puede ser el punto de partida para la selección evolutiva de una nueva función. [37] [38]
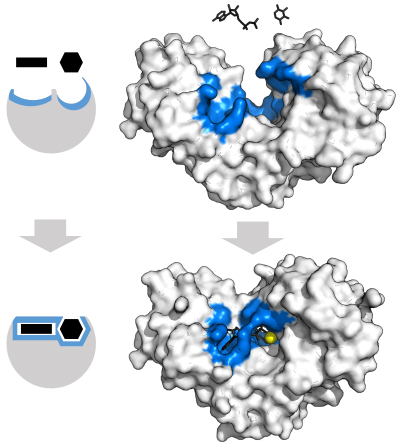
Para explicar la especificidad observada de las enzimas, en 1894 Emil Fischer propuso que tanto la enzima como el sustrato poseen formas geométricas complementarias específicas que encajan exactamente entre sí. [39] Esto se conoce a menudo como el modelo de "cerradura y llave". [1] : 8.3.2 Este modelo temprano explica la especificidad de las enzimas, pero no explica la estabilización del estado de transición que alcanzan las enzimas. [40]
En 1958, Daniel Koshland sugirió una modificación al modelo de llave y cerradura: dado que las enzimas son estructuras bastante flexibles, el sitio activo se remodela continuamente por interacciones con el sustrato a medida que el sustrato interactúa con la enzima. [41] Como resultado, el sustrato no se une simplemente a un sitio activo rígido; las cadenas laterales de aminoácidos que forman el sitio activo se moldean en las posiciones precisas que permiten que la enzima realice su función catalítica. En algunos casos, como las glicosidasas , la molécula de sustrato también cambia de forma ligeramente al entrar en el sitio activo. [42] El sitio activo continúa cambiando hasta que el sustrato está completamente unido, momento en el que se determina la forma final y la distribución de carga. [43] El ajuste inducido puede mejorar la fidelidad del reconocimiento molecular en presencia de competencia y ruido a través del mecanismo de corrección conformacional . [44]
Las enzimas pueden acelerar las reacciones de varias maneras, todas las cuales reducen la energía de activación (ΔG ‡ , energía libre de Gibbs ) [45]
Las enzimas pueden utilizar varios de estos mecanismos simultáneamente. Por ejemplo, las proteasas como la tripsina realizan catálisis covalente utilizando una tríada catalítica , estabilizan la acumulación de carga en los estados de transición utilizando un agujero de oxianión y completan la hidrólisis utilizando un sustrato de agua orientada. [51]
Las enzimas no son estructuras estáticas y rígidas, sino que tienen movimientos dinámicos internos complejos, es decir, movimientos de partes de la estructura de la enzima, como residuos de aminoácidos individuales, grupos de residuos que forman un bucle de proteína o una unidad de estructura secundaria , o incluso un dominio proteico completo . Estos movimientos dan lugar a un conjunto conformacional de estructuras ligeramente diferentes que se interconvierten entre sí en equilibrio . Diferentes estados dentro de este conjunto pueden estar asociados con diferentes aspectos de la función de una enzima. Por ejemplo, diferentes conformaciones de la enzima dihidrofolato reductasa están asociadas con la unión del sustrato, la catálisis, la liberación del cofactor y los pasos de liberación del producto del ciclo catalítico, [52] consistente con la teoría de resonancia catalítica .
La presentación del sustrato es un proceso en el que la enzima se secuestra de su sustrato. Las enzimas pueden secuestrarse en la membrana plasmática lejos de un sustrato en el núcleo o el citosol. O dentro de la membrana, una enzima puede secuestrarse en balsas lipídicas lejos de su sustrato en la región desordenada. Cuando la enzima se libera, se mezcla con su sustrato. Alternativamente, la enzima puede secuestrarse cerca de su sustrato para activarse. Por ejemplo, la enzima puede ser soluble y, al activarse, unirse a un lípido en la membrana plasmática y luego actuar sobre las moléculas en la membrana plasmática.
Los sitios alostéricos son cavidades en la enzima, distintas del sitio activo, que se unen a moléculas en el entorno celular. Estas moléculas luego causan un cambio en la conformación o dinámica de la enzima que se transduce al sitio activo y, por lo tanto, afecta la velocidad de reacción de la enzima. [53] De esta manera, las interacciones alostéricas pueden inhibir o activar las enzimas. Las interacciones alostéricas con metabolitos aguas arriba o aguas abajo en la vía metabólica de una enzima causan una regulación por retroalimentación , alterando la actividad de la enzima de acuerdo con el flujo a través del resto de la vía. [54]

Algunas enzimas no necesitan componentes adicionales para mostrar una actividad completa. Otras requieren que moléculas no proteicas llamadas cofactores estén unidas para la actividad. [55] Los cofactores pueden ser inorgánicos (p. ej., iones metálicos y grupos de hierro-azufre ) u compuestos orgánicos (p. ej., flavina y hemo ). Estos cofactores sirven para muchos propósitos; por ejemplo, los iones metálicos pueden ayudar a estabilizar las especies nucleofílicas dentro del sitio activo. [56] Los cofactores orgánicos pueden ser coenzimas , que se liberan del sitio activo de la enzima durante la reacción, o grupos prostéticos , que están fuertemente unidos a una enzima. Los grupos prostéticos orgánicos pueden estar unidos covalentemente (p. ej., biotina en enzimas como la piruvato carboxilasa ). [57]
Un ejemplo de una enzima que contiene un cofactor es la anhidrasa carbónica , que utiliza un cofactor de zinc unido como parte de su sitio activo. [58] Estos iones o moléculas fuertemente unidos se encuentran generalmente en el sitio activo y están involucrados en la catálisis. [1] : 8.1.1 Por ejemplo, los cofactores flavina y hemo a menudo están involucrados en reacciones redox . [1] : 17
Las enzimas que requieren un cofactor pero no tienen un enlace se denominan apoenzimas o apoproteínas . Una enzima junto con el cofactor o los cofactores necesarios para la actividad se denomina holoenzima (o haloenzima). El término holoenzima también se puede aplicar a las enzimas que contienen múltiples subunidades proteicas, como las ADN polimerasas ; en este caso, la holoenzima es el complejo completo que contiene todas las subunidades necesarias para la actividad. [1] : 8.1.1
Las coenzimas son pequeñas moléculas orgánicas que pueden estar unidas de forma débil o fuerte a una enzima. Las coenzimas transportan grupos químicos de una enzima a otra. [59] Algunos ejemplos son NADH , NADPH y trifosfato de adenosina (ATP). Algunas coenzimas, como el mononucleótido de flavina (FMN), el dinucleótido de flavina y adenina (FAD), el pirofosfato de tiamina (TPP) y el tetrahidrofolato (THF), se derivan de las vitaminas . Estas coenzimas no pueden ser sintetizadas por el cuerpo de novo y los compuestos estrechamente relacionados (vitaminas) deben adquirirse a través de la dieta. Los grupos químicos transportados incluyen:
Dado que las coenzimas se modifican químicamente como consecuencia de la acción enzimática, es útil considerarlas una clase especial de sustratos, o segundos sustratos, que son comunes a muchas enzimas diferentes. Por ejemplo, se sabe que alrededor de 1000 enzimas utilizan la coenzima NADH. [60]
Las coenzimas se regeneran continuamente y sus concentraciones se mantienen a un nivel constante dentro de la célula. Por ejemplo, el NADPH se regenera a través de la vía de las pentosas fosfato y la S -adenosilmetionina por la metionina adenosiltransferasa . Esta regeneración continua significa que se pueden utilizar pequeñas cantidades de coenzimas de forma muy intensiva. Por ejemplo, el cuerpo humano renueva su propio peso en ATP cada día. [61]
Como sucede con todos los catalizadores, las enzimas no alteran la posición del equilibrio químico de la reacción. En presencia de una enzima, la reacción se desarrolla en la misma dirección que sin la enzima, sólo que más rápidamente. [1] : 8.2.3 Por ejemplo, la anhidrasa carbónica cataliza su reacción en cualquier dirección dependiendo de la concentración de sus reactivos: [62]
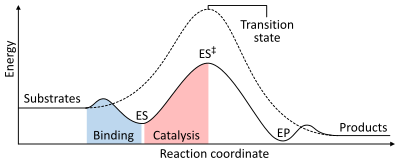
La velocidad de una reacción depende de la energía de activación necesaria para formar el estado de transición que luego se desintegra en productos. Las enzimas aumentan la velocidad de reacción al reducir la energía del estado de transición. En primer lugar, la unión forma un complejo enzima-sustrato (ES) de baja energía. En segundo lugar, la enzima estabiliza el estado de transición de modo que requiere menos energía para alcanzarlo en comparación con la reacción no catalizada (ES ‡ ). Finalmente, el complejo enzima-producto (EP) se disocia para liberar los productos. [1] : 8.3
Las enzimas pueden acoplar dos o más reacciones, de modo que una reacción termodinámicamente favorable puede utilizarse para "impulsar" una reacción termodinámicamente desfavorable, de modo que la energía combinada de los productos sea menor que la de los sustratos. Por ejemplo, la hidrólisis del ATP se utiliza a menudo para impulsar otras reacciones químicas. [63]
La cinética enzimática es la investigación de cómo las enzimas se unen a los sustratos y los convierten en productos. [64] Los datos de velocidad utilizados en los análisis cinéticos se obtienen comúnmente de ensayos enzimáticos . En 1913, Leonor Michaelis y Maud Leonora Menten propusieron una teoría cuantitativa de la cinética enzimática, que se conoce como cinética de Michaelis-Menten . [65] La principal contribución de Michaelis y Menten fue pensar en las reacciones enzimáticas en dos etapas. En la primera, el sustrato se une reversiblemente a la enzima, formando el complejo enzima-sustrato. Esto a veces se llama complejo Michaelis-Menten en su honor. Luego, la enzima cataliza el paso químico en la reacción y libera el producto. Este trabajo fue desarrollado aún más por G. E. Briggs y J. B. S. Haldane , quienes derivaron ecuaciones cinéticas que todavía se usan ampliamente en la actualidad. [66]
Las velocidades de las enzimas dependen de las condiciones de la solución y de la concentración del sustrato . Para encontrar la velocidad máxima de una reacción enzimática, se aumenta la concentración del sustrato hasta que se observa una velocidad constante de formación del producto. Esto se muestra en la curva de saturación de la derecha. La saturación se produce porque, a medida que aumenta la concentración del sustrato, cada vez más enzima libre se convierte en el complejo ES unido al sustrato. A la velocidad máxima de reacción ( Vmax ) de la enzima, todos los sitios activos de la enzima están unidos al sustrato y la cantidad de complejo ES es la misma que la cantidad total de enzima. [1] : 8.4
Vmax es solo uno de varios parámetros cinéticos importantes. La cantidad de sustrato necesaria para alcanzar una velocidad de reacción determinada también es importante. Esto se da por la constante de Michaelis-Menten ( Km ) , que es la concentración de sustrato necesaria para que una enzima alcance la mitad de su velocidad de reacción máxima; generalmente, cada enzima tiene una Km característica para un sustrato determinado. Otra constante útil es kcat , también llamada número de recambio , que es el número de moléculas de sustrato manejadas por un sitio activo por segundo. [1] : 8.4
La eficiencia de una enzima se puede expresar en términos de k cat / K m . Esto también se llama constante de especificidad e incorpora las constantes de velocidad para todos los pasos de la reacción hasta e incluyendo el primer paso irreversible. Debido a que la constante de especificidad refleja tanto la afinidad como la capacidad catalítica, es útil para comparar diferentes enzimas entre sí, o la misma enzima con diferentes sustratos. El máximo teórico para la constante de especificidad se llama límite de difusión y es de aproximadamente 10 8 a 10 9 (M −1 s −1 ). En este punto, cada colisión de la enzima con su sustrato dará como resultado la catálisis, y la velocidad de formación del producto no está limitada por la velocidad de reacción sino por la velocidad de difusión. Las enzimas con esta propiedad se denominan catalíticamente perfectas o cinéticamente perfectas . Ejemplos de tales enzimas son la triosa-fosfato isomerasa , la anhidrasa carbónica , la acetilcolinesterasa , la catalasa , la fumarasa , la β-lactamasa y la superóxido dismutasa . [1] : 8.4.2 El recambio de tales enzimas puede alcanzar varios millones de reacciones por segundo. [1] : 9.2 Pero la mayoría de las enzimas están lejos de ser perfectas: los valores promedio de y son aproximadamente y , respectivamente. [67]
La cinética de Michaelis-Menten se basa en la ley de acción de masas , que se deriva de los supuestos de difusión libre y colisión aleatoria impulsada termodinámicamente. Muchos procesos bioquímicos o celulares se desvían significativamente de estas condiciones, debido al hacinamiento macromolecular y al movimiento molecular restringido. [68] Extensiones más recientes y complejas del modelo intentan corregir estos efectos. [69]
Las velocidades de reacción enzimática se pueden reducir mediante varios tipos de inhibidores enzimáticos. [70] : 73–74
Un inhibidor competitivo y un sustrato no pueden unirse a la enzima al mismo tiempo. [71] A menudo, los inhibidores competitivos se parecen mucho al sustrato real de la enzima. Por ejemplo, el fármaco metotrexato es un inhibidor competitivo de la enzima dihidrofolato reductasa , que cataliza la reducción de dihidrofolato a tetrahidrofolato. [72] La similitud entre las estructuras del dihidrofolato y este fármaco se muestra en la figura adjunta. Este tipo de inhibición se puede superar con una alta concentración de sustrato. En algunos casos, el inhibidor puede unirse a un sitio distinto al sitio de unión del sustrato habitual y ejercer un efecto alostérico para cambiar la forma del sitio de unión habitual. [73]
Un inhibidor no competitivo se une a un sitio distinto al que se une el sustrato. El sustrato sigue uniéndose con su afinidad habitual y, por lo tanto, la Km permanece igual. Sin embargo, el inhibidor reduce la eficiencia catalítica de la enzima, de modo que la Vmax se reduce. A diferencia de la inhibición competitiva, la inhibición no competitiva no se puede superar con una concentración elevada de sustrato. [70] : 76–78
Un inhibidor no competitivo no puede unirse a la enzima libre, sino solo al complejo enzima-sustrato; por lo tanto, este tipo de inhibidores son más eficaces en concentraciones altas de sustrato. En presencia del inhibidor, el complejo enzima-sustrato está inactivo. [70] : 78 Este tipo de inhibición es poco común. [74]
Un inhibidor mixto se une a un sitio alostérico y la unión del sustrato y el inhibidor se afectan mutuamente. La función de la enzima se reduce pero no se elimina cuando se une al inhibidor. Este tipo de inhibidor no sigue la ecuación de Michaelis-Menten. [70] : 76–78
Un inhibidor irreversible inactiva permanentemente la enzima, generalmente formando un enlace covalente con la proteína. [75] La penicilina [76] y la aspirina [77] son medicamentos comunes que actúan de esta manera.
En muchos organismos, los inhibidores pueden actuar como parte de un mecanismo de retroalimentación . Si una enzima produce demasiada cantidad de una sustancia en el organismo, esa sustancia puede actuar como inhibidor de la enzima al comienzo de la vía que la produce, lo que hace que la producción de la sustancia se ralentice o se detenga cuando haya una cantidad suficiente. Esta es una forma de retroalimentación negativa . Las principales vías metabólicas, como el ciclo del ácido cítrico, utilizan este mecanismo. [1] : 17.2.2
Dado que los inhibidores modulan la función de las enzimas, a menudo se utilizan como fármacos. Muchos de estos fármacos son inhibidores competitivos reversibles que se asemejan al sustrato nativo de la enzima, similar al metotrexato mencionado anteriormente; otros ejemplos bien conocidos incluyen las estatinas utilizadas para tratar el colesterol alto , [78] y los inhibidores de la proteasa utilizados para tratar infecciones retrovirales como el VIH . [79] Un ejemplo común de un inhibidor irreversible que se utiliza como fármaco es la aspirina , que inhibe las enzimas COX-1 y COX-2 que producen el mensajero inflamatorio prostaglandina . [77] Otros inhibidores de enzimas son venenos. Por ejemplo, el veneno cianuro es un inhibidor enzimático irreversible que se combina con el cobre y el hierro en el sitio activo de la enzima citocromo c oxidasa y bloquea la respiración celular . [80]
Como las enzimas están formadas por proteínas, sus acciones son sensibles a cambios en muchos factores fisicoquímicos como el pH, la temperatura, la concentración de sustrato, etc.
La siguiente tabla muestra los valores óptimos de pH para varias enzimas. [81]
Las enzimas cumplen una amplia variedad de funciones dentro de los organismos vivos. Son indispensables para la transducción de señales y la regulación celular, a menudo a través de quinasas y fosfatasas . [82] También generan movimiento, con la miosina hidrolizando el trifosfato de adenosina (ATP) para generar la contracción muscular , y también transportan carga alrededor de la célula como parte del citoesqueleto . [83] Otras ATPasas en la membrana celular son bombas de iones involucradas en el transporte activo . Las enzimas también están involucradas en funciones más exóticas, como la luciferasa que genera luz en las luciérnagas . [84] Los virus también pueden contener enzimas para infectar células, como la integrasa y la transcriptasa inversa del VIH , o para la liberación viral de las células, como la neuraminidasa del virus de la influenza . [85]
Una función importante de las enzimas está en los sistemas digestivos de los animales. Las enzimas como las amilasas y las proteasas descomponen moléculas grandes ( almidón o proteínas , respectivamente) en otras más pequeñas, para que puedan ser absorbidas por los intestinos. Las moléculas de almidón, por ejemplo, son demasiado grandes para ser absorbidas desde el intestino, pero las enzimas hidrolizan las cadenas de almidón en moléculas más pequeñas como la maltosa y finalmente la glucosa , que luego pueden absorberse. Diferentes enzimas digieren diferentes sustancias alimenticias. En los rumiantes , que tienen dietas herbívoras , los microorganismos en el intestino producen otra enzima, la celulasa , para descomponer las paredes celulares de celulosa de la fibra vegetal. [86]
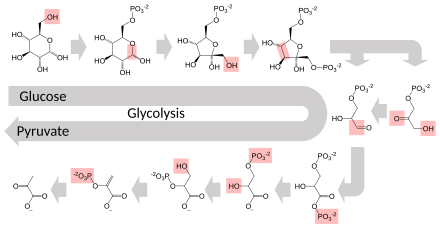
Varias enzimas pueden trabajar juntas en un orden específico, creando vías metabólicas . [1] : 30.1 En una vía metabólica, una enzima toma el producto de otra enzima como sustrato. Después de la reacción catalítica, el producto pasa a otra enzima. A veces más de una enzima puede catalizar la misma reacción en paralelo; esto puede permitir una regulación más compleja: con, por ejemplo, una actividad baja constante proporcionada por una enzima pero una actividad alta inducible de una segunda enzima. [87]
Las enzimas determinan qué pasos se dan en estas vías. Sin enzimas, el metabolismo no seguiría los mismos pasos y no podría regularse para satisfacer las necesidades de la célula. La mayoría de las vías metabólicas centrales se regulan en unos pocos pasos clave, normalmente a través de enzimas cuya actividad implica la hidrólisis del ATP. Debido a que esta reacción libera tanta energía, otras reacciones que son termodinámicamente desfavorables pueden acoplarse a la hidrólisis del ATP, impulsando la serie general de reacciones metabólicas vinculadas. [1] : 30.1
Hay cinco formas principales en que se controla la actividad enzimática en la célula. [1] : 30.1.1
Las enzimas pueden ser activadas o inhibidas por otras moléculas. Por ejemplo, los productos finales de una vía metabólica suelen ser inhibidores de una de las primeras enzimas de la vía (normalmente el primer paso irreversible, llamado paso comprometido), regulando así la cantidad de producto final producido por las vías. Un mecanismo regulador de este tipo se denomina mecanismo de retroalimentación negativa , porque la cantidad de producto final producido está regulada por su propia concentración. [88] : 141–48 El mecanismo de retroalimentación negativa puede ajustar eficazmente la tasa de síntesis de metabolitos intermedios según las demandas de las células. Esto ayuda a la asignación eficaz de materiales y a la economía energética, y evita la fabricación excesiva de productos finales. Al igual que otros dispositivos homeostáticos , el control de la acción enzimática ayuda a mantener un entorno interno estable en los organismos vivos. [88] : 141
Ejemplos de modificación postraduccional incluyen fosforilación , miristoilación y glicosilación . [88] : 149–69 Por ejemplo, en la respuesta a la insulina , la fosforilación de múltiples enzimas, incluyendo la glucógeno sintasa , ayuda a controlar la síntesis o degradación del glucógeno y permite que la célula responda a los cambios en el azúcar en sangre . [89] Otro ejemplo de modificación postraduccional es la escisión de la cadena polipeptídica. La quimotripsina , una proteasa digestiva, se produce en forma inactiva como quimotripsinógeno en el páncreas y se transporta en esta forma al estómago donde se activa. Esto impide que la enzima digiera el páncreas u otros tejidos antes de entrar en el intestino. Este tipo de precursor inactivo de una enzima se conoce como zimógeno [88] : 149–53 o proenzima.
La producción de enzimas ( transcripción y traducción de genes enzimáticos) puede ser mejorada o disminuida por una célula en respuesta a cambios en el entorno celular. Esta forma de regulación genética se llama inducción enzimática . Por ejemplo, las bacterias pueden volverse resistentes a antibióticos como la penicilina porque se inducen enzimas llamadas betalactamasas que hidrolizan el anillo crucial de betalactama dentro de la molécula de penicilina. [90] Otro ejemplo proviene de las enzimas en el hígado llamadas citocromo P450 oxidasas , que son importantes en el metabolismo de los fármacos . La inducción o inhibición de estas enzimas puede causar interacciones farmacológicas . [91] Los niveles de enzimas también pueden regularse cambiando la tasa de degradación enzimática . [1] : 30.1.1 Lo opuesto a la inducción enzimática es la represión enzimática .
Las enzimas pueden compartimentarse, con diferentes vías metabólicas que ocurren en diferentes compartimentos celulares . Por ejemplo, los ácidos grasos son sintetizados por un conjunto de enzimas en el citosol , retículo endoplasmático y Golgi y utilizados por un conjunto diferente de enzimas como fuente de energía en la mitocondria , a través de la β-oxidación . [92] Además, el tráfico de la enzima a diferentes compartimentos puede cambiar el grado de protonación (p. ej., el citoplasma neutro y el lisosoma ácido ) o el estado oxidativo (p. ej., periplasma oxidante o citoplasma reductor ) que a su vez afecta la actividad enzimática. [93] A diferencia de la partición en orgánulos unidos a la membrana, la localización subcelular de la enzima también puede alterarse a través de la polimerización de enzimas en filamentos citoplasmáticos macromoleculares. [94] [95]
En los eucariotas multicelulares , las células de diferentes órganos y tejidos tienen diferentes patrones de expresión genética y, por lo tanto, tienen diferentes conjuntos de enzimas (conocidas como isoenzimas ) disponibles para las reacciones metabólicas. Esto proporciona un mecanismo para regular el metabolismo general del organismo. Por ejemplo, la hexoquinasa , la primera enzima en la vía de la glucólisis , tiene una forma especializada llamada glucoquinasa expresada en el hígado y el páncreas que tiene una menor afinidad por la glucosa pero es más sensible a la concentración de glucosa. [96] Esta enzima está involucrada en la detección del azúcar en sangre y la regulación de la producción de insulina. [97]


Dado que el control estricto de la actividad enzimática es esencial para la homeostasis , cualquier mal funcionamiento (mutación, sobreproducción, subproducción o eliminación) de una sola enzima crítica puede conducir a una enfermedad genética. El mal funcionamiento de un solo tipo de enzima de los miles de tipos presentes en el cuerpo humano puede ser fatal. Un ejemplo de una enfermedad genética fatal debido a la insuficiencia enzimática es la enfermedad de Tay-Sachs , en la que los pacientes carecen de la enzima hexosaminidasa . [98] [99]
Un ejemplo de deficiencia enzimática es el tipo más común de fenilcetonuria . Muchas mutaciones diferentes de un solo aminoácido en la enzima fenilalanina hidroxilasa , que cataliza el primer paso en la degradación de la fenilalanina , dan como resultado la acumulación de fenilalanina y productos relacionados. Algunas mutaciones están en el sitio activo, interrumpiendo directamente la unión y la catálisis, pero muchas están lejos del sitio activo y reducen la actividad al desestabilizar la estructura de la proteína o afectar la oligomerización correcta. [100] [101] Esto puede conducir a una discapacidad intelectual si la enfermedad no se trata. [102] Otro ejemplo es la deficiencia de pseudocolinesterasa , en la que se altera la capacidad del cuerpo para descomponer los fármacos de ésteres de colina. [103] La administración oral de enzimas se puede utilizar para tratar algunas deficiencias enzimáticas funcionales, como la insuficiencia pancreática [104] y la intolerancia a la lactosa . [105]
Otra forma en que el mal funcionamiento de las enzimas puede causar enfermedades son las mutaciones de la línea germinal en los genes que codifican las enzimas reparadoras del ADN . Los defectos en estas enzimas causan cáncer porque las células son menos capaces de reparar las mutaciones en sus genomas . Esto provoca una acumulación lenta de mutaciones y da como resultado el desarrollo de cánceres . Un ejemplo de un síndrome de cáncer hereditario de este tipo es el xeroderma pigmentoso , que causa el desarrollo de cánceres de piel en respuesta a una exposición incluso mínima a la luz ultravioleta . [106] [107]
Al igual que cualquier otra proteína, las enzimas cambian con el tiempo a través de mutaciones y divergencias de secuencia. Dado su papel central en el metabolismo , la evolución de las enzimas juega un papel crítico en la adaptación . Por lo tanto, una pregunta clave es si las enzimas pueden cambiar sus actividades enzimáticas al mismo tiempo y cómo. En general, se acepta que muchas actividades enzimáticas nuevas han evolucionado a través de la duplicación de genes y la mutación de las copias duplicadas, aunque la evolución también puede ocurrir sin duplicación. Un ejemplo de una enzima que ha cambiado su actividad es el antecesor de la metionil aminopeptidasa (MAP) y la creatina amidinohidrolasa ( creatinasa ), que son claramente homólogas pero catalizan reacciones muy diferentes (la MAP elimina la metionina amino-terminal en nuevas proteínas mientras que la creatinasa hidroliza la creatina a sarcosina y urea ). Además, la MAP depende de iones metálicos mientras que la creatinasa no, por lo tanto, esta propiedad también se perdió con el tiempo. [108] Los pequeños cambios de actividad enzimática son extremadamente comunes entre las enzimas. En particular, la especificidad de unión al sustrato (ver arriba) puede cambiar fácil y rápidamente con cambios de un solo aminoácido en sus bolsillos de unión al sustrato. Esto se observa con frecuencia en las principales clases de enzimas, como las quinasas . [109]
En la actualidad, la evolución artificial (in vitro) se utiliza comúnmente para modificar la actividad o especificidad de una enzima para aplicaciones industriales (ver más abajo).
Las enzimas se utilizan en la industria química y en otras aplicaciones industriales cuando se requieren catalizadores extremadamente específicos. Las enzimas en general están limitadas por el número de reacciones que han evolucionado para catalizar y también por su falta de estabilidad en solventes orgánicos y a altas temperaturas. Como consecuencia, la ingeniería de proteínas es un área activa de investigación e implica intentos de crear nuevas enzimas con propiedades novedosas, ya sea mediante diseño racional o evolución in vitro . [110] [111] Estos esfuerzos han comenzado a dar resultados, y ahora se han diseñado algunas enzimas "desde cero" para catalizar reacciones que no ocurren en la naturaleza. [112]