

La estructura primaria de una proteína es la secuencia lineal de aminoácidos en un péptido o proteína . [1] Por convención, la estructura primaria de una proteína se informa comenzando desde el extremo amino terminal (N) hasta el extremo carboxilo terminal (C). La biosíntesis de proteínas la realizan más comúnmente los ribosomas en las células. Los péptidos también se pueden sintetizar en el laboratorio. Las estructuras primarias de las proteínas pueden secuenciarse directamente o inferirse a partir de secuencias de ADN .
Los aminoácidos se polimerizan mediante enlaces peptídicos para formar una larga columna vertebral , con las diferentes cadenas laterales de aminoácidos sobresaliendo a lo largo de ella. En los sistemas biológicos, las proteínas se producen durante la traducción por los ribosomas de una célula . Algunos organismos también pueden producir péptidos cortos mediante síntesis de péptidos no ribosómicos , que a menudo utilizan aminoácidos distintos de los 20 estándar, y pueden ciclarse, modificarse y entrecruzarse.
Los péptidos se pueden sintetizar químicamente mediante diversos métodos de laboratorio. Los métodos químicos suelen sintetizar péptidos en el orden opuesto (comenzando en el extremo C) a la síntesis de proteínas biológicas (comenzando en el extremo N).
La secuencia de proteínas generalmente se indica como una cadena de letras, que enumera los aminoácidos desde el extremo amino terminal hasta el extremo carboxilo terminal. Se puede utilizar un código de tres letras o un código de una sola letra para representar los 20 aminoácidos naturales, así como mezclas o aminoácidos ambiguos (similar a la notación de ácidos nucleicos ). [1] [2] [3]
Los péptidos pueden secuenciarse directamente o inferirse a partir de secuencias de ADN . Actualmente existen grandes bases de datos de secuencias que recopilan secuencias de proteínas conocidas.
En general, los polipéptidos son polímeros no ramificados, por lo que su estructura primaria a menudo puede especificarse mediante la secuencia de aminoácidos a lo largo de su columna vertebral. Sin embargo, las proteínas pueden entrecruzarse, más comúnmente mediante enlaces disulfuro , y la estructura primaria también requiere especificar los átomos de entrecruzamiento, por ejemplo, especificar las cisteínas involucradas en los enlaces disulfuro de la proteína. Otros enlaces cruzados incluyen desmosina .
Los centros quirales de una cadena polipeptídica pueden sufrir racemización . Aunque no cambia la secuencia, sí afecta las propiedades químicas de la secuencia. En particular, los L -aminoácidos que normalmente se encuentran en las proteínas pueden isomerizarse espontáneamente en el átomo para formar D -aminoácidos, que la mayoría de las proteasas no pueden escindir . Además, la prolina puede formar isómeros trans estables en el enlace peptídico.
Además, la proteína puede sufrir una variedad de modificaciones postraduccionales , que se resumen brevemente aquí.
El grupo amino N-terminal de un polipéptido se puede modificar covalentemente, por ejemplo,
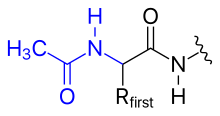
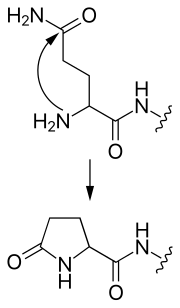
El grupo carboxilato C-terminal de un polipéptido también se puede modificar, por ejemplo,
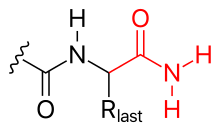
Finalmente, las cadenas laterales peptídicas también pueden modificarse covalentemente, por ejemplo,
La mayoría de las modificaciones polipeptídicas enumeradas anteriormente ocurren postraduccionalmente , es decir, después de que la proteína se ha sintetizado en el ribosoma , lo que ocurre típicamente en el retículo endoplásmico , un orgánulo subcelular de la célula eucariota.
Los químicos han aplicado muchas otras reacciones químicas (p. ej., cianilación) a las proteínas, aunque no se encuentran en los sistemas biológicos.
Además de las enumeradas anteriormente, la modificación más importante de la estructura primaria es la escisión de péptidos (mediante hidrólisis química o mediante proteasas ). Las proteínas suelen sintetizarse en forma de precursor inactivo; normalmente, un segmento N-terminal o C-terminal bloquea el sitio activo de la proteína, inhibiendo su función. La proteína se activa escindiendo el péptido inhibidor.
Algunas proteínas incluso tienen el poder de escindirse a sí mismas. Normalmente, el grupo hidroxilo de una serina (rara vez, treonina) o el grupo tiol de un residuo de cisteína atacará el carbono carbonilo del enlace peptídico anterior, formando un intermedio con enlace tetraédrico [clasificado como hidroxioxazolidina (Ser/Thr) o hidroxitiazolidina ( Cys) intermedio]. Este intermedio tiende a revertir a la forma amida, expulsando al grupo atacante, ya que la forma amida suele verse favorecida por la energía libre (presumiblemente debido a la fuerte estabilización por resonancia del grupo peptídico). Sin embargo, interacciones moleculares adicionales pueden hacer que la forma amida sea menos estable; en cambio, el grupo amino se expulsa, lo que da como resultado un enlace éster (Ser/Thr) o tioéster (Cys) en lugar del enlace peptídico. Esta reacción química se llama desplazamiento de NO acilo.
El enlace éster/tioéster se puede resolver de varias maneras:
La compresión de secuencias de aminoácidos es una tarea comparativamente desafiante. Los compresores de secuencias de aminoácidos especializados existentes son bajos en comparación con los compresores de secuencias de ADN, principalmente debido a las características de los datos. Por ejemplo, modelar inversiones es más difícil debido a la pérdida de información inversa (desde aminoácidos hasta secuencia de ADN). El compresor de datos sin pérdidas actual que proporciona una mayor compresión es el AC2. [5] AC2 mezcla varios modelos de contexto utilizando redes neuronales y codifica los datos mediante codificación aritmética.
La propuesta de que las proteínas eran cadenas lineales de α-aminoácidos fue hecha casi simultáneamente por dos científicos en la misma conferencia de 1902, la 74ª reunión de la Sociedad de Científicos y Médicos Alemanes, celebrada en Karlsbad. Franz Hofmeister hizo la propuesta por la mañana basándose en sus observaciones de la reacción del biuret en las proteínas. A Hofmeister le siguió unas horas más tarde Emil Fischer , quien había acumulado una gran cantidad de detalles químicos que respaldaban el modelo del enlace peptídico. Para completar, la propuesta de que las proteínas contienen enlaces amida fue hecha ya en 1882 por el químico francés E. Grimaux. [6]
A pesar de estos datos y de la evidencia posterior de que las proteínas digeridas proteolíticamente sólo producían oligopéptidos, la idea de que las proteínas eran polímeros de aminoácidos lineales y no ramificados no fue aceptada de inmediato. Algunos científicos muy respetados, como William Astbury, dudaban de que los enlaces covalentes fueran lo suficientemente fuertes como para mantener unidas moléculas tan largas; temían que las agitaciones térmicas hicieran pedazos moléculas tan largas. Hermann Staudinger enfrentó prejuicios similares en la década de 1920 cuando argumentó que el caucho estaba compuesto de macromoléculas . [6]
Así, surgieron varias hipótesis alternativas. La hipótesis de la proteína coloidal afirmaba que las proteínas eran conjuntos coloidales de moléculas más pequeñas. Esta hipótesis fue refutada en la década de 1920 por mediciones de ultracentrifugación realizadas por Theodor Svedberg que mostraron que las proteínas tenían un peso molecular reproducible y bien definido y por mediciones electroforéticas de Arne Tiselius que indicaron que las proteínas eran moléculas individuales. Una segunda hipótesis, la hipótesis del ciclol avanzada por Dorothy Wrinch , propuso que el polipéptido lineal experimentó un reordenamiento químico de ciclol C=O + HN C(OH)-N que entrecruzaba sus grupos amida principales, formando un tejido bidimensional . Varios investigadores propusieron otras estructuras primarias de proteínas, como el modelo de dicetopiperazina de Emil Abderhalden y el modelo de pirrol/piperidina de Troensegaard en 1942. Aunque nunca se les dio mucho crédito, estos modelos alternativos fueron finalmente refutados cuando Frederick Sanger secuenció con éxito la insulina [ cuando ? ] y por la determinación cristalográfica de mioglobina y hemoglobina por Max Perutz y John Kendrew [ ¿cuándo? ] .
Se puede decir que cualquier heteropolímero de cadena lineal tiene una "estructura primaria" por analogía con el uso del término para proteínas, pero este uso es raro en comparación con el uso extremadamente común en referencia a proteínas. En el ARN , que también tiene una estructura secundaria extensa , la cadena lineal de bases generalmente se denomina simplemente "secuencia", como ocurre en el ADN (que generalmente forma una doble hélice lineal con poca estructura secundaria). También se puede considerar que otros polímeros biológicos, como los polisacáridos, tienen una estructura primaria, aunque el uso no es estándar.
La estructura primaria de un polímero biológico determina en gran medida la forma tridimensional ( estructura terciaria ). La secuencia de proteínas se puede utilizar para predecir características locales , como segmentos de estructura secundaria o regiones transmembrana. Sin embargo, la complejidad del plegamiento de proteínas impide actualmente predecir la estructura terciaria de una proteína únicamente a partir de su secuencia. Conocer la estructura de una secuencia homóloga similar (por ejemplo, un miembro de la misma familia de proteínas ) permite una predicción muy precisa de la estructura terciaria mediante modelos de homología . Si se dispone de la secuencia proteica completa, es posible estimar sus propiedades biofísicas generales , como su punto isoeléctrico .
Las familias de secuencias a menudo se determinan mediante agrupación de secuencias , y los proyectos de genómica estructural tienen como objetivo producir un conjunto de estructuras representativas para cubrir el espacio de secuencias de posibles secuencias no redundantes.
{{cite journal}}: Mantenimiento CS1: varios nombres: lista de autores ( enlace )