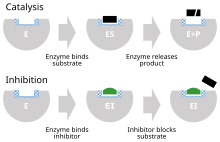
Un inhibidor enzimático es una molécula que se une a una enzima y bloquea su actividad . Las enzimas son proteínas que aceleran las reacciones químicas necesarias para la vida , en las que las moléculas de sustrato se convierten en productos . [1] Una enzima facilita una reacción química específica uniendo el sustrato a su sitio activo , un área especializada de la enzima que acelera el paso más difícil de la reacción .
Un inhibidor enzimático detiene ("inhibe") este proceso, ya sea uniéndose al sitio activo de la enzima (evitando así que el sustrato se una) o uniéndose a otro sitio de la enzima de modo que se bloquee la catálisis de la reacción por parte de la enzima . Los inhibidores enzimáticos pueden unirse de forma reversible o irreversible. Los inhibidores irreversibles forman un enlace químico con la enzima de modo que la enzima se inhibe hasta que se rompe el enlace químico. Por el contrario, los inhibidores reversibles se unen de forma no covalente y pueden abandonar espontáneamente la enzima, permitiendo que la enzima reanude su función. Los inhibidores reversibles producen diferentes tipos de inhibición dependiendo de si se unen a la enzima, al complejo enzima-sustrato o a ambos.
Los inhibidores de enzimas desempeñan un papel importante en todas las células, ya que generalmente son específicos de una enzima cada uno y sirven para controlar la actividad de esa enzima. Por ejemplo, las enzimas en una vía metabólica pueden ser inhibidas por moléculas producidas más adelante en la vía, lo que reduce la producción de moléculas que ya no son necesarias. Este tipo de retroalimentación negativa es una forma importante de mantener el equilibrio en una célula . [2] Los inhibidores de enzimas también controlan enzimas esenciales como las proteasas o nucleasas que, si no se controlan, pueden dañar una célula. Muchos venenos producidos por animales o plantas son inhibidores de enzimas que bloquean la actividad de enzimas cruciales en presas o depredadores .
Muchas moléculas de fármacos son inhibidores de enzimas que inhiben una enzima humana aberrante o una enzima crítica para la supervivencia de un patógeno como un virus , una bacteria o un parásito . Los ejemplos incluyen el metotrexato (utilizado en quimioterapia y en el tratamiento de la artritis reumática ) y los inhibidores de la proteasa utilizados para tratar el VIH/SIDA . Dado que los inhibidores antipatógenos generalmente se dirigen a una sola enzima, dichos fármacos son muy específicos y generalmente producen pocos efectos secundarios en los seres humanos, siempre que no se encuentre ninguna enzima análoga en los seres humanos. (Este suele ser el caso, ya que dichos patógenos y los humanos están genéticamente distantes ). Los inhibidores de enzimas medicinales a menudo tienen constantes de disociación bajas , lo que significa que solo se requiere una cantidad mínima del inhibidor para inhibir la enzima. Una concentración baja del inhibidor enzimático reduce el riesgo de daño hepático y renal y otras reacciones adversas a los medicamentos en humanos. De ahí que el descubrimiento y perfeccionamiento de inhibidores enzimáticos sea un área activa de investigación en bioquímica y farmacología .
Los inhibidores enzimáticos son un conjunto químicamente diverso de sustancias que varían en tamaño desde pequeñas moléculas orgánicas hasta proteínas macromoleculares .
Los inhibidores de moléculas pequeñas incluyen metabolitos primarios esenciales que inhiben las enzimas anteriores que producen esos metabolitos. Esto proporciona un circuito de retroalimentación negativa que previene la sobreproducción de metabolitos y, por lo tanto, mantiene la homeostasis celular (condiciones internas estables). [3] [2] Los inhibidores de enzimas de molécula pequeña también incluyen metabolitos secundarios , que no son esenciales para el organismo que los produce, pero proporcionan al organismo una ventaja evolutiva, ya que pueden usarse para repeler depredadores u organismos competidores o inmovilizar a sus presas. . [4] Además, muchos medicamentos son inhibidores de enzimas de molécula pequeña que se dirigen a enzimas modificadoras de la enfermedad en el paciente [1] : 5 o enzimas en patógenos que son necesarias para el crecimiento y la reproducción del patógeno. [5]
Además de las moléculas pequeñas, algunas proteínas actúan como inhibidores de enzimas. El ejemplo más destacado son las serpinas ( inhibidores de la serina proteasa ) , que son producidas por los animales para proteger contra la activación inadecuada de enzimas y por las plantas para prevenir la depredación . [6] Otra clase de proteínas inhibidoras son los inhibidores de ribonucleasas , que se unen a las ribonucleasas en una de las interacciones proteína-proteína más estrechas que se conocen . [7] Un caso especial de inhibidores de enzimas proteicas son los zimógenos que contienen un péptido N-terminal autoinhibidor que se une al sitio activo de la enzima que bloquea intramolecularmente su actividad como mecanismo protector contra la catálisis incontrolada. El péptido N-terminal es escindido (dividido) del precursor de la enzima zimógeno por otra enzima para liberar una enzima activa. [8]
El sitio de unión de los inhibidores a las enzimas suele ser el mismo sitio que se une al sustrato de la enzima. Estos inhibidores del sitio activo se conocen como inhibidores ortostéricos (orientación "regular"). [9] El mecanismo de inhibición ortostérica es simplemente evitar la unión del sustrato a la enzima mediante competencia directa, lo que a su vez evita que la enzima catalice la conversión de sustratos en productos. Alternativamente, el inhibidor puede unirse a un sitio alejado del sitio activo de la enzima. Estos se conocen como inhibidores alostéricos (orientación "alternativa"). [9] Los mecanismos de inhibición alostérica son variados e incluyen cambiar la conformación (forma) de la enzima de modo que ya no pueda unirse al sustrato ( cinéticamente indistinguible de la inhibición ortostérica competitiva) [10] o, alternativamente, estabilizar la unión del sustrato a la enzima pero bloquear la enzima en una conformación que ya no es catalíticamente activa. [11]
Los inhibidores reversibles se unen a enzimas con interacciones no covalentes, como enlaces de hidrógeno , interacciones hidrófobas y enlaces iónicos . [12] Múltiples enlaces débiles entre el inhibidor y el sitio activo de la enzima se combinan para producir una unión fuerte y específica.
A diferencia de los inhibidores irreversibles, los inhibidores reversibles generalmente no sufren reacciones químicas cuando se unen a la enzima y pueden eliminarse fácilmente mediante dilución o diálisis . Un caso especial son los inhibidores covalentes reversibles que forman un enlace químico con la enzima, pero el enlace se puede romper para que la inhibición sea completamente reversible. [13]
Los inhibidores reversibles generalmente se clasifican en cuatro tipos, según los introdujo Cleland en 1963. [14] Se clasifican según el efecto del inhibidor sobre la Vmax ( velocidad máxima de reacción catalizada por la enzima) y Km (la concentración de sustrato). lo que da como resultado la mitad de la actividad enzimática máxima) a medida que se varía la concentración del sustrato de la enzima. [15] [16]
En la inhibición competitiva, el sustrato y el inhibidor no pueden unirse a la enzima al mismo tiempo. [17] : 134 Esto generalmente se debe a que el inhibidor tiene afinidad por el sitio activo de una enzima al que también se une el sustrato; el sustrato y el inhibidor compiten por el acceso al sitio activo de la enzima. Este tipo de inhibición puede superarse mediante concentraciones suficientemente altas de sustrato ( Vmax permanece constante), es decir, superando al inhibidor. [17] : 134–135 Sin embargo, la K m aparente aumentará a medida que se necesita una mayor concentración del sustrato para alcanzar el punto K m , o la mitad de la V máx . Los inhibidores competitivos suelen tener una estructura similar al sustrato real (consulte, por ejemplo, la figura "metotrexato versus folato" en la sección "Medicamentos"). [17] : 134
En la inhibición no competitiva, el inhibidor se une sólo al complejo enzima-sustrato. [17] : 139 Este tipo de inhibición hace que Vmax disminuya (la velocidad máxima disminuye como resultado de la eliminación del complejo activado) y Km disminuya (debido a una mejor eficiencia de unión como resultado del principio de Le Chatelier y la eliminación efectiva del El complejo ES disminuye así la Km , lo que indica una mayor afinidad de unión). [18] La inhibición no competitiva es rara. [17] : 139 [19]
En la inhibición no competitiva, la unión del inhibidor a la enzima reduce su actividad pero no afecta la unión del sustrato. [16] Este tipo de inhibidor se une con igual afinidad a la enzima libre que al complejo enzima-sustrato. Se puede considerar que tiene la capacidad de inhibidores competitivos y no competitivos, pero sin preferencia por ninguno de los dos tipos. Como resultado, el grado de inhibición depende únicamente de la concentración del inhibidor. V max disminuirá debido a la incapacidad de que la reacción se desarrolle de manera tan eficiente, pero K m seguirá siendo el mismo ya que la unión real del sustrato, por definición, seguirá funcionando correctamente. [20]
En la inhibición mixta, el inhibidor puede unirse a la enzima independientemente de que el sustrato ya se haya unido o no. Por tanto, la inhibición mixta es una combinación de inhibición competitiva y no competitiva. [16] Además, la afinidad del inhibidor por la enzima libre y el complejo enzima-sustrato puede diferir. [17] : 136–139 Al aumentar las concentraciones de sustrato [S], este tipo de inhibición puede reducirse (debido a la contribución competitiva), pero no superarse por completo (debido al componente no competitivo). [21] : 381–382 Aunque es posible que los inhibidores de tipo mixto se unan en el sitio activo, este tipo de inhibición generalmente resulta de un efecto alostérico donde el inhibidor se une a un sitio diferente en una enzima. La unión del inhibidor a este sitio alostérico cambia la conformación (es decir, la estructura terciaria o forma tridimensional) de la enzima de modo que se reduce la afinidad del sustrato por el sitio activo. [22]
Estos cuatro tipos de inhibición también se pueden distinguir por el efecto del aumento de la concentración del sustrato [S] sobre el grado de inhibición causado por una determinada cantidad de inhibidor. Para la inhibición competitiva, el grado de inhibición se reduce aumentando [S], para la inhibición no competitiva el grado de inhibición no cambia y para la inhibición no competitiva (también llamada anticompetitiva) el grado de inhibición aumenta con [S]. [23]
La inhibición reversible puede describirse cuantitativamente en términos de la unión del inhibidor a la enzima y al complejo enzima-sustrato, y sus efectos sobre las constantes cinéticas de la enzima. [24] : 6 En el esquema clásico de Michaelis-Menten (que se muestra en el diagrama "esquemático del mecanismo de inhibición"), una enzima (E) se une a su sustrato (S) para formar el complejo enzima-sustrato ES. Tras la catálisis, este complejo se descompone para liberar el producto P y la enzima libre. [24] : 55 El inhibidor (I) puede unirse a E o ES con las constantes de disociación Ki o Ki ' , respectivamente. [24] : 87
Cuando una enzima tiene múltiples sustratos, los inhibidores pueden mostrar diferentes tipos de inhibición según el sustrato que se considere. Esto se debe a que el sitio activo contiene dos sitios de unión diferentes dentro del sitio activo, uno para cada sustrato. Por ejemplo, un inhibidor podría competir con el sustrato A por el primer sitio de unión, pero ser un inhibidor no competitivo con respecto al sustrato B en el segundo sitio de unión. [26]
Los inhibidores enzimáticos tradicionalmente reversibles se han clasificado en competitivos, no competitivos o no competitivos, según sus efectos sobre el Km y el Vmax . [14] Estos tres tipos de inhibición resultan respectivamente de la unión del inhibidor solo a la enzima E en ausencia del sustrato S, al complejo enzima-sustrato ES, o a ambos. La división de estas clases surge de un problema en su derivación y resulta en la necesidad de utilizar dos constantes de vinculación diferentes para un evento de vinculación. [27] Se supone además que la unión del inhibidor a la enzima da como resultado una inhibición del 100% y no se considera la posibilidad de una inhibición parcial. [27] La forma común del término inhibidor también oscurece la relación entre la unión del inhibidor a la enzima y su relación con cualquier otro término de unión, ya sea la ecuación de Michaelis-Menten o una curva dosis-respuesta asociada con la unión del receptor de ligando. Para demostrar la relación se puede realizar la siguiente reordenación: [28]
Este reordenamiento demuestra que, de manera similar a la ecuación de Michaelis-Menten, la velocidad máxima de reacción depende de la proporción de la población de enzimas que interactúa con su sustrato.
fracción de la población de enzimas unida por el sustrato
fracción de la población de enzimas unida por el inhibidor
el efecto del inhibidor es el resultado del porcentaje de la población de enzimas que interactúa con el inhibidor. El único problema con esta ecuación en su forma actual es que supone una inhibición absoluta de la enzima con la unión del inhibidor, cuando en realidad puede haber una amplia gama de efectos desde una inhibición del 100% de la rotación del sustrato hasta ninguna inhibición. Para tener en cuenta esto , la ecuación se puede modificar fácilmente para permitir diferentes grados de inhibición al incluir un término delta Vmax . [29] : 361
o
Este término puede luego definir la actividad enzimática residual presente cuando el inhibidor interactúa con enzimas individuales en la población. Sin embargo, la inclusión de este término tiene el valor agregado de permitir la posibilidad de activación si el término Vmax secundario resulta ser mayor que el término inicial. Para tener en cuenta también la posibilidad de activación, la notación se puede reescribir reemplazando el inhibidor "I" con un término modificador (estimulador o inhibidor) indicado aquí como "X". [28] : ecuación 13
Si bien esta terminología resulta en una forma simplificada de abordar los efectos cinéticos relacionados con la velocidad máxima de la ecuación de Michaelis-Menten, resalta problemas potenciales con el término utilizado para describir los efectos relacionados con el Km . La Km relacionada con la afinidad de la enzima por el sustrato debería, en la mayoría de los casos , estar relacionada con cambios potenciales en el sitio de unión de la enzima que resultarían directamente de las interacciones del inhibidor de la enzima. Como tal, un término similar al término delta V max propuesto anteriormente para modular V max debería ser apropiado en la mayoría de las situaciones: [28] : ecuación 14
![Gráficos 2D de concentración de 1/[S] (eje x) y 1/V (eje y) que demuestran que a medida que cambia la concentración de inhibidor, las líneas de inhibidor competitivo se cruzan en un solo punto en el eje y, los inhibidores no competitivos se cruzan en el eje x, y los inhibidores mixtos intersectan un punto que no está en ninguno de los ejes](http://upload.wikimedia.org/wikipedia/commons/thumb/b/ba/Inhibition_diagrams-1-.png/220px-Inhibition_diagrams-1-.png)
Un inhibidor enzimático se caracteriza por su constante de disociación Ki , la concentración a la que el inhibidor ocupa la mitad de la enzima. En la inhibición no competitiva, el inhibidor también puede unirse al complejo enzima-sustrato, y la presencia de un sustrato unido puede cambiar la afinidad del inhibidor por la enzima, dando como resultado una segunda constante de disociación Ki ' . Por tanto, Ki y Ki ' son las constantes de disociación del inhibidor para la enzima y para el complejo enzima-sustrato, respectivamente. [30] : Glosario La constante Ki del inhibidor de enzima se puede medir directamente mediante varios métodos; Un método especialmente preciso es la calorimetría de titulación isotérmica , en la que el inhibidor se titula en una solución de enzima y se mide el calor liberado o absorbido. [31] Sin embargo, la otra constante de disociación Ki ' es difícil de medir directamente, ya que el complejo enzima-sustrato tiene una vida corta y sufre una reacción química para formar el producto. Por lo tanto, K i ' generalmente se mide indirectamente, observando la actividad enzimática bajo diversas concentraciones de sustrato e inhibidor, y ajustando los datos mediante regresión no lineal [32] a una ecuación de Michaelis-Menten modificada . [21]
donde los factores modificadores α y α' están definidos por la concentración del inhibidor y sus dos constantes de disociación
Por tanto, en presencia del inhibidor, las Km y Vmax efectivas de la enzima se convierten en ( α / α') Km y (1/α') Vmax , respectivamente. Sin embargo, la ecuación de Michaelis-Menten modificada supone que la unión del inhibidor a la enzima ha alcanzado el equilibrio, lo que puede ser un proceso muy lento para inhibidores con constantes de disociación subnanomolares. En estos casos, la inhibición se vuelve efectivamente irreversible, por lo que es más práctico tratar estos inhibidores de unión fuerte como irreversibles (ver más abajo).
Los efectos de diferentes tipos de inhibidores enzimáticos reversibles sobre la actividad enzimática se pueden visualizar utilizando representaciones gráficas de la ecuación de Michaelis-Menten, como los diagramas de Lineweaver-Burk , Eadie-Hofstee o Hanes-Woolf . [17] : 140–144 Los tres diagramas de Lineweaver-Burk que se muestran en la figura de los diagramas de Lineweaver-Burk proporcionan una ilustración . En el diagrama superior, las líneas de inhibición competitiva se cruzan en el eje y , lo que ilustra que dichos inhibidores no afectan la V máx . En el diagrama inferior, las líneas de inhibición no competitiva se cruzan en el eje x , lo que muestra que estos inhibidores no afectan la Km . Sin embargo, dado que puede resultar difícil estimar Ki y Ki ' con precisión a partir de dichos gráficos, [33] es aconsejable estimar estas constantes utilizando métodos de regresión no lineal más confiables . [33]
El mecanismo de inhibición parcialmente competitiva es similar al de la no competitiva, excepto que el complejo EIS tiene actividad catalítica, que puede ser menor o incluso mayor (activación parcialmente competitiva) que la del complejo enzima-sustrato (ES). Esta inhibición normalmente muestra un Vmax más bajo , pero un valor de Km no afectado . [18]
La inhibición por sustrato o producto es cuando un sustrato o producto de una enzima también actúa como inhibidor. Esta inhibición puede seguir patrones competitivos, no competitivos o mixtos. En la inhibición del sustrato hay una disminución progresiva de la actividad a altas concentraciones de sustrato, potencialmente debido a una enzima que tiene dos sitios de unión al sustrato en competencia. Con un sustrato bajo, se ocupa el sitio de alta afinidad y se sigue una cinética normal. Sin embargo, en concentraciones más altas, el segundo sitio inhibidor queda ocupado, inhibiendo la enzima. [34] La inhibición del producto (ya sea el producto de la propia enzima o un producto de una enzima aguas abajo en su ruta metabólica) es a menudo una característica reguladora en el metabolismo y puede ser una forma de retroalimentación negativa . [2]
La inhibición lenta y estricta ocurre cuando el complejo inicial enzima-inhibidor EI sufre una isomería conformacional (un cambio de forma) a un segundo complejo más estrechamente unido, EI*, pero el proceso de inhibición general es reversible. Esto se manifiesta como una inhibición enzimática que aumenta lentamente. En estas condiciones, la cinética tradicional de Michaelis-Menten da un valor falso para Ki , que depende del tiempo. El verdadero valor de Ki se puede obtener mediante un análisis más complejo de las constantes de velocidad de encendido ( k on ) y off ( k off ) para la asociación del inhibidor con una cinética similar a la de la inhibición irreversible . [17] : 168
Los inhibidores análogos de múltiples sustratos son inhibidores selectivos de alta afinidad que se pueden preparar para enzimas que catalizan reacciones con más de un sustrato capturando la energía de unión de cada uno de esos sustratos en una molécula. [35] [36] Por ejemplo, en las reacciones de transferencia de formilo de la biosíntesis de purina , se preparó sintéticamente un potente inhibidor del aducto de múltiples sustratos (MAI) para la glicinamida ribonucleótido (GAR) TFasa uniendo análogos del sustrato GAR y el N-10. -formil tetrahidrofolato cofactor juntos para producir dideazafolato de ribonucleótido de tioglicinamida (TGDDF), [37] o enzimáticamente a partir del sustrato GAR natural para producir GDDF. [38] Aquí la constante de disociación subnanomolar (KD) de TGDDF fue mayor de lo previsto, presumiblemente debido a las ventajas entrópicas obtenidas y/o a las interacciones positivas adquiridas a través de los átomos que unen los componentes. También se ha observado que los MAI se producen en las células mediante reacciones de profármacos como la isoniazida [39] o ligandos inhibidores de enzimas (por ejemplo, PTC124 ) [40] con cofactores celulares como el dinucleótido de nicotinamida y adenina (NADH) y el trifosfato de adenosina ( ATP) respectivamente. [41]
Como las enzimas han evolucionado para unirse estrechamente a sus sustratos, y la mayoría de los inhibidores reversibles se unen en el sitio activo de las enzimas, no sorprende que algunos de estos inhibidores sean sorprendentemente similares en estructura a los sustratos de sus objetivos. Los inhibidores de la dihidrofolato reductasa (DHFR) son ejemplos destacados. [42] Otros ejemplos de estos sustratos imitadores son los inhibidores de la proteasa , una clase terapéuticamente eficaz de fármacos antirretrovirales utilizados para tratar el VIH/SIDA . [43] [44] La estructura del ritonavir , un inhibidor de la proteasa peptidomimético (imitador de péptidos) que contiene tres enlaces peptídicos , como se muestra en la figura de "inhibición competitiva" anterior. Como este fármaco se parece al péptido que es el sustrato de la proteasa del VIH, compite con el sustrato en el sitio activo de la enzima. [45]
Los inhibidores enzimáticos suelen estar diseñados para imitar el estado de transición o el intermedio de una reacción catalizada por una enzima. [46] Esto asegura que el inhibidor aproveche el efecto estabilizador del estado de transición de la enzima, lo que resulta en una mejor afinidad de unión (menor Ki ) que los diseños basados en sustrato. Un ejemplo de tal inhibidor del estado de transición es el fármaco antiviral oseltamivir ; este fármaco imita la naturaleza plana del ion oxonio del anillo en la reacción de la enzima viral neuraminidasa . [47]
Sin embargo, no todos los inhibidores se basan en las estructuras de los sustratos. Por ejemplo, la estructura de otro inhibidor de la proteasa del VIH, tipranavir , no se basa en un péptido y no tiene ninguna similitud estructural obvia con un sustrato proteico. Estos inhibidores no peptídicos pueden ser más estables que los inhibidores que contienen enlaces peptídicos, porque no serán sustratos para las peptidasas y es menos probable que se degraden. [48]
En el diseño de fármacos es importante considerar las concentraciones de sustratos a los que están expuestas las enzimas diana. Por ejemplo, algunos inhibidores de la proteína quinasa tienen estructuras químicas similares al ATP, uno de los sustratos de estas enzimas. [49] Sin embargo, los fármacos que son simples inhibidores competitivos tendrán que competir con las altas concentraciones de ATP en la célula. Las proteínas quinasas también pueden inhibirse por competencia en los sitios de unión donde las quinasas interactúan con sus proteínas sustrato, y la mayoría de las proteínas están presentes dentro de las células en concentraciones mucho más bajas que la concentración de ATP. Como consecuencia, si dos inhibidores de la proteína quinasa se unen en el sitio activo con afinidad similar, pero solo uno tiene que competir con el ATP, entonces el inhibidor competitivo en el sitio de unión a la proteína inhibirá la enzima de manera más efectiva. [50]
Los inhibidores irreversibles se unen covalentemente a una enzima y, por lo tanto, este tipo de inhibición no puede revertirse fácilmente. [51] Los inhibidores irreversibles a menudo contienen grupos funcionales reactivos como mostazas nitrogenadas , aldehídos , haloalcanos , alquenos , aceptores de Michael , fenilsulfonatos o fluorofosfonatos . [52] Estos grupos electrófilos reaccionan con las cadenas laterales de aminoácidos para formar aductos covalentes . [51] Los residuos modificados son aquellos con cadenas laterales que contienen nucleófilos como grupos hidroxilo o sulfhidrilo ; estos incluyen los aminoácidos serina (que reacciona con el DFP , consulte el diagrama de "reacción del DFP") y también cisteína , treonina o tirosina . [53]
La inhibición irreversible es diferente de la inactivación irreversible de enzimas. [54] Los inhibidores irreversibles generalmente son específicos de una clase de enzima y no inactivan todas las proteínas; no funcionan destruyendo la estructura de las proteínas sino alterando específicamente el sitio activo de su objetivo. Por ejemplo, los extremos de pH o temperatura suelen provocar la desnaturalización de toda la estructura de las proteínas, pero se trata de un efecto no específico. De manera similar, algunos tratamientos químicos no específicos destruyen la estructura de las proteínas: por ejemplo, calentarlas en ácido clorhídrico concentrado hidrolizará los enlaces peptídicos que mantienen unidas las proteínas, liberando aminoácidos libres. [55]
Los inhibidores irreversibles muestran una inhibición dependiente del tiempo y, por lo tanto, su potencia no puede caracterizarse por un valor de IC50 . Esto se debe a que la cantidad de enzima activa en una concentración determinada de inhibidor irreversible será diferente dependiendo de cuánto tiempo se preincuba el inhibidor con la enzima. En su lugar, se utilizan valores de kobs/[I], [ 56 ] donde kobs es la tasa de inactivación de pseudoprimer orden observada (obtenida trazando el registro del % de actividad frente al tiempo) y [ I ] es la concentración de inhibidor. El parámetro k obs /[ I ] es válido siempre que el inhibidor no sature la unión con la enzima (en cuyo caso k obs = k inact ) donde k inact es la tasa de inactivación.
Los inhibidores irreversibles primero forman un complejo no covalente reversible con la enzima (EI o ESI). Posteriormente, se produce una reacción química entre la enzima y el inhibidor para producir el "complejo sin salida" EI* modificado covalentemente (un complejo covalente irreversible). La velocidad a la que se forma EI* se denomina tasa de inactivación o k inact . [13] Dado que la formación de EI puede competir con ES, la unión de inhibidores irreversibles se puede prevenir mediante la competencia con un sustrato o con un segundo inhibidor reversible. Este efecto protector es una buena evidencia de una reacción específica del inhibidor irreversible con el sitio activo.
Los pasos de unión e inactivación de esta reacción se investigan incubando la enzima con inhibidor y analizando la cantidad de actividad restante con el tiempo. La actividad disminuirá de manera dependiente del tiempo, generalmente después de una caída exponencial . Al ajustar estos datos a una ecuación de velocidad se obtiene la velocidad de inactivación a esta concentración de inhibidor. Esto se hace en varias concentraciones diferentes de inhibidor. Si se trata de un complejo EI reversible, la tasa de inactivación será saturable y al ajustar esta curva se obtendrá k inact y Ki . [57]
Otro método que se utiliza mucho en estos análisis es la espectrometría de masas . Aquí, la medición precisa de la masa de la enzima nativa no modificada y la enzima inactivada da el aumento de masa causado por la reacción con el inhibidor y muestra la estequiometría de la reacción. [58] Esto generalmente se hace usando un espectrómetro de masas MALDI-TOF . [59] En una técnica complementaria, la identificación de masas peptídicas implica la digestión de la proteína nativa y modificada con una proteasa como la tripsina . Esto producirá un conjunto de péptidos que se pueden analizar utilizando un espectrómetro de masas. El péptido que cambie de masa tras la reacción con el inhibidor será el que contenga el sitio de modificación. [60]
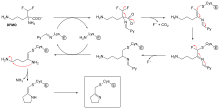
No todos los inhibidores irreversibles forman aductos covalentes con sus dianas enzimáticas. Algunos inhibidores reversibles se unen tan estrechamente a su enzima objetivo que son esencialmente irreversibles. Estos inhibidores de unión estrecha pueden mostrar una cinética similar a la de los inhibidores covalentes irreversibles. En estos casos, algunos de estos inhibidores se unen rápidamente a la enzima en un complejo EI de baja afinidad y este luego sufre una reordenación más lenta hasta un complejo EI* muy estrechamente unido (consulte el diagrama del "mecanismo de inhibición irreversible"). Este comportamiento cinético se llama unión lenta. [62] Este lento reordenamiento después de la unión a menudo implica un cambio conformacional a medida que la enzima "se aprieta" alrededor de la molécula inhibidora. Ejemplos de inhibidores de unión lenta incluyen algunos fármacos importantes, como metotrexato , [63] alopurinol , [64] y la forma activada de aciclovir . [sesenta y cinco]
El diisopropilfluorofosfato (DFP) es un ejemplo de inhibidor de proteasa irreversible (consulte el diagrama de "reacción de DFP"). La enzima hidroliza el enlace fósforo-flúor, pero el residuo de fosfato permanece unido a la serina en el sitio activo , desactivándola. [67] De manera similar, el DFP también reacciona con el sitio activo de la acetilcolina esterasa en las sinapsis de las neuronas y, en consecuencia, es una potente neurotoxina, con una dosis letal de menos de 100 mg. [68]
La inhibición del suicidio es un tipo inusual de inhibición irreversible en la que la enzima convierte el inhibidor en una forma reactiva en su sitio activo. [69] Un ejemplo es el inhibidor de la biosíntesis de poliaminas , α-difluorometilornitina (DFMO), que es un análogo del aminoácido ornitina y se utiliza para tratar la tripanosomiasis africana (enfermedad del sueño). La ornitina descarboxilasa puede catalizar la descarboxilación de DFMO en lugar de ornitina (consulte el diagrama "Mecanismo inhibidor de DFMO"). Sin embargo, a esta reacción de descarboxilación le sigue la eliminación de un átomo de flúor, que convierte este intermedio catalítico en una imina conjugada , una especie altamente electrófila. Esta forma reactiva de DFMO luego reacciona con un residuo de cisteína o lisina en el sitio activo para inactivar irreversiblemente la enzima. [61]
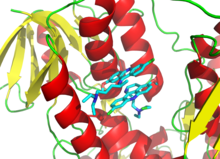
Dado que la inhibición irreversible a menudo implica la formación inicial de un complejo inhibidor enzimático (EI) no covalente, [13] a veces es posible que un inhibidor se una a una enzima de más de una manera. Por ejemplo, en la figura que muestra la tripanotiona reductasa del parásito protozoario humano Trypanosoma cruzi , dos moléculas de un inhibidor llamado mostaza quinacrina están unidas a su sitio activo. La molécula superior está unida de forma reversible, pero la inferior está unida covalentemente ya que ha reaccionado con un residuo de aminoácido a través de su grupo mostaza nitrogenada . [70]
Los inhibidores de enzimas se encuentran en la naturaleza [71] y también se producen artificialmente en el laboratorio. [72] Los inhibidores de enzimas naturales regulan muchos procesos metabólicos y son esenciales para la vida. [3] [1] Además, los venenos producidos naturalmente son a menudo inhibidores de enzimas que han evolucionado para usarse como agentes tóxicos contra depredadores, presas y organismos competidores. [4] Estas toxinas naturales incluyen algunas de las sustancias más venenosas conocidas. [73] Los inhibidores artificiales se utilizan a menudo como fármacos, pero también pueden ser insecticidas como el malatión , herbicidas como el glifosato , [74] o desinfectantes como el triclosán . Otros inhibidores enzimáticos artificiales bloquean la acetilcolinesterasa , una enzima que descompone la acetilcolina , y se utilizan como agentes nerviosos en la guerra química . [75]
La inhibición enzimática es una característica común del control de la vía metabólica en las células. [3] El flujo metabólico a través de una vía a menudo está regulado por los metabolitos de una vía que actúan como inhibidores y potenciadores de las enzimas en esa misma vía. La vía glucolítica es un ejemplo clásico. [76] Esta vía catabólica consume glucosa y produce ATP , NADH y piruvato . Un paso clave para la regulación de la glucólisis es una reacción temprana en la vía catalizada por la fosfofructoquinasa-1 (PFK1). Cuando los niveles de ATP aumentan, el ATP se une a un sitio alostérico en PFK1 para disminuir la velocidad de la reacción enzimática; La glucólisis se inhibe y la producción de ATP disminuye. Este control de retroalimentación negativa ayuda a mantener una concentración constante de ATP en la célula. Sin embargo, las vías metabólicas no se regulan únicamente mediante la inhibición, ya que la activación enzimática es igualmente importante. Con respecto a PFK1, la fructosa 2,6-bisfosfato y el ADP son ejemplos de metabolitos que son activadores alostéricos. [77]
La inhibición enzimática fisiológica también puede producirse mediante inhibidores de proteínas específicos. Este mecanismo ocurre en el páncreas , que sintetiza muchas enzimas precursoras digestivas conocidas como zimógenos . Muchos de estos son activados por la proteasa tripsina , por lo que es importante inhibir la actividad de la tripsina en el páncreas para evitar que el órgano se digiera a sí mismo. Una forma de controlar la actividad de la tripsina es la producción de una proteína inhibidora de tripsina potente y específica en el páncreas. Este inhibidor se une firmemente a la tripsina, impidiendo la actividad de la tripsina que de otro modo sería perjudicial para el órgano. [78] Aunque el inhibidor de tripsina es una proteína, evita ser hidrolizado como sustrato por la proteasa al excluir el agua del sitio activo de la tripsina y desestabilizar el estado de transición. [79] Otros ejemplos de proteínas inhibidoras de enzimas fisiológicas incluyen el inhibidor barstar de la ribonucleasa barnasa bacteriana . [80]

Los animales y las plantas han evolucionado para sintetizar una amplia gama de productos venenosos, incluidos metabolitos secundarios , [81] péptidos y proteínas [82] que pueden actuar como inhibidores. Las toxinas naturales suelen ser pequeñas moléculas orgánicas y son tan diversas que probablemente existan inhibidores naturales para la mayoría de los procesos metabólicos. [83] Los procesos metabólicos a los que se dirigen los venenos naturales abarcan más que enzimas en las vías metabólicas y también pueden incluir la inhibición de las funciones de receptores, canales y proteínas estructurales en una célula. Por ejemplo, el paclitaxel (taxol), una molécula orgánica que se encuentra en el tejo del Pacífico , se une firmemente a los dímeros de tubulina e inhibe su ensamblaje en microtúbulos en el citoesqueleto . [84]
Muchos venenos naturales actúan como neurotoxinas que pueden causar parálisis y provocar la muerte y sirven como defensa contra los depredadores o para cazar y capturar presas. Algunos de estos inhibidores naturales, [85] a pesar de sus atributos tóxicos, son valiosos para usos terapéuticos en dosis más bajas. [86] Un ejemplo de neurotoxina son los glicoalcaloides , de especies vegetales de la familia Solanaceae (incluye patata , tomate y berenjena ), que son inhibidores de la acetilcolinesterasa . La inhibición de esta enzima provoca un aumento incontrolado del neurotransmisor acetilcolina, parálisis muscular y posteriormente la muerte. La neurotoxicidad también puede resultar de la inhibición de los receptores; por ejemplo, la atropina de la belladona ( Atropa belladonna ) que funciona como antagonista competitivo de los receptores muscarínicos de acetilcolina . [87]
Aunque muchas toxinas naturales son metabolitos secundarios, estos venenos también incluyen péptidos y proteínas. Un ejemplo de péptido tóxico es la alfa-amanitina , que se encuentra en parientes del hongo de la muerte . Se trata de un potente inhibidor enzimático que, en este caso, impide que la enzima ARN polimerasa II transcriba el ADN. [88] La toxina de algas microcistina también es un péptido y es un inhibidor de las proteínas fosfatasas . [89] Esta toxina puede contaminar los suministros de agua después de la proliferación de algas y es un carcinógeno conocido que también puede causar hemorragia hepática aguda y la muerte en dosis más altas. [90]
Las proteínas también pueden ser venenos naturales o antinutrientes , como los inhibidores de tripsina (que se analizan en la sección "regulación metabólica" anterior) que se encuentran en algunas legumbres . [91] Una clase menos común de toxinas son las enzimas tóxicas: actúan como inhibidores irreversibles de sus enzimas objetivo y funcionan modificando químicamente sus enzimas sustrato. Un ejemplo es la ricina , una toxina proteica extremadamente potente que se encuentra en el aceite de ricino . [92] Esta enzima es una glicosidasa que inactiva los ribosomas. [93] Dado que la ricina es un inhibidor catalítico irreversible, esto permite que una sola molécula de ricina mate una célula. [94]

Los usos más comunes de los inhibidores de enzimas son como medicamentos para tratar enfermedades. Muchos de estos inhibidores se dirigen a una enzima humana y tienen como objetivo corregir una condición patológica. Por ejemplo, la aspirina es un fármaco muy utilizado que actúa como inhibidor suicida de la enzima ciclooxigenasa . [95] Esta inhibición, a su vez, suprime la producción de prostaglandinas proinflamatorias y, por lo tanto, la aspirina puede usarse para reducir el dolor, la fiebre y la inflamación. [95]
En 2017, [update]se estima que el 29 % de los medicamentos aprobados son inhibidores de enzimas [96] de los cuales aproximadamente una quinta parte son inhibidores de quinasas . [96] Una clase notable de objetivos farmacológicos de quinasa son los receptores tirosina quinasas , que son enzimas esenciales que regulan el crecimiento celular ; su sobreactivación puede provocar cáncer. Por lo tanto, los inhibidores de la quinasa como el imatinib se utilizan con frecuencia para tratar tumores malignos. [97] Las Janus quinasas son otro ejemplo notable de dianas enzimáticas farmacológicas. Los inhibidores de las Janus quinasas bloquean la producción de citoquinas inflamatorias y, por lo tanto, estos inhibidores se usan para tratar una variedad de enfermedades inflamatorias que incluyen artritis , asma y enfermedad de Crohn . [98]
Un ejemplo de la similitud estructural de algunos inhibidores con los sustratos de las enzimas a las que se dirigen se ve en la figura que compara el medicamento metotrexato con el ácido fólico . El ácido fólico es la forma oxidada del sustrato de la dihidrofolato reductasa , una enzima potentemente inhibida por el metotrexato. El metotrexato bloquea la acción de la dihidrofolato reductasa y, por tanto, detiene la biosíntesis de timidina . [42] Este bloque de la biosíntesis de nucleótidos es selectivamente tóxico para las células de rápido crecimiento, por lo que el metotrexato se utiliza a menudo en la quimioterapia contra el cáncer. [99]
Un tratamiento común para la disfunción eréctil es el sildenafil (Viagra). [100] Este compuesto es un potente inhibidor de la fosfodiesterasa tipo 5 específica de cGMP , la enzima que degrada la molécula de señalización monofosfato de guanosina cíclico . [101] Esta molécula de señalización desencadena la relajación del músculo liso y permite que la sangre fluya hacia el cuerpo cavernoso , lo que provoca una erección. Dado que el fármaco disminuye la actividad de la enzima que detiene la señal, hace que esta dure más tiempo.
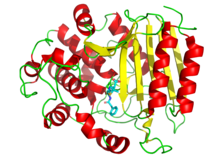
También se utilizan fármacos para inhibir las enzimas necesarias para la supervivencia de los patógenos. Por ejemplo, las bacterias están rodeadas por una gruesa pared celular hecha de un polímero en forma de red llamado peptidoglicano . Muchos antibióticos , como la penicilina y la vancomicina, inhiben las enzimas que producen y luego entrecruzan las hebras de este polímero. [102] [103] Esto hace que la pared celular pierda fuerza y las bacterias exploten. En la figura, se muestra una molécula de penicilina (mostrada en forma de bola y palo) unida a su objetivo, la transpeptidasa de la bacteria Streptomyces R61 (la proteína se muestra como un diagrama de cinta ).
El diseño de fármacos antibióticos se facilita cuando una enzima que es esencial para la supervivencia del patógeno está ausente o es muy diferente en los humanos. [104] Los humanos no producen peptidoglicano, por lo tanto, los antibióticos que inhiben este proceso son selectivamente tóxicos para las bacterias. [105] La toxicidad selectiva también se produce en los antibióticos al explotar las diferencias en la estructura de los ribosomas en las bacterias, [106] o cómo producen ácidos grasos . [107]
Los medicamentos que inhiben las enzimas necesarias para la replicación de los virus son eficaces en el tratamiento de infecciones virales. [108] Los medicamentos antivirales incluyen inhibidores de la proteasa utilizados para tratar el VIH/SIDA [109] y la hepatitis C , [110] inhibidores de la transcriptasa inversa dirigidos al VIH/SIDA, [111] inhibidores de la neuraminidasa dirigidos a la influenza , [112] e inhibidores de la terminasa dirigidos al citomegalovirus humano. . [113]
Muchos pesticidas son inhibidores de enzimas. [114] La acetilcolinesterasa (AChE) es una enzima que se encuentra en animales, desde insectos hasta humanos. Es esencial para el funcionamiento de las células nerviosas a través de su mecanismo de descomposición del neurotransmisor acetilcolina en sus constituyentes, acetato y colina . [115] Esto es algo inusual entre los neurotransmisores, ya que la mayoría, incluida la serotonina , la dopamina y la norepinefrina , se absorben desde la hendidura sináptica en lugar de escindirse. Un gran número de inhibidores de la AChE se utilizan tanto en medicina como en agricultura. [116] Los inhibidores competitivos reversibles, como el edrofonio , la fisostigmina y la neostigmina , se utilizan en el tratamiento de la miastenia gravis [117] y en la anestesia para revertir el bloqueo muscular. [118] Los pesticidas carbamatos también son ejemplos de inhibidores reversibles de la AChE. Los pesticidas organofosforados como el malatión , el paratión y el clorpirifos inhiben irreversiblemente la acetilcolinesterasa. [119]
El herbicida glifosato es un inhibidor de la 3-fosfoshikimato 1-carboxiviniltransferasa , [120] otros herbicidas, como las sulfonilureas, inhiben la enzima acetolactato sintasa . [121] Ambas enzimas son necesarias para que las plantas produzcan aminoácidos de cadena ramificada . Los herbicidas inhiben muchas otras enzimas, incluidas las enzimas necesarias para la biosíntesis de lípidos y carotenoides y los procesos de fotosíntesis y fosforilación oxidativa . [122]

Los nuevos fármacos son el producto de un largo proceso de desarrollo de fármacos , cuyo primer paso suele ser el descubrimiento de un nuevo inhibidor enzimático. [123] Hay dos enfoques principales para descubrir estos inhibidores. [124]
El primer método general es el diseño racional de fármacos basado en la imitación del estado de transición de la reacción química catalizada por la enzima. [125] El inhibidor diseñado a menudo se parece mucho al sustrato, excepto que la porción del sustrato que sufre una reacción química se reemplaza por un grupo funcional químicamente estable que se asemeja al estado de transición. Dado que la enzima ha evolucionado para estabilizar el estado de transición, los análogos del estado de transición generalmente poseen una mayor afinidad por la enzima en comparación con el sustrato y, por lo tanto, son inhibidores eficaces. [46]
La segunda forma de descubrir nuevos inhibidores de enzimas es la detección de alto rendimiento de grandes bibliotecas de compuestos estructuralmente diversos para identificar moléculas impactantes que se unen a la enzima. Este método se ha ampliado para incluir la detección virtual de bases de datos de diversas moléculas utilizando computadoras, [126] [127] , a las que luego sigue la confirmación experimental de la unión de los resultados de la detección virtual. [128] Los enfoques complementarios que pueden proporcionar nuevos puntos de partida para los inhibidores incluyen el descubrimiento de pistas basado en fragmentos [129] y las bibliotecas químicas codificadas por ADN (DEL). [130]
Los resultados de cualquiera de los enfoques anteriores se pueden optimizar para obtener aglutinantes de alta afinidad que inhiban eficazmente la enzima. [131] Se pueden utilizar métodos informáticos para predecir la orientación de unión y la afinidad de un inhibidor por una enzima, como el acoplamiento molecular [132] y la mecánica molecular, para ayudar en el proceso de optimización. [133] Se utilizan nuevos inhibidores para obtener estructuras cristalográficas de la enzima en un complejo inhibidor/enzima para mostrar cómo la molécula se une al sitio activo, lo que permite realizar cambios en el inhibidor para optimizar la unión en un proceso conocido como estructura- diseño de fármacos basado en [1] : 66 Este ciclo de prueba y mejora se repite hasta que se produce un inhibidor suficientemente potente.
En algunos casos, el inhibidor puede unirse a un sitio distinto de la enzima que está en comunicación alostérica con la bolsa de unión del sustrato. En muchos casos, los compuestos alostéricos competitivos por sustrato dan como resultado cambios conformacionales en la enzima que cambian la capacidad de la enzima para unirse al sustrato.
La inactivación enzimática se explica generalmente como un proceso químico que involucra varios fenómenos como agregación, disociación en subunidades o desnaturalización (cambios conformacionales), que ocurren simultáneamente durante la inactivación de una enzima específica.
Figura 1C: Éxito clínico de clases privilegiadas de familias de proteínas (% de medicamentos aprobados dirigidos a cada clase objetivo): reductasa 7,62, quinasa 5,94, proteasa 3,35, hidrolasa 2,76, NPTasa 2,09, transferasa 1,92, liasa 1,59, isomerasa 1,51, fosfodiesterasa 1,50, Citocromo p450 0,84, Borrador epigenético 0,33, Objetivos enzimáticos totales de medicamentos aprobados = 29,45%
{{cite book}}: CS1 maint: location missing publisher (link)Capítulo 10.2.1: Inhibidores de la sulfonilurea acetolactato sintasa