
Las proteínas son grandes biomoléculas y macromoléculas que comprenden una o más cadenas largas de residuos de aminoácidos . Las proteínas realizan una amplia gama de funciones dentro de los organismos, incluyendo la catálisis de reacciones metabólicas , la replicación del ADN , la respuesta a estímulos , el suministro de estructura a células y organismos y el transporte de moléculas de un lugar a otro. Las proteínas se diferencian entre sí principalmente en su secuencia de aminoácidos, que está dictada por la secuencia de nucleótidos de sus genes , y que generalmente da como resultado el plegamiento de la proteína en una estructura 3D específica que determina su actividad.
Una cadena lineal de residuos de aminoácidos se denomina polipéptido . Una proteína contiene al menos un polipéptido largo. Los polipéptidos cortos, que contienen menos de 20 a 30 residuos, rara vez se consideran proteínas y comúnmente se denominan péptidos . Los residuos de aminoácidos individuales están unidos entre sí por enlaces peptídicos y residuos de aminoácidos adyacentes. La secuencia de residuos de aminoácidos en una proteína está definida por la secuencia de un gen, que está codificada en el código genético . En general, el código genético especifica 20 aminoácidos estándar; pero en ciertos organismos el código genético puede incluir selenocisteína y, en ciertas arqueas , pirrolisina . Poco después o incluso durante la síntesis, los residuos de una proteína a menudo se modifican químicamente mediante modificación postraduccional , que altera las propiedades físicas y químicas, el plegamiento, la estabilidad, la actividad y, en última instancia, la función de las proteínas. Algunas proteínas tienen grupos no peptídicos unidos, que pueden llamarse grupos prostéticos o cofactores . Las proteínas también pueden trabajar juntas para lograr una función particular y a menudo se asocian para formar complejos proteicos estables .
Una vez formadas, las proteínas solo existen durante un período determinado y luego son degradadas y recicladas por la maquinaria celular a través del proceso de recambio proteico . La vida útil de una proteína se mide en términos de su vida media y cubre un amplio rango. Pueden existir durante minutos o años con una vida útil promedio de 1 a 2 días en las células de los mamíferos. Las proteínas anormales o mal plegadas se degradan más rápidamente ya sea porque están destinadas a ser destruidas o porque son inestables.
Al igual que otras macromoléculas biológicas como los polisacáridos y los ácidos nucleicos , las proteínas son partes esenciales de los organismos y participan en prácticamente todos los procesos dentro de las células . Muchas proteínas son enzimas que catalizan reacciones bioquímicas y son vitales para el metabolismo . Las proteínas también tienen funciones estructurales o mecánicas, como la actina y la miosina en el músculo y las proteínas en el citoesqueleto , que forman un sistema de andamiaje que mantiene la forma celular. Otras proteínas son importantes en la señalización celular, las respuestas inmunes , la adhesión celular y el ciclo celular . En los animales, las proteínas son necesarias en la dieta para proporcionar los aminoácidos esenciales que no se pueden sintetizar . La digestión descompone las proteínas para su uso metabólico.
Las proteínas han sido estudiadas y reconocidas desde el siglo XVIII por Antoine Fourcroy y otros, [1] [2] quienes a menudo las llamaban colectivamente " albúminas " o "materiales albuminosos" ( Eiweisskörper , en alemán). [2] El gluten , por ejemplo, se separó por primera vez del trigo en una investigación publicada alrededor de 1747, y luego se determinó que existía en muchas plantas. [1] En 1789, Antoine Fourcroy reconoció tres variedades distintas de proteínas animales: albúmina , fibrina y gelatina . [3] Las proteínas vegetales (plantas) estudiadas a fines del siglo XVIII y principios del siglo XIX incluían gluten , albúmina vegetal , gliadina y leguminosa . [1]
Las proteínas fueron descritas por primera vez por el químico holandés Gerardus Johannes Mulder y nombradas por el químico sueco Jöns Jacob Berzelius en 1838. [4] [5] [ mejor fuente necesaria ] Mulder realizó un análisis elemental de proteínas comunes y descubrió que casi todas las proteínas tenían la misma fórmula empírica , C 400 H 620 N 100 O 120 P 1 S 1 . [6] Llegó a la conclusión errónea de que podrían estar compuestas de un solo tipo de molécula (muy grande). El término "proteína" para describir estas moléculas fue propuesto por el asociado de Mulder, Berzelius; proteína se deriva de la palabra griega πρώτειος ( proteios ), que significa "primario", [7] "a la cabeza", o "de pie al frente", [2] + -in . Mulder continuó identificando los productos de la degradación de proteínas, como el aminoácido leucina, para el cual encontró un peso molecular (casi correcto) de 131 Da . [6]
Los primeros científicos nutricionistas, como el alemán Carl von Voit, creían que la proteína era el nutriente más importante para mantener la estructura del cuerpo, porque generalmente se creía que "la carne hace carne". [8] Alrededor de 1862, Karl Heinrich Ritthausen aisló el aminoácido ácido glutámico . [9] Thomas Burr Osborne compiló una revisión detallada de las proteínas vegetales en la Estación Experimental Agrícola de Connecticut . Luego, trabajando con [ aclaración necesaria ] Lafayette Mendel y aplicando la ley del mínimo de Liebig , que establece que el crecimiento está limitado por el recurso más escaso, a la alimentación de ratas de laboratorio, se establecieron los aminoácidos nutricionalmente esenciales . El trabajo fue continuado y comunicado por William Cumming Rose .
La dificultad de purificar proteínas en grandes cantidades hizo que fuera muy difícil para los primeros bioquímicos de proteínas estudiarlas. Por lo tanto, los primeros estudios se centraron en proteínas que pudieran purificarse en grandes cantidades, incluidas las de sangre, claras de huevo y varias toxinas, así como enzimas digestivas y metabólicas obtenidas de mataderos. [ aclaración necesaria ] En la década de 1950, la Armour Hot Dog Company purificó 1 kg de ribonucleasa A pancreática bovina pura y la puso a disposición de los científicos de forma gratuita; este gesto ayudó a que la ribonucleasa A se convirtiera en un objetivo importante para el estudio bioquímico durante las décadas siguientes. [6]

La comprensión de las proteínas como polipéptidos , o cadenas de aminoácidos, llegó a través del trabajo de Franz Hofmeister y Hermann Emil Fischer en 1902. [10] [11] El papel central de las proteínas como enzimas en los organismos vivos que catalizaban reacciones no fue apreciado plenamente hasta 1926, cuando James B. Sumner demostró que la enzima ureasa era de hecho una proteína. [12]
A Linus Pauling se le atribuye la predicción exitosa de estructuras secundarias de proteínas regulares basadas en enlaces de hidrógeno , una idea propuesta por primera vez por William Astbury en 1933. [13] El trabajo posterior de Walter Kauzmann sobre desnaturalización , [14] [15] basado en parte en estudios previos de Kaj Linderstrøm-Lang , [16] contribuyó a una comprensión del plegamiento y la estructura de las proteínas mediada por interacciones hidrofóbicas .
La primera proteína cuya cadena de aminoácidos fue secuenciada fue la insulina , por Frederick Sanger , en 1949. Sanger determinó correctamente la secuencia de aminoácidos de la insulina, demostrando así de manera concluyente que las proteínas estaban formadas por polímeros lineales de aminoácidos en lugar de cadenas ramificadas, coloides o cicloles . [17] Ganó el Premio Nobel por este logro en 1958. [18] Los estudios de Christian Anfinsen sobre el proceso de plegamiento oxidativo de la ribonucleasa A, por los que ganó el premio Nobel en 1972, solidificaron la hipótesis termodinámica del plegamiento de proteínas, según la cual la forma plegada de una proteína representa su mínimo de energía libre . [19] [20]

Con el desarrollo de la cristalografía de rayos X , se hizo posible determinar las estructuras de las proteínas, así como sus secuencias. [21] Las primeras estructuras de proteínas que se resolvieron fueron la hemoglobina por Max Perutz y la mioglobina por John Kendrew , en 1958. [22] [23] El uso de computadoras y el aumento de la potencia computacional también respaldaron la secuenciación de proteínas complejas. En 1999, Roger Kornberg logró secuenciar la estructura altamente compleja de la ARN polimerasa utilizando rayos X de alta intensidad de sincrotrones . [21]
Desde entonces, se ha desarrollado la criomicroscopía electrónica (crio-EM) de grandes conjuntos macromoleculares [24] . La crio-EM utiliza muestras de proteínas congeladas en lugar de cristales, y haces de electrones en lugar de rayos X. Provoca menos daño a la muestra, lo que permite a los científicos obtener más información y analizar estructuras más grandes. [21] La predicción computacional de la estructura de proteínas de pequeños dominios estructurales de proteínas [25] también ha ayudado a los investigadores a acercarse a la resolución a nivel atómico de las estructuras de proteínas. A partir de abril de 2024 [actualizar], el Banco de Datos de Proteínas contiene 181.018 estructuras de proteínas de rayos X, 19.809 EM y 12.697 RMN . [26]
Las proteínas se clasifican principalmente por secuencia y estructura, aunque también se utilizan otras clasificaciones. En el caso de las enzimas, el sistema de numeración EC proporciona un esquema de clasificación funcional. De manera similar, la ontología génica clasifica tanto a los genes como a las proteínas por su función biológica y bioquímica, pero también por su ubicación intracelular.
La similitud de secuencias se utiliza para clasificar las proteínas tanto en términos de similitud evolutiva como funcional. Esto puede utilizar proteínas completas o dominios proteicos , especialmente en proteínas multidominio . Los dominios proteicos permiten la clasificación de proteínas mediante una combinación de secuencia, estructura y función, y se pueden combinar de muchas formas diferentes. En un estudio inicial de 170.000 proteínas, aproximadamente dos tercios se asignaron al menos un dominio, y las proteínas más grandes contenían más dominios (por ejemplo, las proteínas de más de 600 aminoácidos tenían un promedio de más de 5 dominios). [27]

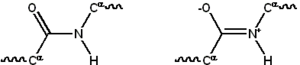
La mayoría de las proteínas consisten en polímeros lineales construidos a partir de series de hasta 20 L -α- aminoácidos diferentes. Todos los aminoácidos proteinogénicos poseen características estructurales comunes, incluido un carbono α al que se unen un grupo amino , un grupo carboxilo y una cadena lateral variable . Solo la prolina difiere de esta estructura básica ya que contiene un anillo inusual en el grupo amina del extremo N, que fuerza a la fracción amida CO–NH a una conformación fija. [28] Las cadenas laterales de los aminoácidos estándar, detalladas en la lista de aminoácidos estándar , tienen una gran variedad de estructuras y propiedades químicas; es el efecto combinado de todas las cadenas laterales de aminoácidos en una proteína lo que finalmente determina su estructura tridimensional y su reactividad química. [29] Los aminoácidos en una cadena polipeptídica están unidos por enlaces peptídicos . Una vez unidos en la cadena proteica, un aminoácido individual se denomina residuo, y la serie enlazada de átomos de carbono, nitrógeno y oxígeno se conoce como la cadena principal o esqueleto proteico. [30] : 19
El enlace peptídico tiene dos formas de resonancia que aportan algún carácter de doble enlace e inhiben la rotación alrededor de su eje, de modo que los carbonos alfa son aproximadamente coplanares . Los otros dos ángulos diedros en el enlace peptídico determinan la forma local asumida por la estructura de la proteína. [30] : 31 El extremo con un grupo amino libre se conoce como el extremo N o amino, mientras que el extremo de la proteína con un grupo carboxilo libre se conoce como el extremo C o carboxi (la secuencia de la proteína se escribe desde el extremo N hasta el extremo C, de izquierda a derecha).
Las palabras proteína , polipéptido y péptido son un poco ambiguas y pueden superponerse en significado. Proteína se usa generalmente para referirse a la molécula biológica completa en una conformación estable , mientras que péptido generalmente se reserva para oligómeros de aminoácidos cortos que a menudo carecen de una estructura 3D estable. Pero el límite entre los dos no está bien definido y generalmente se encuentra cerca de 20-30 residuos. [31] Polipéptido puede referirse a cualquier cadena lineal única de aminoácidos, generalmente independientemente de la longitud, pero a menudo implica la ausencia de una conformación definida .
Las proteínas pueden interactuar con muchos tipos de moléculas, incluidas otras proteínas , lípidos , carbohidratos y ADN . [32] [33] [30] [34]
Se ha estimado que las bacterias de tamaño medio contienen alrededor de 2 millones de proteínas por célula (p. ej., E. coli y Staphylococcus aureus ). Las bacterias más pequeñas, como Mycoplasma o las espiroquetas, contienen menos moléculas, del orden de 50.000 a 1 millón. Por el contrario, las células eucariotas son más grandes y, por lo tanto, contienen muchas más proteínas. Por ejemplo, se ha estimado que las células de levadura contienen alrededor de 50 millones de proteínas y las células humanas del orden de 1 a 3 mil millones. [35] La concentración de copias de proteínas individuales varía desde unas pocas moléculas por célula hasta 20 millones. [36] No todos los genes que codifican proteínas se expresan en la mayoría de las células y su número depende, por ejemplo, del tipo de célula y de los estímulos externos. Por ejemplo, de las aproximadamente 20.000 proteínas codificadas por el genoma humano, solo 6.000 se detectan en las células linfoblastoides . [37]


Las proteínas se ensamblan a partir de aminoácidos utilizando información codificada en los genes. Cada proteína tiene su propia secuencia de aminoácidos única que está especificada por la secuencia de nucleótidos del gen que codifica esta proteína. El código genético es un conjunto de conjuntos de tres nucleótidos llamados codones y cada combinación de tres nucleótidos designa un aminoácido, por ejemplo AUG ( adenina – uracilo – guanina ) es el código para la metionina . Debido a que el ADN contiene cuatro nucleótidos, el número total de codones posibles es 64; por lo tanto, hay cierta redundancia en el código genético, con algunos aminoácidos especificados por más de un codón. [34] : 1002–42 Los genes codificados en el ADN primero se transcriben en ARN premensajero (ARNm) por proteínas como la ARN polimerasa . La mayoría de los organismos luego procesan el pre-ARNm (también conocido como transcripción primaria ) utilizando varias formas de modificación postranscripcional para formar el ARNm maduro, que luego se utiliza como plantilla para la síntesis de proteínas por el ribosoma . En los procariotas, el ARNm puede utilizarse tan pronto como se produce o puede unirse a un ribosoma después de haber salido del nucleoide . Por el contrario, los eucariotas producen ARNm en el núcleo celular y luego lo translocan a través de la membrana nuclear hacia el citoplasma , donde luego tiene lugar la síntesis de proteínas . La tasa de síntesis de proteínas es mayor en los procariotas que en los eucariotas y puede alcanzar hasta 20 aminoácidos por segundo. [38]
El proceso de sintetizar una proteína a partir de una plantilla de ARNm se conoce como traducción . El ARNm se carga en el ribosoma y se lee de a tres nucleótidos por vez haciendo coincidir cada codón con su anticodón de apareamiento de bases ubicado en una molécula de ARN de transferencia , que lleva el aminoácido correspondiente al codón que reconoce. La enzima aminoacil ARNt sintetasa "carga" las moléculas de ARNt con los aminoácidos correctos. El polipéptido en crecimiento a menudo se denomina cadena naciente . Las proteínas siempre se biosintetizan desde el extremo N al extremo C. [34] : 1002–42
El tamaño de una proteína sintetizada se puede medir por el número de aminoácidos que contiene y por su masa molecular total , que normalmente se informa en unidades de daltons (sinónimo de unidades de masa atómica ), o la unidad derivada kilodalton (kDa). El tamaño promedio de una proteína aumenta de Archaea a Bacteria a Eucariota (283, 311, 438 residuos y 31, 34, 49 kDa respectivamente) debido a un mayor número de dominios proteicos que constituyen proteínas en organismos superiores. [39] Por ejemplo, las proteínas de levadura tienen en promedio 466 aminoácidos de longitud y 53 kDa de masa. [31] Las proteínas más grandes conocidas son las titinas , un componente del sarcómero muscular , con una masa molecular de casi 3.000 kDa y una longitud total de casi 27.000 aminoácidos. [40]

Las proteínas cortas también pueden sintetizarse químicamente mediante una familia de métodos conocidos como síntesis de péptidos , que se basan en técnicas de síntesis orgánica como la ligación química para producir péptidos con un alto rendimiento. [41] La síntesis química permite la introducción de aminoácidos no naturales en cadenas polipeptídicas, como la unión de sondas fluorescentes a las cadenas laterales de aminoácidos. [42] Estos métodos son útiles en bioquímica de laboratorio y biología celular , aunque generalmente no para aplicaciones comerciales. La síntesis química es ineficiente para polipéptidos de más de 300 aminoácidos, y las proteínas sintetizadas pueden no asumir fácilmente su estructura terciaria nativa . La mayoría de los métodos de síntesis química proceden del extremo C al extremo N, opuesto a la reacción biológica. [43]


La mayoría de las proteínas se pliegan en estructuras tridimensionales únicas. La forma en la que una proteína se pliega naturalmente se conoce como su conformación nativa . [30] : 36 Aunque muchas proteínas pueden plegarse sin ayuda, simplemente a través de las propiedades químicas de sus aminoácidos, otras requieren la ayuda de chaperonas moleculares para plegarse en sus estados nativos. [30] : 37 Los bioquímicos a menudo se refieren a cuatro aspectos distintos de la estructura de una proteína: [30] : 30–34
Las proteínas no son moléculas completamente rígidas. Además de estos niveles de estructura, las proteínas pueden cambiar entre varias estructuras relacionadas mientras realizan sus funciones. En el contexto de estos reordenamientos funcionales, estas estructuras terciarias o cuaternarias suelen denominarse " conformaciones ", y las transiciones entre ellas se denominan cambios conformacionales. Dichos cambios suelen ser inducidos por la unión de una molécula de sustrato al sitio activo de una enzima , o la región física de la proteína que participa en la catálisis química. En solución, las proteínas también experimentan variaciones en la estructura a través de la vibración térmica y la colisión con otras moléculas. [34] : 368–75

Las proteínas se pueden dividir informalmente en tres clases principales, que se correlacionan con estructuras terciarias típicas: proteínas globulares , proteínas fibrosas y proteínas de membrana . Casi todas las proteínas globulares son solubles y muchas son enzimas. Las proteínas fibrosas suelen ser estructurales, como el colágeno , el componente principal del tejido conectivo, o la queratina , el componente proteico del cabello y las uñas. Las proteínas de membrana a menudo sirven como receptores o proporcionan canales para que las moléculas polares o cargadas pasen a través de la membrana celular . [34] : 165–85
Un caso especial de enlaces de hidrógeno intramoleculares dentro de las proteínas, poco protegidos del ataque del agua y por lo tanto promoviendo su propia deshidratación , se denominan deshidrones . [44]
Muchas proteínas están compuestas por varios dominios proteicos , es decir, segmentos de una proteína que se pliegan en unidades estructurales distintas. Los dominios suelen tener también funciones específicas, como actividades enzimáticas (p. ej. , quinasa ) o sirven como módulos de unión (p. ej., el dominio SH3 se une a secuencias ricas en prolina en otras proteínas).
Las secuencias cortas de aminoácidos dentro de las proteínas a menudo actúan como sitios de reconocimiento para otras proteínas. [45] Por ejemplo, los dominios SH3 normalmente se unen a motivos PxxP cortos (es decir, 2 prolinas [P], separadas por dos aminoácidos no especificados [x], aunque los aminoácidos circundantes pueden determinar la especificidad de unión exacta). Muchos de estos motivos se han recopilado en la base de datos Eukaryotic Linear Motif (ELM).
La topología de una proteína describe el enredo de la estructura principal y la disposición de los contactos dentro de la cadena plegada. [46] Se han aplicado dos marcos teóricos, la teoría de nudos y la topología de circuitos , para caracterizar la topología de proteínas. Poder describir la topología de proteínas abre nuevos caminos para la ingeniería de proteínas y el desarrollo farmacéutico, y aumenta nuestra comprensión de las enfermedades por plegamiento incorrecto de proteínas, como los trastornos neuromusculares y el cáncer.
Las proteínas son los principales actores dentro de la célula, y se dice que llevan a cabo las tareas especificadas por la información codificada en los genes. [31] Con la excepción de ciertos tipos de ARN , la mayoría de las demás moléculas biológicas son elementos relativamente inertes sobre los que actúan las proteínas. Las proteínas constituyen la mitad del peso seco de una célula de Escherichia coli , mientras que otras macromoléculas como el ADN y el ARN constituyen solo el 3% y el 20%, respectivamente. [47] El conjunto de proteínas expresadas en una célula o tipo de célula en particular se conoce como su proteoma .

La característica principal de las proteínas, que también les permite su conjunto diverso de funciones, es su capacidad de unirse a otras moléculas de manera específica y estrecha. La región de la proteína responsable de unirse a otra molécula se conoce como el sitio de unión y a menudo es una depresión o "bolsillo" en la superficie molecular. Esta capacidad de unión está mediada por la estructura terciaria de la proteína, que define el bolsillo del sitio de unión, y por las propiedades químicas de las cadenas laterales de los aminoácidos circundantes. La unión a proteínas puede ser extraordinariamente estrecha y específica; por ejemplo, la proteína inhibidora de la ribonucleasa se une a la angiogenina humana con una constante de disociación subfemtomolar (<10 −15 M) pero no se une en absoluto a su homóloga anfibia onconasa (> 1 M). Cambios químicos extremadamente menores, como la adición de un solo grupo metilo a un socio de unión, a veces pueden ser suficientes para eliminar casi por completo la unión; por ejemplo, la aminoacil ARNt sintetasa específica del aminoácido valina discrimina contra la cadena lateral muy similar del aminoácido isoleucina . [48]
Las proteínas pueden unirse a otras proteínas, así como a sustratos de moléculas pequeñas . Cuando las proteínas se unen específicamente a otras copias de la misma molécula, pueden oligomerizarse para formar fibrillas; este proceso ocurre a menudo en proteínas estructurales que consisten en monómeros globulares que se autoasocian para formar fibras rígidas. Las interacciones proteína-proteína también regulan la actividad enzimática, controlan la progresión a través del ciclo celular y permiten el ensamblaje de grandes complejos proteicos que llevan a cabo muchas reacciones estrechamente relacionadas con una función biológica común. Las proteínas también pueden unirse a las membranas celulares o incluso integrarse en ellas. La capacidad de los socios de unión para inducir cambios conformacionales en las proteínas permite la construcción de redes de señalización enormemente complejas. [34] : 830–49 Como las interacciones entre proteínas son reversibles y dependen en gran medida de la disponibilidad de diferentes grupos de proteínas asociadas para formar agregados que sean capaces de llevar a cabo conjuntos discretos de funciones, el estudio de las interacciones entre proteínas específicas es clave para comprender aspectos importantes de la función celular y, en última instancia, las propiedades que distinguen a los tipos de células particulares. [49] [50]
El papel más conocido de las proteínas en la célula es el de enzimas , que catalizan reacciones químicas. Las enzimas suelen ser muy específicas y aceleran solo una o unas pocas reacciones químicas. Las enzimas llevan a cabo la mayoría de las reacciones implicadas en el metabolismo , así como la manipulación del ADN en procesos como la replicación del ADN , la reparación del ADN y la transcripción . Algunas enzimas actúan sobre otras proteínas para añadir o eliminar grupos químicos en un proceso conocido como modificación postraduccional. Se sabe que unas 4.000 reacciones son catalizadas por enzimas. [51] La aceleración de la velocidad conferida por la catálisis enzimática es a menudo enorme: hasta 10 17 veces más velocidad que la reacción no catalizada en el caso de la orotato descarboxilasa (78 millones de años sin la enzima, 18 milisegundos con la enzima). [52]
Las moléculas unidas y sobre las que actúan las enzimas se denominan sustratos . Aunque las enzimas pueden estar formadas por cientos de aminoácidos, normalmente solo una pequeña fracción de los residuos entran en contacto con el sustrato, y una fracción aún más pequeña (de tres a cuatro residuos en promedio) son los que participan directamente en la catálisis. [53] La región de la enzima que se une al sustrato y contiene los residuos catalíticos se conoce como sitio activo .
Las proteínas dirigentes son miembros de una clase de proteínas que dictan la estereoquímica de un compuesto sintetizado por otras enzimas. [54]
Muchas proteínas están involucradas en el proceso de señalización celular y transducción de señales . Algunas proteínas, como la insulina , son proteínas extracelulares que transmiten una señal desde la célula en la que fueron sintetizadas a otras células en tejidos distantes . Otras son proteínas de membrana que actúan como receptores cuya función principal es unirse a una molécula de señalización e inducir una respuesta bioquímica en la célula. Muchos receptores tienen un sitio de unión expuesto en la superficie celular y un dominio efector dentro de la célula, que puede tener actividad enzimática o puede sufrir un cambio conformacional detectado por otras proteínas dentro de la célula. [33] : 251–81
Los anticuerpos son componentes proteicos de un sistema inmunitario adaptativo cuya función principal es unirse a antígenos o sustancias extrañas en el cuerpo y atacarlos para destruirlos. Los anticuerpos pueden secretarse en el entorno extracelular o anclarse en las membranas de células B especializadas conocidas como células plasmáticas . Mientras que las enzimas están limitadas en su afinidad de unión con sus sustratos por la necesidad de llevar a cabo su reacción, los anticuerpos no tienen tales restricciones. La afinidad de unión de un anticuerpo con su objetivo es extraordinariamente alta. [34] : 275–50
Muchas proteínas de transporte de ligando se unen a pequeñas biomoléculas particulares y las transportan a otras ubicaciones en el cuerpo de un organismo multicelular. Estas proteínas deben tener una alta afinidad de unión cuando su ligando está presente en altas concentraciones, pero también deben liberar el ligando cuando está presente en bajas concentraciones en los tejidos objetivo. El ejemplo canónico de una proteína de unión a ligando es la hemoglobina , que transporta oxígeno desde los pulmones a otros órganos y tejidos en todos los vertebrados y tiene homólogos cercanos en cada reino biológico . [34] : 222–29 Las lectinas son proteínas de unión de azúcar que son altamente específicas para sus fracciones de azúcar. Las lectinas generalmente juegan un papel en los fenómenos de reconocimiento biológico que involucran células y proteínas. [55] Los receptores y las hormonas son proteínas de unión altamente específicas.
Las proteínas transmembrana también pueden servir como proteínas de transporte de ligandos que alteran la permeabilidad de la membrana celular a moléculas pequeñas e iones. La membrana por sí sola tiene un núcleo hidrofóbico a través del cual las moléculas polares o cargadas no pueden difundirse . Las proteínas de membrana contienen canales internos que permiten que dichas moléculas entren y salgan de la célula. Muchas proteínas de canal iónico están especializadas para seleccionar solo un ion en particular; por ejemplo, los canales de potasio y sodio a menudo discriminan solo uno de los dos iones. [33] : 232–34

Las proteínas estructurales confieren rigidez y rigidez a componentes biológicos que de otro modo serían fluidos. La mayoría de las proteínas estructurales son proteínas fibrosas ; por ejemplo, el colágeno y la elastina son componentes críticos del tejido conectivo como el cartílago , y la queratina se encuentra en estructuras duras o filamentosas como el cabello , las uñas , las plumas , las pezuñas y algunos caparazones de animales . [34] : 178–81 Algunas proteínas globulares también pueden desempeñar funciones estructurales, por ejemplo, la actina y la tubulina son globulares y solubles como monómeros, pero se polimerizan para formar fibras largas y rígidas que forman el citoesqueleto , que permite a la célula mantener su forma y tamaño.
Otras proteínas que cumplen funciones estructurales son las proteínas motoras como la miosina , la kinesina y la dineína , que son capaces de generar fuerzas mecánicas. Estas proteínas son cruciales para la motilidad celular de los organismos unicelulares y los espermatozoides de muchos organismos multicelulares que se reproducen sexualmente . También generan las fuerzas ejercidas por los músculos en contracción [34] : 258–64, 272 y desempeñan papeles esenciales en el transporte intracelular.
Una pregunta clave en biología molecular es cómo evolucionan las proteínas, es decir, ¿cómo pueden las mutaciones (o más bien los cambios en la secuencia de aminoácidos ) conducir a nuevas estructuras y funciones? La mayoría de los aminoácidos en una proteína se pueden cambiar sin alterar la actividad o función, como se puede ver en numerosas proteínas homólogas en todas las especies (como se recoge en bases de datos especializadas para familias de proteínas , por ejemplo, PFAM ). [56] Para evitar consecuencias dramáticas de las mutaciones, un gen puede duplicarse antes de que pueda mutar libremente. Sin embargo, esto también puede conducir a la pérdida completa de la función del gen y, por lo tanto, a pseudogenes . [57] Más comúnmente, los cambios de un solo aminoácido tienen consecuencias limitadas, aunque algunos pueden cambiar la función de la proteína sustancialmente, especialmente en enzimas . Por ejemplo, muchas enzimas pueden cambiar su especificidad de sustrato por una o unas pocas mutaciones. [58] Los cambios en la especificidad del sustrato se facilitan por la promiscuidad del sustrato , es decir, la capacidad de muchas enzimas para unirse y procesar múltiples sustratos . Cuando ocurren mutaciones, la especificidad de una enzima puede aumentar (o disminuir) y, por lo tanto, su actividad enzimática. [58] De esta manera, las bacterias (u otros organismos) pueden adaptarse a diferentes fuentes de alimentos, incluidos sustratos no naturales como el plástico. [59]
Los métodos comúnmente utilizados para estudiar la estructura y función de las proteínas incluyen la inmunohistoquímica , la mutagénesis dirigida , la cristalografía de rayos X , la resonancia magnética nuclear y la espectrometría de masas .
Las actividades y estructuras de las proteínas pueden examinarse in vitro , in vivo e in silico . Los estudios in vitro de proteínas purificadas en entornos controlados son útiles para aprender cómo una proteína lleva a cabo su función: por ejemplo, los estudios de cinética enzimática exploran el mecanismo químico de la actividad catalítica de una enzima y su afinidad relativa por varias posibles moléculas de sustrato. Por el contrario, los experimentos in vivo pueden proporcionar información sobre el papel fisiológico de una proteína en el contexto de una célula o incluso de un organismo completo . Los estudios in silico utilizan métodos computacionales para estudiar las proteínas.
Las proteínas pueden purificarse de otros componentes celulares utilizando una variedad de técnicas como ultracentrifugación , precipitación , electroforesis y cromatografía ; el advenimiento de la ingeniería genética ha hecho posible una serie de métodos para facilitar la purificación.
Para realizar un análisis in vitro , una proteína debe purificarse de otros componentes celulares. Este proceso generalmente comienza con la lisis celular , en la que se rompe la membrana de una célula y se libera su contenido interno en una solución conocida como lisado crudo . La mezcla resultante se puede purificar mediante ultracentrifugación , que fracciona los diversos componentes celulares en fracciones que contienen proteínas solubles; lípidos y proteínas de membrana; orgánulos celulares y ácidos nucleicos . La precipitación mediante un método conocido como salinización puede concentrar las proteínas de este lisado. Luego se utilizan varios tipos de cromatografía para aislar la proteína o las proteínas de interés en función de propiedades como el peso molecular, la carga neta y la afinidad de unión. [30] : 21–24 El nivel de purificación se puede monitorear utilizando varios tipos de electroforesis en gel si se conocen el peso molecular y el punto isoeléctrico de la proteína deseada, mediante espectroscopia si la proteína tiene características espectroscópicas distinguibles, o mediante ensayos enzimáticos si la proteína tiene actividad enzimática. Además, las proteínas se pueden aislar según su carga mediante electroenfoque . [60]
En el caso de las proteínas naturales, puede ser necesaria una serie de pasos de purificación para obtener proteínas lo suficientemente puras para aplicaciones de laboratorio. Para simplificar este proceso, a menudo se utiliza la ingeniería genética para añadir características químicas a las proteínas que las hacen más fáciles de purificar sin afectar a su estructura o actividad. Aquí, una "etiqueta" que consiste en una secuencia específica de aminoácidos, a menudo una serie de residuos de histidina (una " etiqueta His "), se une a un extremo de la proteína. Como resultado, cuando el lisado pasa por una columna de cromatografía que contiene níquel , los residuos de histidina se ligan al níquel y se adhieren a la columna mientras que los componentes no etiquetados del lisado pasan sin impedimentos. Se han desarrollado varias etiquetas diferentes para ayudar a los investigadores a purificar proteínas específicas a partir de mezclas complejas. [61]

El estudio de las proteínas in vivo se ocupa a menudo de la síntesis y localización de la proteína dentro de la célula. Aunque muchas proteínas intracelulares se sintetizan en el citoplasma y las proteínas unidas a la membrana o secretadas en el retículo endoplasmático , los detalles de cómo las proteínas se dirigen a orgánulos específicos o estructuras celulares a menudo no están claros. Una técnica útil para evaluar la localización celular utiliza la ingeniería genética para expresar en una célula una proteína de fusión o quimera que consiste en la proteína natural de interés vinculada a un " reportero " como la proteína fluorescente verde (GFP). [62] La posición de la proteína fusionada dentro de la célula se puede visualizar de forma clara y eficiente utilizando microscopía , [63] como se muestra en la figura opuesta.
Otros métodos para determinar la localización celular de las proteínas requieren el uso de marcadores compartimentales conocidos para regiones como el RE, el Golgi, los lisosomas o las vacuolas, las mitocondrias, los cloroplastos, la membrana plasmática, etc. Con el uso de versiones marcadas con fluorescencia de estos marcadores o de anticuerpos contra marcadores conocidos, resulta mucho más sencillo identificar la localización de una proteína de interés. Por ejemplo, la inmunofluorescencia indirecta permitirá la colocalización de la fluorescencia y la demostración de la localización. Los colorantes fluorescentes se utilizan para marcar compartimentos celulares con un propósito similar. [64]
Existen también otras posibilidades. Por ejemplo, la inmunohistoquímica suele utilizar un anticuerpo contra una o más proteínas de interés que se conjugan con enzimas que producen señales luminiscentes o cromogénicas que se pueden comparar entre muestras, lo que permite obtener información de localización. Otra técnica aplicable es la cofracción en gradientes de sacarosa (u otro material) mediante centrifugación isopícnica . [65] Si bien esta técnica no prueba la colocalización de un compartimento de densidad conocida y la proteína de interés, sí aumenta la probabilidad y es más adecuada para estudios a gran escala.
Por último, el método de referencia para la localización celular es la microscopía inmunoelectrónica . Esta técnica también utiliza un anticuerpo contra la proteína de interés, junto con las técnicas clásicas de microscopía electrónica. La muestra se prepara para el examen microscópico electrónico normal y luego se trata con un anticuerpo contra la proteína de interés que está conjugado con un material extremadamente electrodenso, generalmente oro. Esto permite la localización tanto de los detalles ultraestructurales como de la proteína de interés. [66]
Mediante otra aplicación de la ingeniería genética conocida como mutagénesis dirigida , los investigadores pueden alterar la secuencia de proteínas y, por lo tanto, su estructura, localización celular y susceptibilidad a la regulación. Esta técnica incluso permite la incorporación de aminoácidos no naturales a las proteínas, utilizando ARNt modificados, [67] y puede permitir el diseño racional de nuevas proteínas con propiedades novedosas. [68]
El complemento total de proteínas presentes en un momento dado en una célula o tipo de célula se conoce como su proteoma , y el estudio de estos conjuntos de datos a gran escala define el campo de la proteómica , llamado así por analogía con el campo relacionado de la genómica . Las técnicas experimentales clave en proteómica incluyen la electroforesis 2D , [69] que permite la separación de muchas proteínas, la espectrometría de masas , [70] que permite la identificación rápida de alto rendimiento de proteínas y la secuenciación de péptidos (con mayor frecuencia después de la digestión en gel ), los microarreglos de proteínas , que permiten la detección de los niveles relativos de las diversas proteínas presentes en una célula, y el cribado de dos híbridos , que permite la exploración sistemática de las interacciones proteína-proteína . [71] El complemento total de tales interacciones biológicamente posibles se conoce como interactoma . [72] Un intento sistemático de determinar las estructuras de las proteínas que representan cada pliegue posible se conoce como genómica estructural . [73]
Descubrir la estructura terciaria de una proteína, o la estructura cuaternaria de sus complejos, puede proporcionar pistas importantes sobre cómo la proteína realiza su función y cómo puede verse afectada, es decir, en el diseño de fármacos . Como las proteínas son demasiado pequeñas para ser vistas bajo un microscopio óptico , se deben emplear otros métodos para determinar su estructura. Los métodos experimentales comunes incluyen la cristalografía de rayos X y la espectroscopia de RMN , los cuales pueden producir información estructural con resolución atómica . Sin embargo, los experimentos de RMN pueden proporcionar información a partir de la cual se puede estimar un subconjunto de distancias entre pares de átomos, y las conformaciones posibles finales para una proteína se determinan resolviendo un problema de geometría de distancia . La interferometría de polarización dual es un método analítico cuantitativo para medir la conformación general de la proteína y los cambios conformacionales debidos a interacciones u otros estímulos. El dicroísmo circular es otra técnica de laboratorio para determinar la composición interna de lámina β / hélice α de las proteínas. La microscopía crioelectrónica se utiliza para producir información estructural de menor resolución sobre complejos proteicos muy grandes, incluidos los virus ensamblados ; [33] : 340–41 una variante conocida como cristalografía electrónica también puede producir información de alta resolución en algunos casos, especialmente para cristales bidimensionales de proteínas de membrana. [74] Las estructuras resueltas generalmente se depositan en el Protein Data Bank (PDB), un recurso de libre acceso del cual se pueden obtener datos estructurales sobre miles de proteínas en forma de coordenadas cartesianas para cada átomo de la proteína. [75]
Se conocen muchas más secuencias de genes que estructuras de proteínas. Además, el conjunto de estructuras resueltas está sesgado hacia las proteínas que pueden someterse fácilmente a las condiciones requeridas en la cristalografía de rayos X , uno de los principales métodos de determinación de la estructura. En particular, las proteínas globulares son comparativamente fáciles de cristalizar en preparación para la cristalografía de rayos X. Las proteínas de membrana y los complejos proteicos grandes, por el contrario, son difíciles de cristalizar y están subrepresentados en el PDB. [76] Las iniciativas de genómica estructural han intentado remediar estas deficiencias mediante la resolución sistemática de estructuras representativas de las principales clases de plegamiento. Los métodos de predicción de la estructura de proteínas intentan proporcionar un medio para generar una estructura plausible para proteínas cuyas estructuras no se han determinado experimentalmente. [77]

Como complemento del campo de la genómica estructural, la predicción de la estructura de proteínas desarrolla modelos matemáticos eficientes de proteínas para predecir computacionalmente las formaciones moleculares en teoría, en lugar de detectar estructuras con observaciones de laboratorio. [78] El tipo más exitoso de predicción de estructura, conocido como modelado de homología , se basa en la existencia de una estructura "plantilla" con similitud de secuencia con la proteína que se está modelando; el objetivo de la genómica estructural es proporcionar una representación suficiente en estructuras resueltas para modelar la mayoría de las que quedan. [79] Aunque producir modelos precisos sigue siendo un desafío cuando solo se encuentran disponibles estructuras plantilla distantemente relacionadas, se ha sugerido que la alineación de secuencias es el cuello de botella en este proceso, ya que se pueden producir modelos bastante precisos si se conoce una alineación de secuencias "perfecta". [80] Muchos métodos de predicción de estructura han servido para informar el campo emergente de la ingeniería de proteínas , en el que ya se han diseñado nuevos pliegues de proteínas. [81] Además, las proteínas (en eucariotas ~33%) contienen grandes segmentos no estructurados pero biológicamente funcionales y pueden clasificarse como proteínas intrínsecamente desordenadas . [82] Por lo tanto, predecir y analizar el desorden de las proteínas es una parte importante de la caracterización de la estructura de las proteínas. [83]
Se ha desarrollado una amplia gama de métodos computacionales para analizar la estructura, función y evolución de las proteínas. El desarrollo de tales herramientas ha sido impulsado por la gran y creciente cantidad de datos genómicos y proteómicos disponibles para una variedad de organismos, incluido el genoma humano . No existen los recursos para estudiar todas las proteínas experimentalmente, por lo que solo unas pocas se someten a experimentos de laboratorio mientras que las herramientas computacionales se utilizan para extrapolar a proteínas similares. Tales proteínas homólogas se pueden identificar de manera eficiente en organismos distantemente relacionados mediante la alineación de secuencias . Las secuencias de genomas y genes se pueden buscar mediante una variedad de herramientas para ciertas propiedades. Las herramientas de perfilado de secuencias pueden encontrar sitios de enzimas de restricción , marcos de lectura abiertos en secuencias de nucleótidos y predecir estructuras secundarias . Se pueden construir árboles filogenéticos y desarrollar hipótesis evolutivas utilizando software especial como Clustal W con respecto a la ascendencia de los organismos modernos y los genes que expresan. El campo de la bioinformática ahora es indispensable para el análisis de genes y proteínas.
Un problema computacional más complejo es la predicción de interacciones intermoleculares, como en el acoplamiento molecular , [84] el plegamiento de proteínas , la interacción proteína-proteína y la reactividad química. Los modelos matemáticos para simular estos procesos dinámicos involucran la mecánica molecular , en particular, la dinámica molecular . En este sentido, las simulaciones in silico descubrieron el plegamiento de pequeños dominios proteicos α-helicoidales como la cabeza de la villina , [85] la proteína accesoria del VIH [86] y los métodos híbridos que combinan la dinámica molecular estándar con las matemáticas de la mecánica cuántica han explorado los estados electrónicos de las rodopsinas . [87]
Más allá de la dinámica molecular clásica, los métodos de dinámica cuántica permiten la simulación de proteínas con detalles atómicos y una descripción precisa de los efectos mecánicos cuánticos. Algunos ejemplos son el método Hartree multicapa multiconfiguración dependiente del tiempo (MCTDH) y el enfoque de ecuaciones jerárquicas de movimiento (HEOM), que se han aplicado a criptocromos de plantas [88] y complejos de recolección de luz de bacterias [89] , respectivamente. Tanto las simulaciones mecánicas cuánticas como las clásicas de sistemas a escala biológica son extremadamente exigentes desde el punto de vista computacional, por lo que las iniciativas de computación distribuida (por ejemplo, el proyecto Folding@home [90] ) facilitan el modelado molecular al explotar los avances en el procesamiento paralelo de GPU y las técnicas de Monte Carlo .
El contenido total de nitrógeno de la materia orgánica está formado principalmente por los grupos amino de las proteínas. El nitrógeno total Kjeldahl ( TKN ) es una medida de nitrógeno ampliamente utilizada en el análisis de aguas (residuales), suelo, alimentos, piensos y materia orgánica en general. Como sugiere el nombre, se aplica el método Kjeldahl . Hay métodos más sensibles disponibles. [91] [92]
La mayoría de los microorganismos y plantas pueden biosintetizar los 20 aminoácidos estándar , mientras que los animales (incluidos los humanos) deben obtener algunos de los aminoácidos de la dieta . [47] Los aminoácidos que un organismo no puede sintetizar por sí solo se denominan aminoácidos esenciales . Las enzimas clave que sintetizan ciertos aminoácidos no están presentes en los animales, como la aspartoquinasa , que cataliza el primer paso en la síntesis de lisina , metionina y treonina a partir del aspartato . Si los aminoácidos están presentes en el medio ambiente, los microorganismos pueden conservar energía absorbiendo los aminoácidos de su entorno y regulando a la baja sus vías biosintéticas.
En los animales, los aminoácidos se obtienen a través del consumo de alimentos que contienen proteínas. Las proteínas ingeridas se descomponen luego en aminoácidos a través de la digestión , que generalmente implica la desnaturalización de la proteína mediante la exposición al ácido y la hidrólisis por enzimas llamadas proteasas . Algunos aminoácidos ingeridos se utilizan para la biosíntesis de proteínas, mientras que otros se convierten en glucosa a través de la gluconeogénesis o se introducen en el ciclo del ácido cítrico . Este uso de proteínas como combustible es particularmente importante en condiciones de inanición , ya que permite que las propias proteínas del cuerpo se utilicen para sustentar la vida, en particular las que se encuentran en los músculos . [93]
En animales como perros y gatos , las proteínas mantienen la salud y la calidad de la piel al promover el crecimiento del folículo piloso y la queratinización, y por lo tanto reducir la probabilidad de problemas de la piel que produzcan malos olores. [94] Las proteínas de mala calidad también tienen un papel en la salud gastrointestinal, aumentando el potencial de flatulencia y compuestos olorosos en los perros porque cuando las proteínas llegan al colon en un estado no digerido, se fermentan produciendo gas de sulfuro de hidrógeno, indol y escatol. [95] Los perros y los gatos digieren mejor las proteínas animales que las de las plantas, pero los productos de origen animal de baja calidad se digieren mal, incluida la piel, las plumas y el tejido conectivo. [95]
Las propiedades mecánicas de las proteínas son muy diversas y a menudo son fundamentales para su función biológica, como en el caso de proteínas como la queratina y el colágeno . [96] Por ejemplo, la capacidad del tejido muscular de expandirse y contraerse continuamente está directamente relacionada con las propiedades elásticas de su composición proteica subyacente. [97] [98] Más allá de las proteínas fibrosas, la dinámica conformacional de las enzimas [99] y la estructura de las membranas biológicas , entre otras funciones biológicas, están gobernadas por las propiedades mecánicas de las proteínas. Fuera de su contexto biológico, las propiedades mecánicas únicas de muchas proteínas, junto con su relativa sostenibilidad en comparación con los polímeros sintéticos , las han convertido en objetivos deseables para el diseño de materiales de próxima generación. [100] [101]
El módulo de Young , E, se calcula como la tensión axial σ sobre la deformación resultante ε. Es una medida de la rigidez relativa de un material. En el contexto de las proteínas, esta rigidez a menudo se correlaciona directamente con la función biológica. Por ejemplo, el colágeno , que se encuentra en el tejido conectivo , los huesos y el cartílago , y la queratina , que se encuentra en las uñas, las garras y el cabello, han observado rigideces que son varios órdenes de magnitud más altas que las de la elastina , [102] que se cree que da elasticidad a estructuras como los vasos sanguíneos , el tejido pulmonar y el tejido de la vejiga , entre otros. [103] [104] En comparación con esto, las proteínas globulares , como la albúmina de suero bovino , que flotan relativamente libremente en el citosol y a menudo funcionan como enzimas (y, por lo tanto, experimentan cambios conformacionales frecuentes) tienen módulos de Young comparativamente mucho más bajos. [105] [106]
El módulo de Young de una proteína individual se puede determinar mediante simulación de dinámica molecular . Utilizando campos de fuerza atómicos, como CHARMM o GROMOS , o campos de fuerza de grano grueso como Martini, [107] una molécula de proteína individual se puede estirar mediante una fuerza uniaxial mientras se registra la extensión resultante para calcular la deformación. [108] [109] Experimentalmente, se pueden utilizar métodos como la microscopía de fuerza atómica para obtener datos similares. [110]
A nivel macroscópico, el módulo de Young de las redes de proteínas reticuladas se puede obtener mediante pruebas mecánicas más tradicionales . A continuación se muestran los valores observados experimentalmente para algunas proteínas.
Además de servir como enzimas dentro de la célula, las proteínas globulares a menudo actúan como moléculas de transporte clave. Por ejemplo, las albúminas séricas , un componente clave de la sangre , son necesarias para el transporte de una multitud de pequeñas moléculas por todo el cuerpo. [114] Debido a esto, el comportamiento dependiente de la concentración de estas proteínas en solución está directamente relacionado con la función del sistema circulatorio . Una forma de cuantificar este comportamiento es a través de la viscosidad de la solución.
La viscosidad, η, se da generalmente como una medida de la resistencia de un fluido a la deformación. Se puede calcular como la relación entre la tensión aplicada y la tasa de cambio de la deformación cortante resultante, es decir, la tasa de deformación. La viscosidad de mezclas líquidas complejas, como la sangre, a menudo depende en gran medida de la temperatura y la concentración de soluto. [115] Para la albúmina sérica, específicamente la albúmina sérica bovina , se puede utilizar la siguiente relación entre la viscosidad y la temperatura y la concentración . [116]
Donde c es la concentración, T es la temperatura, R es la constante de los gases y α, β, B , D y Δ E son todas constantes de propiedades basadas en el material. Esta ecuación tiene la forma de una ecuación de Arrhenius , que asigna a la viscosidad una dependencia exponencial de la temperatura y la concentración.
{{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace )