
La biosíntesis de proteínas (o síntesis de proteínas ) es un proceso biológico central que ocurre dentro de las células y equilibra la pérdida de proteínas celulares (a través de la degradación o exportación ) mediante la producción de nuevas proteínas. Las proteínas desempeñan una serie de funciones críticas como enzimas , proteínas estructurales u hormonas . La síntesis de proteínas es un proceso muy similar tanto para procariotas como para eucariotas , pero existen algunas diferencias claras. [1]
La síntesis de proteínas se puede dividir a grandes rasgos en dos fases: transcripción y traducción . Durante la transcripción, una sección de ADN que codifica una proteína, conocida como gen , se convierte en una molécula plantilla llamada ARN mensajero (ARNm). Esta conversión la llevan a cabo enzimas, conocidas como ARN polimerasas , en el núcleo de la célula . [2] En eucariotas, este ARNm se produce inicialmente en una forma prematura ( pre-ARNm ) que sufre modificaciones postranscripcionales para producir ARNm maduro . El ARNm maduro se exporta desde el núcleo celular a través de poros nucleares al citoplasma de la célula para que se produzca la traducción. Durante la traducción, los ribosomas leen el ARNm y utilizan la secuencia de nucleótidos del ARNm para determinar la secuencia de aminoácidos . Los ribosomas catalizan la formación de enlaces peptídicos covalentes entre los aminoácidos codificados para formar una cadena polipeptídica .
Después de la traducción, la cadena polipeptídica debe plegarse para formar una proteína funcional; por ejemplo, para funcionar como enzima, la cadena polipeptídica debe plegarse correctamente para producir un sitio activo funcional . Para adoptar una forma tridimensional funcional, la cadena polipeptídica debe formar primero una serie de estructuras subyacentes más pequeñas llamadas estructuras secundarias . La cadena polipeptídica en estas estructuras secundarias luego se pliega para producir la estructura terciaria tridimensional general . Una vez plegada correctamente, la proteína puede sufrir una mayor maduración a través de diferentes modificaciones postraduccionales , que pueden alterar la capacidad de funcionamiento de la proteína, su ubicación dentro de la célula (por ejemplo, citoplasma o núcleo) y su capacidad para interactuar con otras proteínas . [3]
La biosíntesis de proteínas tiene un papel clave en las enfermedades, ya que los cambios y errores en este proceso, a través de mutaciones subyacentes del ADN o el mal plegamiento de las proteínas , suelen ser las causas subyacentes de una enfermedad. Las mutaciones del ADN cambian la secuencia posterior del ARNm, que luego altera la secuencia de aminoácidos codificada por el ARNm. Las mutaciones pueden hacer que la cadena polipeptídica sea más corta al generar una secuencia de parada que provoca la terminación temprana de la traducción. Alternativamente, una mutación en la secuencia del ARNm cambia el aminoácido específico codificado en esa posición en la cadena polipeptídica. Este cambio de aminoácidos puede afectar la capacidad de la proteína para funcionar o plegarse correctamente. [4] Las proteínas mal plegadas tienen tendencia a formar densos grupos de proteínas , que a menudo están implicados en enfermedades, particularmente trastornos neurológicos , como el Alzheimer y el Parkinson . [5]
La transcripción se produce en el núcleo utilizando el ADN como plantilla para producir ARNm . En eucariotas , esta molécula de ARNm se conoce como pre-ARNm ya que sufre modificaciones postranscripcionales en el núcleo para producir una molécula de ARNm madura. Sin embargo, en los procariotas no se requieren modificaciones postranscripcionales, por lo que la molécula de ARNm madura se produce inmediatamente mediante la transcripción. [1]
Inicialmente, una enzima conocida como helicasa actúa sobre la molécula de ADN. El ADN tiene una estructura de doble hélice antiparalela compuesta por dos cadenas de polinucleótidos complementarias , unidas por enlaces de hidrógeno entre los pares de bases. La helicasa rompe los enlaces de hidrógeno provocando que una región del ADN, correspondiente a un gen, se desenrolle, separando las dos hebras de ADN y exponiendo una serie de bases. A pesar de que el ADN es una molécula de doble hebra, sólo una de las hebras actúa como plantilla para la síntesis de pre-ARNm; esta hebra se conoce como hebra plantilla. La otra cadena de ADN (que es complementaria a la cadena plantilla) se conoce como cadena codificante. [6]
Tanto el ADN como el ARN tienen direccionalidad intrínseca , lo que significa que hay dos extremos distintos de la molécula. Esta propiedad de direccionalidad se debe a las subunidades de nucleótidos subyacentes asimétricas, con un grupo fosfato en un lado del azúcar pentosa y una base en el otro. Los cinco carbonos del azúcar pentosa están numerados desde 1' (donde 'significa primo) hasta 5'. Por lo tanto, los enlaces fosfodiéster que conectan los nucleótidos se forman uniendo el grupo hidroxilo en el carbono 3' de un nucleótido al grupo fosfato en el carbono 5' de otro nucleótido. Por lo tanto, la cadena codificante de ADN corre en la dirección 5' a 3' y la cadena de ADN molde complementaria corre en la dirección opuesta de 3' a 5'. [1]
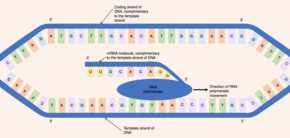
La enzima ARN polimerasa se une a la cadena plantilla expuesta y lee el gen en la dirección 3' a 5'. Simultáneamente, la ARN polimerasa sintetiza una única hebra de pre-ARNm en la dirección 5' a 3' catalizando la formación de enlaces fosfodiéster entre nucleótidos activados (libres en el núcleo) que son capaces de aparearse bases complementarias con la hebra plantilla. . Detrás de la ARN polimerasa en movimiento, las dos hebras de ADN se vuelven a unir, por lo que sólo 12 pares de bases de ADN quedan expuestos a la vez. [6] La ARN polimerasa construye la molécula de pre-ARNm a una velocidad de 20 nucleótidos por segundo, lo que permite la producción de miles de moléculas de pre-ARNm a partir del mismo gen en una hora. A pesar de la rápida velocidad de síntesis, la enzima ARN polimerasa contiene su propio mecanismo de corrección. Los mecanismos de corrección permiten que la ARN polimerasa elimine los nucleótidos incorrectos (que no son complementarios a la cadena plantilla de ADN) de la molécula de pre-ARNm en crecimiento mediante una reacción de escisión. [1] Cuando las ARN polimerasas alcanzan una secuencia de ADN específica que finaliza la transcripción, la ARN polimerasa se desprende y se completa la síntesis de pre-ARNm. [6]
La molécula de pre-ARNm sintetizada es complementaria a la cadena de ADN molde y comparte la misma secuencia de nucleótidos que la cadena de ADN codificante. Sin embargo, existe una diferencia crucial en la composición de nucleótidos de las moléculas de ADN y ARNm. El ADN está compuesto por las bases: guanina , citosina , adenina y timina (G, C, A y T). El ARN también está compuesto por cuatro bases: guanina, citosina, adenina y uracilo . En las moléculas de ARN, la base del ADN, timina, es reemplazada por uracilo, que puede formar pares de bases con adenina. Por tanto, en la molécula de pre-ARNm, todas las bases complementarias que serían timina en la cadena de ADN codificante son reemplazadas por uracilo. [7]

Una vez que se completa la transcripción, la molécula de pre-ARNm sufre modificaciones postranscripcionales para producir una molécula de ARNm madura.
Hay 3 pasos clave dentro de las modificaciones postranscripcionales: [ cita necesaria ]
La tapa 5' se agrega al extremo 5' de la molécula de pre-ARNm y está compuesta por un nucleótido de guanina modificado mediante metilación . El propósito de la tapa 5' es evitar la descomposición de las moléculas de ARNm maduras antes de la traducción; la tapa también ayuda a la unión del ribosoma al ARNm para iniciar la traducción [8] y permite diferenciar el ARNm de otros ARN en la célula. [1] Por el contrario, la cola 3' Poly(A) se agrega al extremo 3' de la molécula de ARNm y está compuesta por 100-200 bases de adenina. [8] Estas distintas modificaciones del ARNm permiten a la célula detectar que el mensaje completo del ARNm está intacto si tanto la tapa 5' como la cola 3' están presentes. [1]
Esta molécula de pre-ARNm modificada luego se somete al proceso de empalme de ARN. Los genes están compuestos por una serie de intrones y exones , los intrones son secuencias de nucleótidos que no codifican una proteína, mientras que los exones son secuencias de nucleótidos que codifican directamente una proteína. Los intrones y exones están presentes tanto en la secuencia de ADN subyacente como en la molécula de pre-ARNm, por lo tanto, para producir una molécula de ARNm madura que codifique una proteína, debe producirse un empalme. [6] Durante el empalme, los intrones intervinientes se eliminan de la molécula de pre-ARNm mediante un complejo multiproteico conocido como espliceosoma (compuesto por más de 150 proteínas y ARN). [9] Esta molécula de ARNm madura luego se exporta al citoplasma a través de poros nucleares en la envoltura del núcleo.

Durante la traducción, los ribosomas sintetizan cadenas polipeptídicas a partir de moléculas plantilla de ARNm. En los eucariotas, la traducción se produce en el citoplasma de la célula, donde se encuentran los ribosomas, ya sea flotando libremente o adheridos al retículo endoplásmico . En los procariotas, que carecen de núcleo, los procesos tanto de transcripción como de traducción ocurren en el citoplasma. [10]
Los ribosomas son máquinas moleculares complejas , formadas por una mezcla de proteínas y ARN ribosomal , dispuestas en dos subunidades (una grande y otra pequeña), que rodean la molécula de ARNm. El ribosoma lee la molécula de ARNm en dirección 5'-3' y la utiliza como plantilla para determinar el orden de los aminoácidos en la cadena polipeptídica. [11] Para traducir la molécula de ARNm, el ribosoma utiliza moléculas pequeñas, conocidas como ARN de transferencia (ARNt), para entregar los aminoácidos correctos al ribosoma. Cada ARNt está compuesto por 70-80 nucleótidos y adopta una estructura de hoja de trébol característica debido a la formación de enlaces de hidrógeno entre los nucleótidos dentro de la molécula. Hay alrededor de 60 tipos diferentes de ARNt, cada ARNt se une a una secuencia específica de tres nucleótidos (conocida como codón ) dentro de la molécula de ARNm y libera un aminoácido específico. [12]
El ribosoma inicialmente se une al ARNm en el codón de inicio (AUG) y comienza a traducir la molécula. La secuencia de nucleótidos del ARNm se lee en tripletes ; tres nucleótidos adyacentes en la molécula de ARNm corresponden a un solo codón. Cada ARNt tiene una secuencia expuesta de tres nucleótidos, conocida como anticodón, que son complementarios en secuencia a un codón específico que puede estar presente en el ARNm. Por ejemplo, el primer codón encontrado es el codón de inicio compuesto por los nucleótidos AUG. El ARNt correcto con el anticodón (secuencia complementaria de 3 nucleótidos UAC) se une al ARNm mediante el ribosoma. Este ARNt entrega el aminoácido correcto correspondiente al codón del ARNm; en el caso del codón de inicio, este es el aminoácido metionina. Luego, el siguiente codón (adyacente al codón de inicio) se une mediante el ARNt correcto con un anticodón complementario, entregando el siguiente aminoácido al ribosoma. Luego, el ribosoma utiliza su actividad enzimática peptidil transferasa para catalizar la formación del enlace peptídico covalente entre los dos aminoácidos adyacentes. [6]
Luego, el ribosoma se mueve a lo largo de la molécula de ARNm hasta el tercer codón. Luego, el ribosoma libera la primera molécula de ARNt, ya que un solo ribosoma solo puede reunir dos moléculas de ARNt a la vez. Se selecciona el siguiente ARNt complementario con el anticodón correcto complementario al tercer codón, entregando el siguiente aminoácido al ribosoma que está unido covalentemente a la cadena polipeptídica en crecimiento. Este proceso continúa con el ribosoma moviéndose a lo largo de la molécula de ARNm agregando hasta 15 aminoácidos por segundo a la cadena polipeptídica. Detrás del primer ribosoma, hasta 50 ribosomas adicionales pueden unirse a la molécula de ARNm formando un polisoma , lo que permite la síntesis simultánea de múltiples cadenas polipeptídicas idénticas. [6] La terminación de la cadena polipeptídica en crecimiento ocurre cuando el ribosoma encuentra un codón de parada (UAA, UAG o UGA) en la molécula de ARNm. Cuando esto ocurre, ningún ARNt puede reconocerlo y un factor de liberación induce la liberación de la cadena polipeptídica completa del ribosoma. [12] El Dr. Har Gobind Khorana , un científico originario de la India, decodificó las secuencias de ARN de unos 20 aminoácidos. [ cita necesaria ] Recibió el premio Nobel en 1968, junto con otros dos científicos, por su trabajo.

Una vez que se completa la síntesis de la cadena polipeptídica, la cadena polipeptídica se pliega para adoptar una estructura específica que permite a la proteína llevar a cabo sus funciones. La forma básica de estructura proteica se conoce como estructura primaria , que es simplemente la cadena polipeptídica, es decir, una secuencia de aminoácidos unidos covalentemente. La estructura primaria de una proteína está codificada por un gen. Por lo tanto, cualquier cambio en la secuencia del gen puede alterar la estructura primaria de la proteína y todos los niveles posteriores de la estructura de la proteína, cambiando en última instancia la estructura y función generales. [ cita necesaria ]
La estructura primaria de una proteína (la cadena polipeptídica) puede luego plegarse o enrollarse para formar la estructura secundaria de la proteína. Los tipos más comunes de estructura secundaria se conocen como hélice alfa o lámina beta , estas son pequeñas estructuras producidas por enlaces de hidrógeno que se forman dentro de la cadena polipeptídica. Esta estructura secundaria luego se pliega para producir la estructura terciaria de la proteína. La estructura terciaria es la estructura 3D general de las proteínas que está formada por diferentes estructuras secundarias que se pliegan entre sí. En la estructura terciaria, las características clave de la proteína, por ejemplo el sitio activo, se pliegan y forman, lo que permite que la proteína funcione. Finalmente, algunas proteínas pueden adoptar una estructura cuaternaria compleja . La mayoría de las proteínas están formadas por una sola cadena polipeptídica; sin embargo, algunas proteínas están compuestas por múltiples cadenas polipeptídicas (conocidas como subunidades) que se pliegan e interactúan para formar la estructura cuaternaria. Por lo tanto, la proteína global es un complejo de múltiples subunidades compuesto de múltiples subunidades de cadenas polipeptídicas plegadas, por ejemplo, hemoglobina . [13]
Hay eventos que siguen a la biosíntesis de proteínas, como la proteólisis [14] y el plegamiento de proteínas. La proteólisis se refiere a la división de proteínas por proteasas y la descomposición de proteínas en aminoácidos por la acción de enzimas.
Cuando se completa el plegamiento de proteínas hacia el estado 3D funcional y maduro, no es necesariamente el final de la vía de maduración de las proteínas. Una proteína plegada aún puede sufrir un procesamiento adicional mediante modificaciones postraduccionales. Hay más de 200 tipos conocidos de modificación postraduccional; estas modificaciones pueden alterar la actividad de la proteína, la capacidad de la proteína para interactuar con otras proteínas y el lugar donde se encuentra la proteína dentro de la célula, por ejemplo, en el núcleo o el citoplasma de la célula. [15] A través de modificaciones postraduccionales, la diversidad de proteínas codificadas por el genoma se amplía de 2 a 3 órdenes de magnitud . [dieciséis]
Hay cuatro clases clave de modificación postraduccional: [3]
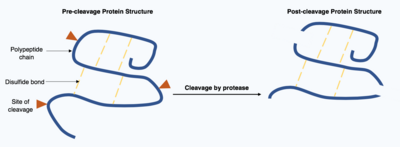
La escisión de proteínas es una modificación postraduccional irreversible llevada a cabo por enzimas conocidas como proteasas . Estas proteasas suelen ser muy específicas y provocan la hidrólisis de un número limitado de enlaces peptídicos dentro de la proteína diana. La proteína acortada resultante tiene una cadena polipeptídica alterada con diferentes aminoácidos al principio y al final de la cadena. Esta modificación postraduccional a menudo altera la función de las proteínas, la proteína puede ser inactivada o activada por la escisión y puede mostrar nuevas actividades biológicas. [17]

Después de la traducción, se pueden agregar pequeños grupos químicos a los aminoácidos dentro de la estructura de la proteína madura. [18] Ejemplos de procesos que agregan grupos químicos a la proteína objetivo incluyen metilación, acetilación y fosforilación .
La metilación es la adición reversible de un grupo metilo a un aminoácido catalizada por enzimas metiltransferasas . La metilación ocurre en al menos 9 de los 20 aminoácidos comunes, sin embargo, ocurre principalmente en los aminoácidos lisina y arginina . Un ejemplo de una proteína que comúnmente está metilada es una histona . Las histonas son proteínas que se encuentran en el núcleo de la célula. El ADN está fuertemente envuelto alrededor de las histonas y se mantiene en su lugar mediante otras proteínas y las interacciones entre las cargas negativas del ADN y las cargas positivas de la histona. Se utiliza un patrón altamente específico de metilación de aminoácidos en las proteínas histonas para determinar qué regiones del ADN están estrechamente enrolladas y no pueden transcribirse y qué regiones están sueltas y pueden transcribirse. [19]
La regulación de la transcripción del ADN basada en histonas también se modifica mediante acetilación. La acetilación es la adición covalente reversible de un grupo acetilo a un aminoácido de lisina mediante la enzima acetiltransferasa . El grupo acetilo se elimina de una molécula donante conocida como acetil coenzima A y se transfiere a la proteína objetivo. [20] Las histonas sufren acetilación en sus residuos de lisina mediante enzimas conocidas como histona acetiltransferasa . El efecto de la acetilación es debilitar las interacciones de carga entre la histona y el ADN, haciendo así que más genes del ADN sean accesibles para la transcripción. [21]
La última modificación de grupo químico postraduccional prevalente es la fosforilación. La fosforilación es la adición covalente reversible de un grupo fosfato a aminoácidos específicos ( serina , treonina y tirosina ) dentro de la proteína. El grupo fosfato se elimina de la molécula donante ATP mediante una proteína quinasa y se transfiere al grupo hidroxilo del aminoácido objetivo, lo que produce adenosina difosfato como biproducto. Este proceso puede revertirse y eliminarse el grupo fosfato mediante la enzima proteína fosfatasa . La fosforilación puede crear un sitio de unión en la proteína fosforilada que le permite interactuar con otras proteínas y generar grandes complejos multiproteicos. Alternativamente, la fosforilación puede cambiar el nivel de actividad de la proteína alterando la capacidad de la proteína para unirse a su sustrato. [1]
Las modificaciones postraduccionales pueden incorporar moléculas grandes y más complejas en la estructura de la proteína plegada. Un ejemplo común de esto es la glicosilación , la adición de una molécula de polisacárido, que se considera ampliamente como la modificación postraduccional más común. [dieciséis]
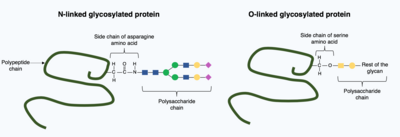
En la glicosilación, las enzimas glicosiltransferasas añaden covalentemente una molécula de polisacárido (conocida como glicano ) a la proteína objetivo y la modifican las glicosidasas en el retículo endoplásmico y el aparato de Golgi . La glicosilación puede tener un papel fundamental en la determinación de la estructura tridimensional plegada final de la proteína objetivo. En algunos casos es necesaria la glicosilación para el correcto plegamiento. "La glicosilación ligada a N promueve el plegamiento de proteínas al aumentar la solubilidad y media la unión de proteínas a las chaperonas proteicas ". Las chaperonas son proteínas responsables de plegar y mantener la estructura de otras proteínas. [1]
En términos generales , existen dos tipos de glicosilación, la glicosilación ligada a N y la glicosilación ligada a O. La glicosilación ligada a N comienza en el retículo endoplásmico con la adición de un glicano precursor. El glicano precursor se modifica en el aparato de Golgi para producir un glicano complejo unido covalentemente al nitrógeno en un aminoácido de asparagina . Por el contrario, la glicosilación ligada a O es la adición covalente secuencial de azúcares individuales al oxígeno en los aminoácidos serina y treonina dentro de la estructura de la proteína madura. [1]
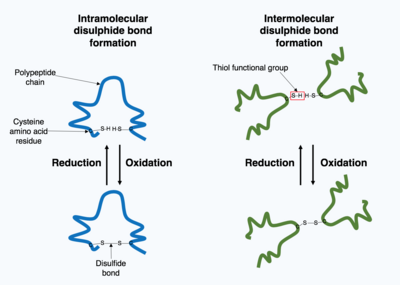
Muchas proteínas producidas dentro de la célula se secretan fuera de la célula para funcionar como proteínas extracelulares . Las proteínas extracelulares están expuestas a una amplia variedad de condiciones. Para estabilizar la estructura de la proteína 3D, se forman enlaces covalentes dentro de la proteína o entre las diferentes cadenas polipeptídicas de la estructura cuaternaria. El tipo más frecuente es el enlace disulfuro (también conocido como puente disulfuro). Se forma un enlace disulfuro entre dos aminoácidos de cisteína utilizando sus grupos químicos de cadena lateral que contienen un átomo de azufre; estos grupos químicos se conocen como grupos funcionales tiol . Los enlaces disulfuro actúan para estabilizar la estructura preexistente de la proteína. Los enlaces disulfuro se forman en una reacción de oxidación entre dos grupos tiol y, por lo tanto, necesitan un ambiente oxidante para reaccionar. Como resultado, los enlaces disulfuro generalmente se forman en el ambiente oxidante del retículo endoplásmico catalizado por enzimas llamadas proteínas disulfuro isomerasas. Rara vez se forman enlaces disulfuro en el citoplasma, ya que es un entorno reductor. [1]
Muchas enfermedades son causadas por mutaciones en genes, debido a la conexión directa entre la secuencia de nucleótidos del ADN y la secuencia de aminoácidos de la proteína codificada. Los cambios en la estructura primaria de la proteína pueden provocar que la proteína se pliegue mal o funcione mal. Se han identificado mutaciones dentro de un solo gen como causa de múltiples enfermedades, incluida la anemia de células falciformes , conocida como trastornos de un solo gen.

La anemia de células falciformes es un grupo de enfermedades causadas por una mutación en una subunidad de la hemoglobina, una proteína que se encuentra en los glóbulos rojos encargada de transportar oxígeno. La más peligrosa de las enfermedades de células falciformes se conoce como anemia de células falciformes. La anemia de células falciformes es el trastorno homocigótico recesivo de un solo gen más común , lo que significa que el individuo afectado debe portar una mutación en ambas copias del gen afectado (una heredada de cada padre) para experimentar la enfermedad. La hemoglobina tiene una estructura cuaternaria compleja y está compuesta por cuatro subunidades polipeptídicas: dos subunidades A y dos subunidades B. [22] Los pacientes con anemia falciforme tienen una mutación sin sentido o de sustitución en el gen que codifica la cadena polipeptídica de la subunidad B de la hemoglobina. Una mutación sin sentido significa que la mutación del nucleótido altera el triplete de codones general de modo que un aminoácido diferente se empareja con el nuevo codón. En el caso de la anemia falciforme, la mutación sin sentido más común es una mutación de un solo nucleótido de timina a adenina en el gen de la subunidad B de la hemoglobina. [23] Esto cambia el codón 6 de codificar el aminoácido ácido glutámico a codificar valina. [22]
Este cambio en la estructura primaria de la cadena polipeptídica de la subunidad B de la hemoglobina altera la funcionalidad del complejo de subunidades múltiples de la hemoglobina en condiciones de bajo nivel de oxígeno. Cuando los glóbulos rojos descargan oxígeno en los tejidos del cuerpo, la proteína hemoglobina mutada comienza a unirse para formar una estructura semisólida dentro del glóbulo rojo. Esto distorsiona la forma de los glóbulos rojos, dando como resultado la característica forma de "hoz" y reduce la flexibilidad celular. Estos glóbulos rojos rígidos y distorsionados pueden acumularse en los vasos sanguíneos creando una obstrucción. El bloqueo impide el flujo sanguíneo a los tejidos y puede provocar la muerte del tejido , lo que causa un gran dolor al individuo. [24]
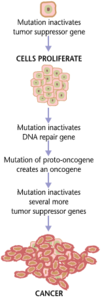
Los cánceres se forman como resultado de mutaciones genéticas y de una traducción inadecuada de proteínas. Además de que las células cancerosas proliferen anormalmente, suprimen la expresión de genes o proteínas antiapoptóticos o proapoptóticos. La mayoría de las células cancerosas ven una mutación en la proteína de señalización Ras, que funciona como un transductor de señales de encendido/apagado en las células. En las células cancerosas, la proteína RAS se vuelve activa de forma persistente, favoreciendo así la proliferación de la célula debido a la ausencia de regulación. [25] Además, la mayoría de las células cancerosas llevan dos copias mutantes del gen regulador p53, que actúa como guardián de los genes dañados e inicia la apoptosis en las células malignas. En su ausencia, la célula no puede iniciar la apoptosis ni enviar señales a otras células para que la destruyan. [26]
A medida que las células tumorales proliferan, permanecen confinadas a un área y se denominan benignas, o se convierten en células malignas que migran a otras áreas del cuerpo. A menudo, estas células malignas secretan proteasas que rompen la matriz extracelular de los tejidos. Esto luego permite que el cáncer entre en su etapa terminal llamada Metástasis, en la que las células ingresan al torrente sanguíneo o al sistema linfático para viajar a una nueva parte del cuerpo. [25]
{{cite book}}: Mantenimiento CS1: ubicación ( enlace )