El metabolismo de las purinas se refiere a las vías metabólicas para sintetizar y descomponer las purinas que están presentes en muchos organismos.
Las purinas se sintetizan biológicamente como nucleótidos y, en particular, como ribotidas, es decir, bases unidas a la ribosa 5-fosfato . Tanto la adenina como la guanina se derivan del nucleótido inosina monofosfato (IMP), que es el primer compuesto de la vía que tiene un sistema de anillos de purina completamente formado.
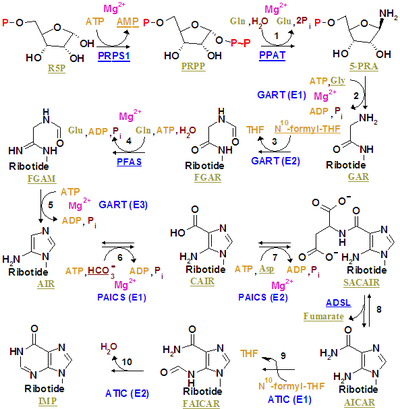
El monofosfato de inosina se sintetiza en una ribosa-fosfato preexistente a través de una vía compleja (como se muestra en la figura de la derecha). La fuente de los átomos de carbono y nitrógeno del anillo de purina, 5 y 4 respectivamente, proviene de múltiples fuentes. El aminoácido glicina aporta todos sus átomos de carbono (2) y nitrógeno (1), con átomos de nitrógeno adicionales de la glutamina (2) y el ácido aspártico (1), y átomos de carbono adicionales de los grupos formilo (2), que se transfieren desde el tetrahidrofolato de coenzima como 10-formiltetrahidrofolato y un átomo de carbono del bicarbonato (1). Los grupos formilo forman carbono 2 y carbono 8 en el sistema de anillos de purina, que son los que actúan como puentes entre dos átomos de nitrógeno.
Un paso regulador clave es la producción de 5-fosfo-α- D -ribosil 1-pirofosfato ( PRPP ) por la ribosa fosfato pirofosfoquinasa, que se activa con fosfato inorgánico e inactiva con ribonucleótidos de purina. No es el paso comprometido para la síntesis de purinas porque el PRPP también se usa en la síntesis de pirimidina y en las vías de recuperación.
El primer paso comprometido es la reacción de PRPP, glutamina y agua a 5'-fosforribosilamina (PRA), glutamato y pirofosfato , catalizada por amidofosforribosiltransferasa , que es activada por PRPP e inhibida por AMP , GMP e IMP .
En el segundo paso, reaccionan PRA , glicina y ATP para crear GAR , ADP y pirofosfato, catalizados por fosforribosilamina-glicina ligasa (GAR sintetasa). Debido a la labilidad química del PRA, que tiene una vida media de 38 segundos a PH 7,5 y 37 °C, los investigadores han sugerido que el compuesto se canaliza desde la amidofosforribosiltransferasa a la GAR sintetasa in vivo. [1]
El tercero está catalizado por la fosforribosilglicinamida formiltransferasa .
El cuarto está catalizado por la fosforribosilformilglicinamidina sintasa .
El quinto está catalizado por la AIR sintetasa (FGAM ciclasa) .
El sexto está catalizado por la fosforribosilaminoimidazol carboxilasa .
El séptimo está catalizado por la fosforribosilaminoimidazolesuccinocarboxamida sintasa .
El ocho está catalizado por la adenilosuccinato liasa .
Los productos AICAR y fumarato siguen dos vías diferentes. AICAR sirve como reactivo para el noveno paso, mientras que el fumarato se transporta al ciclo del ácido cítrico, que luego puede omitir los pasos de evolución del dióxido de carbono para producir malato. La conversión de fumarato en malato es catalizada por la fumarasa. De esta manera, el fumarato conecta la síntesis de purinas con el ciclo del ácido cítrico. [2]
El noveno está catalizado por la fosforribosilaminoimidazolcarboxamida formiltransferasa .
El último paso es catalizado por la inosina monofosfato sintasa .
En eucariotas , el segundo, tercer y quinto paso están catalizados por la proteína biosintética de purina trifuncional adenosina-3 , que está codificada por el gen GART.
Tanto el noveno como el décimo paso se logran mediante una única proteína llamada proteína de biosíntesis de purina bifuncional PURH, codificada por el gen ATIC.
Las purinas son metabolizadas por varias enzimas :
La formación de 5'-fosforribosilamina a partir de glutamina y PRPP catalizada por la PRPP aminotransferasa es el punto de regulación para la síntesis de purinas. La enzima es una enzima alostérica, por lo que se puede convertir de IMP, GMP y AMP en altas concentraciones y se une a la enzima para ejercer inhibición, mientras que el PRPP se une en grandes cantidades a la enzima que causa la activación. Entonces IMP, GMP y AMP son inhibidores, mientras que PRPP es un activador. Entre la formación de 5'-fosforribosil, aminoimidazol e IMP, no se conoce ningún paso de regulación.
Las purinas procedentes del recambio de los ácidos nucleicos celulares (o de los alimentos) también pueden recuperarse y reutilizarse en nuevos nucleótidos.
Cuando un gen defectuoso provoca la aparición de lagunas en el proceso de reciclaje metabólico de purinas y pirimidinas, estas sustancias químicas no se metabolizan adecuadamente y los adultos o niños pueden sufrir cualquiera de los veintiocho trastornos hereditarios, posiblemente algunos más aún desconocidos. Los síntomas pueden incluir gota , anemia, epilepsia, retraso en el desarrollo, sordera, morderse compulsivamente, insuficiencia renal o cálculos, o pérdida de inmunidad.
El metabolismo de las purinas puede tener desequilibrios que pueden surgir de la incorporación de trifosfatos de nucleótidos dañinos en el ADN y el ARN, lo que conduce a alteraciones y mutaciones genéticas y, como resultado, da lugar a varios tipos de enfermedades. Algunas de las enfermedades son:
La modulación del metabolismo de las purinas tiene valor farmacoterapéutico.
Los inhibidores de la síntesis de purinas inhiben la proliferación de las células, especialmente de los leucocitos . Estos inhibidores incluyen la azatioprina , un inmunosupresor utilizado en trasplantes de órganos , enfermedades autoinmunes como la artritis reumatoide o enfermedades inflamatorias intestinales como la enfermedad de Crohn y la colitis ulcerosa .
El micofenolato de mofetilo es un fármaco inmunosupresor que se utiliza para prevenir el rechazo en el trasplante de órganos; inhibe la síntesis de purinas al bloquear la inosina monofosfato deshidrogenasa (IMPDH). [5] El metotrexato también inhibe indirectamente la síntesis de purinas al bloquear el metabolismo del ácido fólico (es un inhibidor de la dihidrofolato reductasa ).
El alopurinol es un fármaco que inhibe la enzima xantina oxidorreductasa y, por tanto, reduce el nivel de ácido úrico en el organismo. Esto puede resultar útil en el tratamiento de la gota, que es una enfermedad causada por el exceso de ácido úrico, que forma cristales en las articulaciones.
Para comprender cómo surgió la vida , se requiere conocimiento de las vías químicas que permiten la formación de los componentes clave de la vida en condiciones prebióticas plausibles . Nam et al. [6] demostraron la condensación directa de nucleobases purina y pirimidina con ribosa para dar ribonucleósidos en microgotas acuosas, un paso clave que conduce a la formación de ARN. Además, Becker et al. presentaron un proceso prebiótico plausible para sintetizar ribonucleósidos de purina. [7]
Los organismos de los tres dominios de la vida, eucariotas , bacterias y arqueas , son capaces de llevar a cabo la biosíntesis de novo de purinas. Esta capacidad refleja la esencialidad de las purinas para la vida. La vía bioquímica de síntesis es muy similar en especies eucariotas y bacterianas, pero es más variable entre especies de arqueas. [8] Se determinó que un conjunto casi completo o completo de genes necesarios para la biosíntesis de purinas estaba presente en 58 de las 65 especies de arqueas estudiadas. [8] Sin embargo, también se identificaron siete especies de arqueas con genes codificadores de purinas total o casi totalmente ausentes. Aparentemente, las especies de arqueas que no pueden sintetizar purinas son capaces de adquirir purinas exógenas para su crecimiento [8] y, por lo tanto, son similares a los mutantes de purinas de eucariotas, por ejemplo, los mutantes de purinas del hongo ascomiceto Neurospora crassa , [9] que también requieren purinas exógenas para su crecimiento. crecimiento.
{{cite book}}: Mantenimiento CS1: falta el editor de la ubicación ( enlace )