
La biología sintética ( SynBio ) es un campo multidisciplinario de la ciencia que se centra en los sistemas y organismos vivos, y aplica principios de ingeniería para desarrollar nuevas partes, dispositivos y sistemas biológicos o para rediseñar sistemas existentes que se encuentran en la naturaleza. [1]
Es una rama de la ciencia que abarca una amplia gama de metodologías de diversas disciplinas, como la bioquímica , la biotecnología , los biomateriales , la ciencia/ingeniería de materiales , la ingeniería genética , la biología molecular , la ingeniería molecular , la biología de sistemas , la ciencia de membranas , la biofísica , la ingeniería química y biológica , la ingeniería eléctrica e informática , la ingeniería de control y la biología evolutiva .
Incluye el diseño y la construcción de módulos biológicos , sistemas biológicos y máquinas biológicas , o el rediseño de sistemas biológicos existentes para fines útiles. [2]
Además, es la rama de la ciencia que se centra en las nuevas capacidades de ingeniería en organismos existentes para rediseñarlos con fines útiles. [3]
Para producir sistemas predecibles y robustos con funcionalidades novedosas que no existen en la naturaleza, también es necesario aplicar el paradigma de ingeniería del diseño de sistemas a los sistemas biológicos. Según la Comisión Europea , esto posiblemente implique un ensamblador molecular basado en sistemas biomoleculares como el ribosoma . [4]
1910: Primer uso identificable del término biología sintética en la publicación de Stéphane Leduc Théorie physico-chimique de la vie et générations spontanées . [5] También señaló este término en otra publicación, La Biologie Synthétique en 1912. [6]
1944 : El científico canadiense-estadounidense Oswald Avery demuestra que el ADN es el material del que están hechos los genes y los cromosomas . Este se convierte en la base sobre la que se construye toda la investigación genética posterior. [7]
1953 : Francis Crick y James Watson publican la estructura del ADN en Nature .
1961 : Jacob y Monod postulan la regulación celular por redes moleculares a partir de su estudio del operón lac en E. coli e imaginaron la capacidad de ensamblar nuevos sistemas a partir de componentes moleculares. [8]
1973 : Cohen, Boyer et al. publican en PNAS la primera clonación y amplificación molecular de ADN en un plásmido, lo que marca el comienzo de la biología sintética. [9]
1978 : Arber , Nathans y Smith ganan el Premio Nobel de Fisiología o Medicina por el descubrimiento de las enzimas de restricción , lo que lleva a Szybalski a ofrecer un comentario editorial en la revista Gene :
El trabajo sobre las nucleasas de restricción no sólo nos permite construir fácilmente moléculas de ADN recombinante y analizar genes individuales, sino que también nos ha llevado a la nueva era de la biología sintética, donde no sólo se describen y analizan los genes existentes, sino que también se pueden construir y evaluar nuevos arreglos genéticos. [10]
1988 : Mullis et al. publican en Science la primera amplificación de ADN mediante la reacción en cadena de la polimerasa (PCR) utilizando una ADN polimerasa termoestable. [11] Esto evitó agregar una nueva ADN polimerasa después de cada ciclo de PCR, simplificando así enormemente la mutagénesis y el ensamblaje del ADN.
2000 : Dos artículos en Nature informan sobre circuitos biológicos sintéticos , un interruptor genético y un reloj biológico, mediante la combinación de genes dentro de células de E. coli . [12] [13]
2003 : Tom Knight inventa los plásmidos BioBrick , las partes de ADN estandarizadas más utilizadas . [14] Estas partes se convertirán en el centro de la competición International Genetically Engineered Machine (iGEM) fundada en el MIT el año siguiente.
2003 : Los investigadores diseñan una vía precursora de la artemisinina en E. coli . [15]
2004 : Se celebra en el MIT la primera conferencia internacional sobre biología sintética, Synthetic Biology 1.0 (SB1.0).
2005 : Los investigadores desarrollan un circuito sensor de luz en E. coli . [16] Otro grupo diseña circuitos capaces de formar patrones multicelulares. [17]
2006 : Los investigadores diseñan un circuito sintético que promueve la invasión bacteriana de células tumorales. [18]
2010 : Los investigadores publican en Science el primer genoma bacteriano sintético, llamado M. mycoides JCVI-syn1.0. [19] [20] El genoma está hecho de ADN sintetizado químicamente mediante recombinación de levadura.
2011 : Se diseñan brazos cromosómicos sintéticos funcionales en levadura. [21]
2012 : Los laboratorios Charpentier y Doudna publican en Science la programación de la inmunidad bacteriana CRISPR-Cas9 para atacar la escisión del ADN. [22] Esta tecnología simplificó y amplió enormemente la edición de genes eucariotas.
2019 : Científicos de la ETH de Zúrich informan sobre la creación del primer genoma bacteriano , llamado Caulobacter ethensis-2.0 , realizado íntegramente por computadora, aunque todavía no existe una forma viable relacionada de C. ethensis-2.0 . [23] [24]
2019 : Los investigadores informan sobre la producción de una nueva forma sintética (posiblemente artificial ) de vida viable , una variante de la bacteria Escherichia coli , al reducir el número natural de 64 codones en el genoma bacteriano a 59 codones, para codificar 20 aminoácidos . [25] [26]
2020 : Los científicos crearon el primer xenobot , un organismo sintético programable derivado de células de rana y diseñado por IA. [27]
2021 : Los científicos informaron que los xenobots pueden autorreplicarse reuniendo células sueltas en el entorno y luego formando nuevos xenobots. [28]
Es un campo cuyo alcance se está expandiendo en términos de integración de sistemas, organismos diseñados y hallazgos prácticos. [1]
Los ingenieros consideran la biología como tecnología (en otras palabras, un sistema dado incluye la biotecnología o su ingeniería biológica ). [29] La biología sintética incluye la amplia redefinición y expansión de la biotecnología, con el objetivo final de poder diseñar y construir sistemas biológicos vivos diseñados que procesen información, manipulen sustancias químicas, fabriquen materiales y estructuras, produzcan energía, proporcionen alimentos y mantengan y mejoren la salud humana, así como avanzar en el conocimiento fundamental de los sistemas biológicos y nuestro medio ambiente. [30]
Los investigadores y las empresas que trabajan en biología sintética están utilizando el poder de la naturaleza para resolver problemas en la agricultura, la industria y la medicina. [3]
Debido a las capacidades más potentes de la ingeniería genética y a los menores costos de síntesis y secuenciación del ADN , el campo de la biología sintética está creciendo rápidamente. En 2016, más de 350 empresas en 40 países participaron activamente en aplicaciones de biología sintética; todas estas empresas tenían un valor neto estimado de $3.9 mil millones en el mercado global. [31] La biología sintética actualmente no tiene una definición generalmente aceptada. A continuación se presentan algunos ejemplos:
Es la ciencia de la ingeniería genética y física emergente para producir formas de vida nuevas (y, por lo tanto, sintéticas). Para desarrollar organismos con características nuevas o mejoradas, este campo de estudio emergente combina conocimientos y técnicas de biología, ingeniería y disciplinas relacionadas para diseñar ADN sintetizado químicamente. [32] [33]
La ingeniería biomolecular incluye enfoques que apuntan a crear un conjunto de unidades funcionales que puedan introducirse para presentar nuevas funciones tecnológicas en células vivas. La ingeniería genética incluye enfoques para construir cromosomas sintéticos u organismos mínimos como Mycoplasma laboratorium .
El diseño biomolecular se refiere a la idea general del diseño de novo y la combinación aditiva de componentes biomoleculares. Cada uno de estos enfoques comparte una tarea similar: desarrollar una entidad más sintética a un nivel más alto de complejidad mediante la manipulación inventiva de una parte más simple en el nivel anterior. [34] [35] La optimización de estas vías exógenas en sistemas no naturales requiere un ajuste fino iterativo de los componentes biomoleculares individuales para seleccionar las concentraciones más altas del producto deseado. [36]
Por otro lado, los "reescritores" son biólogos sintéticos interesados en probar la irreducibilidad de los sistemas biológicos. Debido a la complejidad de los sistemas biológicos naturales, sería más simple reconstruir los sistemas naturales de interés desde cero; para proporcionar sustitutos diseñados que sean más fáciles de comprender, controlar y manipular. [37] Los reescritores se inspiran en la refactorización , un proceso que a veces se utiliza para mejorar el software de computadora.
La bioingeniería, la genómica sintética, la biología sintética de protocélulas, la biología molecular no convencional y las técnicas in silico son las cinco categorías de la biología sintética. [38]
Es necesario revisar las distinciones y analogías entre las categorías de la biología sintética para su valoración social y ética, para distinguir entre cuestiones que afectan a todo el campo y aquellas particulares de uno específico. [38]
El subcampo de la bioingeniería se centra en la creación de nuevas vías metabólicas y reguladoras, y es probablemente el que actualmente atrae la atención de la mayoría de los investigadores y de la financiación. Está motivado principalmente por el deseo de establecer la biotecnología como una disciplina de ingeniería legítima. Al referirse a esta área de la biología sintética, la palabra "bioingeniería" no debe confundirse con la "ingeniería genética tradicional", que implica la introducción de un único transgén en el organismo deseado. Los bioingenieros adaptaron la biología sintética para proporcionar una perspectiva sustancialmente más integrada sobre cómo alterar los organismos o los sistemas metabólicos. [38]
Un ejemplo típico de ingeniería genética de un solo gen es la inserción del gen de la insulina humana en bacterias para crear proteínas transgénicas. La creación de vías de señalización completamente nuevas, que contienen numerosos genes y componentes reguladores (como un circuito oscilador para iniciar la producción periódica de proteína fluorescente verde (GFP) en células de mamíferos), se conoce como bioingeniería como parte de la biología sintética. [38]
Mediante el uso de módulos metabólicos y reguladores simplificados y abstractos, así como otras partes estandarizadas que pueden combinarse libremente para crear nuevas vías o criaturas, la bioingeniería apunta a crear sistemas biológicos innovadores. Además de crear infinitas oportunidades para aplicaciones novedosas, se prevé que esta estrategia haga que la bioingeniería sea más predecible y controlable que la biotecnología tradicional. [38]
La formación de animales con un genoma (mínimo) fabricado químicamente es otra faceta de la biología sintética que se destaca en la genómica sintética. Esta área de la biología sintética ha sido posible gracias a los avances en curso en la tecnología de síntesis de ADN, que ahora hace posible producir moléculas de ADN con miles de pares de bases a un costo razonable. El objetivo es combinar estas moléculas en genomas completos y trasplantarlos a células vivas, reemplazando el genoma de la célula huésped y reprogramando su metabolismo para realizar diferentes funciones. [38]
Los científicos ya habían demostrado el potencial de este enfoque creando virus infecciosos mediante la síntesis de los genomas de múltiples virus. Estos importantes avances en la ciencia y la tecnología desencadenaron las primeras preocupaciones del público sobre los riesgos asociados a esta tecnología. [38]
Un genoma simple también podría funcionar como un "genoma chasis" que podría ampliarse rápidamente mediante la inclusión de genes creados para tareas específicas. Estas "criaturas chasis" serían más adecuadas para la inserción de nuevas funciones que los organismos salvajes, ya que tendrían menos vías biológicas que podrían entrar en conflicto con las nuevas funcionalidades, además de tener sitios de inserción específicos. La genómica sintética se esfuerza por crear criaturas con "arquitecturas" novedosas, de forma muy similar al método de bioingeniería. Adopta una perspectiva integradora u holística del organismo. En este caso, el objetivo es la creación de genomas chasis basados en genes necesarios y otras secuencias de ADN requeridas, en lugar del diseño de vías metabólicas o reguladoras basadas en criterios abstractos. [38]
La generación in vitro de células sintéticas es la rama de las protocélulas de la biología sintética. Para crear estas células artificiales se pueden utilizar vesículas lipídicas, que tienen todos los componentes necesarios para funcionar como un sistema completo. Al final, estas células sintéticas deben cumplir los requisitos para ser consideradas vivas, es decir, la capacidad de autorreplicación, automantenimiento y evolución. La técnica de las protocélulas tiene este objetivo final, pero hay otros pasos intermedios que no cumplen todos los criterios de una célula viva. Para llevar a cabo una función específica, estas vesículas lipídicas contienen extractos celulares o conjuntos más específicos de macromoléculas biológicas y estructuras complejas, como enzimas, ácidos nucleicos o ribosomas. Por ejemplo, los liposomas pueden llevar a cabo reacciones en cadena de la polimerasa particulares o sintetizar una proteína particular. [38]
La biología sintética de protocélulas acerca la vida artificial un paso más a la realidad, ya que, en última instancia, sintetiza no sólo el genoma, sino también todos los componentes de la célula in vitro, a diferencia del enfoque de la genómica sintética, que se basa en obligar a una célula natural a ejecutar las instrucciones codificadas por el genoma sintético introducido. Los biólogos sintéticos de este campo consideran que su trabajo es un estudio básico de las condiciones necesarias para la existencia de la vida y su origen, más que cualquier otra técnica. Sin embargo, la técnica de las protocélulas también se presta bien a otras aplicaciones; de manera similar a otros subproductos de la biología sintética, las protocélulas podrían emplearse para la fabricación de biopolímeros y medicamentos. [38]
El objetivo de la estrategia de la "biología molecular antinatural" es crear nuevas variedades de vida que se basen en un tipo diferente de biología molecular, como nuevos tipos de ácidos nucleicos o un nuevo código genético. La creación de nuevos tipos de nucleótidos que puedan incorporarse a ácidos nucleicos únicos podría lograrse modificando ciertos componentes del ADN o ARN, como las bases o los azúcares de la cadena principal. [38]
El código genético normal se altera insertando codones cuádruples o cambiando algunos codones para codificar nuevos aminoácidos, lo que posteriormente permitiría el uso de aminoácidos no naturales con características únicas en la producción de proteínas. Es un problema científico y tecnológico ajustar la maquinaria enzimática de la célula para ambos enfoques. [38]
Se formaría un nuevo tipo de vida a partir de organismos con un genoma construido a partir de ácidos nucleicos sintéticos o de un sistema de codificación totalmente nuevo para aminoácidos sintéticos. Este nuevo estilo de vida tendría algunas ventajas, pero también algunos peligros nuevos. Al liberarlos en el medio ambiente, no habría transferencia horizontal de genes ni cruzamiento de genes con especies naturales. Además, estos tipos de organismos sintéticos podrían ser creados de manera que necesitaran materiales no naturales para la síntesis de proteínas o ácidos nucleicos, lo que los haría incapaces de prosperar en la naturaleza si escaparan accidentalmente. [38]
Por otra parte, si estos organismos finalmente pudieran sobrevivir fuera del espacio controlado, podrían tener un beneficio particular sobre los organismos naturales porque serían resistentes a los organismos vivos depredadores o a los virus naturales, lo que podría llevar a una propagación descontrolada de los organismos sintéticos. [38]
La biología sintética in silico y las distintas estrategias están interconectadas. El desarrollo de diseños complejos, ya sean vías metabólicas, procesos celulares fundamentales o genomas de chasis, es una de las principales dificultades a las que se enfrentan los cuatro métodos de biología sintética descritos anteriormente. Debido a esto, la biología sintética tiene una rama in silico robusta, similar a la biología de sistemas, que tiene como objetivo crear modelos computacionales para el diseño de componentes biológicos comunes o circuitos sintéticos, que son esencialmente simulaciones de organismos sintéticos. [38]
La aplicación práctica de simulaciones y modelos a través de la bioingeniería u otros campos de la biología sintética es el objetivo a largo plazo de la biología sintética in silico. Muchas de las simulaciones computacionales de organismos sintéticos hasta el momento tienen poca o ninguna analogía directa con los seres vivos. Debido a esto, la biología sintética in silico se considera un grupo separado en este artículo. [38]
Es sensato integrar las cinco áreas bajo el paraguas de la biología sintética como un área de estudio unificada. Aunque se centran en diversas facetas de la vida, como la regulación metabólica, los elementos esenciales o la composición bioquímica, estas cinco estrategias trabajan todas hacia el mismo fin: crear nuevos tipos de organismos vivos. Además, las diversas metodologías comienzan con numerosos enfoques metodológicos, lo que conduce a la diversidad de enfoques de la biología sintética. [38]
La biología sintética es un campo interdisciplinario que se nutre de muchas disciplinas científicas diferentes y se inspira en ellas, no en un solo campo o técnica. Todos los biólogos sintéticos tienen el mismo objetivo subyacente de diseñar y producir nuevas formas de vida, a pesar de que pueden emplear diversas metodologías, técnicas e instrumentos de investigación. Cualquier evaluación de la biología sintética, ya sea que examine consideraciones éticas, legales o de seguridad, debe tener en cuenta el hecho de que, si bien algunas preguntas, riesgos y problemas son exclusivos de cada técnica, en otras circunstancias, debe tomarse en consideración la biología sintética en su conjunto. [38]
La biología sintética se ha dividido tradicionalmente en cuatro enfoques de ingeniería diferentes: de arriba hacia abajo, paralelo, ortogonal y de abajo hacia arriba. [39]
Para replicar comportamientos emergentes de la biología natural y construir vida artificial, se utilizan sustancias químicas no naturales. La otra busca componentes intercambiables de sistemas biológicos para ensamblar y crear sistemas que no funcionan de manera natural. En ambos casos, un objetivo sintético obliga a los investigadores a aventurarse en nuevas áreas para abordar y resolver problemas que no se pueden resolver fácilmente mediante el análisis. Debido a esto, surgen nuevos paradigmas de maneras que el análisis no puede hacer fácilmente. Además de equipos que oscilan, se arrastran y juegan al tres en raya, la biología sintética ha producido instrumentos de diagnóstico que mejoran el tratamiento de pacientes con enfermedades infecciosas. [40]
Se trata de utilizar técnicas de ingeniería metabólica y genética para impartir nuevas funciones a las células vivas. [41] Al comparar genes universales y eliminar los no esenciales para crear un genoma básico, este método busca reducir la complejidad de las células existentes. Estas iniciativas se basan en la hipótesis de una única génesis de la vida celular, el llamado Último Ancestro Común Universal , que apoya la presencia de un genoma mínimo universal que dio origen a todos los seres vivos. Sin embargo, estudios recientes plantean la posibilidad de que las células eucariotas y procariotas que componen el árbol de la vida puedan haber evolucionado a partir de un grupo de células primordiales en lugar de a partir de una sola célula. Como resultado, aunque la búsqueda del "genoma mínimo" se ha vuelto esquiva, la eliminación de una serie de funciones no esenciales perjudica la aptitud de un organismo y conduce a genomas "frágiles". [39]
Este enfoque implica la creación de nuevos sistemas biológicos in vitro reuniendo componentes biomoleculares “no vivos”, [42] a menudo con el objetivo de construir una célula artificial .
La reproducción, la replicación y el ensamblaje son tres principios de autoorganización cruciales que se tienen en cuenta para lograr esto. Las células, que están formadas por un contenedor y un metabolismo, se consideran "hardware" en la definición de reproducción, mientras que la replicación ocurre cuando un sistema duplica una copia perfecta de sí mismo, como en el caso del ADN, que se considera "software". Cuando se agregan vesículas o contenedores (como los coacervados de Oparin) formados por pequeñas gotitas de moléculas orgánicas como lípidos o liposomas, estructuras similares a membranas que comprenden fosfolípidos, se produce el ensamblaje. [39]
El estudio de las protocélulas existe junto con otras iniciativas de biología sintética in vitro que buscan producir células mínimas, vías metabólicas o "proteínas nunca nacidas", así como imitar funciones fisiológicas, incluyendo la división y el crecimiento celular. La mejora in vitro de las vías sintéticas tiene el potencial de tener un efecto en algunos otros sectores de la biología sintética, incluyendo la ingeniería metabólica, a pesar del hecho de que ya no se clasifica como investigación de biología sintética. Esta investigación, que es fundamentalmente esencial, merece un reconocimiento apropiado como investigación de biología sintética. [39]
La ingeniería paralela también se conoce como bioingeniería. El código genético básico es la base de la investigación de ingeniería paralela, que utiliza biomoléculas convencionales como los ácidos nucleicos y los 20 aminoácidos para construir sistemas biológicos. Para una variedad de aplicaciones en bioinformática, bioenergía, biocombustibles, biorremediación, optogenética y medicina, implica la estandarización de componentes de ADN, ingeniería de interruptores, biosensores, circuitos genéticos, puertas lógicas y operadores de comunicación celular. Para dirigir la expresión de dos o más genes y/o proteínas, la mayoría de estas aplicaciones a menudo dependen del uso de uno o más vectores (o plásmidos). Las unidades de ADN pequeñas, circulares y de doble cadena conocidas como plásmidos, que se encuentran principalmente en procariotas pero también pueden detectarse ocasionalmente en células eucariotas, pueden replicarse de forma autónoma del ADN cromosómico. [39]
También se conoce como ingeniería perpendicular. Esta estrategia, también denominada "biología sintética química", busca principalmente alterar o ampliar los códigos genéticos de los sistemas vivos utilizando bases de ADN y/o aminoácidos artificiales. Este subcampo también está conectado a la xenobiología , un campo desarrollado recientemente que combina la química de sistemas, la biología sintética, la exobiología y la investigación sobre los orígenes de la vida. En las últimas décadas, los investigadores han creado compuestos que son estructuralmente similares a las bases canónicas del ADN para ver si esas moléculas "alienígenas" o xeno (XNA) pueden emplearse como portadoras de información genética. De manera similar, las fracciones no canónicas han tomado el lugar del azúcar del ADN ( desoxirribosa ). [39] Para expresar información distinta a los 20 aminoácidos convencionales de las proteínas, el código genético puede alterarse o ampliarse. Un método implica la incorporación de un aminoácido no natural, no canónico o xeno (XAA) específico en una o más proteínas en uno o más lugares precisos utilizando enzimas ortogonales y un adaptador de ARN de transferencia de otro organismo. Mediante el uso de la "evolución dirigida", que implica ciclos repetidos de mutagénesis genética (producción de diversidad genotípica), cribado o selección (de un rasgo fenotípico específico) y amplificación de una variante mejor para la siguiente ronda iterativa, se producen enzimas ortogonales. Numerosos XAA se han incorporado de manera efectiva a las proteínas en criaturas más complejas como gusanos y moscas, así como en bacterias, levaduras y líneas celulares humanas. Como resultado de los cambios en la secuencia de ADN canónico, la evolución dirigida también permite el desarrollo de ribosomas ortogonales, que facilitan la incorporación de XAA a las proteínas o la creación de "vida espejo", o sistemas biológicos que contienen biomoléculas compuestas de enantiómeros con diferentes orientaciones quirales. [39]
Varias tecnologías novedosas que facilitaron el éxito de la biología sintética fueron fundamentales. Entre los conceptos que se incluyen se encuentran la estandarización de las partes biológicas y la abstracción jerárquica para permitir el uso de esas partes en sistemas sintéticos. [43] El ADN sirve como guía para el funcionamiento de los procesos biológicos, como la partitura de una compleja sinfonía de la vida. Nuestra capacidad para comprender y diseñar sistemas biológicos ha sufrido modificaciones significativas como resultado de los avances de las últimas décadas en la lectura (secuenciación) y la escritura (síntesis) de secuencias de ADN. Estos avances han producido técnicas innovadoras para diseñar, ensamblar y modificar genes, materiales, circuitos y vías metabólicas codificados por ADN, lo que permite un control cada vez mayor sobre los sistemas biológicos e incluso sobre organismos enteros. [44]
Las tecnologías básicas incluyen la lectura y escritura de ADN (secuenciación y fabricación). Se necesitan mediciones en múltiples condiciones para lograr un modelado preciso y un diseño asistido por computadora (CAD).
Impulsados por las dramáticas disminuciones en los costos de síntesis de oligonucleótidos ("oligos") y el advenimiento de la PCR, los tamaños de las construcciones de ADN a partir de oligos han aumentado al nivel genómico. [45] En 2000, los investigadores informaron la síntesis del genoma del virus de la hepatitis C de 9,6 kpb (kilo pb) a partir de 60 a 80-meros sintetizados químicamente. [46] En 2002, los investigadores de la Universidad Stony Brook lograron sintetizar el genoma del poliovirus de 7741 pb a partir de su secuencia publicada, produciendo el segundo genoma sintético, que abarcó dos años. [47] En 2003, el genoma de 5386 pb del bacteriófago Phi X 174 se ensambló en aproximadamente dos semanas. [48] En 2006, el mismo equipo, en el Instituto J. Craig Venter , construyó y patentó un genoma sintético de una nueva bacteria mínima, Mycoplasma laboratorium , y estaba trabajando para lograr que funcionara en una célula viva. [49] [50] [51]
En 2007, se informó que varias empresas ofrecían síntesis de secuencias genéticas de hasta 2000 pares de bases (pb) de longitud, por un precio de aproximadamente $1 por pb y un tiempo de respuesta de menos de dos semanas. [52] Los oligonucleótidos cosechados de un chip de ADN fabricado por fotolitografía o inyección de tinta combinados con PCR y corrección de errores de desajuste de ADN permiten cambios económicos a gran escala de codones en sistemas genéticos para mejorar la expresión genética o incorporar nuevos aminoácidos (ver los proyectos de células sintéticas de George M. Church y Anthony Forster. [53] [54] ) . Esto favorece un enfoque de síntesis desde cero.
Además, el sistema CRISPR/Cas ha surgido como una técnica prometedora para la edición genética. Se lo ha descrito como "la innovación más importante en el campo de la biología sintética en casi 30 años". [55] Mientras que otros métodos tardan meses o años en editar secuencias genéticas, CRISPR acelera ese tiempo hasta semanas. [55] Sin embargo, debido a su facilidad de uso y accesibilidad, ha suscitado inquietudes éticas, especialmente en torno a su uso en biohacking . [56] [57] [58]
La secuenciación del ADN determina el orden de las bases de nucleótidos en una molécula de ADN. Los biólogos sintéticos utilizan la secuenciación del ADN en su trabajo de varias maneras. En primer lugar, los esfuerzos de secuenciación genómica a gran escala continúan proporcionando información sobre organismos naturales. Esta información proporciona un sustrato rico a partir del cual los biólogos sintéticos pueden construir piezas y dispositivos. En segundo lugar, la secuenciación puede verificar que el sistema fabricado es el previsto. En tercer lugar, la secuenciación rápida, barata y confiable puede facilitar la detección e identificación rápidas de sistemas y organismos sintéticos. [59]
Es la capacidad de un sistema o componente de funcionar sin referencia a su contexto. [60]
Las partes de ADN estandarizadas más utilizadas [61] : 22–23 son los plásmidos BioBrick , inventados por Tom Knight en 2003. [14] Los Biobricks se almacenan en el Registro de Partes Biológicas Estándar en Cambridge, Massachusetts. El estándar BioBrick ha sido utilizado por decenas de miles de estudiantes en todo el mundo en la competencia internacional Genetically Engineered Machine (iGEM). El estándar de ensamblaje BioBrick 10 promueve la modularidad al permitir que las secuencias codificantes BioBrick se empalmen e intercambien utilizando las enzimas de restricción EcoRI o XbaI (prefijo BioBrick) y SpeI y PstI (sufijo BioBrick). [61] : 22–23
La superposición de secuencias entre dos elementos genéticos ( genes o secuencias codificantes ), denominadas genes superpuestos , puede impedir su manipulación individual. [62] Para aumentar la modularidad del genoma, se ha adoptado en todas las disciplinas de biología sintética la práctica de refactorizar el genoma o mejorar "la estructura interna de un sistema existente para su uso futuro, manteniendo al mismo tiempo la función del sistema externo" [63] . [62] Algunos ejemplos notables de refactorización incluyen el grupo de fijación de nitrógeno [64] y el sistema de secreción tipo III [65] junto con los bacteriófagos T7 [63] y ΦX174. [66]
Si bien el ADN es el más importante para el almacenamiento de información, una gran fracción de las actividades de la célula son llevadas a cabo por proteínas. Las herramientas pueden enviar proteínas a regiones específicas de la célula y unir diferentes proteínas entre sí. La fuerza de interacción entre las proteínas asociadas debe ser ajustable entre una vida útil de segundos (deseable para eventos de señalización dinámica) hasta una interacción irreversible (deseable para la estabilidad del dispositivo o resistente a condiciones severas). Las interacciones como las bobinas enrolladas , [67] la unión del dominio SH3 con el péptido [68] o SpyTag/SpyCatcher [69] ofrecen dicho control. Además, es necesario regular las interacciones proteína-proteína en las células, como con la luz (usando dominios de detección de luz-oxígeno-voltaje ) o pequeñas moléculas permeables a las células mediante dimerización inducida químicamente . [70]
En una célula viva, los motivos moleculares están integrados en una red más grande con componentes ascendentes y descendentes. Estos componentes pueden alterar la capacidad de señalización del módulo de modelado. En el caso de los módulos ultrasensibles, la contribución de sensibilidad de un módulo puede diferir de la sensibilidad que el módulo mantiene de forma aislada. [71] [72]
Los modelos informan el diseño de sistemas biológicos diseñados al predecir mejor el comportamiento del sistema antes de su fabricación. La biología sintética se beneficia de mejores modelos de cómo las moléculas biológicas se unen a los sustratos y catalizan las reacciones, cómo el ADN codifica la información necesaria para especificar la célula y cómo se comportan los sistemas integrados de múltiples componentes. Los modelos multiescala de redes reguladoras de genes se centran en las aplicaciones de la biología sintética. Las simulaciones pueden modelar todas las interacciones biomoleculares en la transcripción , traducción , regulación e inducción de redes reguladoras de genes. [73] [74] [75] [76]
Sólo un modelado extensivo puede permitir la exploración de la expresión genética dinámica en una forma adecuada para la investigación y el diseño debido a las numerosas especies involucradas y la complejidad de sus relaciones. Las simulaciones dinámicas de toda la interconexión biomolecular involucrada en la regulación, el transporte, la transcripción, la inducción y la traducción permiten el detalle a nivel molecular de los diseños. A diferencia del modelado de redes artificiales a posteriori, esto se contrasta. [77]
La microfluídica , en particular la microfluídica de gotas, es una herramienta emergente que se utiliza para construir nuevos componentes y para analizarlos y caracterizarlos. [78] [79] Se emplea ampliamente en ensayos de detección. [80]
Los estudios han considerado los componentes del mecanismo de transcripción del ADN . Un deseo de los científicos que crean circuitos biológicos sintéticos es poder controlar la transcripción del ADN sintético en organismos unicelulares ( procariotas ) y en organismos multicelulares ( eucariotas ). Un estudio probó la capacidad de ajuste de los factores de transcripción sintéticos (sTF) en áreas de salida de transcripción y capacidad cooperativa entre complejos de factores de transcripción múltiples. [81] Los investigadores pudieron mutar regiones funcionales llamadas dedos de zinc , el componente específico del ADN de los sTF, para disminuir su afinidad por sitios específicos de secuencias de ADN operadoras y, por lo tanto, disminuir la actividad específica del sitio asociada del sTF (generalmente regulación transcripcional). Además, utilizaron los dedos de zinc como componentes de los sTF formadores de complejos, que son los mecanismos de traducción eucariotas . [81]
Las iniciativas de biología sintética suelen tener como objetivo rediseñar organismos para que puedan crear un material, como un fármaco o combustible, o adquirir una nueva función, como la capacidad de percibir algo en el entorno. Algunos ejemplos de lo que los investigadores están creando mediante la biología sintética son:
Un biosensor se refiere a un organismo diseñado, generalmente una bacteria, que es capaz de informar sobre algún fenómeno ambiental como la presencia de metales pesados o toxinas. Uno de estos sistemas es el operón Lux de Aliivibrio fischeri , [83] que codifica la enzima que es la fuente de la bioluminiscencia bacteriana , y puede colocarse después de un promotor respondedor para expresar los genes de luminiscencia en respuesta a un estímulo ambiental específico. [84] Uno de estos sensores creados, consistió en un recubrimiento bacteriano bioluminiscente en un chip de computadora fotosensible para detectar ciertos contaminantes del petróleo . Cuando las bacterias detectan el contaminante, emiten luminiscencia. [85] Otro ejemplo de un mecanismo similar es la detección de minas terrestres por una cepa reportera de E. coli diseñada capaz de detectar TNT y su principal producto de degradación DNT , y en consecuencia producir una proteína fluorescente verde ( GFP ). [86]
Los organismos modificados pueden detectar señales ambientales y enviar señales de salida que pueden detectarse y servir para fines de diagnóstico. Se han utilizado cohortes de microbios. [87]
Los biosensores también podrían utilizarse para detectar firmas patógenas, como la del SARS-CoV-2 , y pueden ser portátiles . [88] [89]
Con el fin de detectar y reaccionar a diversos factores ambientales temporales, las células han desarrollado una amplia gama de circuitos reguladores, que van desde los transcripcionales hasta los postraduccionales. Estos circuitos están compuestos por módulos transductores que filtran las señales y activan una respuesta biológica, así como secciones sensibles cuidadosamente diseñadas que fijan analitos y regulan los umbrales de detección de señales. La modularidad y la selectividad están programadas en los circuitos biosensores a nivel transcripcional, traduccional y postraduccional, para lograr el delicado equilibrio de los dos módulos de detección básicos. [90]
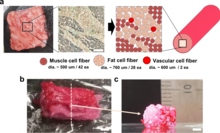
Sin embargo, no todos los productos de nutrición sintética son productos alimenticios para animales; por ejemplo, a partir de 2021, también hay productos de café sintético que se informa que están cerca de comercializarse. [98] [99] [100] Campos similares de investigación y producción basados en la biología sintética que se pueden utilizar para la producción de alimentos y bebidas son:
Las células microbianas fotosintéticas se han utilizado como un paso hacia la producción sintética de seda de araña . [105] [106]
Una computadora biológica se refiere a un sistema biológico diseñado que puede realizar operaciones similares a las de una computadora, que es un paradigma dominante en la biología sintética. Los investigadores construyeron y caracterizaron una variedad de puertas lógicas en varios organismos, [107] y demostraron computación analógica y digital en células vivas. Demostraron que las bacterias pueden ser diseñadas para realizar computación analógica y/o digital. [108] [109] En 2007, en células humanas, la investigación demostró un evaluador lógico universal que opera en células de mamíferos. [110] Posteriormente, los investigadores utilizaron este paradigma para demostrar una terapia de prueba de concepto que utiliza computación digital biológica para detectar y matar células cancerosas humanas en 2011. [111] En 2016, otro grupo de investigadores demostró que los principios de la ingeniería informática se pueden utilizar para automatizar el diseño de circuitos digitales en células bacterianas. [112] En 2017, los investigadores demostraron el sistema 'Lógica y aritmética booleana a través de la escisión de ADN' (BLADE) para diseñar computación digital en células humanas. [113] En 2019, los investigadores implementaron un perceptrón en sistemas biológicos, abriendo el camino para el aprendizaje automático en estos sistemas. [114]
Las células utilizan genes y proteínas que interactúan, denominados circuitos genéticos, para implementar diversas funciones, como responder a señales ambientales, tomar decisiones y comunicarse. Hay tres componentes clave involucrados: ADN, ARN y circuitos genéticos diseñados por biólogos sintéticos que pueden controlar la expresión genética desde varios niveles, incluidos los niveles transcripcional, postranscripcional y traduccional.
La ingeniería metabólica tradicional se ha visto reforzada por la introducción de combinaciones de genes extraños y la optimización mediante la evolución dirigida. Esto incluye la ingeniería de E. coli y levadura para la producción comercial de un precursor del fármaco antipalúdico , la artemisinina . [115]
Aún no se han creado organismos enteros desde cero, aunque las células vivas pueden transformarse con ADN nuevo. Varias formas permiten construir componentes de ADN sintético e incluso genomas sintéticos completos , pero una vez que se obtiene el código genético deseado, se integra en una célula viva que se espera que manifieste las nuevas capacidades o fenotipos deseados mientras crece y prospera. [116] La transformación celular se utiliza para crear circuitos biológicos , que pueden manipularse para producir los resultados deseados. [12] [13]
Al integrar la biología sintética con la ciencia de los materiales , sería posible utilizar las células como fundiciones moleculares microscópicas para producir materiales cuyas propiedades estuvieran codificadas genéticamente. La reingeniería ha producido fibras Curli, el componente amiloide del material extracelular de las biopelículas , como una plataforma para nanomateriales programables . Estas nanofibras se construyeron genéticamente para funciones específicas, incluida la adhesión a sustratos, la creación de plantillas de nanopartículas y la inmovilización de proteínas. [117]

Las proteínas naturales pueden ser diseñadas, por ejemplo, por evolución dirigida , se pueden producir nuevas estructuras proteicas que coincidan o mejoren la funcionalidad de las proteínas existentes. Un grupo generó un haz de hélices que era capaz de unirse al oxígeno con propiedades similares a la hemoglobina , pero no se unía al monóxido de carbono . [119] Se generó una estructura proteica similar para soportar una variedad de actividades de oxidorreductasa [120] mientras que otro formó una ATPasa estructural y secuencialmente novedosa . [121] Otro grupo generó una familia de receptores acoplados a proteína G que podrían ser activados por la pequeña molécula inerte N-óxido de clozapina pero insensibles al ligando nativo , acetilcolina ; estos receptores se conocen como DREADD . [122] También se pueden diseñar nuevas funcionalidades o especificidad de proteínas utilizando enfoques computacionales. Un estudio pudo utilizar dos métodos computacionales diferentes: un método de bioinformática y modelado molecular para extraer bases de datos de secuencias, y un método de diseño de enzimas computacional para reprogramar la especificidad de las enzimas. Ambos métodos dieron como resultado enzimas diseñadas con una especificidad de más de 100 veces para la producción de alcoholes de cadena más larga a partir de azúcar. [123]
Otra investigación común es la expansión del conjunto natural de 20 aminoácidos . Excluyendo los codones de terminación , se han identificado 61 codones , pero solo 20 aminoácidos están codificados generalmente en todos los organismos. Ciertos codones están diseñados para codificar aminoácidos alternativos, incluidos: aminoácidos no estándar como O-metil tirosina ; o aminoácidos exógenos como 4-fluorofenilalanina. Por lo general, estos proyectos hacen uso de pares de ARNt supresores sin sentido recodificados - Aminoacil ARNt sintetasa de otros organismos, aunque en la mayoría de los casos se requiere una ingeniería sustancial. [124]
Otros investigadores estudiaron la estructura y la función de las proteínas reduciendo el conjunto normal de 20 aminoácidos. Se crean bibliotecas de secuencias de proteínas limitadas generando proteínas en las que grupos de aminoácidos pueden reemplazarse por un solo aminoácido. [125] Por ejemplo, varios aminoácidos no polares dentro de una proteína pueden reemplazarse todos por un solo aminoácido no polar. [126] Un proyecto demostró que una versión modificada de la mutasa del corismato aún tenía actividad catalítica cuando solo se usaban nueve aminoácidos. [127]
Los investigadores y las empresas practican la biología sintética para sintetizar enzimas industriales con alta actividad, rendimientos óptimos y efectividad. Estas enzimas sintetizadas tienen como objetivo mejorar productos como detergentes y productos lácteos sin lactosa, así como hacerlos más rentables. [128] Las mejoras de la ingeniería metabólica mediante la biología sintética son un ejemplo de una técnica biotecnológica utilizada en la industria para descubrir productos farmacéuticos y productos químicos fermentativos. La biología sintética puede investigar sistemas de vías modulares en la producción bioquímica y aumentar los rendimientos de la producción metabólica. La actividad enzimática artificial y los efectos posteriores en las tasas de reacción metabólica y los rendimientos pueden desarrollar "nuevas estrategias eficientes para mejorar las propiedades celulares... para la producción bioquímica de importancia industrial". [129]
Los científicos pueden codificar información digital en una sola hebra de ADN sintético . En 2012, George M. Church codificó uno de sus libros sobre biología sintética en ADN. Los 5,3 Mb de datos fueron más de 1000 veces mayores que la mayor cantidad anterior de información almacenada en ADN sintetizado. [130] Un proyecto similar codificó los sonetos completos de William Shakespeare en ADN. [131] De manera más general, algoritmos como NUPACK, [132] ViennaRNA, [133] Ribosome Binding Site Calculator, [134] Cello, [135] y Non-Repetitive Parts Calculator [136] permiten el diseño de nuevos sistemas genéticos.
Se han desarrollado muchas tecnologías para incorporar nucleótidos y aminoácidos artificiales en ácidos nucleicos y proteínas, tanto in vitro como in vivo. Por ejemplo, en mayo de 2014, los investigadores anunciaron que habían introducido con éxito dos nuevos nucleótidos artificiales en el ADN bacteriano. Al incluir nucleótidos artificiales individuales en el medio de cultivo, pudieron intercambiar las bacterias 24 veces; no generaron ARNm ni proteínas capaces de utilizar los nucleótidos artificiales. [137] [138] [139]
La biología sintética despertó el interés de la NASA , ya que podría ayudar a producir recursos para los astronautas a partir de una cartera restringida de compuestos enviados desde la Tierra. [140] [141] [142] En Marte, en particular, la biología sintética podría conducir a procesos de producción basados en recursos locales, lo que la convierte en una herramienta poderosa en el desarrollo de puestos avanzados ocupados con menos dependencia de la Tierra. [140] Se ha trabajado en el desarrollo de cepas de plantas que puedan hacer frente al duro entorno marciano, utilizando técnicas similares a las empleadas para aumentar la resiliencia a ciertos factores ambientales en los cultivos agrícolas. [143]
Un tema importante en la biología sintética es la vida sintética , que se ocupa de organismos hipotéticos creados in vitro a partir de biomoléculas y/o análogos químicos de las mismas . Los experimentos de vida sintética intentan investigar los orígenes de la vida , estudiar algunas de las propiedades de la vida o, más ambiciosamente, recrear la vida a partir de componentes no vivos ( abióticos ). La biología de la vida sintética intenta crear organismos vivos capaces de llevar a cabo funciones importantes, desde la fabricación de productos farmacéuticos hasta la desintoxicación de tierras y agua contaminadas. [145] En medicina, ofrece perspectivas de utilizar partes biológicas de diseño como punto de partida para nuevas clases de terapias y herramientas de diagnóstico. [145]
Una "célula artificial" viviente se ha definido como una célula completamente sintética que puede capturar energía , mantener gradientes iónicos , contener macromoléculas así como almacenar información y tener la capacidad de mutar . [146] Nadie ha sido capaz de crear una célula así. [146]
En 2010, Craig Venter produjo un cromosoma bacteriano completamente sintético y su equipo lo introdujo en células huésped bacterianas vaciadas genómicamente. [19] Las células huésped pudieron crecer y replicarse. [147] [148] El laboratorio Mycoplasma es el único organismo vivo con un genoma completamente diseñado.
En 2014 se presentó el primer organismo vivo con un código genético expandido "artificial"; el equipo utilizó E. coli a la que se le extrajo el genoma y se lo reemplazó por un cromosoma con un código genético expandido. Los nucleósidos añadidos son d5SICS y dNaM . [139]
En mayo de 2019, en un esfuerzo histórico, los investigadores informaron sobre la creación de una nueva forma sintética (posiblemente artificial ) de vida viable , una variante de la bacteria Escherichia coli , al reducir el número natural de 64 codones en el genoma bacteriano a 59 codones en su lugar, para codificar 20 aminoácidos . [25] [26]
En 2017, se inició la colaboración internacional de investigación de código abierto a gran escala Build-a-Cell para la construcción de células vivas sintéticas, [149] seguida por organizaciones nacionales de células sintéticas en varios países, incluidas FabriCell, [150] MaxSynBio [151] y BaSyC. [152] Los esfuerzos europeos en materia de células sintéticas se unificaron en 2019 como iniciativa SynCellEU. [153]
En 2023, los investigadores pudieron crear los primeros embriones humanos sintéticos derivados de células madre. [154]
En el campo de la terapéutica, la biología sintética ha logrado avances significativos en la modificación y simplificación del alcance terapéutico en un período de tiempo relativamente corto. De hecho, nuevas plataformas terapéuticas, desde el descubrimiento de mecanismos patológicos y dianas farmacológicas hasta la fabricación y el transporte de pequeñas moléculas, son posibles gracias a la construcción de componentes biológicos con un diseño lógico y guiado por modelos. [60]
Los dispositivos de biología sintética se han diseñado para actuar como terapias en tratamientos terapéuticos. Es posible controlar virus y organismos completamente creados para atacar patógenos específicos y vías patológicas. Así, en dos estudios independientes 91,92, los investigadores utilizaron bacteriófagos modificados genéticamente para combatir bacterias resistentes a los antibióticos, dándoles características genéticas que atacan específicamente a las defensas bacterianas contra la actividad de los antibióticos y las obstaculizan. [155]
En el tratamiento del cáncer , dado que los medicamentos convencionales suelen atacar indiscriminadamente a los tumores y a los tejidos normales, pueden resultar útiles los virus y organismos creados artificialmente que puedan identificar y conectar su acción terapéutica con señales patológicas. Por ejemplo, se introdujo en adenovirus la actividad de la vía p53 en células humanas para controlar su replicación. [155]
Las bacterias se han utilizado durante mucho tiempo en el tratamiento del cáncer. Bifidobacterium y Clostridium colonizan selectivamente los tumores y reducen su tamaño. [156] Recientemente, los biólogos sintéticos reprogramaron las bacterias para que detecten y respondan a un estado particular del cáncer. La mayoría de las veces, las bacterias se utilizan para administrar una molécula terapéutica directamente al tumor para minimizar los efectos fuera del objetivo. Para dirigirse a las células tumorales, se expresaron péptidos que pueden reconocer específicamente un tumor en las superficies de las bacterias. Los péptidos utilizados incluyen una molécula aficuerpo que se dirige específicamente al receptor 2 del factor de crecimiento epidérmico humano [157] y una adhesina sintética . [158] La otra forma es permitir que las bacterias detecten el microambiente del tumor , por ejemplo la hipoxia, construyendo una puerta lógica AND en las bacterias. [159] Luego, las bacterias solo liberan moléculas terapéuticas objetivo al tumor a través de la lisis [160] o el sistema de secreción bacteriana . [161] La lisis tiene la ventaja de que puede estimular el sistema inmunológico y controlar el crecimiento. Se pueden utilizar múltiples tipos de sistemas de secreción y también otras estrategias. El sistema es inducible por señales externas. Los inductores incluyen sustancias químicas, ondas electromagnéticas o luminosas.
En estas terapias se emplean múltiples especies y cepas. Las bacterias más utilizadas son Salmonella typhimurium , Escherichia coli , Bifidobacteria , Streptococcus , Lactobacillus , Listeria y Bacillus subtilis . Cada una de estas especies tiene sus propias propiedades y son exclusivas de la terapia contra el cáncer en términos de colonización de tejidos, interacción con el sistema inmunológico y facilidad de aplicación.
Plataforma basada en levadura diseñada
Los biólogos sintéticos están desarrollando levaduras vivas modificadas genéticamente que pueden administrar medicamentos biológicos terapéuticos. Cuando se administran por vía oral, estas levaduras vivas actúan como microfábricas y producirán moléculas terapéuticas directamente en el tracto gastrointestinal. Debido a que las levaduras son eucariotas, un beneficio clave es que se pueden administrar junto con antibióticos. La levadura probiótica que expresa el receptor purinérgico P2Y2 humano suprimió la inflamación intestinal en modelos de ratón de enfermedad inflamatoria intestinal. [162] Se ha desarrollado una levadura S. boulardii viva que libera una antitoxina tetraespecífica que neutraliza potentemente la toxina A y la toxina B de Clostridioides difficile . Esta antitoxina terapéutica es una fusión de cuatro anticuerpos de dominio único ( nanocuerpos ) que neutralizan de manera potente y amplia los dos principales factores de virulencia de C. difficile en el sitio de infección en modelos preclínicos. [163] El primer ensayo clínico en humanos de levadura viva modificada genéticamente para el tratamiento de la infección por Clostridioides difficile se prevé para 2024 y será patrocinado por el desarrollador Fzata, Inc.
El sistema inmunológico desempeña un papel importante en el cáncer y puede aprovecharse para atacar las células cancerosas. Las terapias basadas en células se centran en las inmunoterapias , principalmente mediante la ingeniería de células T.
Los receptores de células T fueron diseñados y "entrenados" para detectar epítopos de cáncer . Los receptores de antígenos quiméricos (CAR) están compuestos por un fragmento de un anticuerpo fusionado a dominios de señalización intracelular de células T que pueden activar y desencadenar la proliferación de la célula. La FDA ha aprobado múltiples terapias basadas en CAR de segunda generación. [164]
Los interruptores genéticos se diseñaron para mejorar la seguridad del tratamiento. Los interruptores de apagado se desarrollaron para terminar la terapia si el paciente presentaba efectos secundarios graves. [165] Los mecanismos pueden controlar con mayor precisión el sistema y detenerlo y reactivarlo. [166] [167] Dado que la cantidad de células T es importante para la persistencia y la gravedad de la terapia, el crecimiento de las células T también se controla para marcar la efectividad y la seguridad de las terapias. [168]
Aunque varios mecanismos pueden mejorar la seguridad y el control, las limitaciones incluyen la dificultad de inducir grandes circuitos de ADN en las células y los riesgos asociados con la introducción de componentes extraños, especialmente proteínas, en las células.
El biocombustible más popular es el etanol producido a partir de maíz o caña de azúcar, pero este método de producción de biocombustibles es problemático y limitado debido al alto costo agrícola y las características inadecuadas del etanol como combustible. Un sustituto y una fuente potencial de energía renovable son los microbios cuyas vías metabólicas han sido alteradas para que sean más eficientes en la conversión de biomasa en biocombustibles. Sólo si sus costos de producción pudieran igualarse o incluso superar los de la producción actual de combustibles se podría esperar que estas técnicas tuvieran éxito. En relación con esto, hay varios medicamentos cuyos costosos procedimientos de fabricación impiden que tengan un espectro terapéutico más amplio. La creación de nuevos materiales y la fabricación microbiológica de biomateriales se beneficiarían sustancialmente de las nuevas herramientas de biología artificial. [155]
El sistema de repeticiones palindrómicas cortas agrupadas frecuentemente interespaciadas (CRISPR)/CRISPR asociado (Cas) es un método poderoso de ingeniería genómica en una variedad de organismos debido a su simplicidad, modularidad y escalabilidad. En esta técnica, un ARN guía (gRNA) atrae la nucleasa CRISPR Cas9 a un punto particular en el genoma, causando una rotura de doble cadena. Se pueden utilizar varios procesos de reparación del ADN, incluida la recombinación dirigida por homología y la unión de extremos sin homología, para lograr el cambio deseado en el genoma (es decir, la eliminación o inserción de genes). Además, dCas9 (Cas9 muerto o Cas9 deficiente en nucleasa), un mutante doble de Cas9 (H840A, D10A), se ha utilizado para controlar la expresión génica en bacterias o cuando se vincula a una estimulación del sitio de supresión en levadura. [169]
Para construir y desarrollar sistemas biológicos, los componentes reguladores, incluidos los reguladores, los sitios de unión a los ribosomas (RBS) y los terminadores son cruciales. A pesar de años de estudio, existen muchas variedades y cantidades diferentes de promotores y terminadores para Escherichia coli, pero también para el organismo modelo Saccharomyces cerevisiae, bien investigado, así como para otros organismos de interés, estas herramientas son bastante escasas. Se han inventado numerosas técnicas para la búsqueda e identificación de promotores y terminadores con el fin de superar esta limitación, incluida la minería de genomas, la mutagénesis aleatoria, la ingeniería híbrida, el modelado biofísico, el diseño combinatorio y el diseño racional. [169]
La biología sintética se ha utilizado para los organoides , que son órganos cultivados en laboratorio que tienen aplicaciones en la investigación médica y el trasplante. [170]
La bioimpresión 3D se puede utilizar para reconstruir tejido de varias regiones del cuerpo. El precursor de la adopción de la impresión 3D en la atención médica fue una serie de ensayos realizados por investigadores del Boston Children's Hospital. El equipo construyó vejigas urinarias de reemplazo a mano para siete pacientes construyendo andamios, luego colocando capas de células de los pacientes en los andamios y dejándolos crecer. Los ensayos fueron un éxito ya que los pacientes permanecieron en buen estado de salud 7 años después de la implantación, lo que llevó a un investigador llamado Anthony Atala, MD, a buscar formas de automatizar el proceso. [171] Los pacientes con enfermedad de vejiga en etapa terminal ahora pueden ser tratados mediante el uso de tejidos de vejiga bioingeniería para reconstruir el órgano dañado. [172] Esta tecnología también se puede aplicar potencialmente a los huesos, la piel, el cartílago y el tejido muscular. [173] Aunque un objetivo a largo plazo de la tecnología de bioimpresión 3D es reconstruir un órgano completo, así como minimizar el problema de la falta de órganos para trasplante. [174] Ha habido poco éxito en la bioimpresión de órganos completamente funcionales, por ejemplo, hígado, piel, menisco o páncreas. [175] [176] [177] A diferencia de los stents implantables, los órganos tienen formas complejas y son significativamente más difíciles de bioimprimir. Un corazón bioimpreso, por ejemplo, no solo debe cumplir con los requisitos estructurales, sino también con los requisitos de vascularización, carga mecánica y propagación de señales eléctricas. [178] En 2022, se informó del primer éxito de un ensayo clínico para un trasplante bioimpreso en 3D que se realiza a partir de las propias células del paciente, una oreja externa para tratar la microtia , [179] . [180]
La bioimpresión 3D contribuye a avances significativos en el campo médico de la ingeniería de tejidos al permitir que se realicen investigaciones sobre materiales innovadores llamados biomateriales . Algunas de las sustancias de bioingeniería más notables suelen ser más fuertes que los materiales corporales promedio, incluidos los tejidos blandos y los huesos. Estos componentes pueden actuar como futuros sustitutos, incluso mejoras, de los materiales corporales originales. Además, la Agencia de Reducción de Amenazas de Defensa tiene como objetivo imprimir mini órganos como corazones, hígados y pulmones como potencial para probar nuevos medicamentos con mayor precisión y tal vez eliminar la necesidad de realizar pruebas en animales. [181]
Se están llevando a cabo investigaciones y desarrollos sobre métodos basados en la biología sintética para inducir la regeneración en humanos [¿ relevante? ], así como la creación de órganos artificiales trasplantables .
La biología sintética se puede utilizar para crear nanopartículas que se pueden utilizar para la administración de fármacos, así como para otros fines. [182] Como complemento a la investigación y el desarrollo, se busca y se han creado células sintéticas que imitan las funciones de las células biológicas. Las aplicaciones incluyen la medicina, como las nanopartículas de diseño que hacen que las células sanguíneas se coman, de adentro hacia afuera, porciones de placa aterosclerótica que causan ataques cardíacos. [183] [184] [185] Las microgotas sintéticas para células de algas o los reactores microbianos esferoidales multicelulares sinérgicos de algas y bacterias , por ejemplo, podrían usarse para producir hidrógeno como biotecnología de economía del hidrógeno . [186] [187]
Las células de diseño de mamíferos son diseñadas por humanos para comportarse de una manera específica, como una célula inmune que expresa un receptor sintético diseñado para combatir una enfermedad específica. [188] [189] La electrogenética es una aplicación de la biología sintética que implica la utilización de campos eléctricos para estimular una respuesta en células diseñadas. [190] El control de las células de diseño se puede hacer con relativa facilidad mediante el uso de dispositivos electrónicos comunes, como los teléfonos inteligentes. Además, la electrogenética permite la posibilidad de crear dispositivos que son mucho más pequeños y compactos que los dispositivos que utilizan otros estímulos mediante el uso de electrodos microscópicos. [190] Un ejemplo de cómo se utiliza la electrogenética para beneficiar la salud pública es mediante la estimulación de células de diseño que pueden producir/administrar terapias. [191] Esto se implementó en células Electro HEK, células que contienen canales de calcio dependientes del voltaje que son electrosensibles, lo que significa que el canal de iones puede controlarse por conducción eléctrica entre electrodos y las células Electro HEK. [190] Se ha demostrado que los niveles de expresión del gen artificial que contienen estas células Electro HEK pueden controlarse modificando el voltaje o la duración del pulso eléctrico. Estudios posteriores han ampliado este sistema robusto, uno de los cuales es un sistema de línea celular beta diseñado para controlar la liberación de insulina en función de señales eléctricas. [192]
La creación de nueva vida y la manipulación de la vida existente han suscitado preocupaciones éticas en el campo de la biología sintética y se están debatiendo activamente. [193] [194]
Las preguntas éticas más comunes incluyen:
Los aspectos éticos de la biología sintética tienen tres características principales: bioseguridad , bioprotección y la creación de nuevas formas de vida. [196] Otras cuestiones éticas mencionadas incluyen la regulación de nuevas creaciones, la gestión de patentes de nuevas creaciones, la distribución de beneficios y la integridad de la investigación. [197] [193]
Han surgido cuestiones éticas en relación con las tecnologías de ADN recombinante y organismos genéticamente modificados (OGM), y en muchas jurisdicciones se han establecido amplias regulaciones de la ingeniería genética y la investigación sobre patógenos. Amy Gutmann , ex directora de la Comisión Presidencial de Bioética, sostuvo que deberíamos evitar la tentación de regular excesivamente la biología sintética en general y la ingeniería genética en particular. Según Gutmann, "la parsimonia regulatoria es especialmente importante en las tecnologías emergentes... donde la tentación de sofocar la innovación sobre la base de la incertidumbre y el miedo a lo desconocido es particularmente grande. Los instrumentos contundentes de la restricción estatutaria y regulatoria pueden no sólo inhibir la distribución de nuevos beneficios, sino también ser contraproducentes para la seguridad al impedir que los investigadores desarrollen salvaguardas efectivas". [198]
Una cuestión ética es si es aceptable o no crear nuevas formas de vida, a veces conocido como "jugar a ser Dios". Actualmente, la creación de nuevas formas de vida que no están presentes en la naturaleza es a pequeña escala, los beneficios y peligros potenciales siguen siendo desconocidos, y se garantiza una cuidadosa consideración y supervisión para la mayoría de los estudios. [193] Muchos defensores expresan el gran valor potencial (para la agricultura, la medicina y el conocimiento académico, entre otros campos) de la creación de formas de vida artificiales. La creación de nuevas entidades podría expandir el conocimiento científico mucho más allá de lo que se conoce actualmente a partir del estudio de los fenómenos naturales. Sin embargo, existe la preocupación de que las formas de vida artificiales puedan reducir la "pureza" de la naturaleza (es decir, la naturaleza podría ser corrompida de alguna manera por la intervención y manipulación humanas) y potencialmente influir en la adopción de principios más parecidos a la ingeniería en lugar de ideales centrados en la biodiversidad y la naturaleza. A algunos también les preocupa que si una forma de vida artificial fuera liberada en la naturaleza, podría obstaculizar la biodiversidad al superar a las especies naturales en la búsqueda de recursos (similar a cómo las floraciones de algas matan a las especies marinas). Otra preocupación tiene que ver con el tratamiento ético de las entidades recién creadas si sienten dolor , sensibilidad y autopercepción. Existe un debate en curso sobre si a estas formas de vida se les deben conceder derechos morales o legales, aunque no hay consenso sobre cómo se administrarían o harían cumplir esos derechos.
Las razones éticas y morales que sustentan ciertas aplicaciones de la biología sintética incluyen su potencial mitigación de importantes problemas globales de impactos ambientales perjudiciales de la agricultura convencional (incluida la producción de carne ), el bienestar animal , la seguridad alimentaria y la salud humana , [199] [200] [201] [202] así como la potencial reducción de las necesidades de mano de obra humana y, a través de terapias de enfermedades, la reducción del sufrimiento humano y la prolongación de la vida.
¿Qué es lo más éticamente apropiado al considerar las medidas de bioseguridad? ¿Cómo se puede evitar la introducción accidental de vida sintética en el medio ambiente natural? Se ha dedicado mucha consideración ética y reflexión crítica a estas cuestiones. La bioseguridad no sólo se refiere a la contención biológica; también se refiere a los pasos dados para proteger al público de agentes biológicos potencialmente peligrosos. Aunque estas preocupaciones son importantes y siguen sin respuesta, no todos los productos de la biología sintética presentan problemas de seguridad biológica o consecuencias negativas para el medio ambiente. Se sostiene que la mayoría de las tecnologías sintéticas son benignas e incapaces de prosperar en el mundo exterior debido a sus características "antinaturales", ya que todavía no existe un ejemplo de un microbio transgénico al que se le haya conferido una ventaja de adaptación en la naturaleza.
En general, los controles de peligros existentes , las metodologías de evaluación de riesgos y las regulaciones desarrolladas para los organismos genéticamente modificados (OGM) tradicionales se consideran suficientes para los organismos sintéticos. Los métodos de biocontención "extrínsecos" en un contexto de laboratorio incluyen la contención física a través de cabinas de bioseguridad y cajas de guantes , así como equipo de protección personal . En un contexto agrícola, incluyen distancias de aislamiento y barreras de polen , similares a los métodos de biocontención de OGM . Los organismos sintéticos pueden ofrecer un mayor control de peligros porque pueden diseñarse con métodos de biocontención "intrínsecos" que limitan su crecimiento en un entorno no contenido o previenen la transferencia horizontal de genes a organismos naturales. Los ejemplos de biocontención intrínseca incluyen auxotrofia , interruptores de muerte biológicos , incapacidad del organismo para replicarse o pasar genes modificados o sintéticos a la descendencia, y el uso de organismos xenobiológicos que utilizan bioquímica alternativa, por ejemplo, utilizando ácidos nucleicos xeno artificiales (XNA) en lugar de ADN. [203] [204] En cuanto a la auxotrofia, las bacterias y las levaduras pueden modificarse genéticamente para que no produzcan histidina , un aminoácido importante para toda la vida. Por lo tanto, dichos organismos solo pueden cultivarse en medios ricos en histidina en condiciones de laboratorio, lo que anula los temores de que puedan propagarse a áreas indeseables.
Algunas cuestiones éticas se relacionan con la bioseguridad, ya que las tecnologías biosintéticas podrían utilizarse deliberadamente para causar daño a la sociedad y/o al medio ambiente. Dado que la biología sintética plantea cuestiones éticas y de bioseguridad, la humanidad debe considerar y planificar cómo lidiar con creaciones potencialmente dañinas y qué tipo de medidas éticas podrían emplearse para disuadir a las tecnologías biosintéticas nefastas. Sin embargo, con la excepción de la regulación de las empresas de biología sintética y biotecnología, [205] [206] las cuestiones no se consideran nuevas porque se plantearon durante los debates anteriores sobre el ADN recombinante y los organismos genéticamente modificados (OGM), y ya existen amplias regulaciones de la ingeniería genética y la investigación sobre patógenos en muchas jurisdicciones. [207]
Además, el desarrollo de herramientas de biología sintética ha hecho que sea más fácil para las personas con menos educación, capacitación y acceso a equipos modificar y utilizar organismos patógenos como armas biológicas. Esto aumenta la amenaza del bioterrorismo , especialmente a medida que los grupos terroristas toman conciencia de los importantes trastornos sociales, económicos y políticos causados por pandemias como la COVID-19 . A medida que se desarrollan nuevas técnicas en el campo de la biología sintética, es probable que el riesgo de bioterrorismo siga creciendo. [208] Juan Zárate, quien se desempeñó como Asesor Adjunto de Seguridad Nacional para Combatir el Terrorismo de 2005 a 2009, señaló que "la gravedad y la disrupción extrema de un nuevo coronavirus probablemente estimularán la imaginación de los grupos e individuos más creativos y peligrosos a reconsiderar los ataques bioterroristas". [209]
El proyecto SYNBIOSAFE [210] , financiado por la Unión Europea, ha publicado informes sobre cómo gestionar la biología sintética. En un artículo de 2007 se identificaron cuestiones clave en materia de seguridad, protección, ética y la interfaz entre ciencia y sociedad, que el proyecto definió como educación pública y diálogo permanente entre científicos, empresas, gobierno y especialistas en ética. [211] [212] Las cuestiones clave de seguridad que SYNBIOSAFE identificó implicaban la participación de empresas que venden ADN sintético y de la comunidad de biólogos aficionados que practican el biohacking . Las cuestiones éticas clave se referían a la creación de nuevas formas de vida.
Un informe posterior se centró en la bioseguridad, especialmente en el llamado desafío del doble uso . Por ejemplo, si bien la biología sintética puede conducir a una producción más eficiente de tratamientos médicos, también puede conducir a la síntesis o modificación de patógenos nocivos (por ejemplo, la viruela ). [213] La comunidad de biohacking sigue siendo una fuente de especial preocupación, ya que la naturaleza distribuida y difusa de la biotecnología de código abierto dificulta el seguimiento, la regulación o la mitigación de posibles preocupaciones sobre la bioseguridad y la bioprotección. [214]
COSY, otra iniciativa europea, se centra en la percepción y comunicación pública. [215] [216] [217] Para comunicar mejor la biología sintética y sus ramificaciones sociales a un público más amplio, COSY y SYNBIOSAFE publicaron SYNBIOSAFE , un documental de 38 minutos, en octubre de 2009. [210]
La Asociación Internacional de Biología Sintética ha propuesto la autorregulación [218] . En ella se proponen medidas específicas que la industria de la biología sintética, especialmente las empresas de síntesis de ADN, deberían implementar. En 2007, un grupo dirigido por científicos de las principales empresas de síntesis de ADN publicó un "plan práctico para desarrollar un marco de supervisión eficaz para la industria de síntesis de ADN". [205]
En enero de 2009, la Fundación Alfred P. Sloan financió el Centro Woodrow Wilson , el Centro Hastings y el Instituto J. Craig Venter para examinar la percepción pública, la ética y las implicaciones políticas de la biología sintética. [219]
Los días 9 y 10 de julio de 2009, el Comité de Ciencia, Tecnología y Derecho de las Academias Nacionales convocó un simposio sobre “Oportunidades y desafíos en el campo emergente de la biología sintética”. [220]
Tras la publicación del primer genoma sintético y la cobertura mediática que lo acompañó sobre la creación de "vida", el presidente Barack Obama creó la Comisión Presidencial para el Estudio de Cuestiones Bioéticas para estudiar la biología sintética. [221] La comisión convocó una serie de reuniones y publicó un informe en diciembre de 2010 titulado "Nuevas direcciones: la ética de la biología sintética y las tecnologías emergentes". La comisión afirmó que "si bien el logro de Venter marcó un avance técnico significativo al demostrar que un genoma relativamente grande podía sintetizarse con precisión y sustituirse por otro, no equivalía a la "creación de vida". [222] Señaló que la biología sintética es un campo emergente, que crea riesgos y recompensas potenciales. La comisión no recomendó cambios en las políticas o la supervisión y pidió que se siguiera financiando la investigación y se financiara más para el seguimiento, el estudio de cuestiones éticas emergentes y la educación pública. [207]
La biología sintética, como herramienta principal para los avances biológicos, genera "la posibilidad de desarrollar armas biológicas, posibles impactos negativos imprevistos sobre la salud humana... y cualquier posible impacto ambiental". [223] La proliferación de esa tecnología también podría poner la producción de armas biológicas y químicas al alcance de una gama más amplia de actores estatales y no estatales . [224] Estos problemas de seguridad se pueden evitar regulando los usos industriales de la biotecnología mediante una legislación de políticas. La "Comisión de Bioética del Presidente... en respuesta a la creación anunciada de una célula autorreplicante a partir de un genoma sintetizado químicamente, ha propuesto 18 recomendaciones no sólo para regular la ciencia... sino también para educar al público". [223]
El 13 de marzo de 2012, más de 100 grupos ambientales y de la sociedad civil, incluidos Amigos de la Tierra , el Centro Internacional para la Evaluación de Tecnología y el Grupo ETC , publicaron el manifiesto Principios para la Supervisión de la Biología Sintética . Este manifiesto exige una moratoria mundial sobre la liberación y el uso comercial de organismos sintéticos hasta que se establezcan regulaciones más sólidas y medidas de bioseguridad rigurosas. Los grupos piden específicamente una prohibición total del uso de la biología sintética en el genoma humano o el microbioma humano . [225] [226] Richard Lewontin escribió que algunos de los principios de seguridad para la supervisión discutidos en Principios para la Supervisión de la Biología Sintética son razonables, pero que el principal problema con las recomendaciones del manifiesto es que "el público en general carece de la capacidad para hacer cumplir cualquier realización significativa de esas recomendaciones". [227]
Los peligros de la biología sintética incluyen peligros de bioseguridad para los trabajadores y el público, peligros de bioseguridad derivados de la ingeniería deliberada de organismos para causar daño y peligros ambientales. [228] Los peligros de bioseguridad son similares a los de los campos existentes de la biotecnología, principalmente la exposición a patógenos y sustancias químicas tóxicas, aunque los nuevos organismos sintéticos pueden tener riesgos novedosos. [203] Para la bioseguridad, existe la preocupación de que los organismos sintéticos o rediseñados podrían teóricamente usarse para el bioterrorismo . Los riesgos potenciales incluyen recrear patógenos conocidos desde cero, diseñar patógenos existentes para que sean más peligrosos y diseñar microbios para producir bioquímicos dañinos. [229] Por último, los peligros ambientales incluyen efectos adversos sobre la biodiversidad y los servicios ecosistémicos , incluidos posibles cambios en el uso de la tierra resultantes del uso agrícola de organismos sintéticos. [230] [231] La biología sintética es un ejemplo de una tecnología de doble uso con el potencial de ser utilizada en formas que podrían dañar intencional o involuntariamente a los humanos y/o dañar el medio ambiente. A menudo, "los científicos, las instituciones que los acogen y los organismos de financiación" consideran si la investigación planificada podría ser utilizada indebidamente y, a veces, aplican medidas para reducir la probabilidad de que se haga un uso indebido. [232]
Los sistemas de análisis de riesgos existentes para los OGM se consideran generalmente suficientes para los organismos sintéticos, aunque puede haber dificultades para un organismo construido "de abajo hacia arriba" a partir de secuencias genéticas individuales. [204] [233] La biología sintética generalmente se rige por las regulaciones existentes para los OGM y la biotecnología en general, y cualquier regulación que exista para productos comerciales posteriores, aunque generalmente no hay regulaciones en ninguna jurisdicción que sean específicas para la biología sintética. [234] [235]
{{cite journal}}: Parámetro desconocido |agency=ignorado ( ayuda ){{cite news}}: |last2=tiene nombre genérico ( ayuda ){{cite web}}: Falta o está vacío |title=( ayuda ){{cite book}}: |website=ignorado ( ayuda ){{cite book}}: |website=ignorado ( ayuda )