

Ácido desoxirribonucleico ( / d iː ˈ ɒ k s ɪ ˌ r aɪ b oʊ nj uː ˌ k l iː ɪ k , - ˌ k l eɪ -/ ⓘ ;[1] ADN) es unpolímerocompuesto por dosde polinucleótidosque se enrollan entre sí para formar unadoble hélice. El polímero llevagenéticaspara el desarrollo, funcionamiento, crecimiento yreproducciónde todoslos organismosy de muchosvirus. El ADN yel ácido ribonucleico(ARN) sonácidos nucleicos. Junto conlas proteínas,los lípidosy los carbohidratos complejos (polisacáridos), los ácidos nucleicos son uno de los cuatro tipos principales demacromoléculasesenciales para todas las formas devida.
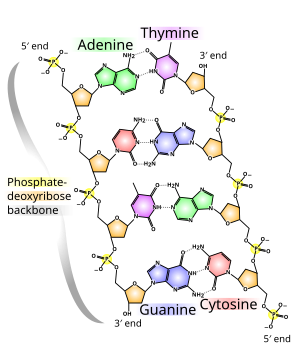
Las dos cadenas de ADN se conocen como polinucleótidos porque están compuestas de unidades monoméricas más simples llamadas nucleótidos . [2] [3] Cada nucleótido está compuesto por una de cuatro nucleobases que contienen nitrógeno ( citosina [C], guanina [G], adenina [A] o timina [T]), un azúcar llamado desoxirribosa y un grupo fosfato . Los nucleótidos están unidos entre sí en una cadena mediante enlaces covalentes (conocidos como enlace fosfodiéster ) entre el azúcar de un nucleótido y el fosfato del siguiente, lo que da como resultado una estructura alterna de azúcar y fosfato . Las bases nitrogenadas de las dos cadenas separadas de polinucleótidos se unen, según las reglas de emparejamiento de bases (A con T y C con G), con enlaces de hidrógeno para formar ADN bicatenario. Las bases nitrogenadas complementarias se dividen en dos grupos, las pirimidinas de un solo anillo y las purinas de doble anillo . En el ADN, las pirimidinas son timina y citosina; las purinas son adenina y guanina.
Ambas cadenas de ADN bicatenario almacenan la misma información biológica . Esta información se replica cuando las dos hebras se separan. Una gran parte del ADN (más del 98% en el caso de los humanos) no es codificante , lo que significa que estas secciones no sirven como patrones para secuencias de proteínas . Las dos hebras de ADN van en direcciones opuestas y, por tanto, son antiparalelas . Unido a cada azúcar hay uno de los cuatro tipos de nucleobases (o bases ). Es la secuencia de estas cuatro nucleobases a lo largo de la columna vertebral la que codifica la información genética. Las cadenas de ARN se crean utilizando cadenas de ADN como plantilla en un proceso llamado transcripción , donde las bases del ADN se intercambian por sus bases correspondientes excepto en el caso de la timina (T), cuyo ARN sustituye al uracilo (U). [4] Según el código genético , estas cadenas de ARN especifican la secuencia de aminoácidos dentro de las proteínas en un proceso llamado traducción .
Dentro de las células eucariotas, el ADN está organizado en estructuras largas llamadas cromosomas . Antes de la división celular típica , estos cromosomas se duplican en el proceso de replicación del ADN, proporcionando un conjunto completo de cromosomas para cada célula hija. Los organismos eucariotas ( animales , plantas , hongos y protistas ) almacenan la mayor parte de su ADN dentro del núcleo celular como ADN nuclear , y algo en las mitocondrias como ADN mitocondrial o en los cloroplastos como ADN de cloroplasto . [5] En cambio, los procariotas ( bacterias y arqueas ) almacenan su ADN únicamente en el citoplasma , en cromosomas circulares . Dentro de los cromosomas eucariotas, las proteínas de la cromatina , como las histonas , compactan y organizan el ADN. Estas estructuras compactadoras guían las interacciones entre el ADN y otras proteínas, ayudando a controlar qué partes del ADN se transcriben.
El ADN es un polímero largo formado por unidades repetidas llamadas nucleótidos . [6] [7] La estructura del ADN es dinámica a lo largo de su longitud, siendo capaz de enrollarse en bucles apretados y otras formas. [8] En todas las especies está compuesto por dos cadenas helicoidales, unidas entre sí por enlaces de hidrógeno . Ambas cadenas están enrolladas alrededor del mismo eje y tienen el mismo paso de 34 ångströms (3,4 nm ). El par de cadenas tiene un radio de 10 Å (1,0 nm). [9] Según otro estudio, cuando se midió en una solución diferente, la cadena de ADN midió entre 22 y 26 Å (2,2 y 2,6 nm) de ancho y una unidad de nucleótido midió 3,3 Å (0,33 nm) de largo. [10] La densidad de flotación de la mayor parte del ADN es de 1,7 g/cm 3 . [11]
El ADN no suele existir como una sola hebra, sino como un par de hebras que se mantienen estrechamente unidas. [9] [12] Estos dos largos hilos se enrollan uno alrededor del otro, en forma de doble hélice . El nucleótido contiene tanto un segmento de la columna vertebral de la molécula (que mantiene unida la cadena) como una nucleobase (que interactúa con la otra cadena de ADN en la hélice). Una nucleobase unida a un azúcar se llama nucleósido , y una base unida a un azúcar y a uno o más grupos fosfato se llama nucleótido . Un biopolímero que comprende múltiples nucleótidos unidos (como en el ADN) se llama polinucleótido . [13]
La columna vertebral de la cadena de ADN está formada por grupos alternos de fosfato y azúcar . [14] El azúcar en el ADN es 2-desoxirribosa , que es un azúcar pentosa (cinco carbonos ). Los azúcares están unidos por grupos fosfato que forman enlaces fosfodiéster entre el tercer y quinto átomo de carbono de los anillos de azúcar adyacentes. Estos se conocen como carbonos del extremo 3' (tres extremos principales) y del extremo 5' (cinco extremos principales), y el símbolo principal se utiliza para distinguir estos átomos de carbono de los de la base con la que la desoxirribosa forma un enlace glicosídico . . [12]
Por lo tanto, cualquier cadena de ADN normalmente tiene un extremo en el que hay un grupo fosfato unido al carbono 5' de una ribosa (el fosforilo 5') y otro extremo en el que hay un grupo hidroxilo libre unido al carbono 3' de una ribosa. ribosa (el 3′ hidroxilo). La orientación de los carbonos 3′ y 5′ a lo largo de la cadena principal de azúcar-fosfato confiere direccionalidad (a veces llamada polaridad) a cada cadena de ADN. En una doble hélice de ácido nucleico , la dirección de los nucleótidos en una hebra es opuesta a su dirección en la otra hebra: las hebras son antiparalelas . Se dice que los extremos asimétricos de las cadenas de ADN tienen una direccionalidad de cinco extremos principales (5') y tres extremos principales (3'), teniendo el extremo 5' un grupo fosfato terminal y el extremo 3' un grupo hidroxilo terminal. Una diferencia importante entre el ADN y el ARN es el azúcar, ya que la 2-desoxirribosa del ADN es reemplazada por la ribosa del azúcar pentosa relacionada en el ARN. [12]
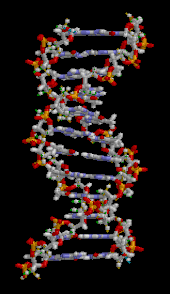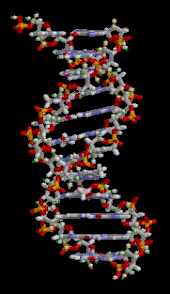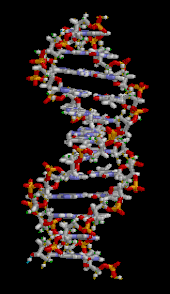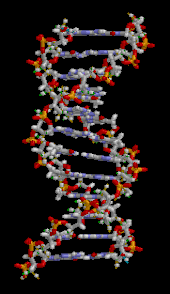
La doble hélice del ADN se estabiliza principalmente mediante dos fuerzas: enlaces de hidrógeno entre nucleótidos e interacciones de apilamiento de bases entre nucleobases aromáticas . [16] Las cuatro bases que se encuentran en el ADN son adenina ( A ), citosina ( C ), guanina ( G ) y timina ( T ). Estas cuatro bases están unidas al azúcar-fosfato para formar el nucleótido completo, como se muestra en el caso del monofosfato de adenosina . La adenina se empareja con timina y la guanina con citosina, formando pares de bases AT y GC . [17] [18]
Las nucleobases se clasifican en dos tipos: las purinas , A y G , que son compuestos heterocíclicos fusionados de cinco y seis miembros , y las pirimidinas , los anillos de seis miembros C y T. [12] Una quinta nucleobase de pirimidina, el uracilo ( U ), generalmente ocupa el lugar de la timina en el ARN y se diferencia de la timina por carecer de un grupo metilo en su anillo. Además del ARN y el ADN, se han creado muchos análogos artificiales de ácidos nucleicos para estudiar las propiedades de los ácidos nucleicos o para su uso en biotecnología. [19]
Las bases modificadas se encuentran en el ADN. El primero de ellos reconocido fue la 5-metilcitosina , que se encontró en el genoma de Mycobacterium tuberculosis en 1925. [20] La razón de la presencia de estas bases no canónicas en los virus bacterianos ( bacteriófagos ) es evitar las enzimas de restricción presentes en las bacterias. Este sistema enzimático actúa al menos en parte como un sistema inmunológico molecular que protege a las bacterias de la infección por virus. [21] Las modificaciones de las bases citosina y adenina, las bases de ADN más comunes y modificadas, desempeñan funciones vitales en el control epigenético de la expresión genética en plantas y animales. [22]
Se sabe que en el ADN se encuentran varias bases no canónicas. [23] La mayoría de estos son modificaciones de las bases canónicas más uracilo.

Las hebras helicoidales gemelas forman la columna vertebral del ADN. Se puede encontrar otra doble hélice trazando los espacios o surcos entre las hebras. Estos huecos son adyacentes a los pares de bases y pueden proporcionar un sitio de unión . Como los cordones no están situados simétricamente entre sí, las ranuras tienen tamaños desiguales. El surco mayor tiene 22 ångströms (2,2 nm) de ancho, mientras que el surco menor tiene 12 Å (1,2 nm) de ancho. [24] Debido al mayor ancho del surco mayor, los bordes de las bases son más accesibles en el surco mayor que en el surco menor. Como resultado, proteínas como los factores de transcripción que pueden unirse a secuencias específicas en el ADN bicatenario generalmente hacen contacto con los lados de las bases expuestas en el surco principal. [25] Esta situación varía en conformaciones inusuales del ADN dentro de la célula (ver más abajo) , pero los surcos mayores y menores siempre se nombran para reflejar las diferencias en ancho que se verían si el ADN se retorciera nuevamente a la forma B ordinaria .
En una doble hélice de ADN, cada tipo de nucleobase de una hebra se une con solo un tipo de nucleobase de la otra hebra. Esto se llama emparejamiento de bases complementarias . Las purinas forman enlaces de hidrógeno con las pirimidinas, la adenina se une sólo a la timina en dos enlaces de hidrógeno y la citosina sólo a la guanina en tres enlaces de hidrógeno. Esta disposición de dos nucleótidos que se unen a través de la doble hélice (de un anillo de seis carbonos a un anillo de seis carbonos) se denomina par de bases de Watson-Crick. El ADN con un alto contenido de GC es más estable que el ADN con un bajo contenido de GC . Un par de bases de Hoogsteen (el enlace de hidrógeno del anillo de 6 carbonos al anillo de 5 carbonos) es una variación poco común del emparejamiento de bases. [26] Como los enlaces de hidrógeno no son covalentes , se pueden romper y volver a unir con relativa facilidad. De este modo, las dos hebras de ADN en una doble hélice pueden separarse como una cremallera, ya sea mediante una fuerza mecánica o una alta temperatura . [27] Como resultado de esta complementariedad de pares de bases, toda la información en la secuencia bicatenaria de una hélice de ADN se duplica en cada hebra, lo cual es vital en la replicación del ADN. Esta interacción reversible y específica entre pares de bases complementarias es fundamental para todas las funciones del ADN en los organismos. [7]
La mayoría de las moléculas de ADN son en realidad dos cadenas de polímeros unidas en forma helicoidal mediante enlaces no covalentes; esta estructura bicatenaria (ADNbc) se mantiene en gran medida gracias a las interacciones de apilamiento de bases dentro de la cadena, que son más fuertes para las pilas G,C . Las dos cadenas pueden separarse (un proceso conocido como fusión) para formar dos moléculas de ADN monocatenario (ADNss). La fusión ocurre a altas temperaturas, poca sal y alto pH (un pH bajo también derrite el ADN, pero como el ADN es inestable debido a la depurinación ácida, rara vez se usa un pH bajo).
La estabilidad de la forma de ADNbc depende no sólo del contenido de GC (% de pares de bases G,C ) sino también de la secuencia (ya que el apilamiento es específico de la secuencia) y también de la longitud (las moléculas más largas son más estables). La estabilidad se puede medir de varias formas; una forma común es la temperatura de fusión (también llamada valor Tm ), que es la temperatura a la que el 50% de las moléculas de doble cadena se convierten en moléculas de una sola cadena; La temperatura de fusión depende de la fuerza iónica y de la concentración de ADN. Como resultado, es tanto el porcentaje de pares de bases de GC como la longitud total de una doble hélice de ADN lo que determina la fuerza de la asociación entre las dos hebras de ADN. Las hélices de ADN largas con un alto contenido de GC tienen hebras que interactúan más fuertemente, mientras que las hélices cortas con un alto contenido de AT tienen hebras que interactúan más débilmente. [28] En biología, las partes de la doble hélice del ADN que necesitan separarse fácilmente, como la caja TATAAT Pribnow en algunos promotores , tienden a tener un alto contenido de AT , lo que hace que las hebras sean más fáciles de separar. [29]
En el laboratorio, la fuerza de esta interacción se puede medir encontrando la temperatura de fusión Tm necesaria para romper la mitad de los enlaces de hidrógeno. Cuando todos los pares de bases en una doble hélice de ADN se funden, las hebras se separan y existen en solución como dos moléculas completamente independientes. Estas moléculas de ADN monocatenario no tienen una forma común única, pero algunas conformaciones son más estables que otras. [30]

En los seres humanos, el genoma nuclear diploide femenino total por célula se extiende por 6,37 pares de gigabases (Gbp), mide 208,23 cm de largo y pesa 6,51 picogramos (pg). [31] Los valores masculinos son 6,27 Gbp, 205,00 cm, 6,41 pg. [31] Cada polímero de ADN puede contener cientos de millones de nucleótidos, como en el cromosoma 1 . El cromosoma 1 es el cromosoma humano más grande con aproximadamente 220 millones de pares de bases , y sería85 mm de largo si está enderezado. [32]
En los eucariotas , además del ADN nuclear , también existe el ADN mitocondrial (ADNmt) que codifica determinadas proteínas utilizadas por las mitocondrias. El ADNmt suele ser relativamente pequeño en comparación con el ADN nuclear. Por ejemplo, el ADN mitocondrial humano forma moléculas circulares cerradas, cada una de las cuales contiene 16.569 [33] [34] pares de bases de ADN, [35] y cada una de estas moléculas normalmente contiene un conjunto completo de genes mitocondriales. Cada mitocondria humana contiene, en promedio, aproximadamente 5 de estas moléculas de ADNmt. [35] Cada célula humana contiene aproximadamente 100 mitocondrias, lo que da un número total de moléculas de ADNmt por célula humana de aproximadamente 500. [35] Sin embargo, la cantidad de mitocondrias por célula también varía según el tipo de célula, y un óvulo puede contener 100.000 mitocondrias. , correspondiente a hasta 1.500.000 copias del genoma mitocondrial (que constituye hasta el 90% del ADN de la célula). [36]
Una secuencia de ADN se denomina secuencia "sentido" si es la misma que la de una copia de ARN mensajero que se traduce en proteína. [37] La secuencia en la hebra opuesta se llama secuencia "antisentido". Tanto las secuencias sentido como las antisentido pueden existir en diferentes partes de la misma hebra de ADN (es decir, ambas hebras pueden contener secuencias tanto sentido como antisentido). Tanto en procariotas como en eucariotas se producen secuencias de ARN antisentido, pero las funciones de estos ARN no están del todo claras. [38] Una propuesta es que los ARN antisentido participan en la regulación de la expresión génica a través del emparejamiento de bases ARN-ARN. [39]
Algunas secuencias de ADN en procariotas y eucariotas, y más en plásmidos y virus , desdibujan la distinción entre cadenas sentido y antisentido al tener genes superpuestos . [40] En estos casos, algunas secuencias de ADN cumplen una doble función: codifican una proteína cuando se lee a lo largo de una cadena y una segunda proteína cuando se lee en la dirección opuesta a lo largo de la otra cadena. En las bacterias , esta superposición puede estar implicada en la regulación de la transcripción genética, [41] mientras que en los virus, los genes superpuestos aumentan la cantidad de información que puede codificarse dentro del pequeño genoma viral. [42]
El ADN se puede torcer como una cuerda en un proceso llamado superenrollamiento del ADN . Con el ADN en su estado "relajado", una hebra normalmente rodea el eje de la doble hélice una vez cada 10,4 pares de bases, pero si el ADN se tuerce las hebras se enrollan más apretadas o más sueltas. [43] Si el ADN se tuerce en la dirección de la hélice, se trata de un superenrollamiento positivo y las bases se mantienen más juntas. Si se tuercen en dirección opuesta, se trata de un superenrollamiento negativo y las bases se separan más fácilmente. En la naturaleza, la mayor parte del ADN tiene un ligero superenrollamiento negativo que es introducido por unas enzimas llamadas topoisomerasas . [44] Estas enzimas también son necesarias para aliviar las tensiones de torsión introducidas en las cadenas de ADN durante procesos como la transcripción y la replicación del ADN . [45]

El ADN existe en muchas conformaciones posibles que incluyen las formas A-DNA , B-DNA y Z-DNA , aunque sólo el B-DNA y el Z-DNA se han observado directamente en organismos funcionales. [14] La conformación que adopta el ADN depende del nivel de hidratación, la secuencia del ADN, la cantidad y dirección del superenrollamiento, las modificaciones químicas de las bases, el tipo y concentración de iones metálicos y la presencia de poliaminas en solución. [46]
Los primeros informes publicados sobre los patrones de difracción de rayos X del ADN-A (y también del ADN-B) utilizaron análisis basados en funciones de Patterson que proporcionaban sólo una cantidad limitada de información estructural para las fibras orientadas del ADN. [47] [48] Wilkins et al. propusieron un análisis alternativo . en 1953 para los patrones de dispersión y difracción de rayos X del ADN B in vivo de fibras de ADN altamente hidratadas en términos de cuadrados de funciones de Bessel . [49] En la misma revista, James Watson y Francis Crick presentaron su análisis de modelado molecular de los patrones de difracción de rayos X del ADN para sugerir que la estructura era una doble hélice. [9]
Aunque la forma de ADN B es más común en las condiciones que se encuentran en las células, [50] no es una conformación bien definida sino una familia de conformaciones de ADN relacionadas [51] que ocurren en los altos niveles de hidratación presentes en las células. Sus correspondientes patrones de difracción y dispersión de rayos X son característicos de paracristales moleculares con un grado significativo de desorden. [52] [53]
En comparación con el ADN-B, la forma del ADN-A es una espiral derecha más ancha , con un surco menor ancho y poco profundo y un surco mayor más estrecho y profundo. La forma A se produce en condiciones no fisiológicas en muestras de ADN parcialmente deshidratadas, mientras que en la célula puede producirse en pares híbridos de cadenas de ADN y ARN, y en complejos enzima-ADN. [54] [55] Los segmentos de ADN donde las bases han sido modificadas químicamente mediante metilación pueden sufrir un cambio mayor en la conformación y adoptar la forma Z. Aquí, los hilos giran alrededor del eje helicoidal en una espiral hacia la izquierda, lo opuesto a la forma B más común. [56] Estas estructuras inusuales pueden ser reconocidas por proteínas de unión a ADN-Z específicas y pueden estar involucradas en la regulación de la transcripción. [57]
Durante muchos años, los exobiólogos han propuesto la existencia de una biosfera en la sombra , una biosfera microbiana postulada de la Tierra que utiliza procesos bioquímicos y moleculares radicalmente diferentes a los de la vida actualmente conocida. Una de las propuestas fue la existencia de formas de vida que utilicen arsénico en lugar de fósforo en el ADN . En 2010 se anunció un informe sobre la posibilidad en la bacteria GFAJ-1 , [58] [59] aunque la investigación fue cuestionada, [59] [60] y la evidencia sugiere que la bacteria previene activamente la incorporación de arsénico en la columna vertebral del ADN y otras biomoléculas. [61]
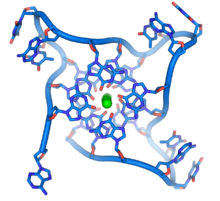
En los extremos de los cromosomas lineales hay regiones especializadas de ADN llamadas telómeros . La función principal de estas regiones es permitir que la célula replique los extremos de los cromosomas utilizando la enzima telomerasa , ya que las enzimas que normalmente replican el ADN no pueden copiar los extremos 3' de los cromosomas. [63] Estas tapas de cromosomas especializadas también ayudan a proteger los extremos del ADN e impiden que los sistemas de reparación del ADN en la célula los traten como daños que deben corregirse. [64] En las células humanas , los telómeros suelen ser longitudes de ADN monocatenario que contienen varios miles de repeticiones de una secuencia TTAGGG simple. [sesenta y cinco]
Estas secuencias ricas en guanina pueden estabilizar los extremos de los cromosomas formando estructuras de conjuntos apilados de unidades de cuatro bases, en lugar de los pares de bases habituales que se encuentran en otras moléculas de ADN. Aquí, cuatro bases de guanina, conocidas como tétrada de guanina , forman una placa plana. Estas unidades planas de cuatro bases luego se apilan una encima de la otra para formar una estructura G-quadruplex estable . [66] Estas estructuras se estabilizan mediante enlaces de hidrógeno entre los bordes de las bases y quelación de un ion metálico en el centro de cada unidad de cuatro bases. [67] También se pueden formar otras estructuras, con el conjunto central de cuatro bases provenientes de una sola hebra doblada alrededor de las bases, o de varias hebras paralelas diferentes, cada una de las cuales contribuye con una base a la estructura central.
Además de estas estructuras apiladas, los telómeros también forman grandes estructuras en bucle llamadas bucles de telómeros o bucles en T. Aquí, el ADN monocatenario se enrolla formando un largo círculo estabilizado por proteínas de unión a los telómeros. [68] Al final del bucle en T, el ADN del telómero monocatenario se sujeta a una región de ADN de doble hebra mediante la hebra del telómero que interrumpe el ADN de doble hélice y el emparejamiento de bases con una de las dos hebras. Esta estructura de triple cadena se llama bucle de desplazamiento o bucle D. [66]
En el ADN, el desgaste se produce cuando existen regiones no complementarias al final de una doble hebra de ADN que de otro modo sería complementaria. Sin embargo, puede producirse ADN ramificado si se introduce una tercera hebra de ADN que contiene regiones contiguas capaces de hibridarse con las regiones deshilachadas de la doble hebra preexistente. Aunque el ejemplo más simple de ADN ramificado involucra sólo tres hebras de ADN, también son posibles complejos que involucran hebras adicionales y múltiples ramas. [69] El ADN ramificado se puede utilizar en nanotecnología para construir formas geométricas; consulte la sección sobre usos en tecnología a continuación.
Se han sintetizado varias nucleobases artificiales y se han incorporado con éxito en el análogo de ADN de ocho bases denominado ADN Hachimoji . Estas bases artificiales, denominadas S, B, P y Z, son capaces de unirse entre sí de forma predecible (S – B y P – Z), mantener la estructura de doble hélice del ADN y transcribirse a ARN. Su existencia podría verse como una indicación de que no hay nada especial en las cuatro nucleobases naturales que evolucionaron en la Tierra. [70] [71] Por otro lado, el ADN está estrechamente relacionado con el ARN , que no solo actúa como transcrito del ADN sino que también realiza como máquinas moleculares muchas tareas en las células. Para ello tiene que plegarse formando una estructura. Se ha demostrado que para permitir crear todas las estructuras posibles se necesitan al menos cuatro bases para el ARN correspondiente , [72] aunque también es posible un número mayor, pero esto iría en contra del principio natural del mínimo esfuerzo .
Los grupos fosfato del ADN le confieren propiedades ácidas similares al ácido fosfórico y puede considerarse un ácido fuerte . Estará completamente ionizado a un pH celular normal, liberando protones que dejan cargas negativas en los grupos fosfato. Estas cargas negativas protegen al ADN de la degradación por hidrólisis al repeler a los nucleófilos que podrían hidrolizarlo. [73]
.jpg/440px-Estrazione_DNA_(cropped).jpg)
El ADN puro extraído de las células forma grumos blancos y fibrosos. [74]
La expresión de los genes está influenciada por cómo el ADN está empaquetado en los cromosomas, en una estructura llamada cromatina . Las modificaciones de bases pueden estar involucradas en el empaquetamiento, y las regiones que tienen una expresión genética baja o nula generalmente contienen altos niveles de metilación de bases de citosina . El empaquetamiento del ADN y su influencia en la expresión génica también pueden ocurrir por modificaciones covalentes del núcleo de la proteína histona alrededor del cual se envuelve el ADN en la estructura de la cromatina o bien por remodelación llevada a cabo por complejos de remodelación de la cromatina (ver Remodelación de la cromatina ). Además, existe una interferencia entre la metilación del ADN y la modificación de histonas, por lo que pueden afectar de manera coordinada a la cromatina y la expresión genética. [75]
Por ejemplo, la metilación de la citosina produce 5-metilcitosina , que es importante para la inactivación X de los cromosomas. [76] El nivel promedio de metilación varía entre organismos: el gusano Caenorhabditis elegans carece de metilación de citosina, mientras que los vertebrados tienen niveles más altos, y hasta el 1% de su ADN contiene 5-metilcitosina. [77] A pesar de la importancia de la 5-metilcitosina, puede desaminarse para dejar una base de timina, por lo que las citosinas metiladas son particularmente propensas a mutaciones . [78] Otras modificaciones de bases incluyen la metilación de adenina en bacterias, la presencia de 5-hidroximetilcitosina en el cerebro , [79] y la glicosilación de uracilo para producir la "base J" en cinetoplástidos . [80] [81]

El ADN puede resultar dañado por muchos tipos de mutágenos , que cambian la secuencia del ADN . Los mutágenos incluyen agentes oxidantes , agentes alquilantes y también radiación electromagnética de alta energía como la luz ultravioleta y los rayos X. El tipo de daño en el ADN producido depende del tipo de mutágeno. Por ejemplo, la luz ultravioleta puede dañar el ADN al producir dímeros de timina , que son enlaces cruzados entre bases de pirimidina. [83] Por otro lado, los oxidantes como los radicales libres o el peróxido de hidrógeno producen múltiples formas de daño, incluidas modificaciones de bases, particularmente de guanosina, y roturas de doble cadena. [84] Una célula humana típica contiene alrededor de 150.000 bases que han sufrido daño oxidativo. [85] De estas lesiones oxidativas, las más peligrosas son las roturas de doble cadena, ya que son difíciles de reparar y pueden producir mutaciones puntuales , inserciones , deleciones de la secuencia de ADN y translocaciones cromosómicas . [86] Estas mutaciones pueden causar cáncer . Debido a los límites inherentes a los mecanismos de reparación del ADN, si los humanos vivieran lo suficiente, eventualmente todos desarrollarían cáncer. [87] [88] Los daños al ADN que ocurren naturalmente , debido a procesos celulares normales que producen especies reactivas de oxígeno, las actividades hidrolíticas del agua celular, etc., también ocurren con frecuencia. Aunque la mayoría de estos daños se reparan, en cualquier célula puede quedar algo de daño en el ADN a pesar de la acción de los procesos de reparación. Estos daños restantes en el ADN se acumulan con la edad en los tejidos posmitóticos de los mamíferos. Esta acumulación parece ser una causa subyacente importante del envejecimiento. [89] [90] [91]
Muchos mutágenos encajan en el espacio entre dos pares de bases adyacentes, esto se llama intercalación . La mayoría de los intercaladores son moléculas aromáticas y planas; los ejemplos incluyen bromuro de etidio , acridinas , daunomicina y doxorrubicina . Para que un intercalador encaje entre pares de bases, las bases deben separarse, distorsionando las hebras de ADN al desenrollarse la doble hélice. Esto inhibe tanto la transcripción como la replicación del ADN, provocando toxicidad y mutaciones. [92] Como resultado, los intercaladores de ADN pueden ser carcinógenos y, en el caso de la talidomida, un teratógeno . [93] Otros, como el epóxido de benzo[ a ]pirenodiol y las aflatoxinas , forman aductos de ADN que inducen errores en la replicación. [94] Sin embargo, debido a su capacidad para inhibir la transcripción y replicación del ADN, otras toxinas similares también se utilizan en quimioterapia para inhibir las células cancerosas de rápido crecimiento . [95]
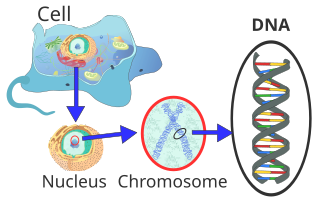
El ADN suele presentarse como cromosomas lineales en los eucariotas y cromosomas circulares en los procariotas . El conjunto de cromosomas de una célula conforma su genoma ; El genoma humano tiene aproximadamente 3 mil millones de pares de bases de ADN dispuestos en 46 cromosomas. [96] La información transportada por el ADN se mantiene en la secuencia de piezas de ADN llamadas genes . La transmisión de información genética en los genes se logra mediante el emparejamiento de bases complementarias. Por ejemplo, en la transcripción, cuando una célula utiliza la información de un gen, la secuencia de ADN se copia en una secuencia de ARN complementaria mediante la atracción entre el ADN y los nucleótidos correctos del ARN. Por lo general, esta copia de ARN se utiliza luego para crear una secuencia de proteína coincidente en un proceso llamado traducción , que depende de la misma interacción entre los nucleótidos de ARN. De forma alternativa, una célula puede copiar su información genética en un proceso llamado replicación del ADN . Los detalles de estas funciones se tratan en otros artículos; aquí la atención se centra en las interacciones entre el ADN y otras moléculas que median la función del genoma.
El ADN genómico se empaqueta de forma apretada y ordenada en el proceso llamado condensación del ADN , para adaptarse a los pequeños volúmenes disponibles de la célula. En los eucariotas, el ADN se localiza en el núcleo celular , con pequeñas cantidades en las mitocondrias y los cloroplastos . En los procariotas, el ADN se encuentra dentro de un cuerpo de forma irregular en el citoplasma llamado nucleoide . [97] La información genética de un genoma se mantiene dentro de los genes, y el conjunto completo de esta información en un organismo se denomina genotipo . Un gen es una unidad de herencia y es una región del ADN que influye en una característica particular de un organismo. Los genes contienen un marco de lectura abierto que se puede transcribir y secuencias reguladoras , como promotores y potenciadores , que controlan la transcripción del marco de lectura abierto.
En muchas especies , sólo una pequeña fracción de la secuencia total del genoma codifica proteínas. Por ejemplo, sólo alrededor del 1,5% del genoma humano consta de exones que codifican proteínas, y más del 50% del ADN humano consta de secuencias repetitivas no codificantes . [98] Las razones de la presencia de tanto ADN no codificante en los genomas eucariotas y las extraordinarias diferencias en el tamaño del genoma , o valor C , entre especies, representan un antiguo enigma conocido como el " enigma del valor C ". [99] Sin embargo, algunas secuencias de ADN que no codifican proteínas aún pueden codificar moléculas de ARN funcionales no codificantes , que participan en la regulación de la expresión genética . [100]

Algunas secuencias de ADN no codificantes desempeñan funciones estructurales en los cromosomas. Los telómeros y centrómeros suelen contener pocos genes, pero son importantes para la función y la estabilidad de los cromosomas. [64] [102] Una forma abundante de ADN no codificante en humanos son los pseudogenes , que son copias de genes que han sido desactivados por mutación. [103] Estas secuencias suelen ser solo fósiles moleculares , aunque ocasionalmente pueden servir como material genético en bruto para la creación de nuevos genes a través del proceso de duplicación y divergencia de genes . [104]
Un gen es una secuencia de ADN que contiene información genética y puede influir en el fenotipo de un organismo. Dentro de un gen, la secuencia de bases a lo largo de una cadena de ADN define una secuencia de ARN mensajero , que luego define una o más secuencias de proteínas. La relación entre las secuencias de nucleótidos de los genes y las secuencias de aminoácidos de las proteínas está determinada por las reglas de traducción , conocidas colectivamente como código genético . El código genético consta de "palabras" de tres letras llamadas codones formadas a partir de una secuencia de tres nucleótidos (por ejemplo, ACT, CAG, TTT).
En la transcripción, los codones de un gen se copian en ARN mensajero mediante la ARN polimerasa . Luego, esta copia de ARN es decodificada por un ribosoma que lee la secuencia de ARN emparejando bases del ARN mensajero para transferir el ARN , que transporta aminoácidos. Como hay 4 bases en combinaciones de 3 letras, hay 64 codones posibles (4 3 combinaciones). Estos codifican los veinte aminoácidos estándar , dando a la mayoría de los aminoácidos más de un codón posible. También hay tres codones de "parada" o "sin sentido" que indican el final de la región codificante; estos son los codones TAG, TAA y TGA (UAG, UAA y UGA en el ARNm).
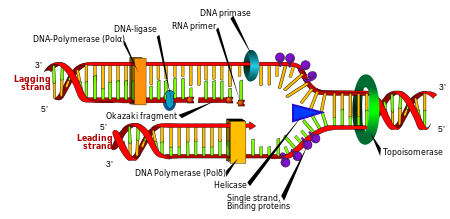
La división celular es esencial para que un organismo crezca, pero, cuando una célula se divide, debe replicar el ADN de su genoma para que las dos células hijas tengan la misma información genética que su madre. La estructura bicatenaria del ADN proporciona un mecanismo simple para la replicación del ADN . Aquí, las dos cadenas se separan y luego una enzima llamada ADN polimerasa recrea la secuencia de ADN complementaria de cada cadena . Esta enzima produce la hebra complementaria encontrando la base correcta mediante el emparejamiento de bases complementarias y uniéndola a la hebra original. Como las ADN polimerasas solo pueden extender una hebra de ADN en dirección 5' a 3', se utilizan diferentes mecanismos para copiar las hebras antiparalelas de la doble hélice. [105] De esta manera, la base de la cadena antigua dicta qué base aparece en la nueva cadena, y la célula termina con una copia perfecta de su ADN.
El ADN extracelular desnudo (eDNA), la mayor parte liberado por la muerte celular, es casi omnipresente en el medio ambiente. Su concentración en el suelo puede llegar a 2 μg/L, y su concentración en ambientes acuáticos naturales puede llegar a 88 μg/L. [106] Se han propuesto varias funciones posibles para el ADNe: puede estar implicado en la transferencia horizontal de genes ; [107] puede proporcionar nutrientes; [108] y puede actuar como un tampón para reclutar o valorar iones o antibióticos. [109] El ADN extracelular actúa como un componente funcional de la matriz extracelular en las biopelículas de varias especies bacterianas. Puede actuar como un factor de reconocimiento para regular la unión y dispersión de tipos de células específicas en la biopelícula; [110] puede contribuir a la formación de biopelículas; [111] y puede contribuir a la fuerza física de la biopelícula y a la resistencia al estrés biológico. [112]
El ADN fetal libre de células se encuentra en la sangre de la madre y puede secuenciarse para determinar una gran cantidad de información sobre el feto en desarrollo. [113]
Bajo el nombre de ADN ambiental, el ADNe ha experimentado un uso cada vez mayor en las ciencias naturales como herramienta de estudio de la ecología , monitoreando los movimientos y la presencia de especies en el agua, el aire o la tierra, y evaluando la biodiversidad de un área. [114] [115]
Las trampas extracelulares de neutrófilos (NET) son redes de fibras extracelulares, compuestas principalmente de ADN, que permiten a los neutrófilos , un tipo de glóbulo blanco, matar patógenos extracelulares y minimizar el daño a las células huésped.
Todas las funciones del ADN dependen de las interacciones con las proteínas. Estas interacciones entre proteínas pueden ser no específicas o la proteína puede unirse específicamente a una única secuencia de ADN. Las enzimas también pueden unirse al ADN y, de ellas, las polimerasas que copian la secuencia de bases del ADN en la transcripción y replicación del ADN son particularmente importantes.

Las proteínas estructurales que se unen al ADN son ejemplos bien comprendidos de interacciones no específicas entre ADN y proteínas. Dentro de los cromosomas, el ADN forma complejos con proteínas estructurales. Estas proteínas organizan el ADN en una estructura compacta llamada cromatina . En los eucariotas, esta estructura implica la unión del ADN a un complejo de pequeñas proteínas básicas llamadas histonas , mientras que en los procariotas están implicados múltiples tipos de proteínas. [116] [117] Las histonas forman un complejo en forma de disco llamado nucleosoma , que contiene dos vueltas completas de ADN bicatenario envuelto alrededor de su superficie. Estas interacciones no específicas se forman a través de residuos básicos en las histonas, formando enlaces iónicos con la columna vertebral de azúcar-fosfato ácido del ADN y, por lo tanto, son en gran medida independientes de la secuencia de bases. [118] Las modificaciones químicas de estos residuos de aminoácidos básicos incluyen metilación , fosforilación y acetilación . [119] Estos cambios químicos alteran la fuerza de la interacción entre el ADN y las histonas, haciendo que el ADN sea más o menos accesible a los factores de transcripción y cambiando la tasa de transcripción. [120] Otras proteínas de unión al ADN no específicas en la cromatina incluyen las proteínas del grupo de alta movilidad, que se unen al ADN doblado o distorsionado. [121] Estas proteínas son importantes para doblar conjuntos de nucleosomas y organizarlos en las estructuras más grandes que forman los cromosomas. [122]
Un grupo distinto de proteínas de unión al ADN son las proteínas de unión al ADN que se unen específicamente al ADN monocatenario. En los seres humanos, la proteína A de replicación es el miembro mejor comprendido de esta familia y se utiliza en procesos en los que se separa la doble hélice, incluida la replicación, recombinación y reparación del ADN. [123] Estas proteínas de unión parecen estabilizar el ADN monocatenario y protegerlo de la formación de bucles de tallo o de ser degradado por nucleasas .

Por el contrario, otras proteínas han evolucionado para unirse a secuencias particulares de ADN. Los más estudiados son los diversos factores de transcripción , que son proteínas que regulan la transcripción. Cada factor de transcripción se une a un conjunto particular de secuencias de ADN y activa o inhibe la transcripción de genes que tienen estas secuencias cerca de sus promotores. Los factores de transcripción hacen esto de dos maneras. En primer lugar, pueden unirse a la ARN polimerasa responsable de la transcripción, ya sea directamente o mediante otras proteínas mediadoras; esto localiza la polimerasa en el promotor y le permite comenzar la transcripción. [125] Alternativamente, los factores de transcripción pueden unirse a enzimas que modifican las histonas en el promotor. Esto cambia la accesibilidad de la plantilla de ADN a la polimerasa. [126]
Como estos objetivos de ADN pueden aparecer en todo el genoma de un organismo, los cambios en la actividad de un tipo de factor de transcripción pueden afectar a miles de genes. [127] En consecuencia, estas proteínas son a menudo el objetivo de los procesos de transducción de señales que controlan las respuestas a los cambios ambientales o la diferenciación y el desarrollo celular . La especificidad de las interacciones de estos factores de transcripción con el ADN proviene de que las proteínas hacen múltiples contactos con los bordes de las bases del ADN, lo que les permite "leer" la secuencia del ADN. La mayoría de estas interacciones con bases se realizan en el surco principal, donde las bases son más accesibles. [25]
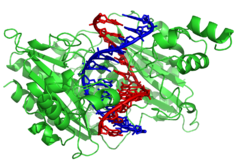
Las nucleasas son enzimas que cortan las cadenas de ADN catalizando la hidrólisis de los enlaces fosfodiéster . Las nucleasas que hidrolizan nucleótidos de los extremos de las cadenas de ADN se denominan exonucleasas , mientras que las endonucleasas cortan dentro de las cadenas. Las nucleasas más utilizadas en biología molecular son las endonucleasas de restricción , que cortan el ADN en secuencias específicas. Por ejemplo, la enzima EcoRV que se muestra a la izquierda reconoce la secuencia de 6 bases 5′-GATATC-3′ y realiza un corte en la línea horizontal. En la naturaleza, estas enzimas protegen a las bacterias contra la infección por fagos al digerir el ADN del fago cuando ingresa a la célula bacteriana, actuando como parte del sistema de modificación de restricción . [129] En tecnología, estas nucleasas de secuencia específica se utilizan en clonación molecular y toma de huellas dactilares de ADN .
Las enzimas llamadas ADN ligasas pueden volver a unir cadenas de ADN cortadas o rotas. [130] Las ligasas son particularmente importantes en la replicación de la cadena retrasada del ADN, ya que unen los segmentos cortos de ADN producidos en la horquilla de replicación en una copia completa de la plantilla de ADN. También se utilizan en la reparación del ADN y la recombinación genética . [130]
Las topoisomerasas son enzimas con actividad tanto nucleasa como ligasa. Estas proteínas cambian la cantidad de superenrollamiento en el ADN. Algunas de estas enzimas funcionan cortando la hélice del ADN y permitiendo que una sección gire, reduciendo así su nivel de superenrollamiento; Luego, la enzima sella la rotura del ADN. [44] Otros tipos de estas enzimas son capaces de cortar una hélice de ADN y luego pasar una segunda hebra de ADN a través de esta ruptura, antes de volver a unirse a la hélice. [131] Las topoisomerasas son necesarias para muchos procesos que involucran al ADN, como la replicación y transcripción del ADN. [45]
Las helicasas son proteínas que son un tipo de motor molecular . Utilizan la energía química de los nucleósidos trifosfato , predominantemente trifosfato de adenosina (ATP), para romper los enlaces de hidrógeno entre las bases y desenrollar la doble hélice del ADN en hebras simples. [132] Estas enzimas son esenciales para la mayoría de los procesos en los que las enzimas necesitan acceder a las bases del ADN.
Las polimerasas son enzimas que sintetizan cadenas de polinucleótidos a partir de nucleósidos trifosfato . La secuencia de sus productos se crea a partir de cadenas de polinucleótidos existentes, que se denominan plantillas . Estas enzimas funcionan añadiendo repetidamente un nucleótido al grupo hidroxilo 3' al final de la cadena polinucleotídica en crecimiento. Como consecuencia, todas las polimerasas funcionan en dirección 5′ a 3′. [133] En el sitio activo de estas enzimas, los pares de bases de nucleósido trifosfato entrantes con la plantilla: esto permite a las polimerasas sintetizar con precisión la hebra complementaria de su plantilla. Las polimerasas se clasifican según el tipo de plantilla que utilizan.
En la replicación del ADN, las ADN polimerasas dependientes de ADN hacen copias de cadenas de polinucleótidos de ADN. Para preservar la información biológica, es esencial que la secuencia de bases en cada copia sea precisamente complementaria a la secuencia de bases en la cadena plantilla. Muchas ADN polimerasas tienen actividad correctora . Aquí, la polimerasa reconoce los errores ocasionales en la reacción de síntesis por la falta de emparejamiento de bases entre los nucleótidos no coincidentes. Si se detecta una falta de coincidencia, se activa una actividad exonucleasa de 3' a 5' y se elimina la base incorrecta. [134] En la mayoría de los organismos, las ADN polimerasas funcionan en un gran complejo llamado replisoma que contiene múltiples subunidades accesorias, como la abrazadera del ADN o las helicasas . [135]
Las ADN polimerasas dependientes de ARN son una clase especializada de polimerasas que copian la secuencia de una cadena de ARN en ADN. Incluyen la transcriptasa inversa , que es una enzima viral implicada en la infección de células por retrovirus , y la telomerasa , que es necesaria para la replicación de los telómeros. [63] [136] Por ejemplo, la transcriptasa inversa del VIH es una enzima para la replicación del virus del SIDA. [136] La telomerasa es una polimerasa inusual porque contiene su propio molde de ARN como parte de su estructura. Sintetiza telómeros en los extremos de los cromosomas. Los telómeros previenen la fusión de los extremos de los cromosomas vecinos y protegen los extremos de los cromosomas del daño. [64]
La transcripción se lleva a cabo mediante una ARN polimerasa dependiente de ADN que copia la secuencia de una cadena de ADN en ARN. Para comenzar a transcribir un gen, la ARN polimerasa se une a una secuencia de ADN llamada promotor y separa las cadenas de ADN. Luego copia la secuencia del gen en una transcripción de ARN mensajero hasta que llega a una región del ADN llamada terminador , donde se detiene y se desprende del ADN. Al igual que con las ADN polimerasas dependientes de ADN humano, la ARN polimerasa II , la enzima que transcribe la mayoría de los genes del genoma humano, opera como parte de un gran complejo proteico con múltiples subunidades reguladoras y accesorias. [137]

Una hélice de ADN no suele interactuar con otros segmentos de ADN, y en las células humanas, los diferentes cromosomas incluso ocupan áreas separadas en el núcleo llamadas " territorios cromosómicos ". [139] Esta separación física de diferentes cromosomas es importante para la capacidad del ADN de funcionar como un depósito estable de información, ya que una de las pocas veces que los cromosomas interactúan es en el cruce cromosómico que ocurre durante la reproducción sexual , cuando ocurre la recombinación genética . El cruce cromosómico ocurre cuando dos hélices de ADN se rompen, intercambian una sección y luego se vuelven a unir.
La recombinación permite a los cromosomas intercambiar información genética y produce nuevas combinaciones de genes, lo que aumenta la eficiencia de la selección natural y puede ser importante en la rápida evolución de nuevas proteínas. [140] La recombinación genética también puede estar implicada en la reparación del ADN, particularmente en la respuesta de la célula a las roturas de doble hebra. [141]
La forma más común de cruce cromosómico es la recombinación homóloga , donde los dos cromosomas involucrados comparten secuencias muy similares. La recombinación no homóloga puede ser perjudicial para las células, ya que puede producir translocaciones cromosómicas y anomalías genéticas. La reacción de recombinación está catalizada por enzimas conocidas como recombinasas , como RAD51 . [142] El primer paso en la recombinación es una rotura de doble cadena causada por una endonucleasa o por un daño al ADN. [143] Una serie de pasos catalizados en parte por la recombinasa conducen a la unión de las dos hélices mediante al menos una unión de Holliday , en la que un segmento de una sola hebra en cada hélice se hibrida con la hebra complementaria en la otra hélice. La unión Holliday es una estructura de unión tetraédrica que se puede mover a lo largo del par de cromosomas, intercambiando una hebra por otra. Luego, la reacción de recombinación se detiene mediante la escisión de la unión y la nueva ligadura del ADN liberado. [144] Sólo las hebras de polaridad similar intercambian ADN durante la recombinación. Hay dos tipos de división: división este-oeste y división norte-sur. La escisión norte-sur corta ambas hebras de ADN, mientras que la escisión este-oeste tiene una hebra de ADN intacta. La formación de una unión Holliday durante la recombinación hace posible la diversidad genética, el intercambio de genes en los cromosomas y la expresión de genomas virales de tipo salvaje.
El ADN contiene la información genética que permite que todas las formas de vida funcionen, crezcan y se reproduzcan. Sin embargo, no está claro durante cuánto tiempo en los 4 mil millones de años de historia de la vida el ADN ha desempeñado esta función, ya que se ha propuesto que las primeras formas de vida pueden haber utilizado el ARN como material genético. [145] [146] El ARN puede haber actuado como la parte central del metabolismo celular temprano , ya que puede transmitir información genética y llevar a cabo catálisis como parte de las ribozimas . [147] Este antiguo mundo de ARN donde el ácido nucleico se habría utilizado tanto para la catálisis como para la genética puede haber influido en la evolución del código genético actual basado en cuatro bases de nucleótidos. Esto ocurriría, ya que el número de bases diferentes en dicho organismo es una compensación entre un pequeño número de bases que aumenta la precisión de la replicación y un gran número de bases que aumentan la eficiencia catalítica de las ribozimas. [148] Sin embargo, no hay evidencia directa de sistemas genéticos antiguos, ya que la recuperación del ADN de la mayoría de los fósiles es imposible porque el ADN sobrevive en el medio ambiente durante menos de un millón de años y se degrada lentamente en fragmentos cortos en solución. [149] Se han hecho afirmaciones sobre ADN más antiguo, en particular un informe sobre el aislamiento de una bacteria viable a partir de un cristal de sal de 250 millones de años, [150] pero estas afirmaciones son controvertidas. [151] [152]
Los componentes básicos del ADN ( adenina , guanina y moléculas orgánicas relacionadas ) pueden haberse formado extraterrestre en el espacio exterior . [153] [154] [155] Los compuestos orgánicos complejos de ADN y ARN de la vida , incluidos uracilo , citosina y timina , también se han formado en el laboratorio en condiciones que imitan las que se encuentran en el espacio exterior , utilizando sustancias químicas de partida, como la pirimidina . encontrado en meteoritos . La pirimidina, al igual que los hidrocarburos aromáticos policíclicos (HAP), la sustancia química más rica en carbono que se encuentra en el universo , puede haberse formado en gigantes rojas o en nubes de gas y polvo cósmico interestelar . [156]
En febrero de 2021, los científicos informaron, por primera vez, de la secuenciación de ADN de restos de animales , un mamut en este caso de más de un millón de años, el ADN más antiguo secuenciado hasta la fecha. [157] [158]
Se han desarrollado métodos para purificar el ADN de organismos, como la extracción con fenol-cloroformo , y para manipularlo en el laboratorio, como los digestos de restricción y la reacción en cadena de la polimerasa . La biología y la bioquímica modernas hacen un uso intensivo de estas técnicas en la tecnología del ADN recombinante. El ADN recombinante es una secuencia de ADN creada por el hombre que se ha ensamblado a partir de otras secuencias de ADN. Pueden transformarse en organismos en forma de plásmidos o en el formato adecuado, utilizando un vector viral . [159] Los organismos genéticamente modificados producidos pueden usarse para producir productos como proteínas recombinantes , utilizadas en investigación médica , [160] o cultivarse en la agricultura . [161] [162]
Los científicos forenses pueden utilizar el ADN de la sangre , el semen , la piel , la saliva o el cabello encontrados en la escena de un crimen para identificar un ADN coincidente de un individuo, como el perpetrador. [163] Este proceso se denomina formalmente perfilado de ADN , también llamado toma de huellas dactilares de ADN . En la elaboración de perfiles de ADN, se comparan entre personas las longitudes de secciones variables de ADN repetitivo, como repeticiones cortas en tándem y minisatélites . Este método suele ser una técnica extremadamente fiable para identificar un ADN coincidente. [164] Sin embargo, la identificación puede ser complicada si la escena está contaminada con ADN de varias personas. [165] El perfil de ADN fue desarrollado en 1984 por el genetista británico Sir Alec Jeffreys , [166] y se utilizó por primera vez en ciencia forense para condenar a Colin Pitchfork en el caso de los asesinatos de Enderby en 1988 . [167]
El desarrollo de la ciencia forense y la capacidad actual de obtener coincidencias genéticas en muestras diminutas de sangre, piel, saliva o cabello ha llevado a reexaminar muchos casos. Ahora se pueden descubrir pruebas que eran científicamente imposibles en el momento del examen original. Combinado con la eliminación de la ley de doble incriminación en algunos lugares, esto puede permitir que se reabran casos en los que juicios anteriores no han logrado producir pruebas suficientes para convencer al jurado. Es posible que se solicite a las personas acusadas de delitos graves que proporcionen una muestra de ADN para fines de comparación. La defensa más obvia contra las coincidencias de ADN obtenidas mediante métodos forenses es afirmar que se ha producido una contaminación cruzada de las pruebas. Esto ha resultado en procedimientos de manejo meticulosos y estrictos con nuevos casos de delitos graves.
Los perfiles de ADN también se utilizan con éxito para identificar positivamente a las víctimas de incidentes con víctimas masivas, [168] cuerpos o partes del cuerpo en accidentes graves y víctimas individuales en fosas comunes de guerra, mediante la comparación con miembros de la familia.
El perfil de ADN también se utiliza en las pruebas de paternidad de ADN para determinar si alguien es el padre biológico o el abuelo de un niño; la probabilidad de paternidad suele ser del 99,99 % cuando el presunto padre está relacionado biológicamente con el niño. Los métodos normales de secuenciación de ADN se aplican después del nacimiento, pero existen nuevos métodos para probar la paternidad mientras la madre aún está embarazada. [169]
Las desoxirribozimas , también llamadas ADNzimas o ADN catalítico, se descubrieron por primera vez en 1994. [170] Son en su mayoría secuencias de ADN monocatenarias aisladas de un gran conjunto de secuencias de ADN aleatorias mediante un enfoque combinatorio llamado selección in vitro o evolución sistemática de ligandos mediante enriquecimiento exponencial. (SELEX). Las ADNzimas catalizan una variedad de reacciones químicas, incluida la escisión de ARN-ADN, la ligadura de ARN-ADN, la fosforilación-desfosforilación de aminoácidos, la formación de enlaces carbono-carbono, etc. Las ADNzimas pueden mejorar la velocidad catalítica de reacciones químicas hasta 100.000.000.000 veces más que la reacción no catalizada. [171] La clase de ADNzimas más estudiada son los tipos de escisión de ARN que se han utilizado para detectar diferentes iones metálicos y diseñar agentes terapéuticos. Se han informado varias ADNzimas específicas de metales, incluida la ADNzima GR-5 (específica de plomo), [170] las ADNzimas CA1-3 (específicas de cobre), [172] la ADNzima 39E (específica de uranilo) y la ADNzima NaA43 ( específico de sodio). [173] La ADNzima NaA43, que según se informa es más de 10.000 veces más selectiva para el sodio que para otros iones metálicos, se utilizó para fabricar un sensor de sodio en tiempo real en las células.
La bioinformática implica el desarrollo de técnicas para almacenar, extraer datos , buscar y manipular datos biológicos, incluidos los datos de secuencias de ácidos nucleicos del ADN . Estos han conducido a avances ampliamente aplicados en ciencias de la computación , especialmente algoritmos de búsqueda de cadenas , aprendizaje automático y teoría de bases de datos . [174] Se desarrollaron algoritmos de búsqueda o coincidencia de cadenas, que encuentran la aparición de una secuencia de letras dentro de una secuencia más grande de letras, para buscar secuencias específicas de nucleótidos. [175] La secuencia de ADN se puede alinear con otras secuencias de ADN para identificar secuencias homólogas y localizar las mutaciones específicas que las hacen distintas. Estas técnicas, especialmente el alineamiento de secuencias múltiples , se utilizan para estudiar las relaciones filogenéticas y la función de las proteínas. [176] Los conjuntos de datos que representan secuencias de ADN de genomas completos, como los producidos por el Proyecto Genoma Humano , son difíciles de utilizar sin las anotaciones que identifican las ubicaciones de los genes y los elementos reguladores en cada cromosoma. Las regiones de la secuencia de ADN que tienen los patrones característicos asociados con genes que codifican proteínas o ARN pueden identificarse mediante algoritmos de búsqueda de genes , que permiten a los investigadores predecir la presencia de productos genéticos particulares y sus posibles funciones en un organismo incluso antes de que hayan sido aislados. experimentalmente. [177] También se pueden comparar genomas completos, lo que puede arrojar luz sobre la historia evolutiva de un organismo en particular y permitir el examen de eventos evolutivos complejos.

La nanotecnología del ADN utiliza las propiedades únicas de reconocimiento molecular del ADN y otros ácidos nucleicos para crear complejos de ADN ramificados y autoensamblados con propiedades útiles. [179] El ADN se utiliza así como material estructural más que como portador de información biológica. Esto ha llevado a la creación de redes periódicas bidimensionales (tanto basadas en mosaicos como utilizando el método del origami de ADN ) y estructuras tridimensionales en forma de poliedros . [180] También se han demostrado dispositivos nanomecánicos y autoensamblaje algorítmico , [181] y estas estructuras de ADN se han utilizado para modelar la disposición de otras moléculas, como nanopartículas de oro y proteínas de estreptavidina . [182] El ADN y otros ácidos nucleicos son la base de los aptámeros , ligandos de oligonucleótidos sintéticos para moléculas diana específicas utilizadas en una variedad de aplicaciones biotecnológicas y biomédicas. [183]
Debido a que el ADN recopila mutaciones a lo largo del tiempo, que luego se heredan, contiene información histórica y, al comparar secuencias de ADN, los genetistas pueden inferir la historia evolutiva de los organismos, su filogenia . [184] Este campo de la filogenética es una herramienta poderosa en la biología evolutiva . Si se comparan secuencias de ADN dentro de una especie, los genetistas de poblaciones pueden conocer la historia de poblaciones particulares. Esto se puede utilizar en estudios que van desde la genética ecológica hasta la antropología .
El ADN como dispositivo de almacenamiento de información tiene un enorme potencial ya que tiene una densidad de almacenamiento mucho mayor en comparación con los dispositivos electrónicos. Sin embargo, los altos costos, los tiempos lentos de lectura y escritura ( latencia de memoria ) y la confiabilidad insuficiente han impedido su uso práctico. [185] [186]


El ADN fue aislado por primera vez por el médico suizo Friedrich Miescher quien, en 1869, descubrió una sustancia microscópica en el pus de vendajes quirúrgicos desechados. Como residía en el núcleo de las células, la llamó "nucleína". [187] [188] En 1878, Albrecht Kossel aisló el componente no proteico de la "nucleína", el ácido nucleico, y más tarde aisló sus cinco nucleobases primarias . [189] [190]
En 1909, Phoebus Levene identificó la unidad de nucleótidos de base, azúcar y fosfato del ARN (entonces denominado "ácido nucleico de levadura"). [191] [192] [193] En 1929, Levene identificó el azúcar desoxirribosa en el "ácido nucleico del timo" (ADN). [194] Levene sugirió que el ADN consistía en una cadena de cuatro unidades de nucleótidos unidas entre sí a través de grupos fosfato ("hipótesis del tetranucleótido"). Levene pensó que la cadena era corta y que las bases se repetían en un orden fijo. En 1927, Nikolai Koltsov propuso que los rasgos heredados se heredarían a través de una "molécula hereditaria gigante" formada por "dos hebras espejo que se replicarían de forma semiconservadora utilizando cada hebra como plantilla". [195] [196] En 1928, Frederick Griffith en su experimento descubrió que los rasgos de la forma "lisa" del neumococo podían transferirse a la forma "áspera" de la misma bacteria mezclando bacterias "lisas" muertas con bacterias vivas "ásperas". " forma. [197] [198] Este sistema proporcionó la primera sugerencia clara de que el ADN transporta información genética.
En 1933, mientras estudiaba los huevos vírgenes de erizo de mar , Jean Brachet sugirió que el ADN se encuentra en el núcleo celular y que el ARN está presente exclusivamente en el citoplasma . En aquel momento, se pensaba que el "ácido nucleico de levadura" (ARN) sólo se encontraba en las plantas, mientras que el "ácido nucleico del timo" (ADN) sólo se encontraba en los animales. Se pensaba que este último era un tetrámero, con la función de amortiguar el pH celular. [199] [200]
En 1937, William Astbury produjo los primeros patrones de difracción de rayos X que demostraron que el ADN tenía una estructura regular. [201]
En 1943, Oswald Avery , junto con sus compañeros de trabajo Colin MacLeod y Maclyn McCarty , identificaron el ADN como el principio transformador , apoyando la sugerencia de Griffith ( experimento Avery-MacLeod-McCarty ). [202] Erwin Chargaff desarrolló y publicó observaciones ahora conocidas como reglas de Chargaff , afirmando que en el ADN de cualquier especie de cualquier organismo, la cantidad de guanina debe ser igual a la citosina y la cantidad de adenina debe ser igual a la timina . [203] [204]

A finales de 1951, Francis Crick comenzó a trabajar con James Watson en el Laboratorio Cavendish de la Universidad de Cambridge , Reino Unido . El papel del ADN en la herencia se confirmó en 1952 cuando Alfred Hershey y Martha Chase en el experimento Hershey-Chase demostraron que el ADN es el material genético del fago T2 de las enterobacterias . [205]
En mayo de 1952, Raymond Gosling , un estudiante de posgrado que trabajaba bajo la supervisión de Rosalind Franklin , tomó una imagen de difracción de rayos X , etiquetada como " Foto 51 ", [206] en altos niveles de hidratación del ADN. Maurice Wilkins les dio esta fotografía a Watson y Crick y fue fundamental para que obtuvieran la estructura correcta del ADN. Franklin les dijo a Crick y Watson que la columna vertebral tenía que estar en el exterior. Antes de eso, Linus Pauling, Watson y Crick tenían modelos erróneos con las cadenas adentro y las bases apuntando hacia afuera. La identificación de Franklin del grupo espacial de los cristales de ADN le reveló a Crick que las dos hebras de ADN eran antiparalelas . [207] En febrero de 1953, Linus Pauling y Robert Corey propusieron un modelo para ácidos nucleicos que contienen tres cadenas entrelazadas, con los fosfatos cerca del eje y las bases en el exterior. [208] Watson y Crick completaron su modelo, que ahora se acepta como el primer modelo correcto de la doble hélice del ADN . El 28 de febrero de 1953, Crick interrumpió la hora del almuerzo de los clientes en el pub The Eagle en Cambridge, Reino Unido, para anunciar que él y Watson habían "descubierto el secreto de la vida". [209]
El número del 25 de abril de 1953 de la revista Nature publicó una serie de cinco artículos que explicaban la estructura de doble hélice del ADN de Watson y Crick y la evidencia que la respalda. [210] La estructura se informó en una carta titulada " ESTRUCTURA MOLECULAR DE LOS ÁCIDOS NUCLEICOS Una estructura para el ácido nucleico desoxirribosa " , en la que decían: "No se nos ha escapado que el emparejamiento específico que hemos postulado sugiere inmediatamente un posible mecanismo de copia para el material genético." [9] Esta carta fue seguida por una carta de Franklin y Gosling, que fue la primera publicación de sus propios datos de difracción de rayos X y de su método de análisis original. [48] [211] Luego siguió una carta de Wilkins y dos de sus colegas, que contenía un análisis de patrones de rayos X de ADN B in vivo y que apoyaba la presencia in vivo de la estructura de Watson y Crick. [49]
En abril de 2023, los científicos, basándose en nuevas pruebas, concluyeron que Rosalind Franklin contribuyó y fue un "actor igual" en el proceso de descubrimiento del ADN, y no otra cosa, como se pudo haber presentado posteriormente después del descubrimiento. [212] [213] [214]
En 1962, tras la muerte de Franklin, Watson, Crick y Wilkins recibieron conjuntamente el Premio Nobel de Fisiología o Medicina . [215] Los Premios Nobel se otorgan únicamente a quienes los reciben vivos. Continúa el debate sobre quién debería recibir el crédito por el descubrimiento. [216]
En una influyente presentación en 1957, Crick expuso el dogma central de la biología molecular , que predijo la relación entre el ADN, el ARN y las proteínas, y articuló la "hipótesis del adaptador". [217] Confirmación final del mecanismo de replicación implícito en la estructura de doble hélice seguida en 1958 mediante el experimento de Meselson-Stahl . [218] Trabajos posteriores de Crick y sus colaboradores demostraron que el código genético se basaba en tripletes de bases no superpuestas, llamados codones , lo que permitió a Har Gobind Khorana , Robert W. Holley y Marshall Warren Nirenberg descifrar el código genético. [219] Estos hallazgos representan el nacimiento de la biología molecular . [220]
En 1986, el análisis de ADN se utilizó por primera vez con fines de investigación criminal cuando la policía del Reino Unido pidió a Alec Jeffreys, de la Universidad de Leicester, que verificara o refutara la "confesión" de un sospechoso de violación y asesinato. En este caso particular, el sospechoso había confesado dos violaciones y asesinatos, pero luego se retractó de su confesión. Las pruebas de ADN en los laboratorios de la universidad pronto refutaron la veracidad de la "confesión" original del sospechoso, y el sospechoso fue exonerado de los cargos de asesinato y violación. [221]
Si viviéramos lo suficiente, tarde o temprano todos padeceríamos cáncer.
Es de esperar una cierta incidencia de fondo irreducible de cáncer, independientemente de las circunstancias: las mutaciones nunca pueden evitarse por completo, porque son una consecuencia ineludible de limitaciones fundamentales en la precisión de la replicación del ADN, como se analiza en Capítulo 5. Si un ser humano pudiera vivir lo suficiente, es inevitable que al menos una de sus células acabe acumulando un conjunto de mutaciones suficientes para que se desarrolle el cáncer.
[pag. 456]
Ich habe mich daher später mit meinen Versuchen an die ganzen Kerne gehalten, die Trennung der Körper, die ich einstweilen ohne weiteres Präjudiz als lösliches und unlösliches Nuclein bezeichnen will, einem günstigeren Material überlassend.
(Por lo tanto, en mis experimentos me limité posteriormente a todo el núcleo, dejando a un material más favorable la separación de las sustancias, que por el momento, sin más prejuicios, designaré como material nuclear soluble e insoluble ("Nucleína") )
{{cite book}}: CS1 maint: location missing publisher (link)En P. 264, Kossel observó proféticamente: Der Erforschung der quantum Verhältnisse der vier stickstoffreichen Basen, der Abhängigkeit ihrer Menge von den psychologischen Zuständen der Zelle, verspricht wichtige Aufschlüsse über die elementaren psychologisch-chemischen Vorgänge. (El estudio de las relaciones cuantitativas de las cuatro bases nitrogenadas —[y] de la dependencia de su cantidad de los estados fisiológicos de la célula—promete importantes conocimientos sobre los procesos fisiológico-químicos fundamentales.)
De la página 313: "Creo que el tamaño de los cromosomas en las glándulas salivales [de Drosophila] se determina mediante la multiplicación de
genonemas
. Con este término designo el hilo axial del cromosoma, en el que los genetistas ubican el combinación lineal de genes;... En el cromosoma normal generalmente hay un solo genonema; antes de la división celular, este genonema se ha dividido en dos cadenas".