Z-DNA es una de las muchas posibles estructuras de doble hélice del ADN . Es una estructura de doble hélice zurda en la que la hélice gira hacia la izquierda en forma de zigzag, en lugar de hacia la derecha, como la forma más común de ADN-B . Se cree que el ADN-Z es una de las tres estructuras de doble hélice biológicamente activas junto con el ADN-A y el ADN-B .
El ADN zurdo fue propuesto por primera vez por Robert Wells y sus colegas como la estructura de un polímero repetido de inosina - citosina . [1] Observaron un espectro de dicroísmo circular "inverso" para dichos ADN e interpretaron esto incorrectamente en el sentido de que las hebras se enroscaban entre sí en forma zurda. La relación entre el ADN-Z y el ADN-B más familiar fue indicada por el trabajo de Pohl y Jovin, [2] quienes demostraron que el dicroísmo circular ultravioleta del poli(dG-dC) estaba casi invertido en una solución de cloruro de sodio 4 M y que la estructura de poli d (I – C) · poli d (I – C) era de hecho una conformación de D-DNA diestra. La sospecha de que esto era el resultado de una conversión de ADN-B a ADN-Z se confirmó examinando los espectros Raman de estas soluciones y los cristales de ADN-Z. [3] Posteriormente, se publicó una estructura cristalina de "Z-DNA" que resultó ser la primera estructura monocristalina de rayos X de un fragmento de ADN (un hexámero de ADN autocomplementario d(CG) 3 ). Se resolvió como una doble hélice izquierda con dos cadenas antiparalelas que se mantenían unidas por pares de bases Watson-Crick (ver Cristalografía de rayos X ). Fue resuelto por Andrew HJ Wang , Alexander Rich y sus compañeros de trabajo en 1979 en el MIT . [4] La cristalización de una unión de ADN B a Z en 2005 [5] proporcionó una mejor comprensión del papel potencial que desempeña el ADN Z en las células. Siempre que se forma un segmento de ADN Z, debe haber uniones B-Z en sus dos extremos, interconectándolo con la forma B de ADN que se encuentra en el resto del genoma .
En 2007, la versión de ARN del Z-DNA, Z-RNA , se describió como una versión transformada de una doble hélice de A-RNA en una hélice izquierda. [6] Sin embargo, la transición del ARN-A al ARN-Z ya se describió en 1984. [7]
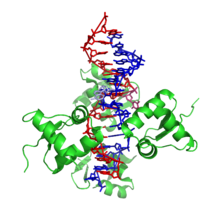
El ADN Z es bastante diferente de las formas diestras. De hecho, el ADN-Z se compara a menudo con el ADN-B para ilustrar las principales diferencias. La hélice del ADN Z es zurda y tiene una estructura que se repite cada dos pares de bases. Los surcos mayor y menor, a diferencia del ADN A y B, muestran poca diferencia en ancho. La formación de esta estructura es generalmente desfavorable, aunque ciertas condiciones pueden favorecerla; como alternancia de secuencia purina - pirimidina (especialmente poli(dGC) 2 ), superenrollamiento negativo de ADN o alto contenido de sal y algunos cationes (todos a temperatura fisiológica, 37 °C y pH 7,3–7,4). El ADN Z puede formar una unión con el ADN B (llamada "caja de conexiones B a Z") en una estructura que implica la extrusión de un par de bases. [8] La conformación del ADN-Z ha sido difícil de estudiar porque no existe como una característica estable de la doble hélice. Más bien, es una estructura transitoria que ocasionalmente es inducida por actividad biológica y luego desaparece rápidamente. [9]
Es posible predecir la probabilidad de que una secuencia de ADN forme una estructura de ADN Z. P. Shing Ho escribió un algoritmo para predecir la propensión del ADN a cambiar de la forma B a la forma Z, ZHunt , en 1984 en el MIT . [10] Este algoritmo fue desarrollado posteriormente por Tracy Camp , P. Christoph Champ, Sandor Maurice y Jeffrey M. Vargason para el mapeo de todo el genoma del ADN-Z (con Ho como investigador principal). [11]
Desde el descubrimiento y cristalización del ADN-Z en 1979, la configuración ha dejado a los científicos desconcertados sobre la vía y el mecanismo desde la configuración del ADN-B a la configuración del ADN-Z. [12] El cambio conformacional del ADN-B a la estructura del ADN-Z se desconocía a nivel atómico, pero en 2010, simulaciones por computadora realizadas por Lee et al. pudieron determinar computacionalmente que la propagación gradual de una transición de B a Z proporcionaría una barrera de energía más baja que el mecanismo concertado previamente hipotetizado. [13] Dado que esto se demostró computacionalmente, la vía aún necesitaría probarse experimentalmente en el laboratorio para una mayor confirmación y validez, en lo que Lee et al. afirma específicamente en su artículo de revista: "El resultado [computacional] actual podría probarse mediante experimentos FRET de molécula única (smFRET) en el futuro". [13] En 2018, la vía del ADN B al ADN Z se demostró experimentalmente mediante ensayos smFRET. [14] Esto se realizó midiendo los valores de intensidad entre los tintes fluorescentes donante y aceptor, también conocidos como fluoróforos , en relación entre sí a medida que intercambian electrones, mientras se etiquetan en una molécula de ADN. [15] [16] Las distancias entre los fluoróforos podrían usarse para calcular cuantitativamente los cambios en la proximidad de los tintes y los cambios conformacionales en el ADN. Se utilizó una proteína de unión de alta afinidad al ADN-Z , hZαADAR1, [17] en concentraciones variables para inducir la transformación de ADN-B a ADN-Z. [14] Los ensayos de smFRET revelaron un estado de transición B*, que se formó cuando la unión de hZαADAR1 se acumuló en la estructura del ADN B y la estabilizó. [14] Este paso se produce para evitar una alta energía de unión, en la que se permite que la estructura del ADN-B experimente un cambio conformacional en la estructura del ADN-Z sin un cambio importante y disruptivo en la energía. Este resultado coincide con los resultados computacionales de Lee et al. demostrando que el mecanismo es paso a paso y su propósito es proporcionar una barrera de energía más baja para el cambio conformacional de la configuración de ADN B a ADN Z. [13] Contrariamente a la idea anterior, las proteínas de unión en realidad no estabilizan la conformación del ADN-Z después de su formación, sino que en realidad promueven la formación del ADN-Z directamente a partir de la conformación B*, que está formada por La estructura del ADN B está unida por proteínas de alta afinidad. [14]
Se ha confirmado un papel biológico del ADN-Z en la regulación de las respuestas al interferón tipo I en estudios de tres enfermedades mendelianas raras bien caracterizadas: discromatosis simétrica hereditaria (OMIM: 127400), síndrome de Aicardi-Goutières (OMIM: 615010) y síndrome estriado bilateral. Necrosis/Distonía. Las familias con transcriptoma ADAR haploide permitieron mapear las variantes Zα directamente a la enfermedad, lo que demuestra que la información genética está codificada en el ADN tanto por su forma como por su secuencia. [18] El papel en la regulación de las respuestas del interferón tipo I en el cáncer también está respaldado por los hallazgos de que el 40% de un panel de tumores dependían de la enzima ADAR para sobrevivir. [19]
En estudios anteriores, el ADN Z se relacionó tanto con la enfermedad de Alzheimer como con el lupus eritematoso sistémico . Para demostrar esto, se llevó a cabo un estudio sobre el ADN encontrado en el hipocampo de cerebros normales, moderadamente afectados por la enfermedad de Alzheimer y gravemente afectados por la enfermedad de Alzheimer. Mediante el uso del dicroísmo circular , este estudio mostró la presencia de ADN-Z en el ADN de los afectados gravemente. [20] En este estudio también se encontró que la mayor parte del ADN moderadamente afectado estaba en la conformación intermedia BZ. Esto es significativo porque a partir de estos hallazgos se concluyó que la transición del ADN B al ADN Z depende de la progresión de la enfermedad de Alzheimer. [20] Además, el ADN-Z se asocia con el lupus eritematoso sistémico (LES) mediante la presencia de anticuerpos naturales. Se encontraron cantidades significativas de anticuerpos anti-Z-DNA en pacientes con LES y no estaban presentes en otras enfermedades reumáticas. [21] Hay dos tipos de estos anticuerpos. Mediante radioinmunoensayo, se descubrió que uno interactúa con las bases expuestas en la superficie del ADN-Z y el ADN desnaturalizado, mientras que el otro interactúa exclusivamente con la columna vertebral en zigzag de solo el ADN-Z. De manera similar a lo que se encuentra en la enfermedad de Alzheimer, los anticuerpos varían según la etapa de la enfermedad, con anticuerpos máximos en las etapas más activas del LES.
Comúnmente se cree que el ADN-Z proporciona alivio de la tensión torsional durante la transcripción y está asociado con un superenrollamiento negativo . [5] [22] Sin embargo, mientras que el superenrollamiento está asociado tanto con la transcripción como con la replicación del ADN, la formación de ADN-Z está relacionada principalmente con la tasa de transcripción . [23]
Un estudio del cromosoma 22 humano mostró una correlación entre las regiones formadoras de ADN Z y las regiones promotoras del factor nuclear I. Esto sugiere que la transcripción en algunos genes humanos puede estar regulada por la formación de ADN Z y la activación del factor nuclear I. [11]
Se ha demostrado que las secuencias de ADN Z aguas arriba de las regiones promotoras estimulan la transcripción. El mayor aumento de actividad se observa cuando la secuencia de ADN Z se coloca tres vueltas helicoidales después de la secuencia promotora . Además, utilizando la técnica de entrecruzamiento de nucleasas microcócicas, [24] es poco probable que el ADN-Z forme nucleosomas , que a menudo se ubican antes y/o después de una secuencia de formación de ADN-Z. Debido a esta propiedad, se supone que el ADN Z codifica el límite en el posicionamiento de los nucleosomas. Dado que la ubicación de los nucleosomas influye en la unión de los factores de transcripción , se cree que el ADN Z regula la tasa de transcripción. [24]
Desarrollado detrás de la vía de la ARN polimerasa mediante superenrollamiento negativo, se ha demostrado que el ADN Z formado mediante transcripción activa aumenta la inestabilidad genética, creando una propensión a la mutagénesis cerca de los promotores. [25] Un estudio sobre Escherichia coli encontró que las deleciones de genes ocurren espontáneamente en regiones de plásmidos que contienen secuencias formadoras de ADN-Z. [26] En células de mamíferos, se descubrió que la presencia de tales secuencias produce grandes eliminaciones de fragmentos genómicos debido a roturas de doble cadena cromosómica . Ambas modificaciones genéticas se han relacionado con las translocaciones genéticas encontradas en cánceres como la leucemia y el linfoma , ya que las regiones de rotura en las células tumorales se han trazado alrededor de secuencias formadoras de ADN Z. [25] Sin embargo, las deleciones más pequeñas en plásmidos bacterianos se han asociado con un deslizamiento de replicación , mientras que las deleciones más grandes asociadas con células de mamíferos son causadas por una reparación de unión de extremos no homóloga , que se sabe que es propensa a errores. [25] [26]
El efecto tóxico del bromuro de etidio (EtBr) sobre los tripanosomas se debe al cambio de su ADN cinetoplástido a la forma Z. El cambio es causado por la intercalación de EtBr y el posterior aflojamiento de la estructura del ADN que conduce al desenrollamiento del ADN, el cambio a la forma Z y la inhibición de la replicación del ADN. [27]
El primer dominio que se une al ADN-Z con alta afinidad se descubrió en ADAR1 utilizando un enfoque desarrollado por Alan Herbert. [28] [29] Los estudios cristalográficos y de RMN confirmaron los hallazgos bioquímicos de que este dominio se unía al ADN-Z de una manera no específica de secuencia. [30] [31] [32] Se identificaron dominios relacionados en varias otras proteínas mediante homología de secuencia . [29] La identificación del dominio Zα proporcionó una herramienta para otros estudios cristalográficos que conducen a la caracterización del Z-RNA y la unión B-Z. Los estudios biológicos sugirieron que el dominio de unión al ADN Z de ADAR1 puede localizar esta enzima que modifica la secuencia del ARN recién formado en sitios de transcripción activa. [33] [34] El papel de Zα, Z-DNA y Z-RNA en la defensa del genoma contra la invasión de retroelementos Alu en humanos ha evolucionado hasta convertirse en un mecanismo para la regulación de las respuestas inmunes innatas al dsRNA. Las mutaciones en Zα son causales de interferonopatías humanas como el síndrome mendeliano de Aicardi-Goutières. [35] [18] Además, se ha demostrado que los dominios Zα se localizan en los gránulos de estrés debido a su capacidad innata para unirse al ácido nucleico. Además, diferentes dominios Zα se unen a la conformación Z del ácido nucleico de manera diferente, lo que proporciona vías importantes para la orientación específica en el descubrimiento de fármacos.
A medida que el Z-DNA se ha investigado más a fondo, se ha descubierto que la estructura del Z-DNA puede unirse a las proteínas de unión del Z-DNA a través de la dispersión de Londres y los enlaces de hidrógeno . [36] Un ejemplo de una proteína de unión al ADN-Z es la proteína E3L de vaccinia , que es un producto del gen E3L e imita una proteína de mamífero que se une al ADN-Z. [37] [38] La proteína E3L no solo tiene afinidad por el ADN-Z, sino que también se ha descubierto que desempeña un papel en el nivel de gravedad de la virulencia en ratones causada por el virus vaccinia, un tipo de poxvirus . Dos componentes críticos de la proteína E3L que determinan la virulencia son el extremo N y el extremo C. El extremo N está formado por una secuencia similar a la del dominio Zα, también llamado dominio z-alfa de adenosina desaminasa , mientras que el extremo C está compuesto por un motivo de unión a ARN bicatenario. [37] A través de la investigación realizada por Kim, Y. et al. en el Instituto de Tecnología de Massachusetts, se demostró que reemplazar el extremo N de la proteína E3L con una secuencia del dominio Zα, que contiene 14 residuos de unión al ADN Z similares a E3L, tenía poco o ningún efecto sobre la patogenicidad del virus en ratones. [37] En contraste, Kim, Y. et al. También descubrió que la eliminación de los 83 residuos del extremo N de E3L daba lugar a una disminución de la virulencia. Esto respalda su afirmación de que el extremo N que contiene los residuos de unión del ADN Z es necesario para la virulencia. [37] En general, estos hallazgos muestran que los residuos de unión Z-DNA similares dentro del extremo N de la proteína E3L y el dominio Zα son los factores estructurales más importantes que determinan la virulencia causada por el virus vaccinia, mientras que los residuos de aminoácidos no involucrados en La unión del ADN-Z tiene poco o ningún efecto. Una implicación futura de estos hallazgos incluye la reducción de la unión del ADN-Z de E3L en las vacunas que contienen el virus vaccinia para minimizar las reacciones negativas al virus en humanos. [37]
Además, Alexander Rich y Jin-Ah Kwon descubrieron que E3L actúa como un transactivador de los genes humanos IL-6, NF-AT y p53. Sus resultados muestran que las células HeLa que contienen E3L tenían una mayor expresión de los genes humanos IL-6, NF-AT y p53 y las mutaciones puntuales o deleciones de ciertos residuos de aminoácidos de unión al ADN Z disminuyeron esa expresión. [36] Específicamente, se encontró que las mutaciones en Tyr 48 y Pro 63 reducen la transactivación de los genes mencionados anteriormente, como resultado de la pérdida de los enlaces de hidrógeno y las fuerzas de dispersión de Londres entre E3L y el ADN Z. [36] En general, estos resultados muestran que la disminución de los enlaces y las interacciones entre el ADN-Z y las proteínas de unión al ADN-Z disminuye tanto la virulencia como la expresión génica, lo que muestra la importancia de tener enlaces entre el ADN-Z y la proteína de unión a E3L.

