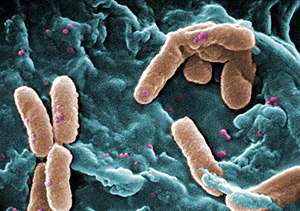
Pseudomonas aeruginosa
Este patógeno oportunista de individuos inmunocomprometidos, P. aeruginosa infecta los pulmones y las vías respiratorias, las vías urinarias, los tejidos, (heridas), y también causa otras sepsis (infecciones generalizadas en el organismo).
[4] Pseudomonas puede causar neumonías a grupos,[5] lo que en ocasiones precisa ayuda mecánica para superar dichas neumonías, siendo uno de los microorganismos más frecuentes aislados en muchos estudios.
Sin embargo, la investigación indica que el ácido salicílico puede inhibir la producción de piocianina.
[7] La fibrosis quística está también predispuesta a la infección con P. aeruginosa de los pulmones.
El más común causante de altas fiebres en infecciones es P. aeruginosa[cita requerida].
Los estudios con mutantes quorum sensibles PAO210 (rhlI), PAO214 (lasI), y PAO216 (lasI rhlI) demostraron que todas las razas eran patogénicas a Arabidopsis, que naturalmente no secretan ácido rosmarínico como un exudado de raíces.
Sin embargo, PAO214 fue la única raza patogénica que emitió exudado dulce, y biofilme de PAO214 pareció comparable con biofilmes formados en razas salvajes de P.
Cuando P. aeruginosa se aísla de sitios estériles (sangre, hueso, colecciones profundas) debe tomarse con mucha seriedad y en la mayor parte de los casos requiere tratamiento rápido.
El monitoreo terapéutico (TDM por sus siglas en inglés: Therapeutic Drug Monitoring) de los aminoglucósidos (p.