La reparación del ADN es un conjunto de procesos mediante los cuales una célula identifica y corrige los daños en las moléculas de ADN que codifican su genoma . [1] En las células humanas, tanto las actividades metabólicas normales como los factores ambientales como la radiación pueden causar daños en el ADN, lo que da como resultado decenas de miles de lesiones moleculares individuales por célula por día. [2] Muchas de estas lesiones causan daño estructural a la molécula de ADN y pueden alterar o eliminar la capacidad de la célula para transcribir el gen que codifica el ADN afectado. Otras lesiones inducen mutaciones potencialmente dañinas en el genoma de la célula, que afectan la supervivencia de sus células hijas después de que experimenta mitosis . Como consecuencia, el proceso de reparación del ADN está constantemente activo ya que responde al daño en la estructura del ADN. Cuando los procesos de reparación normales fallan, y cuando no se produce la apoptosis celular , puede producirse un daño irreparable en el ADN. Esto puede eventualmente conducir a tumores malignos o cáncer según la hipótesis de los dos golpes .
La velocidad de reparación del ADN depende de varios factores, entre ellos el tipo de célula, la edad de la célula y el entorno extracelular. Una célula que ha acumulado una gran cantidad de daño en el ADN o que ya no puede repararlo de manera eficaz puede entrar en uno de tres estados posibles:
La capacidad de reparación del ADN de una célula es vital para la integridad de su genoma y, por lo tanto, para el funcionamiento normal de ese organismo. Muchos genes que inicialmente se demostró que influían en la longevidad han resultado estar involucrados en la reparación y protección del ADN dañado. [3]
El Premio Nobel de Química de 2015 fue otorgado a Tomas Lindahl , Paul Modrich y Aziz Sancar por su trabajo sobre los mecanismos moleculares de los procesos de reparación del ADN. [4] [5]
El daño del ADN, debido a factores ambientales y procesos metabólicos normales dentro de la célula, ocurre a una tasa de 10.000 a 1.000.000 de lesiones moleculares por célula por día. [2] Si bien esto constituye como máximo solo el 0,0003125% de los aproximadamente 3.2 mil millones de bases del genoma humano, las lesiones no reparadas en genes críticos (como los genes supresores de tumores ) pueden impedir la capacidad de una célula para llevar a cabo su función y aumentar apreciablemente la probabilidad de formación de tumores y contribuir a la heterogeneidad tumoral .
La gran mayoría de los daños en el ADN afectan a la estructura primaria de la doble hélice, es decir, las bases mismas se modifican químicamente. Estas modificaciones pueden, a su vez, alterar la estructura helicoidal regular de las moléculas al introducir enlaces químicos no nativos o aductos voluminosos que no encajan en la doble hélice estándar. A diferencia de las proteínas y el ARN , el ADN generalmente carece de estructura terciaria y, por lo tanto, no se producen daños o alteraciones a ese nivel. Sin embargo, el ADN está superenrollado y enrollado alrededor de proteínas de "empaquetamiento" llamadas histonas (en eucariotas), y ambas superestructuras son vulnerables a los efectos del daño en el ADN.
El daño del ADN se puede subdividir en dos tipos principales:
La replicación del ADN dañado antes de la división celular puede dar lugar a la incorporación de bases erróneas en el lado opuesto de las dañadas. Las células hijas que heredan estas bases erróneas son portadoras de mutaciones de las cuales la secuencia original del ADN es irrecuperable (excepto en el raro caso de una mutación inversa , por ejemplo, a través de una conversión génica ).
Existen varios tipos de daños al ADN debido a procesos celulares endógenos:
Los daños causados por agentes exógenos se presentan de muchas formas. Algunos ejemplos son:
El daño por rayos UV, la alquilación/metilación, el daño por rayos X y el daño oxidativo son ejemplos de daño inducido. El daño espontáneo puede incluir la pérdida de una base, la desaminación, el fruncimiento del anillo de azúcar y el desplazamiento tautomérico. El daño constitutivo (espontáneo) del ADN causado por oxidantes endógenos se puede detectar como un nivel bajo de fosforilación de la histona H2AX en células no tratadas. [9]
En las células humanas, y en las células eucariotas en general, el ADN se encuentra en dos ubicaciones celulares: dentro del núcleo y dentro de las mitocondrias . El ADN nuclear (n-ADN) existe como cromatina durante las etapas no replicativas del ciclo celular y se condensa en estructuras agregadas conocidas como cromosomas durante la división celular . En cualquiera de los dos estados, el ADN está muy compactado y enrollado alrededor de proteínas con forma de cuentas llamadas histonas . Siempre que una célula necesita expresar la información genética codificada en su n-ADN, se desenreda la región cromosómica requerida, se expresan los genes ubicados allí y luego la región se condensa nuevamente a su conformación en reposo. El ADN mitocondrial (mtADN) se encuentra dentro de los orgánulos de las mitocondrias , existe en múltiples copias y también está estrechamente asociado con varias proteínas para formar un complejo conocido como nucleoide. Dentro de las mitocondrias, las especies reactivas de oxígeno (ROS), o radicales libres , subproductos de la producción constante de trifosfato de adenosina (ATP) a través de la fosforilación oxidativa , crean un ambiente altamente oxidativo que se sabe que daña el ADNmt. Una enzima fundamental para contrarrestar la toxicidad de estas especies es la superóxido dismutasa , que está presente tanto en las mitocondrias como en el citoplasma de las células eucariotas.
La senescencia, un proceso irreversible en el que la célula ya no se divide , es una respuesta protectora al acortamiento de los extremos de los cromosomas, llamados telómeros . Los telómeros son largas regiones de ADN no codificante repetitivo que tapan los cromosomas y sufren una degradación parcial cada vez que una célula sufre una división (véase el límite de Hayflick ). [10] En contraste, la quiescencia es un estado reversible de latencia celular que no está relacionado con el daño del genoma (véase el ciclo celular ). La senescencia en las células puede servir como una alternativa funcional a la apoptosis en los casos en que el organismo requiere la presencia física de una célula por razones espaciales, [11] lo que sirve como un mecanismo de "último recurso" para evitar que una célula con ADN dañado se replique de forma inapropiada en ausencia de señalización celular pro-crecimiento . La división celular no regulada puede conducir a la formación de un tumor (véase cáncer ), que es potencialmente letal para un organismo. Por lo tanto, la inducción de la senescencia y la apoptosis se considera parte de una estrategia de protección contra el cáncer. [12]
Es importante distinguir entre daño y mutación del ADN, los dos tipos principales de error en el ADN. El daño y la mutación del ADN son fundamentalmente diferentes. El daño produce anomalías físicas en el ADN, como roturas de cadena simple y doble, residuos de 8-hidroxidesoxiguanosina y aductos de hidrocarburos aromáticos policíclicos. El daño del ADN puede ser reconocido por enzimas y, por lo tanto, puede repararse correctamente si hay información redundante, como la secuencia intacta en la cadena de ADN complementaria o en un cromosoma homólogo, disponible para copiar. Si una célula conserva el daño del ADN, se puede prevenir la transcripción de un gen y, por lo tanto, también se bloqueará la traducción a una proteína. También se puede bloquear la replicación o la célula puede morir.
A diferencia de los daños en el ADN, una mutación es un cambio en la secuencia de bases del ADN. Una mutación no puede ser reconocida por las enzimas una vez que el cambio de base está presente en ambas cadenas de ADN, y por lo tanto, no puede ser reparada. A nivel celular, las mutaciones pueden causar alteraciones en la función y regulación de las proteínas. Las mutaciones se replican cuando la célula se replica. En una población de células, la frecuencia de las células mutantes aumentará o disminuirá según los efectos de la mutación en la capacidad de la célula para sobrevivir y reproducirse.
Aunque son claramente diferentes entre sí, el daño y la mutación del ADN están relacionados porque el daño del ADN a menudo causa errores en la síntesis del ADN durante la replicación o reparación; estos errores son una fuente importante de mutación.
Dadas estas propiedades del daño y la mutación del ADN, se puede ver que el daño del ADN es un problema especial en las células que no se dividen o que se dividen lentamente, donde el daño no reparado tenderá a acumularse con el tiempo. Por otro lado, en las células que se dividen rápidamente, el daño no reparado del ADN que no mata a la célula bloqueando la replicación tenderá a causar errores de replicación y, por lo tanto, mutación. La gran mayoría de las mutaciones que no son neutrales en su efecto son perjudiciales para la supervivencia de una célula. Por lo tanto, en una población de células que componen un tejido con células replicantes, las células mutantes tenderán a perderse. Sin embargo, las mutaciones infrecuentes que proporcionan una ventaja de supervivencia tenderán a expandirse clonalmente a expensas de las células vecinas en el tejido. Esta ventaja para la célula es desventajosa para todo el organismo porque dichas células mutantes pueden dar lugar al cáncer. Por lo tanto, el daño del ADN en las células que se dividen con frecuencia, debido a que da lugar a mutaciones, es una causa importante de cáncer. En contraste, el daño del ADN en las células que se dividen con poca frecuencia es probablemente una causa importante de envejecimiento. [13]
Las células no pueden funcionar si el daño del ADN corrompe la integridad y la accesibilidad de la información esencial del genoma (pero las células siguen siendo superficialmente funcionales cuando faltan o están dañados genes no esenciales). Según el tipo de daño infligido a la estructura de doble hélice del ADN, se han desarrollado diversas estrategias de reparación para restaurar la información perdida. Si es posible, las células utilizan la cadena complementaria no modificada del ADN o la cromátida hermana como plantilla para recuperar la información original. Sin acceso a una plantilla, las células utilizan un mecanismo de recuperación propenso a errores conocido como síntesis por translesión como último recurso.
Los daños en el ADN alteran la configuración espacial de la hélice, y la célula puede detectar dichas alteraciones. Una vez localizado el daño, las moléculas reparadoras del ADN específicas se unen en el lugar del daño o cerca de él, lo que induce a otras moléculas a unirse y formar un complejo que permite que se lleve a cabo la reparación.
Se sabe que las células eliminan tres tipos de daño a su ADN revirtiéndolo químicamente. Estos mecanismos no requieren una plantilla, ya que los tipos de daño que contrarrestan pueden ocurrir en solo una de las cuatro bases. Estos mecanismos de reversión directa son específicos del tipo de daño incurrido y no implican la rotura de la cadena principal de fosfodiéster. La formación de dímeros de pirimidina tras la irradiación con luz UV da como resultado un enlace covalente anormal entre bases de pirimidina adyacentes. El proceso de fotorreactivación revierte directamente este daño mediante la acción de la enzima fotoliasa , cuya activación depende obligatoriamente de la energía absorbida de la luz azul/UV ( longitud de onda de 300-500 nm ) para promover la catálisis. [14] La fotoliasa, una antigua enzima presente en bacterias , hongos y la mayoría de los animales , ya no funciona en los humanos, [15] quienes, en cambio, utilizan la reparación por escisión de nucleótidos para reparar el daño causado por la irradiación UV. Otro tipo de daño, la metilación de las bases de guanina, es revertido directamente por la enzima metil guanina metil transferasa (MGMT), cuyo equivalente bacteriano se llama ogt . Este es un proceso costoso porque cada molécula de MGMT puede usarse solo una vez; es decir, la reacción es estequiométrica en lugar de catalítica . [16] Una respuesta generalizada a los agentes metilantes en bacterias se conoce como respuesta adaptativa y confiere un nivel de resistencia a los agentes alquilantes tras una exposición sostenida mediante la regulación positiva de las enzimas de reparación de la alquilación. [17] El tercer tipo de daño del ADN revertido por las células es cierta metilación de las bases citosina y adenina.

Cuando sólo una de las dos cadenas de una doble hélice presenta un defecto, la otra cadena puede utilizarse como plantilla para guiar la corrección de la cadena dañada. Para reparar el daño en una de las dos moléculas pareadas de ADN, existen varios mecanismos de reparación por escisión que eliminan el nucleótido dañado y lo reemplazan por un nucleótido intacto complementario al que se encuentra en la cadena de ADN intacta. [16]
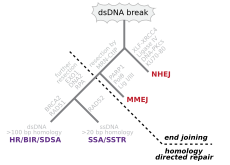
Las roturas de doble cadena, en las que se cortan ambas hebras de la doble hélice, son particularmente peligrosas para la célula porque pueden conducir a reordenamientos del genoma . De hecho, cuando una rotura de doble cadena va acompañada de un enlace cruzado que une las dos hebras en el mismo punto, ninguna de las dos cadenas puede utilizarse como plantilla para los mecanismos de reparación, de modo que la célula no podrá completar la mitosis cuando se divida a continuación y morirá o, en casos raros, sufrirá una mutación. [22] [23] Existen tres mecanismos para reparar las roturas de doble cadena (DSB): unión de extremos no homólogos (NHEJ), unión de extremos mediada por microhomología (MMEJ) y recombinación homóloga (HR): [16] [24]

En un sistema in vitro , la MMEJ se produjo en células de mamíferos en niveles de 10 a 20 % de HR cuando también estaban disponibles los mecanismos de HR y NHEJ. [32]
El extremófilo Deinococcus radiodurans tiene una notable capacidad para sobrevivir al daño del ADN causado por la radiación ionizante y otras fuentes. Al menos dos copias del genoma, con roturas aleatorias del ADN, pueden formar fragmentos de ADN mediante el annealing . Los fragmentos parcialmente superpuestos se utilizan luego para la síntesis de regiones homólogas a través de un D-loop móvil que puede continuar la extensión hasta que se encuentran cadenas complementarias. En el paso final, hay un entrecruzamiento mediante recombinación homóloga dependiente de RecA . [35]
Las topoisomerasas introducen roturas tanto de cadena simple como de cadena doble durante el cambio del estado de superenrollamiento del ADN , lo que es especialmente común en regiones cercanas a una horquilla de replicación abierta. Estas roturas no se consideran daños en el ADN porque son un intermediario natural en el mecanismo bioquímico de la topoisomerasa y son reparadas inmediatamente por las enzimas que las crearon.
Otro tipo de rotura de doble cadena de ADN se origina en los sitios termosensibles o termolábiles del ADN. Estos sitios de ADN no son roturas de doble cadena iniciales. Sin embargo, se convierten en roturas de doble cadena después de tratarlos con temperaturas elevadas. La irradiación ionizante puede inducir una forma altamente compleja de daño del ADN, como daño agrupado. Consiste en diferentes tipos de lesiones del ADN en varias ubicaciones de la hélice del ADN. Algunas de estas lesiones ubicadas cerca probablemente puedan convertirse en roturas de doble cadena mediante la exposición a altas temperaturas. Pero la naturaleza exacta de estas lesiones y sus interacciones aún no se conoce [36].
La síntesis por translesión (TLS) es un proceso de tolerancia al daño del ADN que permite que la maquinaria de replicación del ADN replique lesiones pasadas del ADN, como dímeros de timina o sitios AP . [37] Implica cambiar las polimerasas de ADN regulares por polimerasas de translesión especializadas (es decir, la ADN polimerasa IV o V, de la familia de la polimerasa Y), a menudo con sitios activos más grandes que pueden facilitar la inserción de bases opuestas a los nucleótidos dañados. Se cree que el cambio de polimerasa está mediado, entre otros factores, por la modificación postraduccional del factor de procesividad de replicación PCNA . Las polimerasas de síntesis por translesión a menudo tienen baja fidelidad (alta propensión a insertar bases incorrectas) en plantillas no dañadas en relación con las polimerasas regulares. Sin embargo, muchas son extremadamente eficientes para insertar bases correctas opuestas a tipos específicos de daño. Por ejemplo, Pol η media la omisión sin errores de las lesiones inducidas por la irradiación UV , mientras que Pol ι introduce mutaciones en estos sitios. Se sabe que la pol η agrega la primera adenina a través del fotodímero T^T usando el apareamiento de bases Watson-Crick y la segunda adenina se agregará en su conformación syn usando el apareamiento de bases Hoogsteen . Desde una perspectiva celular, correr el riesgo de la introducción de mutaciones puntuales durante la síntesis de translesión puede ser preferible a recurrir a mecanismos más drásticos de reparación del ADN, que pueden causar aberraciones cromosómicas graves o muerte celular. En resumen, el proceso involucra polimerasas especializadas que evitan o reparan lesiones en lugares donde la replicación del ADN se detiene. Por ejemplo, la ADN polimerasa humana eta puede evitar lesiones complejas del ADN como el entrecruzamiento intracatenario de guanina-timina, G[8,5-Me]T, aunque puede causar mutaciones dirigidas y semidirigidas. [38] Paromita Raychaudhury y Ashis Basu [39] estudiaron la toxicidad y mutagénesis de la misma lesión en Escherichia coli replicando un plásmido modificado con G[8,5-Me]T en E. coli con knockouts específicos de la ADN polimerasa. La viabilidad fue muy baja en una cepa que carecía de pol II, pol IV y pol V, las tres ADN polimerasas inducibles por SOS, lo que indica que la síntesis de translesión se lleva a cabo principalmente por estas ADN polimerasas especializadas. El antígeno nuclear de células en proliferación (PCNA) proporciona una plataforma de derivación a estas polimerasas. En circunstancias normales, el PCNA unido a las polimerasas replica el ADN. En un sitio de lesión , el PCNA es ubiquitinado o modificado por las proteínas RAD6/ RAD18 . para proporcionar una plataforma para que las polimerasas especializadas eviten la lesión y reanuden la replicación del ADN. [40] [41] Después de la síntesis de translesión, se requiere extensión. Esta extensión puede ser realizada por una polimerasa replicativa si el TLS está libre de errores, como en el caso de Pol η, pero si el TLS da como resultado un desajuste, se necesita una polimerasa especializada para extenderlo; Pol ζ . Pol ζ es única en el sentido de que puede extender desajustes terminales, mientras que las polimerasas más procesivas no pueden. Entonces, cuando se encuentra una lesión, la horquilla de replicación se detendrá, PCNA cambiará de una polimerasa procesiva a una polimerasa TLS como Pol ι para arreglar la lesión, luego PCNA puede cambiar a Pol ζ para extender el desajuste, y por último PCNA cambiará a la polimerasa procesiva para continuar la replicación.
Las células expuestas a la radiación ionizante , la luz ultravioleta o los productos químicos son propensas a adquirir múltiples sitios de lesiones voluminosas del ADN y roturas de doble cadena. Además, los agentes que dañan el ADN pueden dañar otras biomoléculas como las proteínas , los carbohidratos , los lípidos y el ARN . La acumulación de daño, para ser específicos, las roturas de doble cadena o los aductos que bloquean las horquillas de replicación , se encuentran entre las señales de estimulación conocidas para una respuesta global al daño del ADN. [42] La respuesta global al daño es un acto dirigido hacia la propia preservación de las células y desencadena múltiples vías de reparación macromolecular, derivación de lesiones, tolerancia o apoptosis . Las características comunes de la respuesta global son la inducción de múltiples genes , la detención del ciclo celular y la inhibición de la división celular .
El empaquetamiento del ADN eucariota en cromatina presenta una barrera para todos los procesos basados en ADN que requieren el reclutamiento de enzimas a sus sitios de acción. Para permitir la reparación del ADN, la cromatina debe ser remodelada . En eucariotas, los complejos de remodelación de cromatina dependientes de ATP y las enzimas modificadoras de histonas son dos factores predominantes empleados para lograr este proceso de remodelación. [43]
La relajación de la cromatina ocurre rápidamente en el sitio de un daño del ADN. [44] [45] En uno de los primeros pasos, la proteína quinasa activada por estrés, c-Jun N-terminal quinasa (JNK) , fosforila SIRT6 en la serina 10 en respuesta a roturas de doble cadena u otro daño del ADN. [46] Esta modificación postraduccional facilita la movilización de SIRT6 a los sitios de daño del ADN, y es necesaria para el reclutamiento eficiente de la poli (ADP-ribosa) polimerasa 1 (PARP1) a los sitios de rotura del ADN y para la reparación eficiente de DSB. [46] La proteína PARP1 comienza a aparecer en los sitios de daño del ADN en menos de un segundo, con la mitad de la acumulación máxima dentro de los 1,6 segundos después de que ocurre el daño. [47] PARP1 sintetiza cadenas poliméricas de adenosina difosfato ribosa (poli (ADP-ribosa) o PAR) sobre sí misma. A continuación, el remodelador de cromatina ALC1 se une rápidamente al producto de la acción de PARP1, una cadena de poli-ADP ribosa, y ALC1 completa la llegada al daño del ADN dentro de los 10 segundos posteriores a la ocurrencia del daño. [45] Aproximadamente la mitad de la relajación máxima de la cromatina, presumiblemente debido a la acción de ALC1, ocurre a los 10 segundos. [45] Esto luego permite el reclutamiento de la enzima reparadora de ADN MRE11 , para iniciar la reparación del ADN, dentro de los 13 segundos. [47]
γH2AX, la forma fosforilada de H2AX también está involucrada en los primeros pasos que conducen a la descondensación de la cromatina después de las roturas de doble cadena de ADN. La variante de histona H2AX constituye aproximadamente el 10% de las histonas H2A en la cromatina humana. [48] γH2AX (H2AX fosforilada en la serina 139) puede detectarse tan pronto como 20 segundos después de la irradiación de las células (con formación de rotura de doble cadena de ADN), y la mitad de la acumulación máxima de γH2AX ocurre en un minuto. [48] La extensión de la cromatina con γH2AX fosforilada es de aproximadamente dos millones de pares de bases en el sitio de una rotura de doble cadena de ADN. [48] γH2AX no causa, por sí misma, la descondensación de la cromatina, pero dentro de los 30 segundos posteriores a la irradiación, la proteína RNF8 puede detectarse en asociación con γH2AX. [49] RNF8 media la descondensación extensa de la cromatina, a través de su interacción posterior con CHD4 , [50] un componente del complejo de remodelación de nucleosomas y desacetilasa NuRD .
DDB2 se presenta en un complejo heterodimérico con DDB1 . Este complejo se une a su vez a la proteína ubiquitina ligasa CUL4A [51] y a PARP1 . [52] Este complejo más grande se asocia rápidamente con el daño inducido por UV dentro de la cromatina, y la asociación máxima se completa en 40 segundos. [51] La proteína PARP1, unida tanto a DDB1 como a DDB2, se PARila (crea una cadena de poli-ADP ribosa) en DDB2 que atrae a la proteína remodeladora de ADN ALC1 . [52] La acción de ALC1 relaja la cromatina en el sitio del daño UV al ADN. Esta relajación permite que otras proteínas en la vía de reparación por escisión de nucleótidos ingresen a la cromatina y reparen los daños del dímero de pirimidina de ciclobutano inducidos por UV .
Después de una rápida remodelación de la cromatina , se activan los puntos de control del ciclo celular para permitir que se produzca la reparación del ADN antes de que progrese el ciclo celular. En primer lugar, se activan dos quinasas , ATM y ATR , dentro de los 5 o 6 minutos posteriores al daño del ADN. A esto le sigue la fosforilación de la proteína del punto de control del ciclo celular Chk1 , que inicia su función, aproximadamente 10 minutos después del daño del ADN. [53]
Después del daño del ADN, se activan los puntos de control del ciclo celular . La activación del punto de control detiene el ciclo celular y le da tiempo a la célula para reparar el daño antes de continuar dividiéndose. Los puntos de control del daño del ADN ocurren en los límites G1 / S y G2 / M. También existe un punto de control intra- S . La activación del punto de control está controlada por dos quinasas maestras , ATM y ATR . ATM responde a las roturas de doble cadena de ADN y las interrupciones en la estructura de la cromatina, [54] mientras que ATR responde principalmente a las horquillas de replicación estancadas . Estas quinasas fosforilan objetivos corriente abajo en una cascada de transducción de señales , que finalmente conduce a la detención del ciclo celular. También se ha identificado una clase de proteínas mediadoras de puntos de control que incluyen BRCA1 , MDC1 y 53BP1 . [55] Estas proteínas parecen ser necesarias para transmitir la señal de activación del punto de control a las proteínas corriente abajo.
El punto de control de daño del ADN es una vía de transducción de señales que bloquea la progresión del ciclo celular en G1, G2 y metafase y reduce la velocidad de progresión de la fase S cuando el ADN está dañado. Provoca una pausa en el ciclo celular que permite que la célula tenga tiempo de reparar el daño antes de continuar dividiéndose.
Las proteínas de punto de control se pueden separar en cuatro grupos: proteína quinasa similar a la fosfatidilinositol 3-quinasa (PI3K) , grupo similar al antígeno nuclear de células proliferantes (PCNA), dos quinasas de serina/treonina (S/T) y sus adaptadores. En el centro de todas las respuestas de puntos de control inducidas por daño del ADN se encuentra un par de grandes quinasas de proteína que pertenecen al primer grupo de quinasas de proteína similares a PI3K: las quinasas ATM ( Ataxia telangiectasia mutada ) y ATR (Ataxia- y Rad-relacionadas), cuya secuencia y funciones se han conservado bien en la evolución. Toda respuesta al daño del ADN requiere ATM o ATR porque tienen la capacidad de unirse a los cromosomas en el sitio del daño del ADN, junto con proteínas accesorias que son plataformas en las que se pueden ensamblar los componentes de respuesta al daño del ADN y los complejos de reparación del ADN.
Un objetivo importante de ATM y ATR es p53 , ya que es necesario para inducir la apoptosis después del daño del ADN. [56] El inhibidor de la quinasa dependiente de ciclina p21 es inducido por mecanismos dependientes e independientes de p53 y puede detener el ciclo celular en los puntos de control G1/S y G2/M desactivando los complejos ciclina / quinasa dependiente de ciclina . [57]
La respuesta SOS son los cambios en la expresión génica en Escherichia coli y otras bacterias en respuesta a un daño extenso del ADN. El sistema SOS procariota está regulado por dos proteínas clave: LexA y RecA . El homodímero LexA es un represor transcripcional que se une a secuencias operadoras comúnmente conocidas como cajas SOS. En Escherichia coli se sabe que LexA regula la transcripción de aproximadamente 48 genes, incluidos los genes lexA y recA. [58] Se sabe que la respuesta SOS está muy extendida en el dominio Bacteria, pero está mayormente ausente en algunos filos bacterianos, como las espiroquetas . [59] Las señales celulares más comunes que activan la respuesta SOS son regiones de ADN monocatenario (ssDNA), que surgen de horquillas de replicación estancadas o roturas de doble cadena, que son procesadas por la helicasa del ADN para separar las dos cadenas de ADN. [42] En la etapa de iniciación, la proteína RecA se une al ssDNA en una reacción impulsada por hidrólisis de ATP que crea filamentos RecA–ssDNA. Los filamentos RecA–ssDNA activan la actividad de la autoproteasa LexA , que finalmente conduce a la escisión del dímero LexA y la posterior degradación de LexA. La pérdida del represor LexA induce la transcripción de los genes SOS y permite una mayor inducción de señales, inhibición de la división celular y un aumento en los niveles de proteínas responsables del procesamiento del daño.
En Escherichia coli , las cajas SOS son secuencias de 20 nucleótidos de longitud cerca de promotores con estructura palindrómica y un alto grado de conservación de secuencia. En otras clases y filos, la secuencia de las cajas SOS varía considerablemente, con diferente longitud y composición, pero siempre está altamente conservada y es una de las señales cortas más fuertes del genoma. [59] El alto contenido de información de las cajas SOS permite la unión diferencial de LexA a diferentes promotores y permite la sincronización de la respuesta SOS. Los genes de reparación de lesiones se inducen al comienzo de la respuesta SOS. Las polimerasas de translesión propensas a errores, por ejemplo, UmuCD'2 (también llamada ADN polimerasa V), se inducen más tarde como último recurso. [60] Una vez que el daño del ADN se repara o se evita mediante polimerasas o mediante recombinación, la cantidad de ADN monocatenario en las células disminuye; al reducir la cantidad de filamentos de RecA, disminuye la actividad de escisión del homodímero LexA, que luego se une a las cajas SOS cerca de los promotores y restaura la expresión genética normal.
Las células eucariotas expuestas a agentes que dañan el ADN también activan vías defensivas importantes al inducir múltiples proteínas involucradas en la reparación del ADN, el control de los puntos de control del ciclo celular , el tráfico de proteínas y la degradación. Esta respuesta transcripcional de todo el genoma es muy compleja y está estrechamente regulada, lo que permite una respuesta global coordinada al daño. La exposición de la levadura Saccharomyces cerevisiae a agentes que dañan el ADN da como resultado perfiles transcripcionales superpuestos pero distintos. Las similitudes con la respuesta al choque ambiental indican que existe una vía general de respuesta al estrés global a nivel de activación transcripcional. En contraste, diferentes tipos de células humanas responden al daño de manera diferente, lo que indica la ausencia de una respuesta global común. La explicación probable para esta diferencia entre las células de levadura y humanas puede estar en la heterogeneidad de las células de mamíferos . En un animal, diferentes tipos de células se distribuyen entre diferentes órganos que han desarrollado diferentes sensibilidades al daño del ADN. [61]
En general, la respuesta global al daño del ADN implica la expresión de múltiples genes responsables de la reparación posreplicación , la recombinación homóloga, la reparación por escisión de nucleótidos, el punto de control del daño del ADN , la activación transcripcional global, los genes que controlan la descomposición del ARNm y muchos otros. Una gran cantidad de daño a una célula la deja con una decisión importante: sufrir apoptosis y morir, o sobrevivir a costa de vivir con un genoma modificado. Un aumento en la tolerancia al daño puede conducir a una mayor tasa de supervivencia que permitirá una mayor acumulación de mutaciones. La levadura Rev1 y la polimerasa humana η son miembros de la familia Y de polimerasas de ADN de translesión presentes durante la respuesta global al daño del ADN y son responsables de la mutagénesis mejorada durante una respuesta global al daño del ADN en eucariotas. [42]

Los animales de experimentación con deficiencias genéticas en la reparación del ADN a menudo muestran una disminución de la esperanza de vida y una mayor incidencia de cáncer. [13] Por ejemplo, los ratones deficientes en la vía dominante NHEJ y en los mecanismos de mantenimiento de los telómeros contraen linfoma e infecciones con mayor frecuencia y, como consecuencia, tienen una esperanza de vida más corta que los ratones de tipo salvaje. [62] De manera similar, los ratones deficientes en una proteína clave de reparación y transcripción que desenrolla las hélices de ADN tienen una aparición prematura de enfermedades relacionadas con el envejecimiento y el consiguiente acortamiento de la esperanza de vida. [63] Sin embargo, no todas las deficiencias de reparación del ADN crean exactamente los efectos previstos; los ratones deficientes en la vía NER mostraron una esperanza de vida más corta sin tasas de mutación correspondientemente más altas. [64]
La esperanza de vida máxima de los ratones , las ratas topo desnudas y los humanos son respectivamente ~3, ~30 y ~129 años. [65] De estos, la especie de vida más corta, el ratón, expresa genes de reparación del ADN, incluidos genes centrales en varias vías de reparación del ADN, a un nivel inferior al de los humanos y las ratas topo desnudas. [65] Además, varias vías de reparación del ADN en humanos y ratas topo desnudas están reguladas al alza en comparación con el ratón. Estas observaciones sugieren que la reparación elevada del ADN facilita una mayor longevidad . [65]
Si la tasa de daño del ADN excede la capacidad de la célula para repararlo, la acumulación de errores puede abrumar a la célula y dar como resultado senescencia temprana, apoptosis o cáncer. Las enfermedades hereditarias asociadas con un funcionamiento defectuoso de la reparación del ADN dan como resultado un envejecimiento prematuro, [13] mayor sensibilidad a los carcinógenos y, en consecuencia, mayor riesgo de cáncer (ver más abajo). Por otro lado, los organismos con sistemas mejorados de reparación del ADN, como Deinococcus radiodurans , el organismo conocido más resistente a la radiación, exhiben una resistencia notable a los efectos inductores de rotura de doble cadena de la radiactividad , probablemente debido a una mayor eficiencia de la reparación del ADN y especialmente de la NHEJ. [66]
Se han identificado varios genes individuales que influyen en las variaciones en la expectativa de vida dentro de una población de organismos. Los efectos de estos genes dependen en gran medida del medio ambiente, en particular, de la dieta del organismo. La restricción calórica produce reproduciblemente una expectativa de vida prolongada en una variedad de organismos, probablemente a través de vías de detección de nutrientes y una tasa metabólica reducida . Los mecanismos moleculares por los cuales dicha restricción produce una expectativa de vida prolongada aún no están claros (ver [67] para una discusión); sin embargo, el comportamiento de muchos genes que se sabe que están involucrados en la reparación del ADN se altera en condiciones de restricción calórica. Se ha demostrado que varios agentes que se ha informado que tienen propiedades antienvejecimiento atenúan el nivel constitutivo de la señalización mTOR , una evidencia de reducción de la actividad metabólica , y al mismo tiempo reducen el nivel constitutivo de daño al ADN inducido por especies reactivas de oxígeno generadas endógenamente. [68]
Por ejemplo, aumentar la dosis génica del gen SIR-2, que regula el empaquetamiento del ADN en el gusano nematodo Caenorhabditis elegans , puede extender significativamente la esperanza de vida. [69] Se sabe que el homólogo mamífero de SIR-2 induce factores de reparación del ADN implicados en la NHEJ, una actividad que se promueve especialmente en condiciones de restricción calórica. [70] La restricción calórica se ha relacionado estrechamente con la tasa de reparación por escisión de bases en el ADN nuclear de roedores, [71] aunque no se han observado efectos similares en el ADN mitocondrial. [72]
El gen AGE-1 de C. elegans , un efector ascendente de las vías de reparación del ADN, confiere una esperanza de vida considerablemente más larga en condiciones de alimentación libre, pero conduce a una disminución de la aptitud reproductiva en condiciones de restricción calórica. [73] Esta observación apoya la teoría de la pleiotropía de los orígenes biológicos del envejecimiento , que sugiere que los genes que confieren una gran ventaja de supervivencia en etapas tempranas de la vida serán seleccionados incluso si conllevan una desventaja correspondiente en etapas posteriores de la vida.
Los defectos en el mecanismo NER son responsables de varios trastornos genéticos, entre ellos:
El retraso mental a menudo acompaña a los dos últimos trastornos, lo que sugiere una mayor vulnerabilidad de las neuronas del desarrollo.
Otros trastornos de reparación del ADN incluyen:
Todas las enfermedades mencionadas anteriormente se denominan a menudo " progerias segmentarias " (" enfermedades de envejecimiento acelerado ") porque los afectados parecen mayores y experimentan enfermedades relacionadas con el envejecimiento a una edad anormalmente joven, sin manifestar todos los síntomas de la vejez.
Otras enfermedades asociadas con una función reducida de reparación del ADN incluyen la anemia de Fanconi , el cáncer de mama hereditario y el cáncer de colon hereditario .
Debido a las limitaciones inherentes de los mecanismos de reparación del ADN, si los humanos vivieran lo suficiente, todos acabarían desarrollando cáncer. [74] [75] Hay al menos 34 mutaciones genéticas hereditarias de reparación del ADN humano que aumentan el riesgo de cáncer . Muchas de estas mutaciones hacen que la reparación del ADN sea menos eficaz de lo normal. En particular, el cáncer colorrectal hereditario no asociado a poliposis (HNPCC) está fuertemente asociado con mutaciones específicas en la vía de reparación de desajustes del ADN. BRCA1 y BRCA2 , dos genes importantes cuyas mutaciones confieren un riesgo enormemente mayor de cáncer de mama a los portadores, [76] están asociados con una gran cantidad de vías de reparación del ADN, especialmente NHEJ y recombinación homóloga.
Los procedimientos de tratamiento del cáncer, como la quimioterapia y la radioterapia , actúan sobrepasando la capacidad de la célula para reparar los daños en el ADN, lo que provoca la muerte celular. Las células que se dividen más rápidamente (normalmente las cancerosas) son las más afectadas. El efecto secundario es que también se ven afectadas otras células no cancerosas pero que se dividen rápidamente, como las células progenitoras del intestino, la piel y el sistema hematopoyético. Los tratamientos modernos contra el cáncer intentan localizar el daño en el ADN en las células y los tejidos asociados únicamente con el cáncer, ya sea por medios físicos (concentrando el agente terapéutico en la región del tumor) o por medios bioquímicos (explotando una característica exclusiva de las células cancerosas en el cuerpo). En el contexto de las terapias dirigidas a los genes de respuesta al daño en el ADN, este último enfoque se ha denominado "letalidad sintética". [77]
Tal vez el más conocido de estos fármacos de "letalidad sintética" es el inhibidor de la poli(ADP-ribosa) polimerasa 1 ( PARP1 ), olaparib , que fue aprobado por la Administración de Alimentos y Medicamentos en 2015 para el tratamiento de cáncer de ovario con defectos en BRCA en mujeres. Las células tumorales con pérdida parcial de la respuesta al daño del ADN (específicamente, la reparación por recombinación homóloga ) dependen de otro mecanismo, la reparación de rotura de cadena sencilla, que es un mecanismo que consiste, en parte, en el producto del gen PARP1. [78] El olaparib se combina con quimioterapéuticos para inhibir la reparación de rotura de cadena sencilla inducida por el daño del ADN causado por la quimioterapia coadministrada. Las células tumorales que dependen de este mecanismo residual de reparación del ADN no pueden reparar el daño y, por lo tanto, no pueden sobrevivir y proliferar, mientras que las células normales pueden reparar el daño con el mecanismo de recombinación homóloga en funcionamiento.
Actualmente se están investigando muchos otros fármacos para su uso contra otros mecanismos residuales de reparación del ADN que se encuentran comúnmente en el cáncer. Sin embargo, los enfoques terapéuticos de letalidad sintética han sido cuestionados debido a la evidencia emergente de resistencia adquirida, lograda a través de la reconfiguración de las vías de respuesta al daño del ADN y la reversión de defectos previamente inhibidos. [79]
En los últimos años se ha hecho evidente que la respuesta al daño del ADN actúa como una barrera para la transformación maligna de las células preneoplásicas. [80] Estudios previos han demostrado una respuesta elevada al daño del ADN en modelos de cultivo celular con activación de oncogenes [81] y adenomas de colon preneoplásicos. [82] Los mecanismos de respuesta al daño del ADN desencadenan la detención del ciclo celular e intentan reparar las lesiones del ADN o promover la muerte celular/senescencia si la reparación no es posible. El estrés de replicación se observa en células preneoplásicas debido al aumento de las señales de proliferación de las mutaciones oncogénicas. El estrés de replicación se caracteriza por: aumento de la activación del origen/inicio de replicación; aumento de la transcripción y colisiones de complejos de transcripción-replicación; deficiencia de nucleótidos; aumento de las especies reactivas de oxígeno (ROS). [83]
El estrés de replicación, junto con la selección de mutaciones inactivadoras en genes de respuesta al daño del ADN en la evolución del tumor, [84] conduce a la regulación negativa y/o pérdida de algunos mecanismos de respuesta al daño del ADN y, por lo tanto, a la pérdida de la reparación del ADN y/o senescencia/muerte celular programada. En modelos experimentales de ratón, se observó la pérdida de la senescencia celular mediada por la respuesta al daño del ADN después de utilizar un ARN de horquilla corta (shRNA) para inhibir la quinasa de respuesta a la rotura de doble cadena de la ataxia telangiectasia ( ATM ), lo que conduce a un aumento del tamaño del tumor y de la invasividad. [82] Los humanos nacidos con defectos hereditarios en los mecanismos de reparación del ADN (por ejemplo, el síndrome de Li-Fraumeni ) tienen un mayor riesgo de cáncer. [85]
La prevalencia de mutaciones en la respuesta al daño del ADN difiere entre los distintos tipos de cáncer; por ejemplo, el 30% de los carcinomas invasivos de mama tienen mutaciones en genes implicados en la recombinación homóloga. [80] En el cáncer, se observa una regulación negativa en todos los mecanismos de respuesta al daño del ADN (reparación por escisión de bases [BER], reparación por escisión de nucleótidos [NER], reparación por desajuste del ADN [MMR], reparación por recombinación homóloga [HR], unión de extremos no homólogos [NHEJ] y síntesis de ADN por translesión [TLS]). [86] Además de las mutaciones en los genes de reparación del daño del ADN, también surgen mutaciones en los genes responsables de detener el ciclo celular para permitir que se produzca el tiempo suficiente para la reparación del ADN, y algunos genes están implicados tanto en la reparación del daño del ADN como en el control del punto de control del ciclo celular, por ejemplo, ATM y la quinasa del punto de control 2 (CHEK2), un supresor tumoral que a menudo está ausente o regulado negativamente en el cáncer de pulmón de células no pequeñas. [87]
Clásicamente, el cáncer se ha considerado como un conjunto de enfermedades que son impulsadas por anomalías genéticas progresivas que incluyen mutaciones en genes supresores de tumores y oncogenes, y aberraciones cromosómicas. Sin embargo, se ha hecho evidente que el cáncer también es impulsado por alteraciones epigenéticas . [88]
Las alteraciones epigenéticas se refieren a modificaciones funcionalmente relevantes del genoma que no implican un cambio en la secuencia de nucleótidos. Ejemplos de tales modificaciones son los cambios en la metilación del ADN (hipermetilación e hipometilación) y la modificación de histonas , [89] los cambios en la arquitectura cromosómica (causados por la expresión inapropiada de proteínas como HMGA2 o HMGA1 ) [90] y los cambios causados por microARN . Cada una de estas alteraciones epigenéticas sirve para regular la expresión génica sin alterar la secuencia de ADN subyacente . Estos cambios generalmente permanecen a través de las divisiones celulares , duran varias generaciones de células y pueden considerarse epimutaciones (equivalentes a mutaciones).
Si bien se encuentran grandes cantidades de alteraciones epigenéticas en los cánceres, las alteraciones epigenéticas en los genes de reparación del ADN, que causan una expresión reducida de las proteínas de reparación del ADN, parecen ser particularmente importantes. Se cree que dichas alteraciones ocurren en las primeras etapas de la progresión hacia el cáncer y que son una causa probable de la inestabilidad genética característica de los cánceres. [91] [92] [93]
La expresión reducida de los genes de reparación del ADN provoca una reparación deficiente del ADN. Cuando la reparación del ADN es deficiente, los daños en el ADN permanecen en las células a un nivel más alto de lo habitual y estos daños excesivos provocan un aumento de las frecuencias de mutación o epimutación. Las tasas de mutación aumentan sustancialmente en células defectuosas en la reparación de desajustes del ADN [94] [95] o en la reparación recombinatoria homóloga (HRR). [96] Los reordenamientos cromosómicos y la aneuploidía también aumentan en las células defectuosas en HRR. [97]
Los niveles más elevados de daño del ADN no solo provocan un aumento de la mutación, sino también de la epimutación. Durante la reparación de roturas de doble cadena de ADN o la reparación de otros daños del ADN, los sitios de reparación que no se han limpiado por completo pueden provocar el silenciamiento de genes epigenéticos. [98] [99]
La expresión deficiente de las proteínas reparadoras del ADN debido a una mutación hereditaria puede causar un mayor riesgo de cáncer. Las personas con un deterioro hereditario en cualquiera de los 34 genes de reparación del ADN (véase el artículo Trastorno por deficiencia en la reparación del ADN ) tienen un mayor riesgo de cáncer, y algunos defectos causan hasta un 100 % de probabilidades de padecer cáncer a lo largo de la vida (por ejemplo, las mutaciones del gen p53). [100] Sin embargo, estas mutaciones de la línea germinal (que causan síndromes de cáncer de alta penetración) son la causa de solo alrededor del 1 por ciento de los cánceres. [101]

Las deficiencias en las enzimas reparadoras del ADN son causadas ocasionalmente por una mutación somática de nueva aparición en un gen reparador del ADN, pero son causadas con mucha más frecuencia por alteraciones epigenéticas que reducen o silencian la expresión de los genes reparadores del ADN. Por ejemplo, cuando se examinaron 113 cánceres colorrectales en secuencia, solo cuatro tenían una mutación sin sentido en el gen reparador del ADN MGMT , mientras que la mayoría tenía una expresión reducida de MGMT debido a la metilación de la región promotora de MGMT (una alteración epigenética). [102] Cinco estudios diferentes encontraron que entre el 40% y el 90% de los cánceres colorrectales tienen una expresión reducida de MGMT debido a la metilación de la región promotora de MGMT. [103] [104] [105] [106] [107]
De manera similar, de 119 casos de cáncer colorrectal deficiente en reparación de desajustes que carecían de expresión del gen de reparación de ADN PMS2 , PMS2 fue deficiente en 6 debido a mutaciones en el gen PMS2, mientras que en 103 casos la expresión de PMS2 fue deficiente porque su pareja de emparejamiento MLH1 fue reprimida debido a la metilación del promotor (la proteína PMS2 es inestable en ausencia de MLH1). [108] En los otros 10 casos, la pérdida de la expresión de PMS2 probablemente se debió a la sobreexpresión epigenética del microARN , miR-155 , que regula negativamente MLH1. [109]
En otro ejemplo, se encontraron defectos epigenéticos en varios tipos de cáncer (por ejemplo, de mama, de ovario, colorrectal y de cabeza y cuello). Dos o tres deficiencias en la expresión de ERCC1 , XPF o PMS2 ocurren simultáneamente en la mayoría de los 49 cánceres de colon evaluados por Facista et al. [110]
El gráfico de esta sección muestra algunos agentes que dañan el ADN con frecuencia, ejemplos de lesiones del ADN que causan y las vías que se ocupan de estos daños en el ADN. Al menos 169 enzimas se emplean directamente en la reparación del ADN o influyen en los procesos de reparación del ADN. [111] De ellas, 83 se emplean directamente en la reparación de los 5 tipos de daños en el ADN ilustrados en el gráfico. [ cita requerida ]
En el gráfico se muestran algunos de los genes más estudiados que son fundamentales para estos procesos de reparación. Las designaciones de genes que se muestran en rojo, gris o cian indican genes que se alteran epigenéticamente con frecuencia en varios tipos de cáncer. Los artículos de Wikipedia sobre cada uno de los genes resaltados en rojo, gris o cian describen la(s) alteración(es) epigenética(s) y el(los) cáncer(es) en el que se encuentran estas epimutaciones. Los artículos de revisión [112] y los artículos de encuestas experimentales amplias [113] [114] también documentan la mayoría de estas deficiencias de reparación epigenética del ADN en los cánceres.
Los genes resaltados en rojo se reducen o silencian con frecuencia por mecanismos epigenéticos en varios tipos de cáncer. Cuando estos genes tienen una expresión baja o ausente, los daños en el ADN pueden acumularse. Los errores de replicación más allá de estos daños (ver síntesis translesional) pueden conducir a un aumento de mutaciones y, en última instancia, al cáncer. La represión epigenética de los genes de reparación del ADN en vías precisas de reparación del ADN parece ser fundamental para la carcinogénesis .
Los dos genes resaltados en gris, RAD51 y BRCA2 , son necesarios para la reparación recombinatoria homóloga . A veces se expresan en exceso y a veces en defecto en ciertos tipos de cáncer. Como se indica en los artículos de Wikipedia sobre RAD51 y BRCA2 , estos tipos de cáncer suelen tener deficiencias epigenéticas en otros genes de reparación del ADN. Estas deficiencias de reparación probablemente causarían un aumento de los daños no reparados en el ADN. La sobreexpresión de RAD51 y BRCA2 observada en estos tipos de cáncer puede reflejar presiones selectivas para la sobreexpresión compensatoria de RAD51 o BRCA2 y el aumento de la reparación recombinatoria homóloga para abordar al menos parcialmente estos daños excesivos en el ADN. En aquellos casos en los que RAD51 o BRCA2 están subexpresados, esto en sí mismo conduciría a un aumento de los daños no reparados en el ADN. Los errores de replicación más allá de estos daños (ver síntesis de translesión) podrían causar un aumento de mutaciones y cáncer, por lo que la subexpresión de RAD51 o BRCA2 sería cancerígena en sí misma.
Los genes resaltados en cian se encuentran en la vía de unión de extremos mediada por microhomología (MMEJ) y se regulan positivamente en el cáncer. MMEJ es una vía de reparación imprecisa propensa a errores adicional para roturas de doble cadena. En la reparación MMEJ de una rotura de doble cadena, una homología de 5 a 25 pares de bases complementarios entre ambas cadenas emparejadas es suficiente para alinear las cadenas, pero generalmente hay extremos desapareados (flaps). MMEJ elimina los nucleótidos adicionales (flaps) donde se unen las cadenas y luego liga las cadenas para crear una doble hélice de ADN intacta. MMEJ casi siempre implica al menos una pequeña deleción, por lo que es una vía mutagénica. [24] FEN1 , la endonucleasa del colgajo en MMEJ, aumenta epigenéticamente por la hipometilación del promotor y se sobreexpresa en la mayoría de los cánceres de mama, [115] próstata, [116] estómago, [117] [118] neuroblastomas, [119] páncreas, [120] y pulmón. [121] PARP1 también se sobreexpresa cuando su sitio ETS de la región promotora está hipometilado epigenéticamente , y esto contribuye a la progresión al cáncer de endometrio [122] y al cáncer de ovario seroso con mutación BRCA. [123] Otros genes en la vía MMEJ también se sobreexpresan en varios cánceres (ver MMEJ para el resumen), y también se muestran en cian.
La actividad diferencial de las vías de reparación del ADN en varias regiones del genoma humano hace que las mutaciones se distribuyan de forma muy desigual en los genomas tumorales. [124] [125] En particular, las regiones del genoma humano ricas en genes y de replicación temprana presentan frecuencias de mutación más bajas que la heterocromatina pobre en genes y de replicación tardía . Un mecanismo subyacente a esto implica la modificación de histona H3K36me3 , que puede reclutar proteínas de reparación de desajustes , [126] reduciendo así las tasas de mutación en las regiones marcadas con H3K36me3 . [127] Otro mecanismo importante se refiere a la reparación por escisión de nucleótidos , que puede ser reclutada por la maquinaria de transcripción, reduciendo las tasas de mutación somática en genes activos [125] y otras regiones de cromatina abiertas. [128]
Los daños en el ADN son muy comunes y se reparan constantemente. Las alteraciones epigenéticas pueden acompañar la reparación del ADN de daños oxidativos o roturas de doble cadena. En las células humanas, el daño oxidativo del ADN ocurre unas 10.000 veces al día y las roturas de doble cadena del ADN ocurren unas 10 a 50 veces por ciclo celular en las células somáticas que se replican (véase daño del ADN (de origen natural) ). La ventaja selectiva de la reparación del ADN es permitir que la célula sobreviva frente al daño del ADN. La ventaja selectiva de las alteraciones epigenéticas que ocurren con la reparación del ADN no está clara. [ cita requerida ]
En el estado estable (con daños endógenos que ocurren y se reparan), hay alrededor de 2400 guaninas dañadas oxidativamente que forman 8-oxo-2'-desoxiguanosina (8-OHdG) en el ADN de la célula de mamífero promedio. [129] 8-OHdG constituye alrededor del 5% de los daños oxidativos comúnmente presentes en el ADN. [130] Las guaninas oxidadas no ocurren aleatoriamente entre todas las guaninas en el ADN. Existe una preferencia de secuencia para la guanina en un sitio CpG metilado (una citosina seguida de guanina a lo largo de su dirección 5' → 3' y donde la citosina está metilada (5-mCpG)). [131] Un sitio 5-mCpG tiene el potencial de ionización más bajo para la oxidación de guanina.

La guanina oxidada tiene potencial de producir errores de apareamiento y es mutagénica. [133] La oxoguanina glicosilasa (OGG1) es la enzima principal responsable de la escisión de la guanina oxidada durante la reparación del ADN. La OGG1 encuentra y se une a una 8-OHdG en unos pocos segundos. [134] Sin embargo, la OGG1 no escinde inmediatamente la 8-OHdG. En las células HeLa, la eliminación máxima de la mitad de la 8-OHdG ocurre en 30 minutos, [135] y en ratones irradiados, las 8-OHdG inducidas en el hígado del ratón se eliminan con una vida media de 11 minutos. [130]
Cuando OGG1 está presente en una guanina oxidada dentro de un sitio CpG metilado, recluta a TET1 a la lesión de 8-OHdG (ver Figura). Esto permite que TET1 desmetile una citosina metilada adyacente. [136] La desmetilación de la citosina es una alteración epigenética. [137]
Como ejemplo, cuando las células epiteliales mamarias humanas fueron tratadas con H 2 O 2 durante seis horas, el 8-OHdG aumentó aproximadamente 3,5 veces en el ADN y esto causó aproximadamente un 80% de desmetilación de las 5-metilcitosinas en el genoma. [132] La desmetilación de CpG en un promotor génico por la actividad de la enzima TET aumenta la transcripción del gen en ARN mensajero. [138] En células tratadas con H 2 O 2 , se examinó un gen en particular, BACE1 . [132] El nivel de metilación de la isla CpG BACE1 se redujo (una alteración epigenética) y esto permitió un aumento de aproximadamente 6,5 veces en la expresión del ARN mensajero BACE1 . [ cita requerida ]
Si bien la incubación de seis horas con H2O2 causa una desmetilación considerable de los sitios 5-mCpG, tiempos más cortos de incubación con H2O2 parecen promover otras alteraciones epigenéticas. El tratamiento de células con H2O2 durante 30 minutos hace que el heterodímero de la proteína de reparación de desajustes MSH2-MSH6 reclute a la ADN metiltransferasa 1 (DNMT1) a los sitios de algunos tipos de daño oxidativo del ADN. [139] Esto podría causar un aumento de la metilación de citosinas (alteraciones epigenéticas) en estas ubicaciones.
Jiang et al. [140] trataron células HEK 293 con agentes que causaban daño oxidativo al ADN ( bromato de potasio (KBrO3) o cromato de potasio (K2CrO4)). La reparación por escisión de bases (BER) del daño oxidativo ocurrió con la enzima de reparación del ADN polimerasa beta localizándose en guaninas oxidadas. La polimerasa beta es la principal polimerasa humana en la BER de parche corto del daño oxidativo al ADN. Jiang et al. [140] también encontraron que la polimerasa beta reclutaba la proteína metiltransferasa del ADN DNMT3b a los sitios de reparación de la BER. Luego evaluaron el patrón de metilación a nivel de nucleótido único en una pequeña región del ADN que incluía la región promotora y la región de transcripción temprana del gen BRCA1 . El daño oxidativo al ADN del bromato moduló el patrón de metilación del ADN (causó alteraciones epigenéticas) en los sitios CpG dentro de la región del ADN estudiada. En las células no tratadas, los CpG ubicados en −189, −134, −29, −19, +16 y +19 del gen BRCA1 tenían citosinas metiladas (la numeración corresponde al sitio de inicio de la transcripción del ARN mensajero y los números negativos indican nucleótidos en la región promotora anterior ). La oxidación inducida por el tratamiento con bromato resultó en la pérdida de la metilación de citosinas en −189, −134, +16 y +19, al tiempo que también condujo a la formación de nueva metilación en los CpG ubicados en −80, −55, −21 y +8 después de que se permitió la reparación del ADN. [ cita requerida ]
Al menos cuatro artículos informan sobre el reclutamiento de la ADN metiltransferasa 1 (DNMT1) a los sitios de roturas de doble cadena de ADN. [141] [142] [98] [143] Durante la reparación recombinatoria homóloga (HR) de la rotura de doble cadena, la participación de DNMT1 hace que las dos cadenas reparadas de ADN tengan diferentes niveles de citosinas metiladas. Una cadena se metila con frecuencia en aproximadamente 21 sitios CpG aguas abajo de la rotura de doble cadena reparada. La otra cadena de ADN pierde la metilación en aproximadamente seis sitios CpG que previamente estaban metilados aguas abajo de la rotura de doble cadena, así como también pierde la metilación en aproximadamente cinco sitios CpG que previamente estaban metilados aguas arriba de la rotura de doble cadena. Cuando el cromosoma se replica, esto da lugar a un cromosoma hijo que está muy metilado aguas abajo del sitio de rotura anterior y uno que no está metilado en la región tanto aguas arriba como aguas abajo del sitio de rotura anterior. En lo que respecta al gen que se rompió por la rotura de doble cadena, la mitad de las células de la progenie expresan ese gen en un nivel alto y en la otra mitad de las células de la progenie la expresión de ese gen está reprimida. Cuando los clones de estas células se mantuvieron durante tres años, los nuevos patrones de metilación se mantuvieron durante ese período de tiempo. [144]
En ratones con una inserción de recombinación dirigida por homología mediada por CRISPR en su genoma, hubo una gran cantidad de metilaciones aumentadas de sitios CpG dentro de la inserción asociada a la ruptura de doble cadena. [145]
La reparación de una rotura de doble cadena mediante unión de extremos no homólogos (NHEJ) puede provocar una pequeña cantidad de desmetilaciones de metilaciones de ADN de citosina preexistentes después de la rotura de doble cadena reparada. [142] Trabajos posteriores de Allen et al. [146] demostraron que la NHEJ de una rotura de doble cadena de ADN en una célula podría dar lugar a que algunas células de la progenie tuvieran expresión reprimida del gen que albergaba la rotura de doble cadena inicial y a que algunas células de la progenie tuvieran una expresión alta de ese gen debido a alteraciones epigenéticas asociadas con la reparación mediante NHEJ. La frecuencia de alteraciones epigenéticas que causan la represión de un gen después de una reparación mediante NHEJ de una rotura de doble cadena de ADN en ese gen puede ser de aproximadamente el 0,9 %. [98]
Los procesos básicos de reparación del ADN están altamente conservados tanto entre procariotas como eucariotas e incluso entre bacteriófagos ( virus que infectan bacterias ); sin embargo, los organismos más complejos con genomas más complejos tienen correspondientemente mecanismos de reparación más complejos. [147] La capacidad de un gran número de motivos estructurales de proteínas para catalizar reacciones químicas relevantes ha jugado un papel significativo en la elaboración de mecanismos de reparación durante la evolución. Para una revisión extremadamente detallada de las hipótesis relacionadas con la evolución de la reparación del ADN, véase. [148]
El registro fósil indica que la vida unicelular comenzó a proliferar en el planeta en algún momento durante el período Precámbrico , aunque no está claro exactamente cuándo surgió la vida moderna reconocible. Los ácidos nucleicos se convirtieron en el único y universal medio de codificar la información genética, lo que requirió mecanismos de reparación del ADN que, en su forma básica, han sido heredados por todas las formas de vida existentes de su ancestro común. La aparición de la atmósfera rica en oxígeno de la Tierra (conocida como la " catástrofe del oxígeno ") debido a los organismos fotosintéticos , así como la presencia de radicales libres potencialmente dañinos en la célula debido a la fosforilación oxidativa , requirió la evolución de mecanismos de reparación del ADN que actúan específicamente para contrarrestar los tipos de daño inducidos por el estrés oxidativo . Sin embargo, el mecanismo por el cual esto sucedió no está claro. [ cita requerida ]
En algunas ocasiones, el daño del ADN no se repara o se repara mediante un mecanismo propenso a errores que da como resultado un cambio de la secuencia original. Cuando esto ocurre, las mutaciones pueden propagarse a los genomas de la progenie de la célula. Si tal evento ocurre en una célula de la línea germinal que eventualmente producirá un gameto , la mutación tiene el potencial de transmitirse a la descendencia del organismo. La tasa de evolución en una especie particular (o, en un gen particular) es una función de la tasa de mutación. Como consecuencia, la velocidad y la precisión de los mecanismos de reparación del ADN tienen una influencia sobre el proceso de cambio evolutivo. [149] La protección y reparación del daño del ADN no influye en la tasa de adaptación por regulación genética y por recombinación y selección de alelos. Por otro lado, la reparación y protección del daño del ADN sí influye en la tasa de acumulación de mutaciones irreparables, ventajosas, que expanden el código y son heredables, y ralentiza el mecanismo evolutivo para la expansión del genoma de organismos con nuevas funcionalidades. La tensión entre la capacidad de evolución y la reparación y protección de la mutación necesita más investigación. [ cita requerida ]
En 2012 se descubrió una tecnología denominada repetición palindrómica corta agrupada y regularmente interespaciada (abreviada como CRISPR -Cas9). La nueva tecnología permite a cualquier persona con formación en biología molecular alterar los genes de cualquier especie con precisión, induciendo daño al ADN en un punto específico y luego alterando los mecanismos de reparación del ADN para insertar nuevos genes. [150] Es más barata, más eficiente y más precisa que otras tecnologías. Con la ayuda de CRISPR–Cas9, los científicos pueden editar partes de un genoma eliminando, agregando o alterando partes en una secuencia de ADN. [ cita requerida ]
Si viviéramos lo suficiente, tarde o temprano todos tendríamos cáncer.
las mutaciones nunca se pueden evitar por completo, porque son una consecuencia ineludible de las limitaciones fundamentales en la precisión de la replicación del ADN, como se analiza en el Capítulo 5. Si un ser humano pudiera vivir lo suficiente, es inevitable que al menos una de sus células eventualmente acumulara un conjunto de mutaciones suficientes para que se desarrollara el cáncer.