
Las bacterias gramnegativas son bacterias que, a diferencia de las grampositivas , no retienen la tinción de violeta de cristal utilizada en el método de tinción de Gram de diferenciación bacteriana. [1] Su característica definitoria es su envoltura celular , que consiste en una delgada pared celular de peptidoglicano intercalada entre una membrana interna ( citoplasmática ) y una membrana externa . [2] Estas bacterias se encuentran en todos los entornos que sustentan la vida en la Tierra .
Dentro de esta categoría, las especies notables incluyen el organismo modelo Escherichia coli , junto con varias bacterias patógenas , como Pseudomonas aeruginosa , Chlamydia trachomatis y Yersinia pestis . Plantean desafíos significativos en el campo médico debido a su membrana externa, que actúa como una barrera protectora contra numerosos antibióticos (incluida la penicilina ), detergentes que normalmente dañarían la membrana celular interna y la enzima antimicrobiana lisozima producida por los animales como parte de su sistema inmunológico innato . Además, el folleto externo de esta membrana contiene un lipopolisacárido (LPS) complejo cuyo componente lipídico A puede desencadenar una reacción tóxica cuando las bacterias son lisadas por células inmunes. Esta reacción puede provocar un choque séptico , lo que resulta en presión arterial baja , insuficiencia respiratoria , reducción del suministro de oxígeno y acidosis láctica . [3]
Se han desarrollado varias clases de antibióticos para atacar a las bacterias gramnegativas, entre las que se incluyen aminopenicilinas , ureidopenicilinas , cefalosporinas , combinaciones de beta-lactámicos e inhibidores de betalactamasas (como piperacilina-tazobactam ), antagonistas del folato , quinolonas y carbapenémicos . Muchos de estos antibióticos también atacan a las bacterias grampositivas. Los antibióticos que atacan específicamente a los organismos gramnegativos incluyen aminoglucósidos , monobactámicos (como aztreonam ) y ciprofloxacino .


Las bacterias gramnegativas convencionales (LPS-diderm) presentan las siguientes características : [ cita requerida ]
Junto con la forma celular, la tinción de Gram es una herramienta de diagnóstico rápido y alguna vez se utilizó para agrupar especies en la subdivisión de Bacteria. Históricamente , el reino Monera se dividió en cuatro divisiones basadas en la tinción de Gram: Firmacutes (+), Gracillicutes (−), Mollicutes (0) y Mendocutes (var.). [4] Desde 1987, la monofilia de las bacterias gramnegativas ha sido refutada con estudios moleculares . [5] Sin embargo, algunos autores, como Cavalier-Smith, todavía las tratan como un taxón monofilético (aunque no un clado ; su definición de monofilia requiere un solo ancestro común pero no requiere holofilia , la propiedad de que todos los descendientes estén abarcados por el taxón ) y se refieren al grupo como un subreino "Negibacteria". [6]
Las bacterias se clasifican tradicionalmente en función de su respuesta a la tinción de Gram en bacterias grampositivas y gramnegativas. Al tener una sola membrana, las bacterias grampositivas también se conocen como bacterias monodérmicas , mientras que las bacterias gramnegativas, que tienen dos membranas, también se conocen como bacterias didérmicas . Tradicionalmente se pensaba que los grupos representan linajes, es decir, la membrana adicional solo evolucionó una vez, de modo que las bacterias gramnegativas están más estrechamente relacionadas entre sí que con cualquier bacteria grampositiva. Si bien esto suele ser cierto, el sistema de clasificación falla en algunos casos, y las agrupaciones de linajes no coinciden con el resultado de la tinción. [7] [8] [9] [10] Por lo tanto, la tinción de Gram no se puede utilizar de manera confiable para evaluar las relaciones familiares de las bacterias. Sin embargo, la tinción a menudo brinda información confiable sobre la composición de la membrana celular, distinguiendo entre la presencia o ausencia de una membrana lipídica externa . [7] [11]
De estos dos grupos estructuralmente distintos de organismos procariotas , se cree que los procariotas monodérmicos son ancestrales. Basándose en diversas observaciones, entre ellas que las bacterias grampositivas son las más sensibles a los antibióticos y que las bacterias gramnegativas son, en general, resistentes a los antibióticos, se ha propuesto que la membrana celular externa de las bacterias gramnegativas (didermas) evolucionó como un mecanismo de protección contra la presión de selección de los antibióticos . [7] [8] [11] [12] Algunas bacterias como Deinococcus , que se tiñen de grampositivas debido a la presencia de una gruesa capa de peptidoglicano , pero que también poseen una membrana celular externa, se sugieren como intermediarias en la transición entre bacterias monodérmicas (grampositivas) y didérmicas (gramnegativas). [7] [12] Las bacterias didérmicas también se pueden diferenciar aún más entre didermas simples que carecen de lipopolisacárido (LPS); las bacterias didermicas arquetípicas, en las que la membrana celular externa contiene lipopolisacárido; y las bacterias didermicas en las que la membrana celular externa está formada por ácido micólico (por ejemplo, Mycobacterium ). [9] [10] [12] [13]
El grupo convencional LPS - diderm de bacterias gramnegativas (por ejemplo, Pseudomonadota , Aquificota , Chlamydiota , Bacteroidota , Chlorobiota , " Cyanobacteria ", Fibrobacterota , Verrucomicrobiota , Planctomycetota , Spirochaetota , Acidobacteriota ; " Hydrobacteria ") se identifican de forma única por unas pocas indeles de firma conservadas (CSI) en la proteína HSP60 ( GroEL ). Además, también se ha descubierto que varios taxones bacterianos (incluidos Negativicutes , Fusobacteriota , Synergistota y Elusimicrobiota ) que forman parte del filo Bacillota (un grupo monodermo) o se ramifican en su proximidad poseen una estructura celular diderma. [10] [12] [13] Carecen de la firma GroEL . [12] La presencia de este CSI en todas las especies secuenciadas de filos bacterianos gramnegativos convencionales que contienen lipopolisacáridos proporciona evidencia de que estos filos de bacterias forman un clado monofilético y que no ha ocurrido ninguna pérdida de la membrana externa de ninguna especie de este grupo. [12]
Las proteobacterias son un superfilo importante de bacterias gramnegativas, entre las que se incluyen E. coli , Salmonella , Shigella y otras Enterobacteriaceae , Pseudomonas , Moraxella , Helicobacter , Stenotrophomonas , Bdellovibrio , bacterias del ácido acético , Legionella , etc. Otros grupos notables de bacterias gramnegativas incluyen las cianobacterias , las espiroquetas , las bacterias verdes del azufre y las bacterias verdes no azufradas [14] . [ cita requerida ]
Los diplococos gramnegativos de relevancia médica incluyen los cuatro tipos que causan una enfermedad de transmisión sexual ( Neisseria gonorrhoeae [15] ), una meningitis ( Neisseria meningitidis [16] ) y síntomas respiratorios ( Moraxella catarrhalis , [17]) . Un cocobacilo Haemophilus influenzae es otro tipo de coco de relevancia médica. [18]
Los bacilos gramnegativos de importancia médica incluyen una multitud de especies. Algunas de ellas causan principalmente problemas respiratorios ( Klebsiella pneumoniae , Legionella pneumophila , Pseudomonas aeruginosa ), principalmente problemas urinarios ( Escherichia coli , Proteus mirabilis , Enterobacter cloacae , Serratia marcescens ) y principalmente problemas gastrointestinales ( Helicobacter pylori , Salmonella enteritidis , Salmonella typhi ). [ cita requerida ]
Las bacterias gramnegativas asociadas con infecciones adquiridas en el hospital incluyen Acinetobacter baumannii , que causa bacteriemia , meningitis secundaria y neumonía asociada al respirador en las unidades de cuidados intensivos de los hospitales .
La transformación es uno de los tres procesos de transferencia horizontal de genes , en el que el material genético exógeno pasa de una bacteria a otra; los otros dos son la conjugación (transferencia de material genético entre dos células bacterianas en contacto directo) y la transducción (inyección de ADN extraño por un virus bacteriófago en la bacteria huésped). [19] [20] En la transformación, el material genético pasa a través del medio intermedio y la captación depende completamente de la bacteria receptora. [19]
En 2014 se sabía que alrededor de 80 especies de bacterias eran capaces de transformarse, divididas aproximadamente de manera uniforme entre bacterias grampositivas y gramnegativas; el número podría ser una sobreestimación ya que varios de los informes están respaldados por artículos individuales. [19] La transformación se ha estudiado en especies de bacterias gramnegativas de importancia médica como Helicobacter pylori , Legionella pneumophila , Neisseria meningitidis , Neisseria gonorrhoeae , Haemophilus influenzae y Vibrio cholerae . [21] También se ha estudiado en especies gramnegativas que se encuentran en el suelo, como Pseudomonas stutzeri , Acinetobacter baylyi , y patógenos vegetales gramnegativos como Ralstonia solanacearum y Xylella fastidiosa . [21]
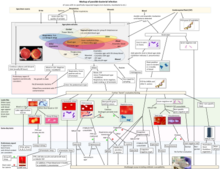
Una de las características únicas de las bacterias gramnegativas es la estructura de la membrana externa bacteriana . La capa externa de esta membrana contiene lipopolisacárido (LPS), cuya porción de lípido A actúa como una endotoxina . [1] Si las bacterias gramnegativas ingresan al sistema circulatorio , el LPS puede desencadenar una respuesta inmune innata , activando el sistema inmunológico y produciendo citocinas (reguladores hormonales). Esto conduce a una inflamación y puede causar una reacción tóxica, resultando en fiebre, aumento de la frecuencia respiratoria y presión arterial baja . Es por eso que algunas infecciones con bacterias gramnegativas pueden provocar un choque séptico potencialmente mortal . [3]
La membrana externa protege a las bacterias de varios antibióticos , colorantes y detergentes que normalmente dañarían la membrana interna o la pared celular (hecha de peptidoglicano ). La membrana externa proporciona a estas bacterias resistencia a la lisozima y la penicilina . El espacio periplásmico (espacio entre las dos membranas celulares) también contiene enzimas que descomponen o modifican los antibióticos. Los medicamentos que se usan comúnmente para tratar las infecciones por gramnegativos incluyen amino, carboxi y ureido penicilinas ( ampicilina , amoxicilina , pipercilina , ticarcilina ). Estos medicamentos se pueden combinar con inhibidores de betalactamasas para combatir la presencia de enzimas que pueden digerir estos medicamentos (conocidas como betalactamasas ) en el espacio periplásmico. Otras clases de fármacos que tienen espectro gramnegativo incluyen cefalosporinas , monobactamas ( aztreonam ), aminoglucósidos, quinolonas , macrólidos , cloranfenicol , antagonistas del folato y carbapenémicos . [22]
Los adjetivos grampositivo y gramnegativo derivan del apellido de Hans Christian Gram , un bacteriólogo danés; como adjetivos epónimos , su letra inicial puede ser G mayúscula o g minúscula , dependiendo de qué guía de estilo (por ejemplo, la de los CDC ), si la hay, rija el documento que se está escribiendo. [23] Esto se explica con más detalle en Tinción de Gram § Nota ortográfica .