Las inserciones y deleciones de firmas conservadas ( CSIs ) en secuencias de proteínas proporcionan una categoría importante de marcadores moleculares para comprender las relaciones filogenéticas. [1] [2] Las CSIs, provocadas por cambios genéticos raros, proporcionan marcadores filogenéticos útiles que generalmente tienen un tamaño definido y están flanqueados en ambos lados por regiones conservadas para garantizar su confiabilidad. Si bien las indels pueden ser inserciones o deleciones arbitrarias, las CSIs se definen solo como aquellas indels de proteínas que están presentes dentro de regiones conservadas de la proteína. [2] [3] [4] [5]
Los CSI que están restringidos a un clado o grupo de especies en particular, generalmente proporcionan buenos marcadores filogenéticos de descendencia evolutiva común. [2] Debido a la rareza y la naturaleza altamente específica de tales cambios, es menos probable que puedan surgir de forma independiente por evolución convergente o paralela (es decir, homoplasia) y, por lo tanto, es probable que representen sinapomorfía . Otros factores de confusión, como las diferencias en las tasas evolutivas en diferentes sitios o entre diferentes especies, generalmente tampoco afectan la interpretación de un CSI. [2] [3] Al determinar la presencia o ausencia de CSI en una especie de grupo externo, se puede inferir si la forma ancestral del CSI fue una inserción o deleción y esto se puede utilizar para desarrollar una relación filogenética arraigada entre organismos. [1] [2]
Los CSI se descubren buscando cambios compartidos en un árbol filogenético construido a partir de secuencias de proteínas. Se ha descubierto que la mayoría de los CSI que se han identificado tienen un alto valor predictivo tras la adición de nuevas secuencias, manteniendo la especificidad para los clados de especies identificados originalmente. Se pueden utilizar para identificar especies conocidas e incluso desconocidas anteriormente que pertenecen a estos grupos en diferentes entornos. [3] En comparación con los órdenes de ramificación de árboles que pueden variar entre métodos, los CSI específicos permiten circunscripciones más concretas que son computacionalmente más baratas de aplicar. [6]

Los CSI específicos de grupo son comúnmente compartidos por diferentes especies que pertenecen a un taxón particular (por ejemplo, género, familia, clase, orden, filo), pero no están presentes en otros grupos. Estos CSI probablemente fueron introducidos en un ancestro del grupo de especies antes de que los miembros del taxón divergieran. Proporcionan medios moleculares para distinguir a los miembros de un taxón particular de todos los demás organismos. [2] [5]
La figura 1 muestra un ejemplo de CSI de 5aa que se encuentra en todas las especies pertenecientes al taxón X. Esta es una característica distintiva de este taxón, ya que no se encuentra en ninguna otra especie. Esta firma probablemente se introdujo en un ancestro común de la especie de este taxón. De manera similar, otras firmas específicas de grupo (no se muestran) podrían ser compartidas por A1 y A2 o B1 y B2, etc., o incluso por X1 y X2 o por X3 y X4, etc. Los grupos A, B, C, D y X, en este diagrama, podrían corresponder a varios filos bacterianos o eucariotas . [7]
En el pasado, se han utilizado CSI específicos de grupo para determinar la relación filogenética de varios filos bacterianos y subgrupos dentro de ellos. Por ejemplo, un inserto de 3 aminoácidos fue compartido de manera única por miembros del filo Thermotogota (anteriormente Thermotogae) en la proteína ribosomal esencial 50S L7/L12 , dentro de una región altamente conservada (82-124 aminoácidos). Esto no está presente en ninguna otra especie de bacteria y podría usarse para caracterizar a los miembros de Thermotogota de todas las demás bacterias. Los CSI específicos de grupo también se utilizaron para caracterizar subgrupos dentro de Thermotogota. [8]

Los CSI principales son aquellos en los que una inserción o deleción conservada es compartida por varios filos principales, pero está ausente en otros filos. [2]
La figura 2 muestra un ejemplo de CSI de 5aa encontrado en una región conservada que está presente comúnmente en las especies pertenecientes a los filos X, Y y Z, pero está ausente en otros filos (A, B y C). Esta firma indica una relación específica de los taxones X, Y y Z y también A, B y C. Con base en la presencia o ausencia de tal indel, en especies de grupos externos (por ejemplo, Archaea), se puede inferir si el indel es una inserción o una deleción, y cuál de estos dos grupos A, B, C o X, Y, Z es ancestral. [7]
Los CSI de línea principal se han utilizado en el pasado para determinar la relación filogenética de varios filos bacterianos. El gran CSI de alrededor de 150-180 aminoácidos dentro de una región conservada de girasa B (entre los aminoácidos 529-751), es compartido comúnmente entre varias especies de Pseudomonadota , Chlamydiota , Planctomycetota y Aquificota . Este CSI está ausente en otros filos bacterianos ancestrales, así como en Archaea . [9] De manera similar, un gran CSI de alrededor de 100 aminoácidos en homólogos de RpoB (entre los aminoácidos 919-1058) está presente en varias especies pertenecientes a Pseudomonadota, Bacteroidota , Chlorobiota , Chlamydiota , Planctomycetota y Aquificota. Este CSI está ausente en otros filos bacterianos ancestrales, así como en Archaea. [10] [11] En ambos casos se puede inferir que los grupos que carecen del CSI son ancestrales.


Una cuestión clave en la filogenia bacteriana es comprender cómo se relacionan entre sí las diferentes especies bacterianas y su orden de ramificación a partir de un ancestro común. Actualmente, la mayoría de los árboles filogenéticos se basan en el ARNr 16S u otros genes/proteínas. Estos árboles no siempre son capaces de resolver cuestiones filogenéticas clave con un alto grado de certeza. [12] [13] [14] [15] [16] Sin embargo, en los últimos años, el descubrimiento y análisis de indeles conservados (CSI) en muchas proteínas distribuidas universalmente han ayudado en esta búsqueda. Se postula que los eventos genéticos que conducen a ellos ocurrieron en puntos de ramificación evolutiva importantes y sus patrones de distribución de especies brindan información valiosa sobre el orden de ramificación y las interrelaciones entre diferentes filos bacterianos. [1] [2] [8]
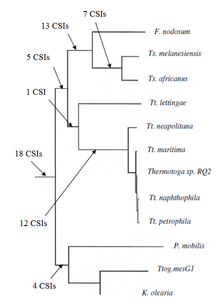
Recientemente se ha caracterizado la relación filogenética del grupo Thermotogota con base en el enfoque CSI. Hasta ahora no se conocían marcadores bioquímicos o moleculares que pudieran distinguir claramente las especies de este filo de todas las demás bacterias. Se descubrieron más de 60 CSI que eran específicos para todo el filo Thermotogota o sus diferentes subgrupos. De estos, 18 CSI están presentes de forma única en varias especies de Thermotogota y proporcionan marcadores moleculares para el filo. Además, había muchos CSI que eran específicos para varios subgrupos de Thermotogota. Otros 12 CSI eran específicos para un clado que consta de varias especies de Thermotogota excepto Tt. Lettingae. Mientras que 14 CSI eran específicos para un clado que consta de los géneros Fervidobacterium y Thermosipho y 18 CSI eran específicos para el género Thermosiphon . [ cita requerida ]
Por último, se informaron 16 CSI que fueron compartidos por algunas o todas las especies de Thermotogota o algunas especies de otros taxones como Archaea , Aquificota , Bacillota , Pseudomonadota , Deinococcota , Fusobacteriota , Dictyoglomota , Chloroflexota y eucariotas . La presencia compartida de algunos de estos CSI podría deberse a la transferencia lateral de genes (LGT) entre estos grupos. Sin embargo, el número de CSI que se comparten comúnmente con otros taxones es mucho menor que los que son específicos de Thermotogota y no exhiben ningún patrón específico. Por lo tanto, no tienen un efecto significativo en la distinción de Thermotogota. [8]
Recientemente, los Thermoproteota mesófilos se han incluido en un nuevo filo de arqueas llamado Nitrososphaerota (anteriormente Thaumarchaeota). Sin embargo, hay muy pocos marcadores moleculares que puedan distinguir a este grupo de arqueas del filo Thermoproteota (anteriormente Crenarchaeota). Se realizó un estudio filogenético detallado utilizando el enfoque CSI para distinguir estos filos en términos moleculares. Se encontraron 6 CSI de forma única en varios Nitrososphaerota, a saber, Cenarchaeum symbiosum , Nitrosopumilus maritimus y varios Thermoproteota marinos no cultivados. Se encontraron 3 CSI que eran comúnmente compartidos entre especies pertenecientes a Nitrososphaerota y Thermoproteota. Además, se encontraron varios CSI que son específicos para diferentes órdenes de Thermoproteota: 3 CSI para Sulfolobales , 5 CSI para Thermoproteales y, por último, 2 CSI comunes para Sulfolobales y Desulfurococcales . Las firmas descritas proporcionan nuevos medios para distinguir Thermoproteota y Nitrososphaerota, además podrían utilizarse como una herramienta para la clasificación e identificación de especies relacionadas. [17]
Los miembros del orden Pasteurellales se distinguen actualmente principalmente en función de su posición en la ramificación del árbol 16srRNA. Actualmente se conocen muy pocos marcadores moleculares que puedan distinguir a los miembros de este orden de otras bacterias. Recientemente se utilizó un enfoque CSI para dilucidar las relaciones filogenéticas entre las especies de este orden; se descubrieron más de 40 CSI que eran compartidos de forma única por todas o la mayoría de las especies. Dentro de este Pasteurellales se forman dos clados principales: el clado I, que abarca Aggregatibacter , Pasteurella , Actinobacillus succinogenes , Mannheimia succiniciproducens , Haemophilus influenzae y Haemophilus somnus , fue respaldado por 13 CSI. El clado II, que abarca Actinobacillus pleuropneumoniae , Actinobacillus minor , Haemophilus ducreyi , Mannheimia haemolytica y Haemophilus parasuis , fue respaldado por 9 CSI. Con base en estos resultados, se propuso dividir Pasteurellales de su familia actual en dos familias diferentes. Además, las firmas descritas proporcionarían nuevos medios para identificar especies de Pasteurellales no descubiertas. [18]
La clase Gammaproteobacteria forma uno de los grupos más grandes de bacterias. Actualmente se distingue de otras bacterias únicamente por árboles filogenéticos basados en ARNr 16s . No se conocen características moleculares exclusivas de la clase o sus diferentes subgrupos. Se realizó un estudio detallado basado en CSI para comprender mejor la filogenia de esta clase. En primer lugar, se creó un árbol filogenético basado en secuencias concatenadas de varias proteínas distribuidas universalmente. El orden de ramificación de los diferentes órdenes de la clase Gammaproteobacteria (desde el más reciente hasta el más temprano en divergencia) fue: Enterobacteriales > Pasteurellales > Vibrionales , Aeromonadales > Alteromonadales > Oceanospirillales , Pseudomonadales > Chromatiales, Legionellales , Methylococcales , Xanthomonadales , Cardiobacteriales , Thiotrichales . Además, se descubrieron 4 CSI que eran exclusivos de la mayoría de las especies de la clase Gammaproteobacteria. Una deleción de 2 aa en la transformilasa AICAR fue compartida de forma única por todas las gammaproteobacterias excepto por Francisella tularensis . Una deleción de 4 aa en la subunidad b de la ARN polimerasa y una deleción de 1 aa en la proteína ribosomal L16 se encontraron de forma única en varias especies pertenecientes a los órdenes Enterobacteriales, Pasteurellales, Vibrionales, Aeromonadales y Alteromonadales, pero no se encontraron en otras gammaproteobacterias. Por último, una deleción de 2 aa en la leucil-ARNt sintetasa estuvo presente comúnmente en los órdenes anteriores de la clase Gammaproteobacteria y en algunos miembros del orden Oceanospirillales. [19] Otro estudio basado en CSI también ha identificado 4 CSI que son exclusivos del orden Xanthomonadales. En conjunto, estos dos hechos muestran que Xanthomonadales es un grupo monofilético ancestral de otras Gammaproteobacteria, lo que demuestra además que Xanthomonadales es una subdivisión independiente y constituye uno de los linajes de ramificación más profunda dentro del clado Gammaproteobacteria. [4] [19]
{{cite web}}: CS1 maint: bot: estado de URL original desconocido ( enlace )