Una proteasa (también llamada peptidasa , proteinasa o enzima proteolítica ) [1] es una enzima que cataliza la proteólisis , descomponiendo las proteínas en polipéptidos más pequeños o aminoácidos individuales y estimulando la formación de nuevos productos proteicos. [2] Lo hacen escindiendo los enlaces peptídicos dentro de las proteínas por hidrólisis , una reacción en la que el agua rompe los enlaces . Las proteasas están involucradas en numerosas vías biológicas, incluida la digestión de proteínas ingeridas, el catabolismo proteico (descomposición de proteínas viejas), [3] [4] y la señalización celular .
En ausencia de aceleradores funcionales, la proteólisis sería muy lenta y tardaría cientos de años . [5] Las proteasas se pueden encontrar en todas las formas de vida y en los virus . Han evolucionado de forma independiente varias veces y diferentes clases de proteasas pueden realizar la misma reacción mediante mecanismos catalíticos completamente diferentes .
Las proteasas se pueden clasificar en siete grandes grupos: [6]
Las proteasas se agruparon por primera vez en 84 familias según su relación evolutiva en 1993, y se clasificaron en cuatro tipos catalíticos: serina , cisteína , aspártico y metaloproteasas . [7] Las proteasas de treonina y glutámico no se describieron hasta 1995 y 2004 respectivamente. El mecanismo utilizado para escindir un enlace peptídico implica hacer que un residuo de aminoácido que tenga la cisteína y la treonina (proteasas) o una molécula de agua (aspártico, glutámico y metaloproteasas) sea nucleófilo para que pueda atacar al grupo carbonilo del péptido . Una forma de hacer un nucleófilo es mediante una tríada catalítica , donde se utiliza un residuo de histidina para activar la serina , la cisteína o la treonina como nucleófilo. Sin embargo, no se trata de una agrupación evolutiva, ya que los tipos de nucleófilos han evolucionado de manera convergente en diferentes superfamilias , y algunas superfamilias muestran una evolución divergente hacia múltiples nucleófilos diferentes. Las metaloproteasas, las proteasas aspárticas y glutámicas utilizan los residuos de su sitio activo para activar una molécula de agua, que luego ataca el enlace escindible. [8]
En 2011 se describió un séptimo tipo catalítico de enzimas proteolíticas, la asparagina péptido liasa . Su mecanismo proteolítico es inusual ya que, en lugar de hidrólisis , realiza una reacción de eliminación . [9] Durante esta reacción, la asparagina catalítica forma una estructura química cíclica que se escinde a sí misma en los residuos de asparagina en las proteínas en las condiciones adecuadas. Dado su mecanismo fundamentalmente diferente, su inclusión como peptidasa puede ser discutible. [9]
Una clasificación actualizada de las superfamilias evolutivas de proteasas se encuentra en la base de datos MEROPS. [10] En esta base de datos, las proteasas se clasifican en primer lugar por "clan" ( superfamilia ) según la estructura, el mecanismo y el orden de los residuos catalíticos (por ejemplo, el clan PA donde P indica una mezcla de familias de nucleófilos). Dentro de cada "clan", las proteasas se clasifican en familias según la similitud de secuencia (por ejemplo, las familias S1 y C3 dentro del clan PA). Cada familia puede contener cientos de proteasas relacionadas (por ejemplo, tripsina , elastasa , trombina y estreptogrisina dentro de la familia S1).
Actualmente se conocen más de 50 clanes, cada uno de los cuales indica un origen evolutivo independiente de la proteólisis. [10]
Alternativamente, las proteasas pueden clasificarse según el pH óptimo en el que son activas:
Las proteasas intervienen en la digestión de cadenas proteicas largas en fragmentos más cortos mediante la división de los enlaces peptídicos que unen los residuos de aminoácidos . Algunas separan los aminoácidos terminales de la cadena proteica ( exopeptidasas , como las aminopeptidasas , la carboxipeptidasa A ); otras atacan los enlaces peptídicos internos de una proteína ( endopeptidasas , como la tripsina , la quimotripsina , la pepsina , la papaína , la elastasa ).
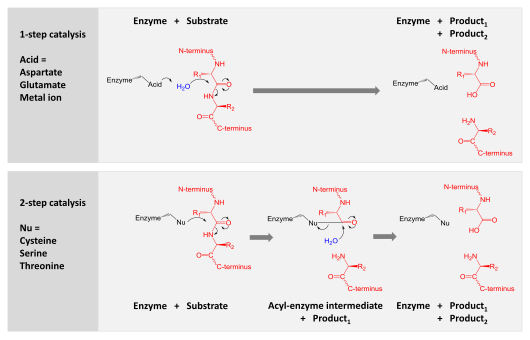
La catálisis se consigue mediante uno de dos mecanismos:
La proteólisis puede ser muy promiscua , de modo que se hidroliza una amplia gama de sustratos proteicos. Este es el caso de las enzimas digestivas como la tripsina , que deben ser capaces de escindir el conjunto de proteínas ingeridas en fragmentos peptídicos más pequeños. Las proteasas promiscuas normalmente se unen a un solo aminoácido en el sustrato y, por lo tanto, solo tienen especificidad para ese residuo. Por ejemplo, la tripsina es específica para las secuencias ...K\... o ...R\... ('\'= sitio de escisión). [12]
Por el contrario, algunas proteasas son muy específicas y solo escinden sustratos con una secuencia determinada. La coagulación sanguínea (como la trombina ) y el procesamiento de poliproteínas virales (como la proteasa TEV ) requieren este nivel de especificidad para lograr eventos de escisión precisos. Esto se logra mediante proteasas que tienen una hendidura o túnel de unión largo con varios bolsillos que se unen a residuos específicos. Por ejemplo, la proteasa TEV es específica para la secuencia ...ENLYFQ\S... ('\'=sitio de escisión). [13]
Las proteasas, al ser proteínas en sí mismas, son escindidas por otras moléculas de proteasa, a veces de la misma variedad. Esto actúa como un método de regulación de la actividad de la proteasa. Algunas proteasas son menos activas después de la autólisis (p. ej. , la proteasa TEV ), mientras que otras son más activas (p. ej., el tripsinógeno ).
Las proteasas se encuentran en todos los organismos, desde procariotas hasta eucariotas y virus . Estas enzimas están involucradas en una multitud de reacciones fisiológicas, desde la digestión simple de proteínas de los alimentos hasta cascadas altamente reguladas (por ejemplo, la cascada de coagulación sanguínea , el sistema del complemento , las vías de apoptosis y la cascada de activación de la profenoloxidasa de invertebrados). Las proteasas pueden romper enlaces peptídicos específicos ( proteólisis limitada ), dependiendo de la secuencia de aminoácidos de una proteína, o descomponer completamente un péptido en aminoácidos ( proteólisis ilimitada ). La actividad puede ser un cambio destructivo (eliminación de la función de una proteína o digestión de la misma hasta sus componentes principales), puede ser una activación de una función o puede ser una señal en una vía de señalización.
Los genomas de las plantas codifican cientos de proteasas, en su mayoría de función desconocida. Las que tienen una función conocida están involucradas en gran medida en la regulación del desarrollo . [14] Las proteasas vegetales también desempeñan un papel en la regulación de la fotosíntesis . [15]
Las proteasas se utilizan en todo el organismo para diversos procesos metabólicos. Las proteasas ácidas secretadas en el estómago (como la pepsina ) y las serina proteasas presentes en el duodeno ( tripsina y quimotripsina ) nos permiten digerir la proteína de los alimentos. Las proteasas presentes en el suero sanguíneo ( trombina , plasmina , factor de Hageman , etc.) desempeñan un papel importante en la coagulación sanguínea, así como en la lisis de los coágulos y en el correcto funcionamiento del sistema inmunitario. Otras proteasas están presentes en los leucocitos ( elastasa , catepsina G ) y desempeñan varias funciones diferentes en el control metabólico. Algunos venenos de serpientes también son proteasas, como la hemotoxina de la víbora de foseta , e interfieren en la cascada de coagulación sanguínea de la víctima. Las proteasas determinan la vida útil de otras proteínas que desempeñan importantes funciones fisiológicas, como las hormonas, los anticuerpos u otras enzimas. Este es uno de los mecanismos reguladores de “activación” y “desactivación” más rápidos en la fisiología de un organismo.
Mediante una acción cooperativa compleja, las proteasas pueden catalizar reacciones en cascada , que dan como resultado una amplificación rápida y eficiente de la respuesta de un organismo a una señal fisiológica.
Las bacterias secretan proteasas para hidrolizar los enlaces peptídicos en las proteínas y, por lo tanto, descomponerlas en sus aminoácidos constituyentes . Las proteasas bacterianas y fúngicas son particularmente importantes para los ciclos globales de carbono y nitrógeno en el reciclaje de proteínas, y dicha actividad tiende a estar regulada por señales nutricionales en estos organismos. [16] El impacto neto de la regulación nutricional de la actividad de las proteasas entre las miles de especies presentes en el suelo se puede observar a nivel de la comunidad microbiana general a medida que las proteínas se descomponen en respuesta a la limitación de carbono, nitrógeno o azufre. [17]
Las bacterias contienen proteasas responsables del control general de la calidad de las proteínas (por ejemplo, el proteasoma AAA+ ) al degradar proteínas desdobladas o mal plegadas .
Una proteasa bacteriana secretada también puede actuar como exotoxina y ser un ejemplo de factor de virulencia en la patogénesis bacteriana (por ejemplo, toxina exfoliativa ). Las proteasas exotóxicas bacterianas destruyen las estructuras extracelulares.
Los genomas de algunos virus codifican una poliproteína masiva , que necesita una proteasa para escindirla en unidades funcionales (por ejemplo, el virus de la hepatitis C y los picornavirus ). [18] Estas proteasas (por ejemplo, la proteasa TEV ) tienen una alta especificidad y solo escinden un conjunto muy restringido de secuencias de sustrato. Por lo tanto, son un objetivo común para los inhibidores de proteasas . [19] [20]
Las arqueas utilizan proteasas para regular diversos procesos celulares, desde la señalización celular , el metabolismo , la secreción y el control de calidad de las proteínas. [21] [22] Solo se encuentran dos proteasas dependientes de ATP en las arqueas: la proteasa LonB asociada a la membrana y un complejo de proteosoma 20S soluble . [21]
El campo de investigación de las proteasas es enorme. Desde 2004, se han publicado aproximadamente 8000 artículos relacionados con este campo cada año. [23] Las proteasas se utilizan en la industria, la medicina y como herramienta básica de investigación biológica. [24] [25]
Las proteasas digestivas forman parte de muchos detergentes para ropa y también se utilizan ampliamente en la industria del pan como mejorador del pan . Una variedad de proteasas se utilizan médicamente tanto por su función nativa (por ejemplo, controlar la coagulación sanguínea) como por funciones completamente artificiales ( por ejemplo, para la degradación dirigida de proteínas patógenas). Las proteasas altamente específicas, como la proteasa TEV y la trombina, se utilizan comúnmente para escindir proteínas de fusión y etiquetas de afinidad de manera controlada. Las soluciones vegetales que contienen proteasas llamadas cuajo vegetariano se han utilizado durante cientos de años en Europa y Oriente Medio para hacer quesos kosher y halal . El cuajo vegetariano de Withania coagulans se ha utilizado durante miles de años como remedio ayurvédico para la digestión y la diabetes en el subcontinente indio. También se utiliza para hacer Paneer .
La actividad de las proteasas es inhibida por los inhibidores de proteasas . [26] Un ejemplo de inhibidores de proteasas es la superfamilia de las serpinas . Incluye la alfa 1-antitripsina (que protege al cuerpo de los efectos excesivos de sus propias proteasas inflamatorias ), la alfa 1-antiquimotripsina (que hace lo mismo), el inhibidor de C1 (que protege al cuerpo de la activación excesiva desencadenada por proteasas de su propio sistema de complemento ), la antitrombina (que protege al cuerpo de la coagulación excesiva ), el inhibidor del activador del plasminógeno-1 (que protege al cuerpo de la coagulación inadecuada al bloquear la fibrinólisis desencadenada por proteasas ) y la neuroserpina . [27]
Los inhibidores de proteasa naturales incluyen la familia de proteínas lipocalinas , que desempeñan un papel en la regulación y diferenciación celular. Se ha descubierto que los ligandos lipofílicos , unidos a las proteínas lipocalinas, poseen propiedades inhibidoras de proteasas tumorales. Los inhibidores de proteasa naturales no deben confundirse con los inhibidores de proteasa utilizados en la terapia antirretroviral. Algunos virus , entre ellos el VIH/SIDA , dependen de las proteasas en su ciclo reproductivo. Por ello, los inhibidores de proteasa se desarrollan como agentes terapéuticos antivirales .
Otros inhibidores naturales de la proteasa se utilizan como mecanismos de defensa. Ejemplos comunes son los inhibidores de tripsina que se encuentran en las semillas de algunas plantas, la más conocida para los humanos es la soja, un importante cultivo alimentario, donde actúan para disuadir a los depredadores. La soja cruda es tóxica para muchos animales, incluidos los humanos, hasta que se desnaturalizan los inhibidores de la proteasa que contiene.
Evaluar las competencias relativas de las enzimas que catalizan la hidrólisis de enlaces peptídicos internos y C-terminales [...]