Las serina proteasas (o serina endopeptidasas ) son enzimas que rompen los enlaces peptídicos en las proteínas . La serina actúa como aminoácido nucleofílico en el sitio activo de la enzima . [1] Se encuentran en todas partes, tanto en eucariotas como en procariotas . Las serina proteasas se dividen en dos grandes categorías según su estructura: similares a la quimotripsina (similares a la tripsina) o similares a la subtilisina . [2]
El sistema de clasificación de proteasas MEROPS cuenta con 16 superfamilias (a partir de 2013), cada una de las cuales contiene muchas familias . Cada superfamilia utiliza la tríada o díada catalítica en un pliegue proteico diferente y, por lo tanto, representa una evolución convergente del mecanismo catalítico . La mayoría pertenece a la familia S1 del clan PA (superfamilia) de proteasas.
Para las superfamilias , P: superfamilia, que contiene una mezcla de familias de clases de nucleófilos , S: serina proteasas puras. superfamilia. Dentro de cada superfamilia, las familias se designan por su nucleófilo catalítico, (S: serina proteasas).

Las serina proteasas se caracterizan por una estructura distintiva, que consta de dos dominios de barril beta que convergen en el sitio catalítico activo. Estas enzimas se pueden clasificar además en función de su especificidad de sustrato como similares a la tripsina, similares a la quimotripsina o similares a la elastasa. [5]
Las proteasas similares a la tripsina rompen los enlaces peptídicos que siguen a un aminoácido con carga positiva ( lisina o arginina ). [6] Esta especificidad está impulsada por el residuo que se encuentra en la base del bolsillo S1 de la enzima (generalmente un ácido aspártico o ácido glutámico con carga negativa ).
El bolsillo S1 de las enzimas similares a la quimotripsina es más hidrófobo que el de las proteasas similares a la tripsina. Esto da como resultado una especificidad para residuos hidrófobos de tamaño mediano a grande, como la tirosina , la fenilalanina y el triptófano .
Entre ellos se encuentran la trombina , el plasminógeno activador de tejidos y la plasmina . Se ha descubierto que desempeñan funciones en la coagulación y la digestión, así como en la fisiopatología de trastornos neurodegenerativos como la demencia inducida por Alzheimer y Parkinson. En los venenos de serpiente se encuentran muchas isoformas de serina proteasas similares a la trombina altamente tóxicas. [7]
Las proteasas similares a la elastasa tienen una hendidura S1 mucho más pequeña que las proteasas similares a la tripsina o la quimotripsina. En consecuencia, tienden a preferirse residuos como la alanina , la glicina y la valina .
La subtilisina es una proteasa de serina en procariotas . La subtilisina no está relacionada evolutivamente con el clan de la quimotripsina, pero comparte el mismo mecanismo catalítico que utiliza una tríada catalítica para crear una serina nucleófila . Este es el ejemplo clásico utilizado para ilustrar la evolución convergente , ya que el mismo mecanismo evolucionó dos veces de forma independiente durante la evolución .
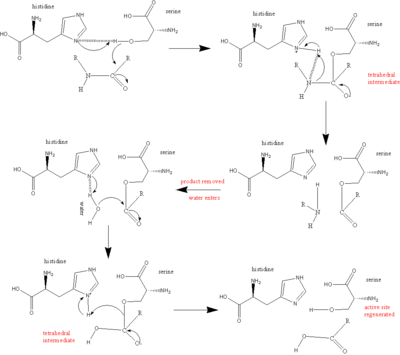
El principal actor del mecanismo catalítico de las serina proteasas es la tríada catalítica. La tríada se encuentra en el sitio activo de la enzima, donde se produce la catálisis, y se conserva en todas las superfamilias de enzimas serina proteasas. La tríada es una estructura coordinada que consta de tres aminoácidos : His 57, Ser 195 (de ahí el nombre de "serina proteasa") y Asp 102. Estos tres aminoácidos clave desempeñan un papel esencial en la capacidad de escisión de las proteasas. Si bien los aminoácidos miembros de la tríada se encuentran lejos unos de otros en la secuencia de la proteína, debido al plegamiento, estarán muy cerca unos de otros en el corazón de la enzima. La geometría particular de los miembros de la tríada es muy característica de su función específica: se demostró que la posición de solo cuatro puntos de la tríada caracteriza la función de la enzima que los contiene. [8]
En el caso de la catálisis, se produce un mecanismo ordenado en el que se generan varios intermediarios. La catálisis de la escisión del péptido puede verse como una catálisis de ping-pong , en la que se une un sustrato (en este caso, el polipéptido que se está escindiendo), se libera un producto (la "mitad" del extremo C del péptido con el grupo amino visible), se une otro sustrato (en este caso, agua) y se libera otro producto (la "mitad" del extremo N del péptido con el grupo carboxilo visible).
Cada aminoácido de la tríada realiza una tarea específica en este proceso:
La reacción completa se puede resumir de la siguiente manera:
Se descubrió que aminoácidos adicionales de la proteasa, Gly 193 y Ser 195 , están involucrados en la creación de lo que se llama un agujero de oxianión . Tanto Gly 193 como Ser 195 pueden donar hidrógenos de la cadena principal para la formación de puentes de hidrógeno. Cuando se genera el intermedio tetraédrico del paso 1 y el paso 3, el ion de oxígeno negativo, habiendo aceptado los electrones del doble enlace carbonílico , encaja perfectamente en el agujero de oxianión. En efecto, las serina proteasas se unen preferentemente al estado de transición y se favorece la estructura general, lo que reduce la energía de activación de la reacción. Esta "unión preferencial" es responsable de gran parte de la eficiencia catalítica de la enzima.
Los organismos hospedadores deben garantizar que la actividad de las serina proteasas esté adecuadamente regulada, lo que se logra mediante la activación inicial de las proteasas y la secreción de inhibidores.
Los zimógenos son los precursores normalmente inactivos de una enzima. Si las enzimas digestivas estuvieran activas cuando se sintetizan, comenzarían inmediatamente a devorar los órganos y tejidos que las sintetizan. La pancreatitis aguda es una afección en la que se produce una activación prematura de las enzimas digestivas en el páncreas, lo que da lugar a una autodigestión (autólisis). También complica las investigaciones post mortem , ya que el páncreas a menudo se digiere a sí mismo antes de que pueda evaluarse visualmente.
Los zimógenos son estructuras grandes e inactivas que tienen la capacidad de desintegrarse o transformarse en enzimas activadas más pequeñas. La diferencia entre los zimógenos y las enzimas activadas radica en que el sitio activo para la catálisis de los zimógenos está distorsionado. Como resultado, el polipéptido sustrato no puede unirse de manera efectiva y no se produce la proteólisis . Solo después de la activación, durante la cual la conformación y la estructura del zimógeno cambian y el sitio activo se abre, puede ocurrir la proteólisis .
Como se puede observar, la activación del tripsinógeno a tripsina es esencial, ya que activa su propia reacción, así como la reacción tanto de la quimotripsina como de la elastasa . Por lo tanto, es esencial que esta activación no se produzca de forma prematura. Existen varias medidas de protección que toma el organismo para evitar la autodigestión:
Existen ciertos inhibidores que se asemejan al intermediario tetraédrico y, por lo tanto, llenan el sitio activo, impidiendo que la enzima funcione correctamente. La tripsina, una enzima digestiva potente, se genera en el páncreas. Los inhibidores impiden la autodigestión del páncreas.
Las serina proteasas se combinan con inhibidores de serina proteasas , que desactivan su actividad cuando ya no son necesarios. [9] [ fuente autopublicada ? ]
Las serina proteasas son inhibidas por un grupo diverso de inhibidores , incluidos inhibidores químicos sintéticos con fines de investigación o terapéuticos, y también inhibidores proteínicos naturales. Una familia de inhibidores naturales llamados "serpinas" (abreviatura de inhibidores de serina proteasa ) pueden formar un enlace covalente con la serina proteasa, inhibiendo su función. Las serpinas mejor estudiadas son la antitrombina y la alfa 1-antitripsina , estudiadas por su papel en la coagulación / trombosis y enfisema / A1AT , respectivamente. Los inhibidores artificiales irreversibles de moléculas pequeñas incluyen AEBSF y PMSF .
Se ha identificado una familia de inhibidores de la serina peptidasa de los artrópodos , llamada pacifastina , en langostas y cangrejos de río , y puede funcionar en el sistema inmunológico de los artrópodos . [10]
Las mutaciones pueden provocar una disminución o un aumento de la actividad de las enzimas. Esto puede tener diferentes consecuencias, dependiendo del funcionamiento normal de la serina proteasa. Por ejemplo, las mutaciones en la proteína C pueden provocar una deficiencia de proteína C y predisponer a la trombosis . Además, algunas proteasas desempeñan un papel vital en la activación de la fusión entre la célula huésped y el virus al preparar la proteína Spike del virus para que muestre la proteína denominada "proteína de fusión" ( TMPRSS2 activa la fusión del SARS-CoV-2 ). Las serina proteasas exógenas del veneno de serpiente causan una amplia gama de coagulopatías cuando se inyectan en un huésped debido a la falta de regulación de su actividad. [7]
La determinación de los niveles de serina proteasa puede ser útil en el contexto de enfermedades particulares.
Debido a su actividad catalítica, algunas proteasas de serina poseen potentes propiedades antimicrobianas. Varios estudios in vitro han demostrado la eficacia de algunas proteasas para reducir la virulencia al escindir las proteínas de superficie virales. La entrada viral en las células huésped está mediada por la interacción de estas proteínas de superficie con la célula huésped. Cuando estas proteínas se fragmentan o se inactivan en la superficie viral, la entrada viral se ve afectada, lo que conduce a una reducción de la infectividad de un amplio espectro de microorganismos patológicamente relevantes como la influenza , hRSV y otros. [11] [12]