Una proteína quinasa activada por mitógenos ( MAPK o MAP quinasa ) es un tipo de proteína quinasa específica de serina/treonina que participa en la dirección de las respuestas celulares a una amplia gama de estímulos, como mitógenos , estrés osmótico , choque térmico y citocinas proinflamatorias . Regulan funciones celulares, incluida la proliferación , la expresión genética , la diferenciación , la mitosis , la supervivencia celular y la apoptosis . [1]
Las quinasas MAP se encuentran sólo en eucariotas , pero son bastante diversas y se encuentran en todos los animales, hongos y plantas, e incluso en una variedad de eucariotas unicelulares. [ cita requerida ]
Las MAPK pertenecen al grupo de las quinasas CMGC (CDK/MAPK/GSK3/CLK). Los parientes más cercanos de las MAPK son las quinasas dependientes de ciclina (CDK). [2]
La primera proteína quinasa activada por mitógenos que se descubrió fue ERK1 ( MAPK3 ) en mamíferos. Dado que ERK1 y su pariente cercano ERK2 ( MAPK1 ) están involucrados en la señalización del factor de crecimiento, la familia se denominó "activada por mitógenos". Con el descubrimiento de otros miembros, incluso de organismos distantes (por ejemplo, plantas), se ha vuelto cada vez más claro que el nombre es un nombre inapropiado, ya que la mayoría de las MAPK están realmente involucradas en la respuesta a estímulos de estrés abiótico potencialmente dañinos (hiperosmosis, estrés oxidativo, daño del ADN, baja osmolaridad, infección, etc.). Debido a que las plantas no pueden "huir" del estrés, las plantas terrestres tienen el mayor número de genes MAPK por organismo jamás encontrado [ cita requerida ] . Por lo tanto, el papel de las quinasas ERK1/2 de mamíferos como reguladores de la proliferación celular no es una función genérica, sino altamente especializada.
La mayoría de las MAPK comparten una serie de características, como la activación dependiente de dos eventos de fosforilación , una arquitectura de vías de tres niveles y sitios de reconocimiento de sustratos similares. Estas son las quinasas MAP "clásicas". Pero también hay algunas MAPK atípicas antiguas del grupo, como se describió anteriormente, que no tienen sitios de fosforilación dual, solo forman vías de dos niveles y carecen de las características requeridas por otras MAPK para la unión del sustrato. Por lo general, se las denomina MAPK "atípicas". [3] Todavía no está claro si las MAPK atípicas forman un solo grupo en oposición a las clásicas. [ Se necesita aclaración ]
La familia de quinasas MAPK de mamíferos incluye tres subfamilias:
Generalmente, las ERK son activadas por factores de crecimiento y mitógenos , mientras que el estrés celular y las citocinas inflamatorias activan las JNK y las p38. [4]

Las proteínas quinasas activadas por mitógenos son catalíticamente inactivas en su forma básica. Para activarse, requieren (potencialmente múltiples) eventos de fosforilación en sus bucles de activación. Esto lo llevan a cabo enzimas especializadas del grupo de las proteínas quinasas STE. De esta manera, la dinámica de las proteínas puede inducir un cambio conformacional en la estructura de la proteína a través de la alosteria de largo alcance .
En el caso de las quinasas MAP clásicas, el bucle de activación contiene un motivo característico TxY (treonina-x-tirosina) (TEY en ERK1 y ERK2 de mamíferos , TDY en ERK5 , TPY en JNK , TGY en quinasas p38 ) que necesita ser fosforilado tanto en los residuos de treonina como de tirosina para bloquear el dominio de la quinasa en una conformación catalíticamente competente. In vivo e in vitro , la fosforilación de la tirosina a menudo precede a la fosforilación de la treonina, aunque la fosforilación de cualquiera de los residuos puede ocurrir en ausencia del otro. [ cita requerida ]
Esta fosforilación del bucle de activación en tándem (que se propuso que era distributiva o procesiva, dependiendo del entorno celular) es realizada por miembros de la familia de las proteínas quinasas Ste7, también conocidas como quinasas MAP2 . Las quinasas MAP2, a su vez, también son activadas por fosforilación, por una serie de diferentes quinasas de serina-treonina corriente arriba ( quinasas MAP3 ). Debido a que las quinasas MAP2 muestran muy poca actividad en sustratos distintos de su MAPK afín, las vías MAPK clásicas forman vías de varios niveles, pero relativamente lineales. Estas vías pueden transmitir de manera efectiva estímulos desde la membrana celular (donde se activan muchas MAP3K) al núcleo (donde solo pueden ingresar las MAPK) o a muchos otros objetivos subcelulares. [ cita requerida ]
En comparación con las vías MAPK clásicas de tres niveles, algunas quinasas MAP atípicas parecen tener un sistema más antiguo, de dos niveles. Recientemente se ha demostrado que ERK3 (MAPK6) y ERK4 (MAPK4) son fosforiladas directamente y, por lo tanto, activadas por las quinasas PAK (relacionadas con otras quinasas MAP3). [6] A diferencia de las quinasas MAP clásicas, estas MAPK atípicas requieren solo un único residuo en sus bucles de activación para ser fosforiladas. Los detalles de la activación de NLK y ERK7 (MAPK15) siguen siendo desconocidos.
La inactivación de las MAPK la llevan a cabo varias fosfatasas . Una familia muy conservada de fosfatasas dedicadas son las denominadas fosfatasas quinasas MAP (MKP), un subgrupo de las fosfatasas de especificidad dual (DUSP). [7] Como su nombre lo indica, estas enzimas son capaces de hidrolizar el fosfato tanto de los residuos de fosfotirosina como de fosfotreonina. Dado que la eliminación de cualquiera de los grupos fosfato reducirá en gran medida la actividad de las MAPK, aboliendo esencialmente la señalización, algunas fosfatasas de tirosina también participan en la inactivación de las quinasas MAP (por ejemplo, las fosfatasas HePTP , STEP y PTPRR en mamíferos).

Como se mencionó anteriormente, las MAPK suelen formar vías de varios niveles, recibiendo entrada varios niveles por encima de la quinasa MAP real. En contraste con el mecanismo de activación relativamente simple, dependiente de la fosforilación de las MAPK y MAP2K , las MAP3K tienen una regulación sorprendentemente compleja. Muchas de las MAP3K más conocidas , como c-Raf , MEKK4 o MLK3 requieren múltiples pasos para su activación. Estas son enzimas típicamente controladas alostéricamente, fuertemente bloqueadas en un estado inactivo por múltiples mecanismos. El primer paso en el camino hacia su activación consiste en aliviar su autoinhibición por un ligando más pequeño (como Ras para c-Raf , GADD45 para MEKK4 [10] o Cdc42 para MLK3 [11] ). Esto comúnmente (pero no siempre) sucede en la membrana celular, donde están unidos la mayoría de sus activadores (nótese que las proteínas G pequeñas están constitutivamente asociadas a la membrana debido a la prenilación ). A este paso le sigue una homodimerización y heterodimerización de lado a lado de sus dominios de quinasa ahora accesibles. Las estructuras complejas determinadas recientemente revelan que los dímeros se forman en una orientación que deja libres sus dos regiones de unión al sustrato. [12] Es importante destacar que este evento de dimerización también obliga a los dominios de quinasa MAP3 a adoptar una conformación parcialmente activa. La actividad completa solo se logra una vez que estos dímeros se transfosforilan entre sí en sus bucles de activación. El último paso también puede lograrse o ayudarse con proteínas quinasas auxiliares (quinasas MAP4, miembros de la familia Ste20). Una vez que una quinasa MAP3 está completamente activa, puede fosforilar sus sustratos quinasas MAP2, que a su vez fosforilarán sus sustratos de quinasa MAP. [ cita requerida ]

La vía ERK1/2 de los mamíferos es probablemente el sistema MAPK mejor caracterizado. Los activadores aguas arriba más importantes de esta vía son las proteínas Raf ( A-Raf , B-Raf o c-Raf ), los mediadores clave de la respuesta a los factores de crecimiento ( EGF , FGF , PDGF , etc.); pero otras MAP3K como c-Mos y Tpl2/Cot también pueden desempeñar el mismo papel. Todas estas enzimas fosforilan y, por lo tanto, activan las quinasas MKK1 y/o MKK2 , que son activadores altamente específicos para ERK1 y ERK2 . Estas últimas fosforilan una serie de sustratos importantes para la proliferación celular , la progresión del ciclo celular , la división celular y la diferenciación ( quinasas RSK , factor de transcripción Elk-1 , etc.)
A diferencia de la vía ERK1/2 relativamente bien aislada , las quinasas p38 y JNK de mamíferos tienen la mayoría de sus activadores compartidos a nivel de MAP3K ( MEKK1 , MEKK4 , ASK1 , TAK1 , MLK3 , TAOK1 , etc.). Además, algunas enzimas MAP2K pueden activar tanto p38 como JNK ( MKK4 ), mientras que otras son más específicas para JNK ( MKK7 ) o p38 ( MKK3 y MKK6 ). Debido a estos interbloqueos, hay muy pocos estímulos, si es que hay alguno, que puedan provocar la activación de JNK sin activar simultáneamente p38 o revertirlo. [13] Las vías de señalización JNK y p38 responden a estímulos de estrés, como citocinas , irradiación ultravioleta , choque térmico y choque osmótico , y están involucradas en la adaptación al estrés , la apoptosis o la diferenciación celular . Las JNK tienen una serie de sustratos dedicados que sólo ellas pueden fosforilar ( c-Jun , NFAT4 , etc.), mientras que las p38 también tienen algunos objetivos únicos (por ejemplo, las quinasas MAPKAP MK2 y MK3 ), lo que garantiza la necesidad de ambas para responder a estímulos estresantes.
ERK5 es parte de una vía bastante bien separada en los mamíferos. Su único activador específico aguas arriba MKK5 se activa en respuesta a las quinasas MAP3 MEKK2 y MEKK3 . La especificidad de estas interacciones es proporcionada por la arquitectura única de MKK5 y MEKK2/3, ambas conteniendo dominios PB1 N-terminales, permitiendo la heterodimerización directa entre sí. [14] El dominio PB1 de MKK5 también contribuye a la interacción ERK5-MKK5: proporciona una interfaz especial (además del motivo D encontrado en MKK5) a través de la cual MKK5 puede reconocer específicamente su sustrato ERK5. [15] Aunque los detalles a nivel molecular son poco conocidos, MEKK2 y MEKK3 responden a ciertas señales de desarrollo para dirigir la formación de endotelio y la morfogénesis cardíaca . Si bien también están implicadas en el desarrollo cerebral, la letalidad embrionaria de la inactivación de ERK5 debido a anomalías cardíacas subraya su papel central en la vasculogénesis de los mamíferos . [16] Es notable que la eliminación condicional de ERK5 en animales adultos también es letal, debido a la alteración generalizada de las barreras endoteliales . [17] Se cree que las mutaciones en los componentes anteriores de la vía ERK5 (el complejo CCM) son la base de las malformaciones cavernosas cerebrales en humanos.

Las vías MAPK de los hongos también están bien estudiadas. En la levadura, la MAPK Fus3 es responsable de la detención del ciclo celular y el apareamiento en respuesta a la estimulación con feromonas. El factor alfa de la feromona es detectado por un receptor transmembrana de siete . El reclutamiento y la activación de los componentes de la vía Fus3 dependen estrictamente de la activación de la proteína G heterotrimérica . La vía MAPK de apareamiento consta de tres niveles (Ste11-Ste7-Fus3), pero las quinasas MAP2 y MAP3 se comparten con otra vía, la Kss1 o vía de crecimiento filamentoso. Si bien Fus3 y Kss1 son quinasas de tipo ERK estrechamente relacionadas, las células de levadura aún pueden activarlas por separado, con la ayuda de una proteína de andamiaje Ste5 que es reclutada selectivamente por las proteínas G de la vía de apareamiento. El truco es que Ste5 puede asociarse con Fus3 y "desbloquearlo" para Ste7 como sustrato en un complejo terciario, mientras que no hace lo mismo para Kss1, dejando que la vía de crecimiento filamentoso se active solo en ausencia del reclutamiento de Ste5. [18]
Los hongos también tienen una vía que recuerda a la señalización JNK/p38 de los mamíferos. Se trata de la vía Hog1: se activa por una alta osmolaridad (en Saccharomyces cerevisiae ) o por una serie de otros factores de estrés abióticos (en Schizosaccharomyces pombe ). La quinasa MAP2 de esta vía se llama Pbs2 (relacionada con MKK3/4/6/7 de los mamíferos), las quinasas MAP3 dedicadas a la activación son Ssk2 y SSk22. El sistema en S. cerevisiae se activa mediante un sofisticado módulo de osmosensibilidad que consiste en las proteínas Sho1 y Sln1, pero aún no está claro cómo otros estímulos pueden provocar la activación de Hog1. La levadura también muestra una serie de otras vías MAPK sin homólogos cercanos en animales, como la vía de integridad de la pared celular (Mpk1/Slt2) o la vía de esporulación (Smk1). [19]
A pesar del alto número de genes MAPK, las vías MAPK de plantas superiores fueron estudiadas menos que las de animales o hongos. Aunque su señalización parece muy compleja, las quinasas MPK3, MPK4 y MPK6 de Arabidopsis thaliana son mediadores clave de las respuestas al choque osmótico , estrés oxidativo , respuesta al frío e involucradas en respuestas antipatógenas. [20] [21] [22] El modelo de inmunidad mediada por MAPK de Asai et al. 2002 pasa la señal de reconocimiento del efector de FLS2 ⇨ MEKK1 ⇨ MKK4 o MKK5 ⇨ MPK3 y MPK6 ⇨ WRKY22 o WRKY29. [22] Sin embargo, el trabajo de Mészáros et al. 2006 y Suarez-Rodriguez et al. 2007 da otras órdenes para esta vía y es posible que estas sean vías paralelas operando simultáneamente. [22] También están involucrados en la morfogénesis , ya que los mutantes MPK4 muestran un enanismo severo . [23]

Los miembros de la familia MAPK se pueden encontrar en todos los organismos eucariotas examinados hasta ahora. En particular, tanto las quinasas MAP clásicas como las atípicas se pueden rastrear hasta la raíz de la radiación de los principales grupos eucariotas. Las plantas terrestres contienen cuatro grupos de MAPK clásicas (MAPK-A, MAPK-B, MAPK-C y MAPK-D) que están involucradas en la respuesta a una gran cantidad de estreses abióticos. [25] Sin embargo, ninguno de estos grupos se puede equiparar directamente con los grupos de MAPK clásicas que se encuentran en los opistocontos (hongos y animales). En estos últimos, los principales subgrupos de MAPK clásicas forman la rama similar a ERK/Fus3 (que se subdivide en los metazoos en los subgrupos ERK1/2 y ERK5), y las quinasas similares a p38/Hog1 (que también se han dividido en los subgrupos p38 y JNK en animales multicelulares). [26] Además, existen varias MAPK tanto en hongos como en animales, cuyos orígenes son menos claros, ya sea debido a una alta divergencia (por ejemplo, NLK) o debido a que posiblemente sean una rama temprana de toda la familia MAPK (ERK3, ERK4, ERK7). En los vertebrados, debido a las duplicaciones gemelas del genoma completo después de la división cefalocordado/vertebrado, [27] existen varios parálogos en cada grupo. Por lo tanto, ERK1 y ERK2 corresponden a la quinasa de Drosophila rolled , JNK1, JNK2 y JNK3 son todos ortólogos de la canasta de genes en Drosophila . Aunque entre el grupo p38, p38 alfa y beta son claramente pares parálogos, y también lo son p38 gamma y delta en vertebrados, el momento de la división de bases es menos claro, dado que muchos metazoos ya poseen múltiples homólogos de p38 (hay tres quinasas de tipo p38 en Drosophila , Mpk2 ( p38a ), p38b y p38c ). La proteína única ERK5 parece cumplir una función muy especializada (esencial para el desarrollo vascular en vertebrados) dondequiera que esté presente. Este linaje ha sido eliminado en protostomas , junto con sus componentes de la vía ascendente (MEKK2/3, MKK5), aunque están claramente presentes en cnidarios , esponjas e incluso en ciertos organismos unicelulares (por ejemplo, el coanoflagelado Monosiga brevicollis ) estrechamente relacionados con los orígenes de los animales multicelulares. [28]
La división entre las quinasas MAP clásicas y algunas atípicas ocurrió bastante temprano. Esto es sugerido no solo por la alta divergencia entre los genes existentes, sino también por los descubrimientos recientes de MAPK atípicas en eucariotas basales primitivos. La secuenciación del genoma de Giardia lamblia reveló la presencia de dos genes MAPK, uno de ellos similar a las MAPK de mamíferos ya conocidas (ERK, p38, etc.), el otro mostrando similitudes con la proteína ERK7 de mamíferos. [29] La situación es similar en la ameba multicelular Dictyostelium discoideum , donde la proteína ddERK1 parece ser una MAPK clásica, mientras que ddERK2 se parece más a nuestras proteínas ERK7 y ERK3/4. [30] Las MAPK atípicas también se pueden encontrar en plantas superiores, aunque son poco conocidas. De manera similar a la situación en los mamíferos, la mayoría de los aspectos de las MAPK atípicas no están caracterizados debido a la falta de atención a la investigación en esta área.
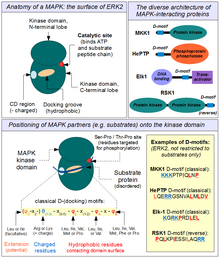
Como es típico para el grupo de quinasas CMGC, el sitio catalítico de las quinasas MAP tiene una secuencia de consenso muy laxa para los sustratos . Como todos sus parientes, solo requieren que los aminoácidos serina / treonina diana sean seguidos por un aminoácido pequeño, preferiblemente prolina ("quinasas dirigidas por prolina"). Pero como los sitios SP/TP son extremadamente comunes en todas las proteínas, se han desarrollado mecanismos adicionales de reconocimiento de sustrato para asegurar la fidelidad de la señalización. [31] A diferencia de sus parientes más cercanos, las quinasas dependientes de ciclina (CDK), donde los sustratos son reconocidos por la subunidad de ciclina , las MAPK se asocian con sus sustratos a través de regiones de unión auxiliares en sus dominios de quinasa. La región más importante de este tipo consiste en el surco de acoplamiento hidrofóbico y la región CD cargada negativamente. Juntos reconocen el llamado acoplamiento MAPK o motivos D (también llamado motivo de interacción de quinasa / KIM). Los motivos D consisten esencialmente en uno o dos aminoácidos cargados positivamente, seguidos de residuos hidrófobos alternados (principalmente leucinas), típicamente aguas arriba del sitio de fosforilación por 10-50 aminoácidos. [32] Muchos de los sustratos MAPK conocidos contienen motivos D que no solo pueden unirse a, sino que también proporcionan reconocimiento específico por ciertas MAPK. Los motivos D no están restringidos a sustratos: las quinasas MAP2 también contienen motivos en sus extremos N que son absolutamente necesarios para la interacción MAP2K-MAPK y la activación de MAPK. [33] De manera similar, tanto las fosfatasas quinasas MAP de especificidad dual como las fosfatasas de tirosina específicas de MAP se unen a las quinasas MAP a través del mismo sitio de acoplamiento. [34] [35] Los motivos D incluso se pueden encontrar en ciertos reguladores y andamiajes de la vía MAPK (por ejemplo, en las proteínas JIP de mamíferos).
También existen otros sitios de unión al sustrato menos caracterizados. Uno de estos sitios (el sitio DEF) está formado por el bucle de activación (cuando está en la conformación activa) y el inserto específico de la quinasa MAP debajo de él. Este sitio puede acomodar péptidos con una secuencia de consenso FxFP, típicamente aguas abajo del sitio de fosforilación. [36] Nótese que el último sitio solo se puede encontrar en proteínas que necesitan reconocer selectivamente las quinasas MAP activas, por lo que se encuentran casi exclusivamente en sustratos. Diferentes motivos pueden cooperar entre sí, como en la familia Elk de factores de transcripción, que poseen tanto un motivo D como un motivo FxFP. La presencia de un motivo FxFP en la proteína de andamiaje KSR1 también sirve para convertirla en un sustrato de ERK1/2, lo que proporciona un mecanismo de retroalimentación negativa para establecer la fuerza correcta de activación de ERK1/2.
Desde el descubrimiento de Ste5 en la levadura, los científicos han buscado descubrir elementos de la vía de andamiaje no enzimáticos similares en los mamíferos. De hecho, hay varias proteínas implicadas en la señalización de ERK, que pueden unirse a múltiples elementos de la vía: MP1 se une tanto a MKK1/2 como a ERK1/2, KSR1 y KSR2 pueden unirse a B-Raf o c-Raf, MKK1/2 y ERK1/2. También se descubrieron proteínas análogas para la vía JNK: se demostró que las familias de proteínas JIP1 / JIP2 y JIP3 /JIP4 se unen a MLK, MKK7 y cualquier quinasa JNK. Desafortunadamente, a diferencia de Ste5 de levadura, los mecanismos por los cuales regulan la activación de MAPK son considerablemente menos conocidos. Si bien Ste5 en realidad forma un complejo ternario con Ste7 y Fus3 para promover la fosforilación de este último, las proteínas de andamiaje conocidas en mamíferos parecen funcionar mediante mecanismos muy diferentes. Por ejemplo, KSR1 y KSR2 son en realidad quinasas MAP3 y están relacionadas con las proteínas Raf. [37] Aunque las KSR por sí solas muestran una actividad insignificante de la quinasa MAP3, las proteínas KSR aún pueden participar en la activación de las quinasas Raf formando heterodímeros lado a lado con ellas, proporcionando un par alostérico para activar cada enzima. [38] Las JIP, por otro lado, son aparentemente proteínas de transporte, responsables del enriquecimiento de los componentes de señalización de MAPK en ciertos compartimentos de células polarizadas. [39] En este contexto, la fosforilación dependiente de JNK de JIP1 (y posiblemente JIP2) proporciona una señal para que las JIP liberen los componentes de la vía ascendente unidos a JIP e inactivos, impulsando así un fuerte ciclo de retroalimentación positiva local. [40] Este sofisticado mecanismo acopla el transporte dependiente de kinesina a la activación local de JNK, no solo en mamíferos, sino también en la mosca de la fruta Drosophila melanogaster . [41]
Dado que la vía de señalización de ERK está involucrada en la proliferación celular tanto fisiológica como patológica, es natural que los inhibidores de ERK1/2 representen una clase deseable de agentes antineoplásicos . De hecho, muchas de las mutaciones "impulsoras" proto-oncogénicas están vinculadas a la señalización de ERK1/2, como las tirosina quinasas receptoras constitutivamente activas (mutantes) , las proteínas Ras o Raf . Aunque no se desarrollaron inhibidores de MKK1/2 o ERK1/2 para uso clínico, los inhibidores de quinasas que también inhiben las quinasas Raf (por ejemplo, Sorafenib ) son agentes antineoplásicos exitosos contra varios tipos de cáncer. [42] [43] El inhibidor de MEK cobimetinib se ha investigado en modelos preclínicos de cáncer de pulmón en combinación con la inhibición de la vía PI3K , donde los dos fármacos conducen a una respuesta sinérgica. [44] [45]
Las quinasas JNK están implicadas en el desarrollo de la resistencia a la insulina en individuos obesos [46], así como en la excitotoxicidad de los neurotransmisores después de condiciones isquémicas. La inhibición de JNK1 mejora la resistencia a la insulina en ciertos modelos animales. Los ratones que fueron modificados genéticamente para carecer de un gen JNK3 funcional (la isoforma principal en el cerebro) muestran una mayor tolerancia isquémica y recuperación del accidente cerebrovascular. [47] Aunque se están desarrollando inhibidores de JNK de moléculas pequeñas, ninguno de ellos demostró ser efectivo en pruebas humanas todavía. Un inhibidor de JNK basado en péptidos (AM-111, un péptido con motivo D retroinverso de JIP1, anteriormente conocido como XG-102) también está en desarrollo clínico para la pérdida auditiva neurosensorial . [48]
En el pasado, se creía que la p38 era un objetivo perfecto para los fármacos antiinflamatorios. Sin embargo, el fracaso de más de una docena de compuestos químicamente diferentes en la fase clínica sugiere que las quinasas p38 podrían ser objetivos terapéuticos deficientes en las enfermedades autoinmunes . Se descubrió que muchos de estos compuestos eran hepatotóxicos en diversos grados y que la tolerancia al efecto antiinflamatorio se desarrollaba en cuestión de semanas. [49] Un enfoque alternativo es evaluar el potencial para dirigirse a las MAPK anteriores, como ASK1 . [50] Los estudios en modelos animales de artritis inflamatoria han arrojado resultados prometedores, y recientemente se ha descubierto que la ASK1 es única entre las MAPK en el sentido de que es inducible por citocinas inflamatorias como TNF-α . [50]