La fosfolipasa D (EC 3.1.4.4, lipofosfodiesterasa II, lecitinasa D, colina fosfatasa, PLD ; nombre sistemático fosfatidilcolina fosfatidohidrolasa ) es una enzima de la superfamilia de fosfolipasas que cataliza la siguiente reacción
Las fosfolipasas se encuentran ampliamente y se pueden encontrar en una amplia gama de organismos, incluidas bacterias, levaduras, plantas, animales y virus. [1] [2] El sustrato principal de la fosfolipasa D es la fosfatidilcolina , que hidroliza para producir la molécula señal ácido fosfatídico (PA), y colina soluble en un proceso dependiente del colesterol llamado presentación del sustrato . [3] Las plantas contienen numerosos genes que codifican diversas isoenzimas PLD , con pesos moleculares que oscilan entre 90 y 125 kDa . [4] Las células de mamífero codifican dos isoformas de fosfolipasa D: PLD1 y PLD2 . [5] La fosfolipasa D juega un papel importante en muchos procesos fisiológicos , incluido el tráfico de membranas , la reorganización del citoesqueleto , la endocitosis mediada por receptores , la exocitosis y la migración celular . [6] A través de estos procesos, se ha implicado aún más en la fisiopatología de múltiples enfermedades : en particular, la progresión del Parkinson y el Alzheimer , así como en varios cánceres . [4] [6] La PLD también puede ayudar a establecer el umbral de sensibilidad a la anestesia y la fuerza mecánica. [7] [8]
La actividad de tipo PLD fue reportada por primera vez en 1947 por Donald J. Hanahan e IL Chaikoff. [1] Sin embargo, no fue hasta 1975 que se esclareció el mecanismo de acción hidrolítico en células de mamíferos . Las isoformas vegetales de PLD se purificaron primero a partir de repollo y ricino ; Finalmente, PLDα se clonó y caracterizó a partir de una variedad de plantas, incluidos arroz, maíz y tomate. [1] Los PLD de plantas se han clonado en tres isoformas: PLDα, PLDβ y PLDγ. [9] Más de medio siglo de estudios bioquímicos han implicado la actividad de la fosfolipasa D y la PA en una amplia gama de procesos y enfermedades fisiológicos , incluida la inflamación , la diabetes , la fagocitosis , la señalización neuronal y cardíaca y la oncogénesis . [10]
Estrictamente hablando, la fosfolipasa D es una transfosfatidilasa: media en el intercambio de grupos de cabeza polares unidos covalentemente a lípidos unidos a membranas . Utilizando agua como nucleófilo , esta enzima cataliza la escisión del enlace fosfodiéster en fosfolípidos estructurales como la fosfatidilcolina y la fosfatidiletanolamina . [4] Los productos de esta hidrólisis son el ácido fosfatídico (PA) lipídico unido a la membrana y la colina , que se difunde en el citosol . Como la colina tiene poca actividad de segundo mensajero , la actividad de PLD se transduce principalmente mediante la producción de PA. [6] [11] La PA está muy implicada en la transducción de señales intracelulares . [12] Además, algunos miembros de la superfamilia PLD pueden emplear alcoholes primarios como etanol o 1-butanol en la escisión del fosfolípido , catalizando eficazmente el intercambio del grupo de cabeza de lípidos polares . [4] [9] Otros miembros de esta familia son capaces de hidrolizar otros sustratos fosfolípidos, como la cardiolipina , o incluso el enlace fosfodiéster que constituye la columna vertebral del ADN . [5]
Muchas de las funciones celulares de la fosfolipasa D están mediadas por su producto principal, el ácido fosfatídico (PA). La PA es un fosfolípido cargado negativamente , cuyo pequeño grupo de cabeza promueve la curvatura de la membrana . [5] Por lo tanto, se cree que facilita la fusión y fisión membrana - vesícula de una manera análoga a la endocitosis mediada por clatrina . [5] La PA también puede reclutar proteínas que contienen su correspondiente dominio de unión , una región caracterizada por regiones ricas en aminoácidos básicos . Además, la PA se puede convertir en otros lípidos , como el ácido lisofosfatídico (liso-PA) o el diacilglicerol , moléculas señal que tienen una multitud de efectos en las vías celulares posteriores . [9] La PA y sus derivados lipídicos están implicados en innumerables procesos que incluyen el tráfico de vesículas intracelulares , la endocitosis , la exocitosis , la dinámica del citoesqueleto de actina , la diferenciación de la proliferación celular y la migración . [5]

El PLD de mamíferos interactúa directamente con quinasas como PKC , ERK , TYK y controla la señalización que indica que estas quinasas activan el PLD. [13] Como la colina es muy abundante en la célula, la actividad de PLD no afecta significativamente los niveles de colina y es poco probable que la colina desempeñe algún papel en la señalización.
El ácido fosfatídico es una molécula señal y actúa para reclutar SK1 en las membranas . La PA tiene una vida extremadamente corta y es rápidamente hidrolizada por la enzima fosfatidato fosfatasa para formar diacilglicerol (DAG). DAG también puede convertirse en PA mediante la DAG quinasa . Aunque PA y DAG son interconvertibles, no actúan en las mismas vías . Los estímulos que activan PLD no activan las enzimas aguas abajo de DAG y viceversa.
Es posible que, aunque PA y DAG sean interconvertibles, se puedan mantener grupos separados de lípidos de señalización y no señalización. Los estudios han sugerido que la señalización de DAG está mediada por DAG poliinsaturado, mientras que la PA derivada de PLD es monoinsaturada o saturada . Por lo tanto, la PA saturada/monoinsaturada funcional se puede degradar hidrolizándola para formar DAG saturado/monoinsaturado no funcional, mientras que la DAG poliinsaturada funcional se puede degradar convirtiéndola en PA poliinsaturada no funcional. [14] [15] [16]
Recientemente se identificó que una lisofosfolipasa D llamada autotaxina tiene un papel importante en la proliferación celular a través de su producto, el ácido lisofosfatídico (LPA).
Los PLD de plantas y animales tienen una estructura molecular consistente , caracterizada por sitios de catálisis rodeados por una variedad de secuencias reguladoras . [4] El sitio activo de los PLD consta de cuatro secuencias de aminoácidos altamente conservadas (I-IV), de las cuales los motivos II y IV están particularmente conservados. Estos dominios estructurales contienen la secuencia catalítica distintiva HxKxxxxD (HKD), donde H , K y D son los aminoácidos histidina (H), lisina (K), ácido aspártico (D), mientras que x representa aminoácidos no conservativos . [4] [5] Estos dos motivos HKD confieren actividad hidrolítica a PLD y son fundamentales para su actividad enzimática tanto in vitro como in vivo . [5] [10] La hidrólisis del enlace fosfodiéster se produce cuando estas secuencias de HKD están en la proximidad correcta .
Las proteínas humanas que contienen este motivo incluyen:
La PLD hidrolizante de PC es un homólogo de la cardiolipina sintasa , [17] [18] fosfatidilserina sintasa , las PLD bacterianas y las proteínas virales . Cada uno de estos parece poseer una duplicación de dominio que es evidente por la presencia de dos motivos HKD que contienen residuos de histidina , lisina y asparagina bien conservados que pueden contribuir al sitio activo del ácido aspártico . Una endonucleasa de Escherichia coli (nuc) y proteínas similares parecen ser homólogas de PLD pero poseen sólo uno de estos motivos. [19] [20] [21] [22]
Los genes PLD codifican además dominios reguladores altamente conservados : la secuencia de consenso de Phox (PX) , el dominio de homología de pleckstrina (PH) y un sitio de unión para fosfatidilinositol 4,5-bisfosfato (PIP 2 ). [2]
Se ha propuesto que la hidrólisis catalizada por PLD se produzca en dos etapas mediante un mecanismo de " ping-pong ". En este esquema, los residuos de histidina de cada motivo HKD atacan sucesivamente al sustrato fosfolípido . Al funcionar como nucleófilos , los restos imidazol constituyentes de las histidinas forman enlaces covalentes transitorios con el fosfolípido , produciendo un intermediario de vida corta que puede hidrolizarse fácilmente con agua en un paso posterior . [4] [12]
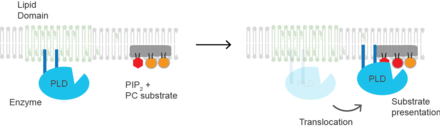
Presentación del sustrato Para PLD2 de mamíferos, la base molecular de la activación es la presentación del sustrato. La enzima reside inactiva en microdominios lipídicos ricos en esfingomielina y sin sustrato de PC. [23] El aumento de PIP2 o una disminución del colesterol hace que la enzima se transloque a microdominios de PIP2 cerca de su sustrato PC. Por lo tanto, la PLD se activa principalmente por localización dentro de la membrana plasmática en lugar de por un cambio conformacional de la proteína. Alteración de los dominios lipídicos por anestésicos. [24] o fuerza mecánica. [23] La proteína también puede sufrir un cambio conformacional tras la unión de PIP2, pero esto no se ha demostrado experimentalmente y constituiría un mecanismo de activación distinto de la presentación del sustrato.
Se han identificado dos isoformas principales de fosfolipasa D en células de mamíferos : PLD1 y PLD2 (53% de homología de secuencia ), [25] cada una codificada por genes distintos . [5] La actividad PLD parece estar presente en la mayoría de los tipos de células , con las posibles excepciones de los leucocitos periféricos y otros linfocitos . [10] Ambas isoformas de PLD requieren PIP 2 como cofactor para la actividad . [5] PLD1 y PLD2 exhiben diferentes localizaciones subcelulares que cambian dinámicamente en el curso de la transducción de señales . Se ha observado actividad PLD dentro de la membrana plasmática , el citosol , el RE y el complejo de Golgi . [10]
PLD1 es una proteína de 120 kDa que se encuentra principalmente en las membranas internas de las células. Está presente principalmente en el complejo de Golgi , endosomas , lisosomas y gránulos secretores . [5] Tras la unión de un estímulo extracelular , PLD1 se transporta a la membrana plasmática . Sin embargo, la actividad basal de PLD1 es baja y, para transducir la señal extracelular, primero debe ser activada por proteínas como Arf , Rho , Rac y la proteína quinasa C. [5] [6] [11]
La actividad de la fosfolipasa D está ampliamente regulada por hormonas , neurotransmisores , lípidos , pequeñas GTPasas monoméricas y otras moléculas pequeñas que se unen a sus dominios correspondientes en la enzima. [4] En la mayoría de los casos, la transducción de señales está mediada por la producción de ácido fosfatídico , que funciona como mensajero secundario . [4]
Los fosfolípidos específicos son reguladores de la actividad PLD en células vegetales y animales. [1] [4] La mayoría de los PLD requieren fosfatidilinositol 4,5-bisfosfato (PIP 2 ), como cofactor para su actividad. [2] [4] PIP 2 y otros fosfoinosítidos son modificadores importantes de la dinámica citoesquelética y del transporte de membrana y pueden transportar PLD a su sustrato PC. [26] Los PLD regulados por estos fosfolípidos suelen participar en la transducción de señales intracelulares . [4] Su actividad depende de la unión de estos fosfoinosítidos cerca del sitio activo . [4] En plantas y animales, este sitio de unión se caracteriza por la presencia de una secuencia conservada de aminoácidos básicos y aromáticos . [4] [12] En plantas como Arabidopsis thaliana , esta secuencia está constituida por un motivo RxxxxxKxR junto con su repetición invertida , donde R es arginina y K es lisina . Su proximidad al sitio activo garantiza un alto nivel de actividad de PLD1 y PLD2 , y promueve la translocación de PLD1 a las membranas diana en respuesta a señales extracelulares . [4]
El calcio actúa como cofactor en las isoformas de PLD que contienen el dominio C2 . La unión de Ca 2+ al dominio C2 conduce a cambios conformacionales en la enzima que fortalecen la unión enzima-sustrato , al tiempo que debilitan la asociación con fosfoinosítidos . En algunas isoenzimas vegetales , como la PLDβ, el Ca 2+ puede unirse directamente al sitio activo , aumentando indirectamente su afinidad por el sustrato al fortalecer la unión del activador PIP 2 . [4]
Se cree que la secuencia consenso de pbox (PX) media la unión de fosfatos de fosfatidilinositol adicionales; en particular, el fosfatidilinositol 5-fosfato (PtdIns5P), un lípido que se cree que es necesario para la endocitosis , puede ayudar a facilitar la reinternalización de PLD1 desde la membrana plasmática . [1]
El dominio de homología de Pleckstrin (PH) altamente conservado es un dominio estructural de aproximadamente 120 aminoácidos de longitud. Se une a fosfatidilinosítidos como el fosfatidilinositol (3,4,5)-trifosfato (PIP 3 ) y el fosfatidilinositol (4,5)-bifosfato (PIP 2 ). También puede unirse a proteínas G heterotriméricas a través de su subunidad βγ . También se cree que la unión a este dominio facilita la reinternalización de la proteína al aumentar su afinidad por las balsas lipídicas endocitoticas . [1]
En las células animales , pequeños factores proteicos son importantes reguladores adicionales de la actividad del PLD. Estas pequeñas GTPasas monoméricas son miembros de las familias Rho y ARF de la superfamilia Ras . Algunas de estas proteínas, como Rac1 , Cdc42 y RhoA , activan alostéricamente la PLD1 de los mamíferos , aumentando directamente su actividad. En particular, la translocación del factor de ribosilación de ADP (ARF) citosólico a la membrana plasmática es esencial para la activación de PLD. [1] [4]
La fosfolipasa D metaboliza el etanol en fosfatidiletanol (PEtOH) en un proceso denominado transfosfatidilación. Utilizando genética de moscas, se demostró que el PEtOH media la respuesta hiperactiva del alcohol en las moscas de la fruta. [27] Y se demostró que la transfosfatidilación de etanol está regulada positivamente en alcohólicos y sus familiares. [28] Este mecanismo de transfosfatidilación de etanol surgió recientemente como una teoría alternativa para el efecto del alcohol en los canales iónicos. Muchos canales iónicos están regulados por lípidos aniónicos. [29] y se cree que la competencia del PETOH con lípidos de señalización endógenos media el efecto del etanol en los canales iónicos en algunos casos y no la unión directa del etanol libre al canal. [27]
PLD2 es un mecanosensor y directamente sensible a la alteración mecánica de los lípidos GM1 agrupados. [3] La interrupción mecánica (cizallamiento del fluido) luego indica a la célula que se diferencie. PLD2 también activa los canales TREK-1, un canal de potasio en la vía analgésica. [30]
La fosfolipasa D es un regulador de varios procesos celulares críticos, incluido el transporte de vesículas , la endocitosis , la exocitosis , la migración celular y la mitosis . [6] La desregulación de estos procesos es común en la carcinogénesis , [6] y, a su vez, las anomalías en la expresión de PLD se han implicado en la progresión de varios tipos de cáncer . [2] [5] Se ha observado una mutación conductora que confiere una actividad elevada de PLD2 en varios cánceres de mama malignos . [5] La expresión elevada de PLD también se ha correlacionado con el tamaño del tumor en el carcinoma colorrectal , el carcinoma gástrico y el cáncer renal . [5] [6] Sin embargo, las vías moleculares a través de las cuales la PLD impulsa la progresión del cáncer aún no están claras. [5] Una posible hipótesis plantea un papel fundamental para la fosfolipasa D en la activación de mTOR , un supresor de la apoptosis de las células cancerosas . [5] La capacidad de PLD para suprimir la apoptosis en células con actividad tirosina quinasa elevada lo convierte en un oncogén candidato en cánceres donde dicha expresión es típica. [6]
La fosfolipasa D también puede desempeñar un papel fisiopatológico importante en la progresión de enfermedades neurodegenerativas , principalmente a través de su capacidad como transductor de señales en procesos celulares indispensables como la reorganización del citoesqueleto y el tráfico de vesículas . [25] Se ha demostrado que la desregulación de PLD por la proteína α-sinucleína conduce a la pérdida específica de neuronas dopaminérgicas en los mamíferos . La α-sinucleína es el componente estructural principal de los cuerpos de Lewy , agregados de proteínas que son el sello distintivo de la enfermedad de Parkinson . [5] La desinhibición de PLD por la α-sinucleína puede contribuir al fenotipo nocivo del Parkinson . [5]
También se ha sospechado una actividad anormal de PLD en la enfermedad de Alzheimer , donde se ha observado que interactúa con la presenilina 1 (PS-1), el componente principal del complejo γ-secretasa responsable de la escisión enzimática de la proteína precursora de amiloide (APP). Las placas extracelulares del producto β-amiloide son una característica definitoria de los cerebros con enfermedad de Alzheimer . [5] Se ha demostrado que la acción de PLD1 sobre PS-1 afecta el tráfico intracelular del precursor amiloide a este complejo . [5] [25] La fosfolipasa D3 (PLD3), un miembro no clásico y mal caracterizado de la superfamilia PLD , también se ha asociado con la patogénesis de esta enfermedad. [31]