El microbioma humano es el conjunto de toda la microbiota que reside en los tejidos y biofluidos humanos, junto con los sitios anatómicos correspondientes en los que residen, [1] incluidos el tracto gastrointestinal , la piel , las glándulas mamarias , el líquido seminal , el útero , los folículos ováricos , los pulmones , la saliva , la mucosa oral , la conjuntiva y el tracto biliar . Los tipos de microbiota humana incluyen bacterias , arqueas , hongos , protistas y virus . Aunque los microanimales también pueden vivir en el cuerpo humano, normalmente se los excluye de esta definición. En el contexto de la genómica , el término microbioma humano se utiliza a veces para referirse a los genomas colectivos de microorganismos residentes; [2] sin embargo, el término metagenoma humano tiene el mismo significado. [1]
El cuerpo humano alberga muchos microorganismos, con aproximadamente el mismo orden de magnitud de células no humanas que de células humanas. [3] Algunos microorganismos que los humanos albergan son comensales , lo que significa que coexisten sin dañar a los humanos; otros tienen una relación mutualista con sus huéspedes humanos. [2] : 700 [4] Por el contrario, algunos microorganismos no patógenos pueden dañar a los huéspedes humanos a través de los metabolitos que producen, como la trimetilamina , que el cuerpo humano convierte en N-óxido de trimetilamina a través de la oxidación mediada por FMO3 . [5] [6] Ciertos microorganismos realizan tareas que se sabe que son útiles para el huésped humano, pero el papel de la mayoría de ellos no se entiende bien. Aquellos que se espera que estén presentes, y que en circunstancias normales no causan enfermedades, a veces se consideran flora normal o microbiota normal . [2]
Durante los primeros años de vida, el establecimiento de una microbiota humana diversa y equilibrada desempeña un papel fundamental en la conformación de la salud a largo plazo de un individuo. [7] Los estudios han demostrado que la composición de la microbiota intestinal durante la infancia está influenciada por varios factores, incluido el modo de parto, la lactancia materna y la exposición a factores ambientales. [8] Hay varias especies beneficiosas de bacterias y probióticos potenciales presentes en la leche materna . [9] La investigación ha destacado los efectos beneficiosos de una microbiota saludable en los primeros años de vida, como la promoción del desarrollo del sistema inmunológico, la regulación del metabolismo y la protección contra microorganismos patógenos. [10] Comprender la compleja interacción entre la microbiota humana y la salud en los primeros años de vida es crucial para desarrollar intervenciones y estrategias que respalden el desarrollo óptimo de la microbiota y mejoren los resultados generales de salud en las personas. [11]
El Proyecto Microbioma Humano (HMP) se propuso secuenciar el genoma de la microbiota humana, centrándose especialmente en la microbiota que habita normalmente en la piel, la boca, la nariz, el tracto digestivo y la vagina. [2] Alcanzó un hito en 2012 cuando publicó sus resultados iniciales. [12]
Aunque se la conoce ampliamente como flora o microflora , este término es inapropiado en términos técnicos, ya que la palabra flora de la raíz se refiere a las plantas, y biota se refiere al conjunto total de organismos en un ecosistema particular. Recientemente, se aplica el término más apropiado microbiota , aunque su uso no ha eclipsado el uso y reconocimiento arraigados de flora con respecto a bacterias y otros microorganismos. Ambos términos se están utilizando en diferentes literaturas. [4]
Se estima que el número de células bacterianas en el cuerpo humano es de alrededor de 38 billones, mientras que la estimación para las células humanas es de alrededor de 30 billones. [13] [14] [15] [16] Se estima que el número de genes bacterianos es de 2 millones, 100 veces el número de aproximadamente 20.000 genes humanos . [17] [18] [19]
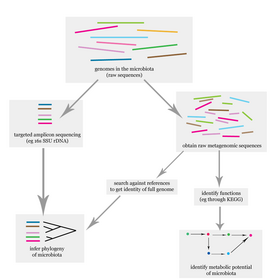
El problema de dilucidar el microbioma humano es esencialmente identificar a los miembros de una comunidad microbiana, que incluye bacterias, eucariotas y virus. [20] Esto se hace principalmente mediante estudios basados en ácido desoxirribonucleico (ADN), aunque también se realizan estudios basados en ácido ribonucleico (ARN), proteínas y metabolitos. [20] [21] Los estudios del microbioma basados en ADN generalmente se pueden clasificar como estudios de amplicones específicos o, más recientemente, estudios metagenómicos de escopeta . El primero se centra en genes marcadores conocidos específicos y es principalmente informativo taxonómicamente, mientras que el segundo es un enfoque metagenómico completo que también se puede utilizar para estudiar el potencial funcional de la comunidad. [20] Uno de los desafíos que está presente en los estudios del microbioma humano, pero no en otros estudios metagenómicos, es evitar incluir el ADN del huésped en el estudio. [22]
Además de simplemente dilucidar la composición del microbioma humano, una de las principales preguntas que involucra al microbioma humano es si existe un "núcleo", es decir, si existe un subconjunto de la comunidad que es compartido por la mayoría de los humanos. [23] [24] Si existe un núcleo, entonces sería posible asociar ciertas composiciones de la comunidad con estados patológicos, que es uno de los objetivos del HMP. Se sabe que el microbioma humano (como la microbiota intestinal) es altamente variable tanto dentro de un mismo sujeto como entre diferentes individuos, un fenómeno que también se observa en ratones. [4]
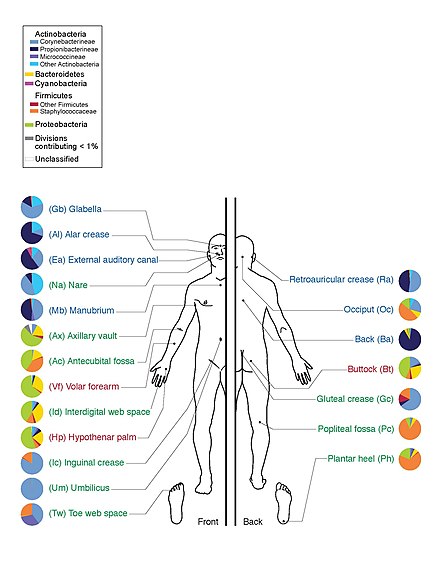
El 13 de junio de 2012, el director de los Institutos Nacionales de Salud (NIH), Francis Collins , anunció un hito importante del HMP . [12] El anuncio estuvo acompañado de una serie de artículos coordinados publicados en Nature [25] [26] y varias revistas en la Biblioteca Pública de Ciencias (PLoS) el mismo día. Al mapear la composición microbiana normal de humanos sanos utilizando técnicas de secuenciación del genoma, los investigadores del HMP han creado una base de datos de referencia y los límites de la variación microbiana normal en humanos. De 242 voluntarios estadounidenses sanos, se recolectaron más de 5000 muestras de tejidos de 15 (hombres) a 18 (mujeres) sitios del cuerpo como boca, nariz, piel, intestino delgado (heces) y vagina. Todo el ADN, humano y microbiano, se analizó con máquinas de secuenciación de ADN. Los datos del genoma microbiano se extrajeron identificando el ARN ribosómico específico bacteriano, 16S rRNA . Los investigadores calcularon que más de 10.000 especies microbianas ocupan el ecosistema humano y han identificado entre el 81 y el 99% de los géneros . [27]
El análisis estadístico es esencial para validar los resultados obtenidos ( se puede utilizar ANOVA para dimensionar las diferencias entre los grupos); si se combina con herramientas gráficas, el resultado se visualiza y comprende fácilmente. [28]
Una vez ensamblado un metagenoma, es posible inferir el potencial funcional del microbioma. Los desafíos computacionales para este tipo de análisis son mayores que para los genomas individuales, porque generalmente los ensambladores de metagenomas tienen una calidad inferior y muchos genes recuperados no están completos o están fragmentados. Después del paso de identificación de genes, los datos se pueden utilizar para realizar una anotación funcional mediante el alineamiento múltiple de los genes objetivo contra bases de datos de ortólogos . [29]
Es una técnica que explota primers para apuntar a una región genética específica y permite determinar las filogenias microbianas . La región genética se caracteriza por una región altamente variable que puede conferir una identificación detallada; está delimitada por regiones conservadas, que funcionan como sitios de unión para los primers utilizados en PCR . El gen principal utilizado para caracterizar bacterias y arqueas es el gen 16S rRNA , mientras que la identificación de hongos se basa en Internal Transcribed Spacer (ITS). La técnica es rápida y no tan costosa y permite obtener una clasificación de baja resolución de una muestra microbiana; es óptima para muestras que pueden estar contaminadas por ADN del huésped. La afinidad de los primers varía entre todas las secuencias de ADN, lo que puede resultar en sesgos durante la reacción de amplificación; de hecho, las muestras de baja abundancia son susceptibles a errores de sobreamplificación, ya que los otros microorganismos contaminantes resultan estar sobrerrepresentados en caso de aumentar los ciclos de PCR. Por lo tanto, la optimización de la selección de primers puede ayudar a disminuir tales errores, aunque requiere un conocimiento completo de los microorganismos presentes en la muestra y sus abundancias relativas. [30]
El análisis de genes marcadores puede verse influenciado por la elección del cebador; en este tipo de análisis, es deseable utilizar un protocolo bien validado (como el utilizado en el Proyecto del Microbioma de la Tierra ). Lo primero que hay que hacer en un análisis de amplicones de genes marcadores es eliminar los errores de secuenciación; muchas plataformas de secuenciación son muy fiables, pero la mayor parte de la aparente diversidad de secuencias se debe todavía a errores durante el proceso de secuenciación. Para reducir este fenómeno, un primer enfoque es agrupar las secuencias en unidades taxonómicas operativas (OTU): este proceso consolida secuencias similares (normalmente se adopta un umbral de similitud del 97%) en una única característica que se puede utilizar en pasos de análisis posteriores; sin embargo, este método descartaría los SNP porque se agruparían en una única OTU. Otro enfoque es la oligotipificación , que incluye información específica de la posición de la secuenciación del ARNr 16s para detectar pequeñas variaciones de nucleótidos y de la discriminación entre taxones distintos estrechamente relacionados. Estos métodos dan como resultado una tabla de secuencias de ADN y recuentos de las diferentes secuencias por muestra en lugar de OTU. [30]
Otro paso importante en el análisis es asignar un nombre taxonómico a las secuencias microbianas en los datos. Esto se puede hacer utilizando métodos de aprendizaje automático que pueden alcanzar una precisión a nivel de género de alrededor del 80%. Otros paquetes de análisis populares brindan soporte para la clasificación taxonómica utilizando coincidencias exactas con bases de datos de referencia y deberían proporcionar una mayor especificidad, pero una sensibilidad pobre. Los microorganismos no clasificados deben revisarse más a fondo para buscar secuencias de orgánulos. [30]
Muchos métodos que explotan la inferencia filogenética utilizan el gen 16SRNA para Archea y Bacteria y el gen 18SRNA para Eucariotas. Los métodos comparativos filogenéticos (PCS) se basan en la comparación de múltiples rasgos entre microorganismos; el principio es: cuanto más estrechamente relacionados estén, mayor número de rasgos compartirán. Por lo general, los PCS se combinan con mínimos cuadrados generalizados filogenéticos (PGLS) u otro análisis estadístico para obtener resultados más significativos. La reconstrucción del estado ancestral se utiliza en estudios de microbioma para imputar valores de rasgos para taxones cuyos rasgos son desconocidos. Esto se realiza comúnmente con PICRUSt , que se basa en las bases de datos disponibles. Los investigadores eligen las variables filogenéticas según el tipo de estudio: a través de la selección de algunas variables con información biológica significativa, es posible reducir la dimensión de los datos a analizar. [31]
La distancia filogenética se suele realizar con UniFrac o herramientas similares, como el índice de Soresen o la D de Rao, para cuantificar las diferencias entre las distintas comunidades. Todos estos métodos se ven afectados negativamente por la transmisión horizontal de genes (HGT), ya que puede generar errores y conducir a la correlación de especies distantes. Existen diferentes formas de reducir el impacto negativo de la HGT: el uso de múltiples genes o herramientas computacionales para evaluar la probabilidad de posibles eventos HGT. [31]
Las comunidades microbianas se desarrollan en una dinámica muy compleja que puede verse y analizarse como un ecosistema. Las interacciones ecológicas entre microbios gobiernan su cambio, equilibrio y estabilidad, y pueden representarse mediante un modelo de dinámica poblacional. [32] El estudio en curso de las características ecológicas del microbioma está creciendo rápidamente y permite comprender las propiedades fundamentales del microbioma. Comprender las reglas subyacentes de la comunidad microbiana podría ayudar a tratar enfermedades relacionadas con comunidades microbianas inestables. Una pregunta muy básica es si diferentes humanos, que comparten diferentes comunidades microbianas, tienen la misma dinámica microbiana subyacente. [33] Cada vez hay más evidencia e indicaciones de que la dinámica es realmente universal. [34] Esta pregunta es un paso básico que permitirá a los científicos desarrollar estrategias de tratamiento basadas en la dinámica compleja de las comunidades microbianas humanas. Hay propiedades más importantes sobre las que se deben tener en cuenta las consideraciones para desarrollar estrategias de intervención para controlar la dinámica microbiana humana. [35] Controlar las comunidades microbianas podría dar como resultado la solución de enfermedades muy malas y dañinas.

Las poblaciones de microbios (como bacterias y levaduras ) habitan la piel y las superficies mucosas en varias partes del cuerpo. Su papel forma parte de la fisiología humana normal y saludable, sin embargo, si las cantidades de microbios crecen más allá de sus rangos típicos (a menudo debido a un sistema inmunológico comprometido) o si los microbios pueblan (por ejemplo, debido a una mala higiene o una lesión) áreas del cuerpo normalmente no colonizadas o estériles (como la sangre, o el tracto respiratorio inferior, o la cavidad abdominal), puede resultar en enfermedad (causando, respectivamente, bacteriemia/sepsis, neumonía y peritonitis). [36]
El Proyecto del Microbioma Humano descubrió que los individuos albergan miles de tipos de bacterias y que los diferentes sitios del cuerpo tienen sus propias comunidades distintivas. La piel y la vagina mostraron una diversidad menor que la boca y el intestino, que son los que muestran la mayor riqueza. La composición bacteriana de un sitio determinado del cuerpo varía de persona a persona, no solo en tipo, sino también en abundancia. Las bacterias de la misma especie que se encuentran en toda la boca son de múltiples subtipos y prefieren habitar lugares claramente diferentes en la boca. Incluso los enterotipos del intestino humano, que antes se creían bien comprendidos, pertenecen a un amplio espectro de comunidades con límites taxonómicos difusos. [37] [38]
Se estima que entre 500 y 1.000 especies de bacterias viven en el intestino humano, pero pertenecen a sólo unos pocos filos: dominan Bacillota y Bacteroidota , pero también hay Pseudomonadota , Verrucomicrobiota , Actinobacteriota , Fusobacteriota y " Cianobacteria ". [39]
En la boca viven varios tipos de bacterias, como Actinomyces viscosus y A. naeslundii , donde forman parte de una sustancia pegajosa llamada placa . Si no se elimina con el cepillado, se endurece y forma sarro . Las mismas bacterias también secretan ácidos que disuelven el esmalte dental , lo que provoca caries . [ cita requerida ]
La microflora vaginal está formada principalmente por diversas especies de lactobacilos . Durante mucho tiempo se creyó que la más común de estas especies era Lactobacillus acidophilus , pero más tarde se demostró que L. iners es de hecho la más común, seguida de L. crispatus . Otros lactobacilos que se encuentran en la vagina son L. jensenii, L. delbruekii y L. gasseri . La alteración de la flora vaginal puede provocar infecciones como vaginosis bacteriana y candiadiasis. [40]
Las arqueas están presentes en el intestino humano, pero, en contraste con la enorme variedad de bacterias en este órgano, el número de especies de arqueas es mucho más limitado. [41] El grupo dominante son los metanógenos , particularmente Methanobrevibacter smithii y Methanosphaera stadtmanae . [42] Sin embargo, la colonización por metanógenos es variable, y solo alrededor del 50% de los humanos tienen poblaciones fácilmente detectables de estos organismos. [43]
Hasta 2007, no se conocían ejemplos claros de patógenos arqueológicos, [44] [45] aunque se ha propuesto una relación entre la presencia de algunos metanógenos y la enfermedad periodontal humana . [46] El sobrecrecimiento bacteriano del intestino delgado con predominio de metano (SIBO) también es causado predominantemente por metanógenos, y en particular por Methanobrevibacter smithii . [47]
Los hongos, en particular las levaduras , están presentes en el intestino humano. [48] [49] [50] [51] Las más estudiadas de ellas son las especies de Candida debido a su capacidad de volverse patógenas en huéspedes inmunodeprimidos e incluso sanos. [49] [50] [51] Las levaduras también están presentes en la piel, [48] como las especies de Malassezia , donde consumen aceites secretados por las glándulas sebáceas . [52] [53]
Los virus, especialmente los virus bacterianos ( bacteriófagos ), colonizan diversos sitios del cuerpo. Estos sitios colonizados incluyen la piel, [54] el intestino, [55] los pulmones, [56] y la cavidad oral. [57] Las comunidades de virus se han asociado con algunas enfermedades y no reflejan simplemente las comunidades bacterianas. [58] [59] [60]
En enero de 2024, los biólogos informaron el descubrimiento de " obeliscos ", una nueva clase de elementos similares a los viroides , y "oblins", su grupo relacionado de proteínas, en el microbioma humano. [61] [62]
Un estudio de 20 sitios de la piel en cada uno de diez humanos sanos encontró 205 géneros identificados en 19 filos bacterianos, con la mayoría de las secuencias asignadas a cuatro filos: Actinomycetota (51,8%), Bacillota (24,4%), Pseudomonadota (16,5%) y Bacteroidota (6,3%). [63] Una gran cantidad de géneros de hongos están presentes en la piel humana sana, con cierta variabilidad según la región del cuerpo; sin embargo, durante las condiciones patológicas, ciertos géneros tienden a dominar en la región afectada. [48] Por ejemplo, Malassezia es dominante en la dermatitis atópica y Acremonium es dominante en los cueros cabelludos afectados por caspa. [48]
La piel actúa como una barrera para impedir la invasión de microbios patógenos. La piel humana contiene microbios que residen en la piel o sobre ella y pueden ser residentes o transitorios. Los tipos de microorganismos residentes varían en relación con el tipo de piel del cuerpo humano. La mayoría de los microbios residen en células superficiales de la piel o prefieren asociarse con glándulas. Estas glándulas, como las glándulas sebáceas o sudoríparas, proporcionan a los microbios agua, aminoácidos y ácidos grasos. Además, las bacterias residentes que se asocian con las glándulas sebáceas suelen ser grampositivas y pueden ser patógenas. [2]
Una pequeña cantidad de bacterias y hongos están normalmente presentes en la conjuntiva . [48] [64] Las clases de bacterias incluyen cocos Gram-positivos (p. ej., Staphylococcus y Streptococcus ) y bacilos y cocos Gram-negativos (p. ej., Haemophilus y Neisseria ). [64] Los géneros de hongos incluyen Candida , Aspergillus y Penicillium . [48] Las glándulas lagrimales secretan continuamente, manteniendo la conjuntiva húmeda, mientras que el parpadeo intermitente lubrica la conjuntiva y lava el material extraño. Las lágrimas contienen bactericidas como la lisozima , por lo que los microorganismos tienen dificultad para sobrevivir a la lisozima y asentarse en las superficies epiteliales .
En los seres humanos, la composición del microbioma gastrointestinal se establece durante el nacimiento. [69] El nacimiento por cesárea o parto vaginal también influye en la composición microbiana del intestino. Los bebés nacidos por vía vaginal tienen una microbiota intestinal beneficiosa y no patógena similar a la que se encuentra en la madre. [70] Sin embargo, la microbiota intestinal de los bebés nacidos por cesárea alberga más bacterias patógenas como Escherichia coli y Staphylococcus y lleva más tiempo desarrollar una microbiota intestinal beneficiosa y no patógena. [71]
La relación entre algunas microbiotas intestinales y los humanos no es meramente comensal (una coexistencia no dañina), sino más bien una relación mutualista . [2] Algunos microorganismos intestinales humanos benefician al huésped fermentando la fibra dietética en ácidos grasos de cadena corta (AGCC), como el ácido acético y el ácido butírico , que luego son absorbidos por el huésped. [4] [72] Las bacterias intestinales también juegan un papel en la síntesis de vitamina B y vitamina K , así como en el metabolismo de ácidos biliares , esteroles y xenobióticos . [2] [72] La importancia sistémica de los AGCC y otros compuestos que producen son como las hormonas y la propia flora intestinal parece funcionar como un órgano endocrino , [72] y la desregulación de la flora intestinal se ha correlacionado con una serie de condiciones inflamatorias y autoinmunes. [4] [73]
La composición de la microbiota intestinal humana cambia con el tiempo, cuando cambia la dieta y a medida que cambia la salud general. [4] [73] Una revisión sistemática de 15 ensayos controlados aleatorios humanos de julio de 2016 encontró que ciertas cepas disponibles comercialmente de bacterias probióticas de los géneros Bifidobacterium y Lactobacillus ( B. longum , B. breve , B. infantis , L. helveticus , L. rhamnosus , L. plantarum y L. casei ), cuando se toman por vía oral en dosis diarias de 10 9 –10 10 unidades formadoras de colonias (UFC) durante 1 a 2 meses, poseen eficacia de tratamiento (es decir, mejora los resultados conductuales) en ciertos trastornos del sistema nervioso central , incluyendo ansiedad , depresión , trastorno del espectro autista y trastorno obsesivo-compulsivo , y mejoran ciertos aspectos de la memoria . [74]
El sistema genitourinario parece tener una microbiota, [75] [76] lo cual es un hallazgo inesperado a la luz del uso de larga data de métodos de cultivo microbiológico clínico estándar para detectar bacterias en la orina cuando las personas muestran signos de una infección del tracto urinario ; es común que estas pruebas no muestren bacterias presentes. [77] Parece que los métodos de cultivo comunes no detectan muchos tipos de bacterias y otros microorganismos que normalmente están presentes. [77] A partir de 2017, se utilizaron métodos de secuenciación para identificar estos microorganismos para determinar si existen diferencias en la microbiota entre las personas con problemas del tracto urinario y las que están sanas. [75] [76] [78] Para evaluar adecuadamente el microbioma de la vejiga en oposición al sistema genitourinario, la muestra de orina debe recolectarse directamente de la vejiga, lo que a menudo se hace con un catéter . [79]
La microbiota vaginal se refiere a aquellas especies y géneros que colonizan la vagina. Estos organismos juegan un papel importante en la protección contra infecciones y el mantenimiento de la salud vaginal. [80] Los microorganismos vaginales más abundantes encontrados en mujeres premenopáusicas son del género Lactobacillus , que suprimen los patógenos produciendo peróxido de hidrógeno y ácido láctico. [50] [80] [81] La composición y las proporciones de las especies bacterianas varían dependiendo de la etapa del ciclo menstrual . [82] [83] [ necesita actualización ] La etnia también influye en la flora vaginal. La aparición de lactobacilos productores de peróxido de hidrógeno es menor en mujeres afroamericanas y el pH vaginal es más alto. [84] Otros factores influyentes como las relaciones sexuales y los antibióticos se han relacionado con la pérdida de lactobacilos. [81] Además, los estudios han encontrado que las relaciones sexuales con condón parecen cambiar los niveles de lactobacilos y aumentan el nivel de Escherichia coli dentro de la flora vaginal. [81] Los cambios en la microbiota vaginal normal y saludable son una indicación de infecciones, [85] como candidiasis o vaginosis bacteriana . [81] Candida albicans inhibe el crecimiento de especies de Lactobacillus , mientras que las especies de Lactobacillus que producen peróxido de hidrógeno inhiben el crecimiento y la virulencia de Candida albicans tanto en la vagina como en el intestino. [48] [50] [51]
Los géneros de hongos que se han detectado en la vagina incluyen Candida , Pichia , Eurotium , Alternaria , Rhodotorula y Cladosporium , entre otros. [48]
Hasta hace poco, se consideraba que la placenta era un órgano estéril, pero se han identificado especies y géneros de bacterias comensales no patógenas que residen en el tejido placentario. [86] [87] [88] Sin embargo, la existencia de un microbioma en la placenta es controvertida, ya que ha sido criticada en varias investigaciones. El llamado "microbioma placentario" probablemente se deriva de la contaminación de los reactivos, ya que las muestras de baja biomasa se contaminan fácilmente. [89] [90] [91]
Hasta hace poco, se consideraba que el tracto reproductivo superior de la mujer era un entorno estéril. Una variedad de microorganismos habitan en el útero de mujeres sanas y asintomáticas en edad reproductiva. El microbioma del útero difiere significativamente del de la vagina y el tracto gastrointestinal. [92]
El ambiente presente en la boca humana permite el crecimiento de los microorganismos característicos que allí se encuentran. Proporciona una fuente de agua y nutrientes, así como una temperatura moderada. [2] Los microbios residentes de la boca se adhieren a los dientes y las encías para resistir el lavado mecánico desde la boca hasta el estómago, donde los microbios sensibles al ácido son destruidos por el ácido clorhídrico . [2] [50]
Las bacterias anaeróbicas en la cavidad oral incluyen: Actinomyces , Arachnia , Bacteroides , Bifidobacterium , Eubacterium , Fusobacterium , Lactobacillus , Leptotrichia , Peptococcus , Peptostreptococcus , Propionibacterium , Selenomonas , Treponema y Veillonella . [93] [ necesita actualización ] Los géneros de hongos que se encuentran frecuentemente en la boca incluyen Candida , Cladosporium , Aspergillus , Fusarium , Glomus , Alternaria , Penicillium y Cryptococcus , entre otros. [48]
Las bacterias se acumulan en los tejidos bucales duros y blandos formando una biopelícula que les permite adherirse y moverse en el entorno bucal mientras están protegidas de los factores ambientales y los agentes antimicrobianos. [94] La saliva desempeña un papel homeostático clave en la biopelícula, ya que permite la recolonización de las bacterias para su formación y controla el crecimiento mediante el desprendimiento de la acumulación de biopelícula. [95] También proporciona un medio de regulación de la temperatura y los nutrientes. La ubicación de la biopelícula determina el tipo de nutrientes expuestos que recibe. [96]
Las bacterias bucales han desarrollado mecanismos para percibir su entorno y evadir o modificar al huésped. Sin embargo, un sistema de defensa innato del huésped altamente eficiente monitorea constantemente la colonización bacteriana y previene la invasión bacteriana de los tejidos locales. Existe un equilibrio dinámico entre las bacterias de la placa dental y el sistema de defensa innato del huésped. [97]
Esta dinámica entre la cavidad oral del huésped y los microbios orales juega un papel clave en la salud y la enfermedad, ya que proporciona entrada al cuerpo. [98] Un equilibrio saludable presenta una relación simbiótica donde los microbios orales limitan el crecimiento y la adherencia de los patógenos mientras que el huésped proporciona un entorno para que florezcan. [98] [94] Los cambios ecológicos como el cambio del estado inmunológico, el cambio de microbios residentes y la disponibilidad de nutrientes cambian de una relación mutua a una parasitaria, lo que resulta en que el huésped sea propenso a enfermedades orales y sistémicas. [94] Las enfermedades sistémicas como la diabetes y las enfermedades cardiovasculares se han correlacionado con una mala salud bucal. [98] De particular interés es el papel de los microorganismos orales en las dos principales enfermedades dentales: caries dental y enfermedad periodontal . [97] La colonización de patógenos en el periodonto causa una respuesta inmune excesiva que resulta en una bolsa periodontal, un espacio más profundo entre el diente y la encía. [94] Esto actúa como un reservorio protegido rico en sangre con nutrientes para patógenos anaeróbicos. [94] La enfermedad sistémica en varios sitios del cuerpo puede ser resultado de microbios orales que ingresan a la sangre sin pasar por las bolsas periodontales y las membranas orales. [98]
Una higiene bucal adecuada y constante es el método principal para prevenir enfermedades orales y sistémicas. [98] Reduce la densidad de la biopelícula y el crecimiento excesivo de bacterias patógenas potenciales que provocan enfermedades. [96] Sin embargo, una higiene bucal adecuada puede no ser suficiente, ya que el microbioma oral, la genética y los cambios en la respuesta inmunitaria influyen en el desarrollo de infecciones crónicas. [96] El uso de antibióticos podría tratar una infección que ya se está propagando, pero no es eficaz contra las bacterias dentro de las biopelículas. [96]
El microbioma nasal sano está dominado por Corynebacterium y Staphylococcus. El microbioma mucoso desempeña un papel fundamental en la modulación de la infección viral. [99]
Al igual que la cavidad oral, el sistema respiratorio superior e inferior poseen disuasivos mecánicos para eliminar microbios. Las células caliciformes producen moco que atrapa microbios y los expulsa del sistema respiratorio a través de células epiteliales ciliadas en movimiento continuo . [2] Además, el moco nasal que contiene la enzima lisozima genera un efecto bactericida. [2] El tracto respiratorio superior e inferior parece tener su propio conjunto de microbiota. [100] La microbiota bacteriana pulmonar pertenece a 9 géneros bacterianos principales: Prevotella , Sphingomonas , Pseudomonas , Acinetobacter , Fusobacterium , Megasphaera , Veillonella , Staphylococcus y Streptococcus . Algunas de las bacterias consideradas "biota normal" en el tracto respiratorio pueden causar enfermedades graves, especialmente en individuos inmunodeprimidos; Estos incluyen Streptococcus pyogenes , Haemophilus influenzae , Streptococcus pneumoniae , Neisseria meningitidis y Staphylococcus aureus . [ cita requerida ] Los géneros de hongos que componen el micobioma pulmonar incluyen Candida , Malassezia , Neosartorya , Saccharomyces y Aspergillus , entre otros. [ 48 ]
En las personas con fibrosis quística se observan distribuciones inusuales de géneros bacterianos y fúngicos en el tracto respiratorio . [48] [101] Su flora bacteriana a menudo contiene bacterias resistentes a los antibióticos y de crecimiento lento, y la frecuencia de estos patógenos cambia en relación con la edad. [101]
Tradicionalmente se ha considerado que el tracto biliar es normalmente estéril y que la presencia de microorganismos en la bilis es un marcador de un proceso patológico. Esta suposición se confirmó por la falla en la asignación de cepas bacterianas del conducto biliar normal. En 2013 comenzaron a aparecer artículos que mostraban que la microbiota biliar normal es una capa funcional separada que protege al tracto biliar de la colonización por microorganismos exógenos. [102]
Los cuerpos humanos dependen de los innumerables genes bacterianos como fuente de nutrientes esenciales. [103] Tanto los estudios metagenómicos como los epidemiológicos indican funciones vitales para el microbioma humano en la prevención de una amplia gama de enfermedades, desde la diabetes tipo 2 y la obesidad hasta la enfermedad inflamatoria intestinal, la enfermedad de Parkinson e incluso afecciones de salud mental como la depresión. [104] Una relación simbiótica entre la microbiota intestinal y diferentes bacterias puede influir en la respuesta inmunitaria de un individuo. [105] Los metabolitos generados por los microbios intestinales parecen ser factores causales de la diabetes tipo 2. [106] Aunque está en sus inicios, el tratamiento basado en el microbioma también se muestra prometedor, sobre todo para el tratamiento de la infección por C. difficile resistente a los medicamentos [107] y en el tratamiento de la diabetes. [108]
Una presencia abrumadora de la bacteria, C. difficile, conduce a una infección del tracto gastrointestinal, normalmente asociada a disbiosis con la microbiota que se cree que ha sido causada por la administración de antibióticos. El uso de antibióticos erradica la flora intestinal beneficiosa dentro del tracto gastrointestinal, lo que normalmente evita que las bacterias patógenas establezcan el dominio. [109] El tratamiento tradicional para las infecciones por C. difficile incluye un régimen adicional de antibióticos, sin embargo, las tasas de eficacia promedian entre 20 y 30%. [110] Reconociendo la importancia de las bacterias intestinales saludables, los investigadores recurrieron a un procedimiento conocido como trasplante de microbiota fecal (FMT), donde los pacientes que experimentan enfermedades gastrointestinales, como la infección por C. difficile (CDI), reciben contenido fecal de un individuo sano con la esperanza de restaurar una microbiota intestinal que funcione normalmente. [111] El trasplante de microbiota fecal tiene aproximadamente un 85-90% de efectividad en personas con CDI para quienes los antibióticos no han funcionado o en quienes la enfermedad recurre después de los antibióticos. [112] [113] La mayoría de las personas con CDI se recuperan con un tratamiento de FMT. [114] [109] [115]
Aunque el cáncer es generalmente una enfermedad de la genética del huésped y de factores ambientales, los microorganismos están implicados en alrededor del 20% de los cánceres humanos. [116] En particular, en lo que respecta a los factores potenciales del cáncer de colon , la densidad bacteriana es un millón de veces mayor que en el intestino delgado , y se producen aproximadamente 12 veces más cánceres en el colon en comparación con el intestino delgado, lo que posiblemente establezca un papel patógeno para la microbiota en los cánceres de colon y recto . [117] La densidad microbiana puede utilizarse como herramienta de pronóstico en la evaluación de los cánceres colorrectales. [117]
La microbiota puede afectar la carcinogénesis de tres maneras generales: (i) alterando el equilibrio de la proliferación y muerte de células tumorales, (ii) regulando la función del sistema inmunológico, y (iii) influyendo en el metabolismo de factores producidos por el huésped, alimentos y productos farmacéuticos. [116] Los tumores que surgen en superficies limítrofes, como la piel, la orofaringe y los tractos respiratorio, digestivo y urogenital , albergan una microbiota. La presencia sustancial de microbios en un sitio de tumor no establece asociación o vínculos causales. En cambio, los microbios pueden encontrar tensión de oxígeno tumoral o perfil de nutrientes favorables. La disminución de las poblaciones de microbios específicos o el estrés oxidativo inducido también pueden aumentar los riesgos. [116] [117] De los aproximadamente 10 30 microbios en la Tierra, diez están designados por la Agencia Internacional para la Investigación sobre el Cáncer como carcinógenos humanos. [116] Los microbios pueden secretar proteínas u otros factores que impulsan directamente la proliferación celular en el huésped, o pueden regular al alza o a la baja el sistema inmunológico del huésped, lo que incluye impulsar la inflamación aguda o crónica de maneras que contribuyen a la carcinogénesis. [116]
En cuanto a la relación entre la función inmunitaria y el desarrollo de la inflamación, las barreras de la superficie mucosa están sujetas a riesgos ambientales y deben repararse rápidamente para mantener la homeostasis . La resiliencia comprometida del huésped o de la microbiota también reduce la resistencia a la malignidad, posiblemente induciendo inflamación y cáncer. Una vez que se rompen las barreras, los microbios pueden provocar programas proinflamatorios o inmunosupresores a través de varias vías. [116] Por ejemplo, los microbios asociados al cáncer parecen activar la señalización de NF-κΒ dentro del microambiente tumoral. Otros receptores de reconocimiento de patrones, como los miembros de la familia del receptor similar al dominio de oligomerización de unión a nucleótidos (NLR) NOD-2 , NLRP3 , NLRP6 y NLRP12 , pueden desempeñar un papel en la mediación del cáncer colorrectal. [116] Asimismo, Helicobacter pylori parece aumentar el riesgo de cáncer gástrico, debido a que impulsa una respuesta inflamatoria crónica en el estómago. [116] [117]
La enfermedad inflamatoria intestinal consta de dos enfermedades diferentes: la colitis ulcerosa y la enfermedad de Crohn, y ambas enfermedades se presentan con alteraciones en la microbiota intestinal (también conocida como disbiosis ). Esta disbiosis se presenta en forma de disminución de la diversidad microbiana en el intestino, [118] [119] y se correlaciona con defectos en los genes del huésped que cambian la respuesta inmune innata en los individuos. [118]
La progresión de la enfermedad por VIH influye en la composición y función de la microbiota intestinal, con diferencias notables entre las poblaciones VIH-negativas, VIH-positivas y VIH-positivas post- TAR . [ cita requerida ] El VIH disminuye la integridad de la función de barrera epitelial intestinal al afectar las uniones estrechas . Esta degradación permite la translocación a través del epitelio intestinal, lo que se cree que contribuye al aumento de la inflamación observada en personas con VIH. [ 120 ]
La microbiota vaginal desempeña un papel en la infectividad del VIH, con un mayor riesgo de infección y transmisión cuando la mujer tiene vaginosis bacteriana , una afección caracterizada por un equilibrio anormal de bacterias vaginales. [121] La mayor infectividad se observa con el aumento de citocinas proinflamatorias y células CCR5 + CD4+ en la vagina. Sin embargo, se observa una disminución de la infectividad con el aumento de los niveles de Lactobacillus vaginal , que promueve una condición antiinflamatoria. [120]
Los seres humanos que tienen 100 años o más, llamados centenarios , tienen un microbioma intestinal distintivo. Este microbioma se caracteriza por estar enriquecido con microorganismos capaces de sintetizar nuevos ácidos biliares secundarios . [122] Estos ácidos biliares secundarios incluyen varias isoformas del ácido litocólico que pueden contribuir a un envejecimiento saludable. [122]
Con la muerte, el microbioma del cuerpo vivo colapsa y una composición diferente de microorganismos llamada necrobioma se establece como un componente activo importante del complejo proceso de descomposición física. Se cree que sus cambios predecibles a lo largo del tiempo son útiles para ayudar a determinar el momento de la muerte. [123] [124]
Estudios realizados en 2009 cuestionaron si la disminución de la biota (incluida la microfauna ) como resultado de la intervención humana podría obstaculizar la salud humana, los procedimientos de seguridad hospitalaria, el diseño de productos alimenticios y los tratamientos de enfermedades. [125]

Higiene , [127] probióticos , [126] prebióticos , [128] simbióticos , [129] trasplantes de microbiota ( fecal [130] o piel [131] ), antibióticos , [132] ejercicio , [133] [134] dieta , [135] lactancia materna , [136] envejecimiento [137] pueden cambiar el microbioma humano en varios sistemas anatómicos o regiones como la piel y el intestino.
El microbioma humano se transmite entre una madre y sus hijos , así como entre personas que viven en el mismo hogar . [138] [139]
Las investigaciones primarias indican que pueden ocurrir cambios inmediatos en la microbiota cuando una persona migra de un país a otro, como cuando los inmigrantes tailandeses se establecieron en los Estados Unidos [140] o cuando los latinoamericanos inmigraron a los Estados Unidos. [141] Las pérdidas de diversidad de la microbiota fueron mayores en individuos obesos y en los hijos de inmigrantes . [140] [141]
Un estudio de 2024 sugiere que la microbiota intestinal capaz de digerir celulosa se puede encontrar en el microbioma humano, y es menos abundante en las personas que viven en sociedades industrializadas . [142] [143]
Microbioma
Este término se refiere a todo el hábitat, incluidos los microorganismos (bacterias, arqueas, eucariotas inferiores y superiores y virus), sus genomas (es decir, genes) y las condiciones ambientales circundantes. Esta definición se basa en la de "bioma", los factores bióticos y abióticos de entornos determinados. Otros en el campo limitan la definición de microbioma a la colección de genes y genomas de miembros de una microbiota. Se argumenta que esta es la definición de
metagenoma
, que combinado con el entorno constituye el microbioma.
Revisamos la literatura sobre la trimetilamina (TMA), un metabolito generado por la microbiota vinculado al desarrollo de la aterosclerosis.
La trimetilamina es exclusivamente un producto derivado de la microbiota de los nutrientes (lecitina, colina, TMAO, L-carnitina) de la dieta normal, de la que parece originarse dos enfermedades, la trimetilaminuria (o síndrome del olor a pescado) y la enfermedad cardiovascular a través de la propiedad proaterogénica de su forma oxidada derivada del hígado.
de Candida
y otros microorganismos están involucrados en esta complicada infección fúngica, pero
Candida albicans
sigue siendo la más prevalente. En las últimas dos décadas, se ha observado un sobrecrecimiento anormal en los tractos gastrointestinal, urinario y respiratorio, no solo en pacientes inmunodeprimidos, sino también relacionado con infecciones nosocomiales e incluso en individuos sanos. Existe una amplia variedad de factores causales que contribuyen a la infección por hongos, lo que significa que la candidiasis es un buen ejemplo de un síndrome multifactorial.
Además, se informa de infecciones fúngicas GI incluso entre aquellos pacientes con estado inmunológico normal. Las infecciones fúngicas relacionadas con el sistema digestivo pueden ser inducidas tanto por hongos oportunistas comensales como por hongos patógenos exógenos. ...
Candida
sp. también es la especie identificada con mayor frecuencia entre pacientes con IFI gástrica. ... Alguna vez se creyó que el ácido gástrico podía matar los microbios que ingresaban al estómago y que el entorno ecológico único del estómago no era adecuado para la colonización o infección microbiana. Sin embargo, varios estudios que utilizan métodos independientes del cultivo confirmaron que en el estómago existen grandes cantidades de bacterias resistentes al ácido pertenecientes a ocho filos y hasta 120 especies, como
Streptococcus
sp.,
Neisseria
sp. y
Lactobacillus
sp. etc.
26, 27
Además,
Candida albicans
puede crecer bien en ambientes altamente ácidos,
28
y algunos genotipos pueden aumentar la gravedad de las lesiones de la mucosa gástrica.
29
El sobrecrecimiento fúngico en el intestino delgado (SIFO) se caracteriza por la presencia de una cantidad excesiva de organismos fúngicos en el intestino delgado asociados con síntomas gastrointestinales (GI). Se sabe que la candidiasis causa síntomas GI particularmente en pacientes inmunodeprimidos o en aquellos que reciben esteroides o antibióticos. Sin embargo, solo recientemente, ha surgido literatura que indica que un sobrecrecimiento de hongos en el intestino delgado de sujetos no inmunodeprimidos puede causar síntomas GI inexplicables. Dos estudios recientes mostraron que el 26 % (24/94) y el 25,3 % (38/150) de una serie de pacientes con síntomas GI inexplicables tenían SIFO. Los síntomas más comunes observados en estos pacientes fueron eructos, distensión abdominal, indigestión, náuseas, diarrea y gases. ... La interacción entre hongos y bacterias puede actuar de diferentes maneras y puede ser sinérgica, antagónica o simbiótica [29]. Algunas bacterias, como las especies
de Lactobacillus,
pueden interactuar e inhibir tanto la virulencia como el crecimiento de las especies
de Candida
en el intestino al producir peróxido de hidrógeno [30]. Cualquier daño a la barrera mucosa o alteración de la microbiota GI con quimioterapia o uso de antibióticos, procesos inflamatorios, activación de moléculas inmunes y alteración de la reparación epitelial pueden causar sobrecrecimiento fúngico [27].
Lactobacillus
convierten el triptófano en indol-3-aldehído (I3A) a través de enzimas no identificadas [125].
Clostridium sporogenes
convierte el triptófano en IPA [6], probablemente a través de una triptófano desaminasa. ... El IPA también elimina de forma potente los radicales hidroxilo
Se demostró que la producción de IPA depende completamente de la presencia de microflora intestinal y podría establecerse mediante la colonización con la bacteria
Clostridium sporogenes
.
[El ácido indol-3-propiónico (IPA)] se ha identificado previamente en el plasma y el líquido cefalorraquídeo de humanos, pero sus funciones no se conocen. ... En experimentos de competencia cinética con agentes que atrapan radicales libres, la capacidad del IPA para eliminar radicales hidroxilo superó a la de la melatonina, una indolamina considerada el eliminador natural de radicales libres más potente. A diferencia de otros antioxidantes, el IPA no se convirtió en intermediarios reactivos con actividad prooxidante.
Estos probióticos demostraron eficacia para mejorar las conductas relacionadas con trastornos psiquiátricos, como ansiedad, depresión, trastorno del espectro autista (TEA), trastorno obsesivo-compulsivo y capacidades de memoria, incluida la memoria espacial y no espacial. Debido a que muchos de los estudios de ciencia básica demostraron cierta eficacia de los probióticos en la función del sistema nervioso central, estos antecedentes pueden guiar y promover estudios preclínicos y clínicos adicionales. ... Según los análisis cualitativos de los estudios actuales, podemos concluir provisionalmente que B. longum, B. breve, B. infantis, L. helveticus, L. rhamnosus, L. plantarum y L. casei fueron los más eficaces para mejorar la función del SNC, incluidas las funciones asociadas a enfermedades psiquiátricas (ansiedad, depresión, estado de ánimo, respuesta al estrés) y las capacidades de memoria.
{{cite journal}}: CS1 maint: DOI inactive as of June 2024 (link){{cite web}}: CS1 maint: numeric names: authors list (link)