La epigenética del cáncer es el estudio de las modificaciones epigenéticas del ADN de las células cancerosas que no implican un cambio en la secuencia de nucleótidos, sino un cambio en la forma en que se expresa el código genético. Los mecanismos epigenéticos son necesarios para mantener secuencias normales de expresión génica específica de tejido y son cruciales para el desarrollo normal. [1] Pueden ser tan importantes, si no más importantes, que las mutaciones genéticas en la transformación de una célula en cáncer. La alteración de los procesos epigenéticos en los cánceres puede conducir a una pérdida de expresión de genes que ocurre aproximadamente 10 veces más frecuentemente por silenciamiento de la transcripción (causado por la hipermetilación del promotor epigenético de las islas CpG ) que por mutaciones. Como señala Vogelstein et al., en un cáncer colorrectal generalmente hay alrededor de 3 a 6 mutaciones conductoras y 33 a 66 mutaciones autoestopistas o pasajeras. [2] Sin embargo, en los tumores de colon en comparación con la mucosa colónica de apariencia normal adyacente, hay alrededor de 600 a 800 islas CpG fuertemente metiladas en los promotores de genes en los tumores, mientras que estas islas CpG no están metiladas en la mucosa adyacente. [3] [4] [5] La manipulación de alteraciones epigenéticas es muy prometedora para la prevención, detección y terapia del cáncer. [6] [7] En diferentes tipos de cáncer, se pueden perturbar diversos mecanismos epigenéticos, como el silenciamiento de genes supresores de tumores y la activación de oncogenes por patrones de metilación de islas CpG alterados, modificaciones de histonas y desregulación de proteínas de unión al ADN . Hay varios medicamentos que tienen un impacto epigenético, que ahora se utilizan en varias de estas enfermedades.


En las células somáticas, los patrones de metilación del ADN se transmiten en general a las células hijas con alta fidelidad. [8] Por lo general, esta metilación solo ocurre en las citosinas que se encuentran en posición 5' respecto a la guanosina en los dinucleótidos CpG de los eucariotas de orden superior. [9] Sin embargo, la metilación epigenética del ADN difiere entre las células normales y las células tumorales en los seres humanos. El perfil de metilación de CpG "normal" a menudo se invierte en las células que se vuelven tumorígenas. [10] En las células normales, las islas CpG que preceden a los promotores de genes generalmente no están metiladas y tienden a ser transcripcionalmente activas, mientras que otros dinucleótidos CpG individuales en todo el genoma tienden a estar metilados. Sin embargo, en las células cancerosas, las islas CpG que preceden a los promotores de genes supresores de tumores a menudo están hipermetiladas, mientras que la metilación de CpG de las regiones promotoras de oncogenes y las secuencias de repetición parásitas a menudo está disminuida. [11]
La hipermetilación de las regiones promotoras de genes supresores de tumores puede provocar el silenciamiento de esos genes. Este tipo de mutación epigenética permite que las células crezcan y se reproduzcan sin control, lo que conduce a la tumorogénesis. [10] La adición de grupos metilo a las citosinas hace que el ADN se enrolle firmemente alrededor de las proteínas histonas, lo que da como resultado un ADN que no puede experimentar transcripción (ADN silenciado transcripcionalmente). Los genes que se encuentran comúnmente silenciados transcripcionalmente debido a la hipermetilación del promotor incluyen: inhibidor de la cinasa dependiente de ciclina p16 , un inhibidor del ciclo celular; MGMT , un gen de reparación del ADN ; APC , un regulador del ciclo celular; MLH1 , un gen de reparación del ADN; y BRCA1 , otro gen de reparación del ADN. [10] [12] De hecho, las células cancerosas pueden volverse adictas al silenciamiento transcripcional, debido a la hipermetilación del promotor, de algunos genes supresores de tumores clave, un proceso conocido como adicción epigenética. [13]
La hipometilación de dinucleótidos CpG en otras partes del genoma conduce a la inestabilidad cromosómica debido a mecanismos como la pérdida de impronta y la reactivación de elementos transponibles . [14] [15] [16] [17] La pérdida de impronta del gen del factor de crecimiento similar a la insulina (IGF2) aumenta el riesgo de cáncer colorrectal y está asociada con el síndrome de Beckwith-Wiedemann, que aumenta significativamente el riesgo de cáncer en los recién nacidos. [18] En células sanas, los dinucleótidos CpG de densidades más bajas se encuentran dentro de las regiones intergénicas codificantes y no codificantes . La expresión de algunas secuencias repetitivas y la recombinación meiótica en los centrómeros se reprimen a través de la metilación [19]
El genoma completo de una célula cancerosa contiene significativamente menos metilcitosina que el genoma de una célula sana. De hecho, los genomas de células cancerosas tienen entre un 20 y un 50 % menos de metilación en dinucleótidos CpG individuales en todo el genoma. [14] [ 15] [16] [17] Las islas CpG que se encuentran en las regiones promotoras suelen estar protegidas de la metilación del ADN. En las células cancerosas, las islas CpG están hipometiladas [20]. Las regiones que flanquean las islas CpG, llamadas orillas de las islas CpG, son donde se produce la mayor parte de la metilación del ADN en el contexto del dinucleótido CpG. Las células cancerosas se metilan de forma diferencial en las orillas de las islas CpG. En las células cancerosas, la hipermetilación en las orillas de las islas CpG se desplaza hacia las islas CpG, o la hipometilación de las islas CpG se desplaza hacia las orillas de las islas CpG, eliminando los límites epigenéticos nítidos entre estos elementos genéticos. [21] En las células cancerosas, la "hipometilación global" debida a la alteración de las metiltransferasas del ADN (DNMT) puede promover la recombinación mitótica y el reordenamiento cromosómico , lo que en última instancia produce aneuploidía cuando los cromosomas no se separan adecuadamente durante la mitosis . [14] [15] [16] [17]
La metilación de las islas CpG es importante para regular la expresión génica, pero la metilación de las citosinas puede conducir directamente a mutaciones genéticas desestabilizadoras y a un estado celular precanceroso. Las citosinas metiladas hacen que la hidrólisis del grupo amino y la conversión espontánea a timina sean más favorables. Pueden causar un reclutamiento aberrante de proteínas de la cromatina . Las metilaciones de las citosinas cambian la cantidad de absorción de luz ultravioleta de la base del nucleótido, creando dímeros de pirimidina . Cuando la mutación da como resultado la pérdida de heterocigosidad en los sitios de los genes supresores de tumores, estos genes pueden volverse inactivos. Las mutaciones de un solo par de bases durante la replicación también pueden tener efectos perjudiciales. [12]
El ADN eucariota tiene una estructura compleja. Generalmente está envuelto alrededor de proteínas especiales llamadas histonas para formar una estructura llamada nucleosoma . Un nucleosoma consta de 2 conjuntos de 4 histonas: H2A , H2B , H3 y H4 . Además, la histona H1 contribuye al empaquetamiento del ADN fuera del nucleosoma. Ciertas enzimas modificadoras de histonas pueden agregar o eliminar grupos funcionales a las histonas, y estas modificaciones influyen en el nivel de transcripción de los genes envueltos alrededor de esas histonas y el nivel de replicación del ADN. Los perfiles de modificación de histonas de células sanas y cancerosas tienden a diferir.
En comparación con las células sanas, las células cancerosas muestran formas monoacetiladas y trimetiladas disminuidas de la histona H4 (H4ac y H4me3 disminuidos). [22] Además, los modelos de ratón han demostrado que una disminución en la dimetilación asimétrica de la histona H4R3 (H4R3me2a) del promotor p19ARF se correlaciona con casos más avanzados de tumorigénesis y metástasis. [23] En modelos de ratón, la pérdida de acetilación y trimetilación de la histona H4 aumenta a medida que continúa el crecimiento del tumor. [22] La pérdida de acetilación de la lisina 16 de la histona H4 ( H4K16ac ), que es una marca del envejecimiento en los telómeros , pierde específicamente su acetilación. Algunos científicos esperan que esta pérdida particular de acetilación de histonas pueda combatirse con un inhibidor de la histona desacetilasa (HDAC) específico para SIRT1 , un HDAC específico para H4K16. [10] [24]
Otras marcas de histonas asociadas con la tumorogénesis incluyen una mayor desacetilación (disminución de la acetilación) de las histonas H3 y H4, una menor trimetilación de la histona H3 lisina 4 ( H3K4me3 ) y una mayor monometilación de la histona H3 lisina 9 (H3K9me1) y trimetilación de la histona H3 lisina 27 ( H3K27me3 ). Estas modificaciones de histonas pueden silenciar genes supresores de tumores a pesar de la caída de la metilación de la isla CpG del gen (un evento que normalmente activa los genes). [25] [26]
Algunas investigaciones se han centrado en bloquear la acción de BRD4 sobre las histonas acetiladas, lo que ha demostrado aumentar la expresión de la proteína Myc , implicada en varios tipos de cáncer. El proceso de desarrollo del fármaco que se une a BRD4 es digno de mención por el enfoque colaborativo y abierto que está adoptando el equipo. [27]
El gen supresor de tumores p53 regula la reparación del ADN y puede inducir la apoptosis en células desreguladas. E Soto-Reyes y F Recillas-Targa explicaron la importancia de la proteína CTCF en la regulación de la expresión de p53. [28] CTCF, o factor de unión CCCTC, es una proteína con dedos de zinc que aísla al promotor p53 de la acumulación de marcas histónicas represivas. En ciertos tipos de células cancerosas, la proteína CTCF no se une normalmente y el promotor p53 acumula marcas histónicas represivas, lo que hace que la expresión de p53 disminuya. [28]
También pueden producirse mutaciones en la propia maquinaria epigenética, que podrían ser responsables de los cambios en los perfiles epigenéticos de las células cancerosas. Las variantes de histonas de la familia H2A están muy conservadas en los mamíferos y desempeñan papeles fundamentales en la regulación de muchos procesos nucleares alterando la estructura de la cromatina . Una de las variantes clave de H2A, H2A.X, marca el daño del ADN, lo que facilita el reclutamiento de proteínas de reparación del ADN para restaurar la integridad genómica. Otra variante, H2A.Z, desempeña un papel importante tanto en la activación como en la represión de genes. Se detecta un alto nivel de expresión de H2A.Z en muchos cánceres y está significativamente asociado con la proliferación celular y la inestabilidad genómica. [11] La variante de histona macroH2A1 es importante en la patogénesis de muchos tipos de cánceres, por ejemplo, en el carcinoma hepatocelular. [29] Otros mecanismos incluyen una disminución de H4K16ac que puede ser causada por una disminución de la actividad de una acetiltransferasa de histonas (HAT) o un aumento de la desacetilación por SIRT1. [10] Asimismo, una mutación inactivadora por cambio de marco en HDAC2 , una histona desacetilasa que actúa sobre muchas lisinas de cola de histona , se ha asociado con cánceres que muestran patrones alterados de acetilación de histonas. [30] Estos hallazgos indican un mecanismo prometedor para alterar los perfiles epigenéticos a través de la inhibición o mejora enzimática. Un nuevo campo emergente que captura los cambios epigenéticos toxicológicos como resultado de la exposición a diferentes compuestos (fármacos, alimentos y medio ambiente) es la toxicoepigenética. En este campo, existe un creciente interés en el mapeo de los cambios en las modificaciones de las histonas y sus posibles consecuencias. [31]
El daño del ADN , causado por la luz ultravioleta, la radiación ionizante , las toxinas ambientales y los productos químicos metabólicos, también puede provocar inestabilidad genómica y cáncer. La respuesta del daño del ADN a las roturas de doble cadena del ADN (DSB) está mediada en parte por modificaciones de las histonas. En una DSB, el complejo proteico MRE11 - RAD50 - NBS1 (MRN) recluta a la quinasa de ataxia telangiectasia mutada (ATM) que fosforila la serina 129 de la histona 2A. MDC1, mediador del punto de control 1 del daño del ADN, se une al fosfopéptido y la fosforilación de H2AX puede propagarse mediante un ciclo de retroalimentación positiva de reclutamiento y fosforilación de MRN-ATM. TIP60 acetila el γH2AX, que luego se poliubiquitina. RAP80, una subunidad del complejo proteico de susceptibilidad al cáncer de mama tipo 1 de reparación del ADN ( BRCA1 -A), se une a la ubiquitina unida a las histonas. La actividad de BRCA1-A detiene el ciclo celular en el punto de control G2/M , lo que da tiempo para la reparación del ADN, o puede iniciarse la apoptosis . [32]
En los mamíferos, los microARN (miARN) regulan alrededor del 60% de la actividad transcripcional de los genes que codifican proteínas. [33] Algunos miARN también sufren silenciamiento asociado a la metilación en las células cancerosas. [34] [35] Let-7 y miR15/16 desempeñan papeles importantes en la regulación negativa de los oncogenes RAS y BCL2 , y su silenciamiento ocurre en las células cancerosas. [18] Se observó una disminución de la expresión de miR-125b1, un miARN que funciona como supresor de tumores , en los cánceres de próstata, ovario , mama y células gliales . Los experimentos in vitro han demostrado que miR-125b1 se dirige a dos genes, HER2/neu y ESR1 , que están vinculados al cáncer de mama. La metilación del ADN, específicamente la hipermetilación, es una de las principales formas en que el miR-125b1 se silencia epigenéticamente. En pacientes con cáncer de mama, se observó hipermetilación de islas CpG ubicadas proximales al sitio de inicio de la transcripción. La pérdida de la unión de CTCF y un aumento en las marcas de histonas represivas, H3K9me3 y H3K27me3, se correlacionan con la metilación del ADN y el silenciamiento de miR-125b1. Mecanísticamente, CTCF puede funcionar como un elemento límite para detener la propagación de la metilación del ADN. Los resultados de los experimentos realizados por Soto-Reyes et al. [36] indican un efecto negativo de la metilación en la función y expresión de miR-125b1. Por lo tanto, concluyeron que la metilación del ADN tiene un papel en el silenciamiento del gen. Además, algunos miRNA se silencian epigenéticamente en las primeras etapas del cáncer de mama y, por lo tanto, estos miRNA podrían ser potencialmente útiles como marcadores tumorales. [36] El silenciamiento epigenético de genes de miRNA por metilación aberrante del ADN es un evento frecuente en las células cancerosas; Casi un tercio de los promotores de miRNA activos en células mamarias normales se encontraron hipermetilados en células de cáncer de mama, lo que representa una proporción varias veces mayor que la que se observa habitualmente para los genes codificadores de proteínas. [37]
La desregulación del metabolismo permite que las células tumorales generen los componentes básicos necesarios, así como que modulen las marcas epigenéticas para apoyar la iniciación y la progresión del cáncer. Los cambios metabólicos inducidos por el cáncer alteran el panorama epigenético, especialmente las modificaciones en las histonas y el ADN, promoviendo así la transformación maligna, la adaptación a una nutrición inadecuada y la metástasis. Para satisfacer las demandas biosintéticas de las células cancerosas, las vías metabólicas se alteran manipulando oncogenes y genes supresores de tumores simultáneamente. [38] La acumulación de ciertos metabolitos en el cáncer puede dirigirse a las enzimas epigenéticas para alterar globalmente el panorama epigenético. Los cambios metabólicos relacionados con el cáncer conducen a la recodificación específica del locus de las marcas epigenéticas. La epigenética del cáncer puede ser reprogramada con precisión por el metabolismo celular a través de 1) la modulación de la epigenética del cáncer en función de la dosis por metabolitos; 2) el reclutamiento específico de la secuencia de enzimas metabólicas; y 3) la orientación de las enzimas epigenéticas por señales nutricionales. [38] Además de modular la programación metabólica a nivel molecular, existen factores microambientales que pueden influir y afectar la recodificación metabólica. Estas influencias incluyen la respuesta nutricional, inflamatoria e inmunitaria de los tejidos malignos.
El daño del ADN parece ser la principal causa subyacente del cáncer. [39] [40] Si la reparación del ADN es deficiente, el daño del ADN tiende a acumularse. Este daño excesivo del ADN puede aumentar los errores mutacionales durante la replicación del ADN debido a la síntesis de translesión propensa a errores . El daño excesivo del ADN también puede aumentar las alteraciones epigenéticas debido a errores durante la reparación del ADN. [41] [42] Estas mutaciones y alteraciones epigenéticas pueden dar lugar al cáncer (ver neoplasias malignas ).
Las mutaciones de la línea germinal en los genes de reparación del ADN causan solo entre el 2 y el 5 % de los casos de cáncer de colon . [43] Sin embargo, la expresión alterada de microARN, que causa deficiencias en la reparación del ADN, se asocia con frecuencia con cánceres y puede ser un factor causal importante para estos cánceres.
La sobreexpresión de ciertos miRNA puede reducir directamente la expresión de proteínas específicas de reparación del ADN. Wan et al. [44] hicieron referencia a 6 genes de reparación del ADN que son el objetivo directo de los miRNA indicados entre paréntesis: ATM (miR-421), RAD52 (miR-210, miR-373), RAD23B (miR-373), MSH2 (miR-21), BRCA1 (miR-182) y P53 (miR-504, miR-125b). Más recientemente, Tessitore et al. [45] enumeraron otros genes de reparación de ADN que son el objetivo directo de otros miRNA, incluidos ATM (miR-18a, miR-101), DNA-PK (miR-101), ATR (miR-185), Wip1 (miR-16), MLH1, MSH2 y MSH6 (miR-155), ERCC3 y ERCC4 (miR-192) y UNG2 (mir-16, miR-34c y miR-199a). De estos miRNA, miR-16, miR-18a, miR-21, miR-34c, miR-125b, miR-101, miR-155, miR-182, miR-185 y miR-192 se encuentran entre los identificados por Schnekenburger y Diederich [46] como sobreexpresados en el cáncer de colon a través de la hipometilación epigenética. La sobreexpresión de cualquiera de estos miRNA puede provocar una expresión reducida de su gen de reparación del ADN objetivo.
Hasta el 15% de las deficiencias de MLH1 en cánceres de colon esporádicos parecían deberse a la sobreexpresión del microARN miR-155 , que reprime la expresión de MLH1. [47] Sin embargo, se encontró que la mayoría de los 68 cánceres de colon esporádicos con expresión reducida de la proteína de reparación de desajustes de ADN MLH1 eran deficientes debido a la metilación epigenética de la isla CpG del gen MLH1 . [48]
En el 28% de los glioblastomas, la proteína reparadora del ADN MGMT es deficiente pero el promotor MGMT no está metilado. [49] En los glioblastomas sin promotores MGMT metilados , el nivel de microARN miR-181d está inversamente correlacionado con la expresión proteica de MGMT y el objetivo directo de miR-181d es el ARNm 3'UTR de MGMT (la región no traducida principal del ARNm de MGMT). [49] Por lo tanto, en el 28% de los glioblastomas, el aumento de la expresión de miR-181d y la expresión reducida de la enzima reparadora del ADN MGMT pueden ser un factor causal. En el 29–66% [49] [50] de los glioblastomas , la reparación del ADN es deficiente debido a la metilación epigenética del gen MGMT , que reduce la expresión proteica de MGMT.
Las proteínas del grupo A de alta movilidad ( HMGA ), caracterizadas por un gancho AT , son proteínas pequeñas, no histonas, asociadas a la cromatina que pueden modular la transcripción. Los microARN controlan la expresión de las proteínas HMGA , y estas proteínas ( HMGA1 y HMGA2 ) son elementos arquitectónicos que controlan la transcripción de la cromatina. Palmieri et al. [51] demostraron que, en tejidos normales, los genes HGMA1 y HMGA2 son el objetivo (y por lo tanto reducen fuertemente su expresión) de miR-15 , miR-16 , miR-26a , miR-196a2 y Let-7a .
La expresión de HMGA es casi indetectable en tejidos adultos diferenciados, pero está elevada en muchos cánceres. Las proteínas HGMA son polipéptidos de ~100 residuos de aminoácidos caracterizados por una organización de secuencia modular. Estas proteínas tienen tres regiones altamente cargadas positivamente, denominadas ganchos AT , que se unen al surco menor de tramos de ADN ricos en AT en regiones específicas de ADN. Las neoplasias humanas, incluidos el carcinoma de tiroides, próstata, cuello uterino, colorrectal, pancreático y ovárico, muestran un fuerte aumento de las proteínas HMGA1a y HMGA1b. [52] Los ratones transgénicos con HMGA1 dirigido a células linfoides desarrollan un linfoma agresivo, lo que demuestra que la alta expresión de HMGA1 no solo está asociada con cánceres, sino que el gen HMGA1 puede actuar como un oncogén para causar cáncer. [53] Baldassarre et al., [54] demostraron que la proteína HMGA1 se une a la región promotora del gen de reparación de ADN BRCA1 e inhibe la actividad del promotor BRCA1 . También demostraron que, mientras que sólo el 11% de los tumores de mama tenían hipermetilación del gen BRCA1 , el 82% de los cánceres de mama agresivos tienen una baja expresión de la proteína BRCA1, y la mayoría de estas reducciones se debieron a la remodelación de la cromatina por altos niveles de proteína HMGA1.
La proteína HMGA2 se dirige específicamente al promotor de ERCC1 , reduciendo así la expresión de este gen de reparación del ADN. [55] La expresión de la proteína ERCC1 fue deficiente en el 100% de los 47 cánceres de colon evaluados (aunque se desconoce en qué medida estuvo involucrado HGMA2). [56]
Palmieri et al. [51] demostraron que cada uno de los miRNA que se dirigen a los genes HMGA se reducen drásticamente en casi todos los adenomas hipofisarios humanos estudiados, en comparación con la glándula pituitaria normal. En consonancia con la regulación negativa de estos miRNA que se dirigen a HMGA, se observó un aumento en los ARNm específicos de HMGA1 y HMGA2. Tres de estos microRNA (miR-16, miR-196a y Let-7a) [46] [57] tienen promotores metilados y, por lo tanto, baja expresión en el cáncer de colon. Para dos de estos, miR-15 y miR-16, las regiones codificantes están silenciadas epigenéticamente en el cáncer debido a la actividad de la histona desacetilasa . [58] Cuando estos microRNA se expresan a un nivel bajo, las proteínas HMGA1 y HMGA2 se expresan a un nivel alto. HMGA1 y HMGA2 se dirigen a (reducen la expresión de) los genes de reparación del ADN BRCA1 y ERCC1 . De esta manera, la reparación del ADN puede verse reducida, lo que probablemente contribuya a la progresión del cáncer. [40]

El gráfico de esta sección muestra algunos agentes que dañan el ADN con frecuencia, ejemplos de lesiones que causan y las vías que se ocupan de estos daños. Al menos 169 enzimas se emplean directamente en la reparación del ADN o influyen en los procesos de reparación del ADN. [59] De ellas, 83 se emplean directamente en la reparación de los 5 tipos de daños del ADN ilustrados en el gráfico.
En el gráfico se muestran algunos de los genes más estudiados que son fundamentales para estos procesos de reparación. Las designaciones de genes que se muestran en rojo, gris o cian indican genes que se alteran epigenéticamente con frecuencia en varios tipos de cáncer. Los artículos de Wikipedia sobre cada uno de los genes resaltados en rojo, gris o cian describen la(s) alteración(es) epigenética(s) y el(los) cáncer(es) en el que se encuentran estas epimutaciones. Dos amplios artículos de encuesta experimental [60] [61] también documentan la mayoría de estas deficiencias de reparación epigenética del ADN en los cánceres.
Los genes resaltados en rojo se reducen o silencian con frecuencia por mecanismos epigenéticos en varios tipos de cáncer. Cuando estos genes tienen una expresión baja o ausente, los daños en el ADN pueden acumularse. Los errores de replicación más allá de estos daños (ver síntesis por translesión ) pueden conducir a un aumento de mutaciones y, en última instancia, al cáncer. La represión epigenética de los genes de reparación del ADN en vías precisas de reparación del ADN parece ser fundamental para la carcinogénesis .
Los dos genes resaltados en gris , RAD51 y BRCA2 , son necesarios para la reparación recombinatoria homóloga . A veces se expresan en exceso y a veces en defecto en ciertos tipos de cáncer. Como se indica en los artículos de Wikipedia sobre RAD51 y BRCA2 , estos tipos de cáncer suelen tener deficiencias epigenéticas en otros genes de reparación del ADN. Estas deficiencias de reparación probablemente causarían un aumento de los daños no reparados en el ADN. La sobreexpresión de RAD51 y BRCA2 observada en estos tipos de cáncer puede reflejar presiones selectivas para la sobreexpresión compensatoria de RAD51 o BRCA2 y el aumento de la reparación recombinatoria homóloga para abordar al menos parcialmente estos daños excesivos en el ADN. En aquellos casos en los que RAD51 o BRCA2 están subexpresados, esto en sí mismo conduciría a un aumento de los daños no reparados en el ADN. Los errores de replicación más allá de estos daños (ver síntesis de translesión ) podrían causar un aumento de mutaciones y cáncer, por lo que la subexpresión de RAD51 o BRCA2 sería cancerígena en sí misma.
Los genes resaltados en cian se encuentran en la vía de unión de extremos mediada por microhomología (MMEJ) y se regulan positivamente en el cáncer. MMEJ es una vía adicional de reparación imprecisa propensa a errores para roturas de doble cadena. En la reparación MMEJ de una rotura de doble cadena, una homología de 5 a 25 pares de bases complementarios entre ambas cadenas emparejadas es suficiente para alinear las cadenas, pero generalmente hay extremos desapareados (flaps). MMEJ elimina los nucleótidos adicionales (flaps) donde se unen las cadenas y luego liga las cadenas para crear una doble hélice de ADN intacta. MMEJ casi siempre implica al menos una pequeña deleción, por lo que es una vía mutagénica. [62] FEN1 , la endonucleasa del colgajo en MMEJ, aumenta epigenéticamente por la hipometilación del promotor y se sobreexpresa en la mayoría de los cánceres de mama, [63] próstata, [64] estómago, [65] [66] neuroblastomas, [67] páncreas, [68] y pulmón. [69] PARP1 también se sobreexpresa cuando su sitio ETS de la región promotora está hipometilado epigenéticamente, y esto contribuye a la progresión al cáncer de endometrio, [70] cáncer de ovario con mutación BRCA, [71] y cáncer de ovario seroso con mutación BRCA. [72] Otros genes en la vía MMEJ también se sobreexpresan en varios cánceres (ver MMEJ para el resumen), y también se muestran en azul.
Las deficiencias en las proteínas de reparación del ADN que funcionan en vías precisas de reparación del ADN aumentan el riesgo de mutación. Las tasas de mutación aumentan considerablemente en células con mutaciones en la reparación de desajustes del ADN [73] [74] o en la reparación recombinatoria homóloga (HRR). [75] Las personas con mutaciones heredadas en cualquiera de los 34 genes de reparación del ADN tienen un mayor riesgo de cáncer (consulte Defectos de reparación del ADN y mayor riesgo de cáncer ).
En los cánceres esporádicos, ocasionalmente se encuentra que una deficiencia en la reparación del ADN se debe a una mutación en un gen de reparación del ADN, pero con mucha más frecuencia la expresión reducida o ausente de los genes de reparación del ADN se debe a alteraciones epigenéticas que reducen o silencian la expresión génica. Por ejemplo, para 113 cánceres colorrectales examinados en secuencia, solo cuatro tenían una mutación sin sentido en el gen de reparación del ADN MGMT , mientras que la mayoría tenía una expresión reducida de MGMT debido a la metilación de la región promotora MGMT (una alteración epigenética). [76] De manera similar, de 119 casos de cánceres colorrectales deficientes en reparación de desajustes que carecían de la expresión del gen de reparación del ADN PMS2 , la proteína PMS2 fue deficiente en 6 debido a mutaciones en el gen PMS2 , mientras que en 103 casos la expresión de PMS2 fue deficiente porque su pareja de emparejamiento MLH1 fue reprimida debido a la metilación del promotor (la proteína PMS2 es inestable en ausencia de MLH1). [48] En los otros 10 casos, la pérdida de la expresión de PMS2 probablemente se debió a la sobreexpresión epigenética del microARN, miR-155, que regula negativamente MLH1. [47]
Los defectos epigenéticos en los genes de reparación del ADN son frecuentes en los cánceres. En la tabla, se evaluaron múltiples cánceres para detectar la expresión reducida o ausente del gen de reparación del ADN de interés, y la frecuencia que se muestra es la frecuencia con la que los cánceres tenían una deficiencia epigenética de la expresión génica. Es probable que dichas deficiencias epigenéticas surjan en las primeras etapas de la carcinogénesis , ya que también se encuentran con frecuencia (aunque con una frecuencia algo menor) en el defecto de campo que rodea al cáncer del que probablemente surgió el cáncer (consulte la tabla).
Parece que los cánceres pueden iniciarse con frecuencia por una reducción epigenética en la expresión de una o más enzimas de reparación del ADN. La reparación reducida del ADN probablemente permite la acumulación de daños en el ADN. La síntesis de translesión propensa a errores más allá de algunos de estos daños del ADN puede dar lugar a una mutación con una ventaja selectiva. Un parche clonal con una ventaja selectiva puede crecer y superar a las células vecinas, formando un defecto de campo . Si bien no existe una ventaja selectiva obvia para una célula que tenga una reparación reducida del ADN, la epimutación del gen de reparación del ADN puede llevarse como pasajero cuando se replican las células con la mutación selectivamente ventajosa. En las células que llevan tanto la epimutación del gen de reparación del ADN como la mutación con la ventaja selectiva, se acumularán más daños en el ADN y estos, a su vez, podrían dar lugar a más mutaciones con ventajas selectivas aún mayores. Los defectos epigenéticos en la reparación del ADN pueden contribuir así a la alta frecuencia característica de mutaciones en los genomas de los cánceres y causar su progresión carcinogénica.
Los cánceres tienen altos niveles de inestabilidad genómica , asociada con una alta frecuencia de mutaciones . Una alta frecuencia de mutaciones genómicas aumenta la probabilidad de que ocurran mutaciones particulares que activan oncogenes e inactivan genes supresores de tumores, lo que lleva a la carcinogénesis . Sobre la base de la secuenciación del genoma completo , se ha descubierto que los cánceres tienen miles a cientos de miles de mutaciones en sus genomas completos. [87] (Véase también Frecuencias de mutación en cánceres ). En comparación, la frecuencia de mutación en el genoma completo entre generaciones de seres humanos (de padre a hijo) es de unas 70 nuevas mutaciones por generación. [88] [89] En las regiones codificantes de proteínas del genoma, solo hay unas 0,35 mutaciones entre generaciones de padre/hijo (menos de una proteína mutada por generación). [90] La secuenciación del genoma completo en células sanguíneas de un par de gemelos idénticos centenarios de 100 años solo encontró 8 diferencias somáticas, aunque la variación somática que ocurre en menos del 20% de las células sanguíneas no se detectaría. [91]
Si bien los daños en el ADN pueden dar lugar a mutaciones a través de una síntesis de translesión propensa a errores , los daños en el ADN también pueden dar lugar a alteraciones epigenéticas durante procesos de reparación del ADN defectuosos. [41] [42] [92] [93] Los daños en el ADN que se acumulan debido a defectos de reparación del ADN epigenético pueden ser una fuente del aumento de las alteraciones epigenéticas encontradas en muchos genes en los cánceres. En un estudio temprano, que observó un conjunto limitado de promotores transcripcionales, Fernández et al. [94] examinaron los perfiles de metilación del ADN de 855 tumores primarios. Comparando cada tipo de tumor con su tejido normal correspondiente, 729 sitios de islas CpG (55% de los 1322 sitios CpG evaluados) mostraron una metilación diferencial del ADN. De estos sitios, 496 estaban hipermetilados (reprimidos) y 233 estaban hipometilados (activados). Por lo tanto, existe un alto nivel de alteraciones de metilación del promotor epigenético en los tumores. Algunas de estas alteraciones epigenéticas pueden contribuir a la progresión del cáncer.
Se considera que una variedad de compuestos son carcinógenos epigenéticos : dan lugar a una mayor incidencia de tumores, pero no muestran actividad mutágena (los compuestos tóxicos o patógenos que causan tumores como consecuencia de una mayor regeneración también deben excluirse). Algunos ejemplos incluyen dietilestilbestrol , arsenito , hexaclorobenceno y compuestos de níquel .
Muchos teratógenos ejercen efectos específicos sobre el feto por mecanismos epigenéticos. [95] [96] Si bien los efectos epigenéticos pueden preservar el efecto de un teratógeno como el dietilestilbestrol durante toda la vida de un niño afectado, la posibilidad de defectos de nacimiento resultantes de la exposición de los padres o en la segunda y sucesivas generaciones de descendencia generalmente se ha rechazado por razones teóricas y por falta de evidencia. [97] Sin embargo, se ha demostrado una variedad de anomalías mediadas por los hombres, y es probable que existan más. [98] La información de la etiqueta de la FDA para Vidaza, una formulación de 5-azacitidina (un análogo no metilable de la citidina que causa hipometilación cuando se incorpora al ADN) establece que "se debe advertir a los hombres que no engendren un hijo" mientras usan el medicamento, citando evidencia en ratones machos tratados de fertilidad reducida, mayor pérdida de embriones y desarrollo anormal del embrión. [99] En ratas, se observaron diferencias endocrinas en la descendencia de machos expuestos a la morfina. [100] En ratones, se han descrito efectos de segunda generación del dietilestilbesterol que ocurren por mecanismos epigenéticos. [101]
El melanoma es un cáncer de piel mortal que se origina en los melanocitos. Se sabe que varias alteraciones epigenéticas desempeñan un papel en la transición de los melanocitos a células de melanoma. Esto incluye la metilación del ADN que se puede heredar sin realizar cambios en la secuencia de ADN, así como el silenciamiento de los genes supresores de tumores en la epidermis que han estado expuestas a la radiación UV durante períodos de tiempo. [102] El silenciamiento de los genes supresores de tumores conduce a la fotocarcinogénesis que está asociada a alteraciones epigenéticas en la metilación del ADN, las metiltransferasas del ADN y la acetilación de histonas. [102] Estas alteraciones son consecuencia de la desregulación de sus enzimas correspondientes. Varias metiltransferasas y desmetilasas de histonas se encuentran entre estas enzimas. [103]
El cáncer de próstata mata a unos 35.000 hombres al año, y alrededor de 220.000 hombres son diagnosticados con cáncer de próstata por año, solo en América del Norte. [104] El cáncer de próstata es la segunda causa principal de muertes por cáncer en hombres, y durante la vida de un hombre, uno de cada seis hombres tendrá la enfermedad. [104] Las alteraciones en la acetilación de histonas y la metilación del ADN ocurren en varios genes que influyen en el cáncer de próstata, y se han visto en genes involucrados en la respuesta hormonal. [105] Más del 90% de los cánceres de próstata muestran silenciamiento genético por hipermetilación de la isla CpG del promotor del gen GSTP1 , que protege a las células de la próstata del daño genómico causado por diferentes oxidantes o carcinógenos . [106] La reacción en cadena de la polimerasa (PCR) específica de metilación en tiempo real sugiere que muchos otros genes también están hipermetilados. [106] La expresión genética en la próstata puede ser modulada por cambios en la nutrición y el estilo de vida. [107]
El segundo tumor maligno más común en mujeres es el cáncer cervical invasivo (CCI) y más del 50% de todos los cánceres cervicales invasivos (CCI) son causados por el virus del papiloma humano oncogénico 16 ( HPV16 ). [108] Además, la neoplasia intraepitelial del cuello uterino (CIN) es causada principalmente por el HPV16 oncogénico. [108] Como en muchos casos, el factor causal del cáncer no siempre toma una ruta directa desde la infección hasta el desarrollo del cáncer. Los patrones de metilación genómica se han asociado con el cáncer cervical invasivo. Dentro de la región HPV16L1 , 14 sitios CpG probados tienen una metilación significativamente mayor en CIN3+ que en los genomas HPV16 de mujeres sin CIN3 . [108] Solo 2/16 sitios CpG probados en la región reguladora ascendente de HPV16 se encontraron asociados con una mayor metilación en CIN3+. [108] Esto sugiere que la ruta directa de la infección al cáncer a veces se desvía a un estado precanceroso en la neoplasia intraepitelial del cuello uterino. Además, se encontró una mayor metilación del sitio CpG en niveles bajos en la mayoría de los cinco genes nucleares del huésped estudiados, incluidos 5/5 TERT , 1/4 DAPK1 , 2/5 RARB , MAL y CADM1 . [108] Además, 1/3 de los sitios CpG en el ADN mitocondrial se asociaron con una mayor metilación en CIN3+. [108] Por lo tanto, existe una correlación entre CIN3+ y una mayor metilación de los sitios CpG en el marco de lectura abierto L1 de HPV16. [108] Esto podría ser un biomarcador potencial para futuras pruebas de detección de enfermedad cervical cancerosa y precancerosa. [108]
Estudios recientes han demostrado que el gen de la leucemia de linaje mixto (MLL) causa leucemia al reorganizarse y fusionarse con otros genes en diferentes cromosomas, lo que es un proceso bajo control epigenético. [109] Las mutaciones en MLL bloquean las regiones reguladoras correctas en las translocaciones o inserciones asociadas a la leucemia, lo que causa una transformación maligna controlada por los genes HOX. [110] Esto es lo que conduce al aumento de glóbulos blancos. Los genes relacionados con la leucemia son controlados por las mismas vías que controlan la epigenética, la transducción de señales, la regulación transcripcional y el metabolismo energético. Se indicó que las infecciones, los campos electromagnéticos y el aumento de peso al nacer pueden contribuir a ser las causas de la leucemia. [111]
En los EE. UU., se producen alrededor de 15 000 casos nuevos de sarcoma cada año y se estima que en 2014 morirán de sarcoma alrededor de 6200 personas en ese país. [112] Los sarcomas comprenden una gran cantidad de tumores mesenquimales histogenéticamente heterogéneos y poco frecuentes, como, por ejemplo, el condrosarcoma, el sarcoma de Ewing, el leiomiosarcoma, el liposarcoma, el osteosarcoma, el sarcoma sinovial y el rabdomiosarcoma (alveolar y embrionario). En los sarcomas, varios oncogenes y genes supresores de tumores se alteran epigenéticamente, como APC, CDKN1A, CDKN2A, CDKN2B, Ezrin, FGFR1, GADD45A, MGMT, STK3, STK4, PTEN, RASSF1A, WIF1, así como varios miRNA. [113] La expresión de modificadores epigenéticos como la del componente BMI1 del complejo PRC1 está desregulada en el condrosarcoma, el sarcoma de Ewing y el osteosarcoma, y la expresión del componente EZH2 del complejo PRC2 está alterada en el sarcoma de Ewing y el rabdomiosarcoma. De manera similar, la expresión de otro modificador epigenético, la histona desmetilasa LSD1, aumenta en el condrosarcoma, el sarcoma de Ewing, el osteosarcoma y el rabdomiosarcoma. La focalización farmacológica y la inhibición de EZH2 en el sarcoma de Ewing [114] , o de LSD1 en varios sarcomas [115] , inhibe el crecimiento de células tumorales en estos sarcomas.
El cáncer de pulmón es el segundo tipo de cáncer más común y la principal causa de muerte en hombres y mujeres en los Estados Unidos; se estima que hay alrededor de 216.000 casos nuevos y 160.000 muertes debido al cáncer de pulmón. [116]
La iniciación y progresión del carcinoma de pulmón es el resultado de la interacción entre factores genéticos, epigenéticos y ambientales. La mayoría de los casos de cáncer de pulmón se deben a mutaciones genéticas en EGFR , KRAS , STK11 (también conocido como LKB1 ), TP53 (también conocido como p53 ) y CDKN2A (también conocido como p16 o INK4a ) [117] [118] [119] siendo el tipo más común de cáncer de pulmón una inactivación en p16. p16 es una proteína supresora de tumores que se presenta principalmente en humanos. La importancia funcional de las mutaciones se probó en muchas otras especies, incluidos ratones, gatos, perros, monos y vacas. La identificación de estos múltiples clones no superpuestos no fue del todo sorprendente ya que la hibridación de rigurosidad reducida de un zoo blot con la misma sonda también reveló 10-15 fragmentos EcoRI positivos en todas las especies analizadas. [120]
Anteriormente, los perfiles epigenéticos se limitaban a genes individuales que un equipo de investigación en particular examinaba. Sin embargo, recientemente, los científicos han estado avanzando hacia un enfoque más genómico para determinar un perfil genómico completo de células cancerosas en comparación con células sanas. [10]
Los enfoques populares para medir la metilación de CpG en las células incluyen:
Dado que la secuenciación con bisulfito se considera el método de referencia para medir la metilación de CpG, cuando se utiliza uno de los otros métodos, los resultados suelen confirmarse mediante la secuenciación con bisulfito[1]. Los métodos populares para determinar los perfiles de modificación de histonas en células cancerosas frente a células sanas incluyen: [10]
Los investigadores esperan identificar perfiles epigenéticos específicos de varios tipos y subtipos de cáncer con el objetivo de utilizar estos perfiles como herramientas para diagnosticar a los individuos de forma más específica y precisa. [10] Dado que los perfiles epigenéticos cambian, a los científicos les gustaría utilizar los diferentes perfiles epigenómicos para determinar la etapa de desarrollo o el nivel de agresividad de un cáncer particular en los pacientes. Por ejemplo, la hipermetilación de los genes que codifican la proteína quinasa asociada a la muerte (DAPK), p16 y la proteína de membrana epitelial 3 (EMP3) se han relacionado con formas más agresivas de cáncer de pulmón , colorrectal y cerebral . [17] Este tipo de conocimiento puede afectar la forma en que los médicos diagnosticarán y elegirán tratar a sus pacientes.
Otro factor que influirá en el tratamiento de los pacientes es saber qué tan bien responderán a ciertos tratamientos. Los perfiles epigenómicos personalizados de las células cancerosas pueden proporcionar información en este campo. Por ejemplo, MGMT es una enzima que revierte la adición de grupos alquilo al nucleótido guanina . [121] Sin embargo, la alquilación de guanina es el mecanismo por el cual actúan varios fármacos quimioterapéuticos con el fin de alterar el ADN y causar la muerte celular . [122] [123] [124] [125] Por lo tanto, si el gen que codifica MGMT en las células cancerosas está hipermetilado y en efecto silenciado o reprimido, los fármacos quimioterapéuticos que actúan metilando la guanina serán más efectivos que en las células cancerosas que tienen una enzima MGMT funcional.
Los biomarcadores epigenéticos también pueden utilizarse como herramientas para el pronóstico molecular. En muestras de biopsia de tumores primarios y ganglios linfáticos mediastínicos , la hipermetilación de CDKN2A y CDH13 sirve como marcador de un mayor riesgo de recaída más rápida del cáncer y una mayor tasa de mortalidad de los pacientes. [126]
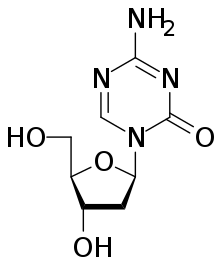
El control epigenético de las regiones proto-onco y las secuencias supresoras de tumores por cambios conformacionales en las histonas desempeña un papel en la formación y progresión del cáncer. [127] Los fármacos que revierten los cambios epigenéticos podrían tener un papel en una variedad de cánceres. [105] [127] [128]
Recientemente, se sabe claramente que las asociaciones entre histotipos específicos de cáncer y cambios epigenéticos pueden facilitar el desarrollo de nuevos fármacos epigenéticos. [129] El desarrollo de fármacos se ha centrado principalmente en modificar la ADN metiltransferasa , la histona acetiltransferasa (HAT) y la histona desacetilasa (HDAC). [130]
Los medicamentos que se dirigen específicamente al patrón de metilación invertido de las células cancerosas incluyen los inhibidores de la metiltransferasa del ADN azacitidina [131] [ 132] y decitabina [133] [134] Estos agentes hipometilantes se utilizan para tratar el síndrome mielodisplásico [135] , un cáncer de la sangre producido por células madre anormales de la médula ósea [12] Estos agentes inhiben los tres tipos de metiltransferasas del ADN activas y se pensaba que eran altamente tóxicos, pero demostraron ser efectivos cuando se usaron en dosis bajas, reduciendo la progresión del síndrome mielodisplásico a leucemia [136] .
Los inhibidores de la histona desacetilasa (HDAC) muestran eficacia en el tratamiento del linfoma de células T. Dos inhibidores de HDAC, vorinostat y romidepsina , han sido aprobados por la Administración de Alimentos y Medicamentos . [137] [138] Sin embargo, dado que estos inhibidores de HDAC alteran el estado de acetilación de muchas proteínas además de la histona de interés, se requiere conocimiento del mecanismo subyacente a nivel molecular de la respuesta del paciente para mejorar la eficiencia del uso de dichos inhibidores como tratamiento. [18] Se ha descubierto que el tratamiento con inhibidores de HDAC promueve la reactivación genética después de que los inhibidores de las metiltransferasas del ADN hayan reprimido la transcripción. [139] El panobinostat está aprobado para ciertas situaciones en el mieloma . [140]
Otros objetivos farmacéuticos en investigación son las histonas lisinas metiltransferasas (KMT) y las proteínas argininas metiltransferasas (PRMT). [141] Un estudio preclínico ha sugerido que la lunasina puede tener efectos epigenéticos potencialmente beneficiosos. [142]
La terapia epigenética del cáncer ha demostrado ser un tratamiento prometedor y posible para las células cancerosas. La inactivación epigenética es un objetivo ideal para las células cancerosas porque se dirige a los genes imprescindibles para controlar el crecimiento celular, específicamente el crecimiento de las células cancerosas. Es crucial que estos genes se reactiven para suprimir el crecimiento del tumor y sensibilizar a las células a las terapias curativas del cáncer. [143] La quimioterapia típica tiene como objetivo matar y eliminar las células cancerosas en el cuerpo. El cáncer iniciado por alteraciones genéticas de las células suele ser permanente y casi imposible de revertir, esto se diferencia del cáncer epigenético porque el cáncer que causa aberraciones epigenéticas tiene la capacidad de revertirse y las células pueden volver a funcionar normalmente. La capacidad de revertir los mecanismos epigenéticos se atribuye al hecho de que la codificación de los genes que se silencian mediante la modificación de histonas y ADN no se altera. [144]
Existen dos tipos principales de alteraciones epigenéticas en las células cancerosas, conocidas como metilación del ADN y modificación de histonas. El objetivo de las terapias epigenéticas es inhibir estas alteraciones. Las ADN metiltransferasas (DNMT) y las histonas desacetilasas (HDAC) son los catalizadores primarios de las modificaciones epigenéticas de las células cancerosas. [145] El objetivo de las terapias epigenéticas es reprimir esta metilación y revertir estas modificaciones para crear un nuevo epigenoma donde las células cancerosas ya no prosperen y la supresión tumoral sea la nueva función. Los fármacos sintéticos se utilizan como herramientas en las terapias epigenéticas debido a su capacidad para inhibir las enzimas que causan modificaciones de histonas y metilaciones del ADN. La terapia combinada es un método de terapia epigenética que implica el uso de más de un fármaco sintético, estos fármacos incluyen un inhibidor de DNMT de dosis baja, así como un inhibidor de HDAC. Juntos, estos fármacos pueden dirigirse al vínculo entre la metilación del ADN y la modificación de histonas. [146]
El objetivo de las terapias epigenéticas para el cáncer en relación con la metilación del ADN es tanto disminuir la metilación del ADN como, a su vez, disminuir el silenciamiento de los genes relacionados con la supresión tumoral. [147] El término asociado con la disminución de la metilación del ADN se conocerá como hipometilación. La Administración de Alimentos y Medicamentos (FDA) ha aprobado actualmente un agente hipometilante que, a través de la realización de ensayos clínicos, ha mostrado resultados prometedores cuando se utiliza para tratar a pacientes con síndrome mielodisplásico (SMD). [148] Este agente hipometilante se conoce como el análogo doozy de la 5-azacitidina y actúa para promover la hipometilación al dirigirse a todas las metiltransferasas del ADN para su degradación. [147]