La evolución molecular describe cómo el ADN y/o ARN heredados cambian a lo largo del tiempo evolutivo y las consecuencias de esto para las proteínas y otros componentes de las células y organismos . La evolución molecular es la base de los enfoques filogenéticos para describir el árbol de la vida . La evolución molecular se superpone con la genética de poblaciones , especialmente en escalas de tiempo más cortas. Los temas de la evolución molecular incluyen los orígenes de nuevos genes, la naturaleza genética de rasgos complejos , la base genética de la adaptación y la especiación , la evolución del desarrollo y los patrones y procesos subyacentes a los cambios genómicos durante la evolución.
La historia de la evolución molecular comienza a principios del siglo XX con la bioquímica comparada y el uso de métodos de "huellas dactilares" como ensayos inmunológicos, electroforesis en gel y cromatografía en papel en la década de 1950 para explorar proteínas homólogas . [1] [2] La llegada de la secuenciación de proteínas permitió a los biólogos moleculares crear filogenias basadas en la comparación de secuencias y utilizar las diferencias entre secuencias homólogas como un reloj molecular para estimar el tiempo transcurrido desde el ancestro común más reciente . [3] [1] La cantidad sorprendentemente grande de divergencia molecular dentro y entre especies inspiró la teoría neutral de la evolución molecular a finales de los años 1960. [4] [5] [6] La teoría neutral también proporcionó una base teórica para el reloj molecular , aunque esto no es necesario para la validez del reloj. Después de la década de 1970, la secuenciación de ácidos nucleicos permitió que la evolución molecular fuera más allá de las proteínas y llegara a secuencias de ARN ribosomal altamente conservadas , la base de una reconceptualización de la historia temprana de la vida . [1] La Sociedad de Biología Molecular y Evolución se fundó en 1982.
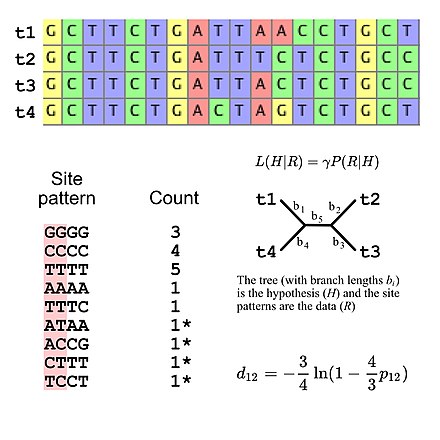
La filogenética molecular utiliza secuencias de ADN , ARN o proteínas para resolver cuestiones de sistemática , es decir, sobre su correcta clasificación científica desde el punto de vista de la historia evolutiva . El resultado de un análisis filogenético molecular se expresa en un árbol filogenético . La inferencia filogenética se lleva a cabo utilizando datos de la secuenciación del ADN . Esto se alinea para identificar qué sitios son homólogos . Un modelo de sustitución describe qué patrones se espera que sean comunes o raros. Luego se utiliza una inferencia computacional sofisticada para generar uno o más árboles plausibles.
Algunos métodos filogenéticos dan cuenta de la variación entre sitios y entre ramas de árboles . Diferentes genes, por ejemplo, hemoglobina versus citocromo c , generalmente evolucionan a diferentes ritmos . [7] Estas tasas son relativamente constantes a lo largo del tiempo (por ejemplo, la hemoglobina no evoluciona al mismo ritmo que el citocromo c, pero las hemoglobinas de humanos, ratones, etc. tienen tasas de evolución comparables), aunque la evolución rápida a lo largo de una rama puede indicar mayor selección direccional en esa rama. [8] La selección purificadora hace que las regiones funcionalmente importantes evolucionen más lentamente, y las sustituciones de aminoácidos que involucran aminoácidos similares ocurren con más frecuencia que las sustituciones diferentes. [7]


La duplicación de genes puede producir múltiples proteínas homólogas (parálogos) dentro de la misma especie. El análisis filogenético de las proteínas ha revelado cómo las proteínas evolucionan y cambian su estructura y función con el tiempo. [9] [10]
Por ejemplo, la ribonucleótido reductasa (RNR) ha desarrollado una multitud de variantes estructurales y funcionales. Los RNR de clase I utilizan una subunidad de ferritina y se diferencian por el metal que utilizan como cofactores. En las RNR de clase II , el radical tiilo se genera utilizando un cofactor de adenosilcobalamina y estas enzimas no requieren subunidades adicionales (a diferencia de las de clase I, que sí las requieren). En los RNR de clase III , el radical tiilo se genera utilizando S-adenosilmetionina unida a un grupo [ 4Fe-4S ]. Es decir, dentro de una única familia de proteínas pueden evolucionar numerosos mecanismos estructurales y funcionales. [11]
En un estudio de prueba de concepto, Bhattacharya y sus colegas convirtieron la mioglobina , una proteína no enzimática de almacenamiento de oxígeno, en una eliminasa Kemp altamente eficiente utilizando solo tres mutaciones . Esto demuestra que sólo se necesitan unas pocas mutaciones para cambiar radicalmente la función de una proteína. [12] La evolución dirigida es el intento de diseñar proteínas utilizando métodos inspirados en la evolución molecular.
El cambio en un locus comienza con una nueva mutación , que podría fijarse debido a alguna combinación de selección natural , deriva genética y conversión genética .

Las mutaciones son cambios permanentes y transmisibles en el material genético ( ADN o ARN ) de una célula o virus . Las mutaciones resultan de errores en la replicación del ADN durante la división celular y por la exposición a radiación , sustancias químicas, otros factores estresantes ambientales, virus o elementos transponibles . Cuando las mutaciones puntuales en un solo par de bases del ADN caen dentro de una región que codifica una proteína , se caracterizan por ser sinónimas (no cambian la secuencia de aminoácidos) o no sinónimas. Otros tipos de mutaciones modifican segmentos más grandes de ADN y pueden provocar duplicaciones, inserciones, eliminaciones, inversiones y translocaciones. [13]
La distribución de tasas para diversos tipos de mutaciones se denomina "espectro de mutación" (ver Apéndice B de [14] ). Mutaciones de diferentes tipos ocurren a ritmos muy variables. Las tasas de mutación puntual para la mayoría de los organismos son muy bajas, aproximadamente de 10 −9 a 10 −8 por sitio por generación, [15] aunque algunos virus tienen tasas de mutación más altas, del orden de 10 −6 por sitio por generación. [16] Las transiciones (A ↔ G o C ↔ T) son más comunes que las transversiones ( purina (adenina o guanina)) ↔ pirimidina (citosina o timina, o en ARN, uracilo)). [17] Quizás el tipo más común de mutación en humanos es un cambio en la longitud de una repetición corta en tándem (p. ej., las repeticiones CAG subyacentes a varias mutaciones asociadas a enfermedades). Estas mutaciones STR pueden ocurrir a tasas del orden de 10 −3 por generación. [18]
Diferentes frecuencias de diferentes tipos de mutaciones pueden desempeñar un papel importante en la evolución a través del sesgo en la introducción de variación (sesgo de llegada), contribuyendo al paralelismo, las tendencias y las diferencias en la navegabilidad de los paisajes adaptativos. [19] [20] El sesgo de mutación hace contribuciones sistemáticas o predecibles a la evolución paralela . [14] Desde la década de 1960, se ha pensado que el contenido genómico de GC refleja tendencias mutacionales. [21] [22] Los sesgos mutacionales también contribuyen al sesgo de uso de codones . [23] Aunque tales hipótesis a menudo se asocian con la neutralidad, resultados teóricos y empíricos recientes han establecido que las tendencias mutacionales pueden influir tanto en la evolución neutral como en la adaptativa a través del sesgo en la introducción de variación (sesgo de llegada).
La selección puede ocurrir cuando un alelo confiere una mayor aptitud , es decir, una mayor capacidad para sobrevivir o reproducirse, al individuo promedio que lo porta. Un enfoque seleccionista enfatiza, por ejemplo, que los sesgos en el uso de codones se deben, al menos en parte, a la capacidad de incluso una selección débil para dar forma a la evolución molecular. [24]
La selección también puede operar a nivel genético a expensas de la aptitud del organismo, lo que resulta en un conflicto intragenómico . Esto se debe a que puede haber una ventaja selectiva para los elementos genéticos egoístas a pesar del costo para el huésped. Ejemplos de tales elementos egoístas incluyen elementos transponibles , controladores meióticos y mitocondrias egoístas .
La selección se puede detectar mediante la relación Ka/Ks , la prueba de McDonald-Kreitman . A menudo se encuentra una rápida evolución adaptativa de genes implicados en conflictos intragenómicos , coevolución sexual antagonista y el sistema inmunológico .
La deriva genética es el cambio de frecuencias alélicas de una generación a la siguiente debido a efectos estocásticos del muestreo aleatorio en poblaciones finitas. Estos efectos pueden acumularse hasta que una mutación se fija en una población . Para mutaciones neutras, la tasa de fijación por generación es igual a la tasa de mutación por replicación. Por tanto, una tasa de mutación relativamente constante produce una tasa constante de cambio por generación (reloj molecular).
También se pueden corregir mutaciones ligeramente perjudiciales con un coeficiente de selección inferior a un valor umbral de 1/el tamaño efectivo de la población . Muchas características genómicas se han atribuido a la acumulación de mutaciones perjudiciales casi neutras como resultado de tamaños de población efectivos pequeños. [25] Con un tamaño de población efectivo más pequeño, una mayor variedad de mutaciones se comportará como si fueran neutrales debido a la ineficiencia de la selección.
La conversión de genes ocurre durante la recombinación, cuando el daño de los nucleótidos se repara utilizando una región genómica homóloga como plantilla. Puede ser un proceso sesgado, es decir, un alelo puede tener una mayor probabilidad de ser donante que el otro en un evento de conversión genética. En particular, la conversión de genes sesgada por GC tiende a aumentar el contenido de GC de los genomas, particularmente en regiones con tasas de recombinación más altas. [26] También hay evidencia de sesgo de GC en el proceso de reparación de desajustes. [27] Se cree que esto puede ser una adaptación a la alta tasa de desaminación de metilcitosina que puede conducir a transiciones C→T.
La dinámica de la conversión genética sesgada se parece a la de la selección natural, en el sentido de que un alelo favorecido tenderá a aumentar exponencialmente en frecuencia cuando sea raro.
El tamaño del genoma está influenciado por la cantidad de ADN repetitivo, así como por la cantidad de genes en un organismo. Algunos organismos, como la mayoría de las bacterias, Drosophila y Arabidopsis, tienen genomas particularmente compactos con poco contenido repetitivo o ADN no codificante. Otros organismos, como los mamíferos o el maíz, tienen grandes cantidades de ADN repetitivo, intrones largos y un espacio sustancial entre genes. La paradoja del valor C se refiere a la falta de correlación entre la "complejidad" del organismo y el tamaño del genoma. Las explicaciones de la llamada paradoja son dobles. En primer lugar, los elementos genéticos repetitivos pueden comprender grandes porciones del genoma de muchos organismos, inflando así el contenido de ADN del genoma haploide. Los elementos genéticos repetitivos a menudo descienden de elementos transponibles .
En segundo lugar, el número de genes no es necesariamente indicativo del número de etapas de desarrollo o tipos de tejidos de un organismo. Un organismo con pocas etapas de desarrollo o tipos de tejidos puede tener una gran cantidad de genes que influyen en los fenotipos que no están en desarrollo, inflando el contenido de genes en relación con las familias de genes del desarrollo.
Las explicaciones neutrales sobre el tamaño del genoma sugieren que cuando el tamaño de la población es pequeño, muchas mutaciones se vuelven casi neutrales. Por lo tanto, en poblaciones pequeñas se puede acumular contenido repetitivo y otro tipo de ADN "basura" sin colocar al organismo en desventaja competitiva. Hay poca evidencia que sugiera que el tamaño del genoma esté sujeto a una fuerte selección generalizada en eucariotas multicelulares. El tamaño del genoma, independientemente del contenido genético, se correlaciona mal con la mayoría de los rasgos fisiológicos y muchos eucariotas, incluidos los mamíferos, albergan cantidades muy grandes de ADN repetitivo.
Sin embargo, es probable que las aves hayan experimentado una fuerte selección para reducir el tamaño del genoma, en respuesta a las cambiantes necesidades energéticas para volar. Las aves, a diferencia de los humanos, producen glóbulos rojos nucleados y los núcleos más grandes conducen a niveles más bajos de transporte de oxígeno. El metabolismo de las aves es mucho mayor que el de los mamíferos, debido en gran parte al vuelo, y las necesidades de oxígeno son elevadas. Por tanto, la mayoría de las aves tienen genomas pequeños y compactos con pocos elementos repetitivos. La evidencia indirecta sugiere que los dinosaurios terópodos no aviares, ancestros de las aves modernas [28] también tenían tamaños de genoma reducidos, en consonancia con la endotermia y las altas necesidades energéticas para la velocidad de carrera. Muchas bacterias también han experimentado una selección por un tamaño de genoma pequeño, ya que el tiempo de replicación y el consumo de energía están estrechamente relacionados con la aptitud.
La hormiga Myrmecia pilosula tiene un solo par de cromosomas [29] , mientras que el helecho lengua de víbora Ophioglossum reticulatum tiene hasta 1260 cromosomas. [30] El número de cromosomas en el genoma de un organismo no necesariamente se correlaciona con la cantidad de ADN en su genoma. La cantidad de recombinación en todo el genoma está controlada directamente por la cantidad de cromosomas, con un cruce por cromosoma o por brazo cromosómico, según la especie. [31]
Los cambios en el número de cromosomas pueden desempeñar un papel clave en la especiación , ya que los diferentes números de cromosomas pueden servir como una barrera para la reproducción en los híbridos. El cromosoma 2 humano se creó a partir de una fusión de dos cromosomas de chimpancé y todavía contiene telómeros centrales así como un segundo centrómero vestigial . La poliploidía , especialmente la alopoliploidía, que ocurre a menudo en las plantas, también puede provocar incompatibilidades reproductivas con las especies parentales. Las mariposas azules Agrodiatus tienen diversos números de cromosomas que van desde n=10 hasta n=134 y, además, tienen una de las tasas de especiación más altas identificadas hasta la fecha. [32]
Los genomas ciliados albergan cada gen en cromosomas individuales.
Además del genoma nuclear , los orgánulos endosimbiontes contienen su propio material genético. El ADN mitocondrial y del cloroplasto varía según los taxones, pero las proteínas unidas a la membrana , especialmente los constituyentes de la cadena de transporte de electrones, se codifican con mayor frecuencia en el orgánulo. Los cloroplastos y las mitocondrias se heredan por vía materna en la mayoría de las especies, ya que los orgánulos deben pasar a través del óvulo . En un caso poco común, se sabe que algunas especies de mejillones heredan mitocondrias de padres a hijos.
Los nuevos genes surgen de varios mecanismos genéticos diferentes, incluida la duplicación de genes , el nacimiento de genes de novo , la retrotransposición , la formación de genes quiméricos , el reclutamiento de una secuencia no codificante en un gen existente y el truncamiento de genes.
La duplicación de genes conduce inicialmente a la redundancia. Sin embargo, las secuencias de genes duplicados pueden mutar para desarrollar nuevas funciones o especializarse de modo que el nuevo gen realice un subconjunto de las funciones ancestrales originales. La retrotransposición duplica genes copiando el ARNm en ADN e insertándolo en el genoma. "Los retrogenes generalmente se insertan en nuevas ubicaciones genómicas y carecen de intrones ". y en ocasiones desarrollan nuevos patrones de expresión y funciones.
Los genes quiméricos se forman cuando la duplicación, deleción o retrotransposición incompleta combinan porciones de dos secuencias codificantes diferentes para producir una nueva secuencia genética. Las quimeras a menudo causan cambios regulatorios y pueden mezclar dominios de proteínas para producir nuevas funciones adaptativas.
El nacimiento de genes de novo puede dar lugar a genes codificadores de proteínas y genes no codificantes a partir de ADN previamente no funcional. [33] Por ejemplo, Levine y sus colegas informaron sobre el origen de cinco nuevos genes en el genoma de D. melanogaster . [34] [35] También se ha demostrado un origen similar de novo de genes en otros organismos como la levadura, [36] el arroz [37] y los seres humanos. [38] Los genes de novo pueden evolucionar a partir de transcripciones espurias que ya se expresan en niveles bajos. [39]
La evolución neutral constructiva (CNE) explica que los sistemas complejos pueden surgir y extenderse a una población a través de transiciones neutrales con los principios de exceso de capacidad, presupresión y trinquete, [40] [41] [42] y se ha aplicado en áreas que van desde "Los orígenes del espliceosoma hasta la compleja interdependencia de las comunidades microbianas ". [43] [44] [45]
La Sociedad de Biología Molecular y Evolución publica las revistas "Molecular Biology and Evolution" y "Genome Biology and Evolution" y celebra una reunión internacional anual. Otras revistas dedicadas a la evolución molecular incluyen Journal of Molecular Evolution y Molecular Phylogenetics and Evolution . La investigación sobre la evolución molecular también se publica en revistas de genética , biología molecular , genómica , sistemática y biología evolutiva .
A este respecto carece de importancia si la selección ha sido insignificante o se ha autoanulado.
Categoría: evolución molecular (kimura 1968)