La deriva genética , también conocida como deriva genética aleatoria , deriva alélica o efecto Wright , [1] es el cambio en la frecuencia de una variante genética ( alelo ) existente en una población debido al azar. [2]
La deriva genética puede hacer que las variantes genéticas desaparezcan por completo y, por tanto, reducir la variación genética . [3] También puede causar que alelos inicialmente raros se vuelvan mucho más frecuentes e incluso fijos.
Cuando existen pocas copias de un alelo, el efecto de la deriva genética es más notable, y cuando existen muchas copias, el efecto es menos notable (debido a la ley de los grandes números ). A mediados del siglo XX, se produjeron intensos debates sobre la importancia relativa de la selección natural frente a los procesos neutrales, incluida la deriva genética. Ronald Fisher , que explicó la selección natural utilizando la genética mendeliana , [4] sostenía la opinión de que la deriva genética desempeña como mucho un papel menor en la evolución , y ésta siguió siendo la opinión dominante durante varias décadas. En 1968, el genetista de poblaciones Motoo Kimura reavivó el debate con su teoría neutral de la evolución molecular , que afirma que la mayoría de los casos en los que un cambio genético se propaga a través de una población (aunque no necesariamente cambios en los fenotipos ) son causados por una deriva genética que actúa sobre mutaciones neutrales . [5] [6] En la década de 1990, se propuso la evolución neutral constructiva que busca explicar cómo surgen sistemas complejos a través de transiciones neutrales. [7] [8]
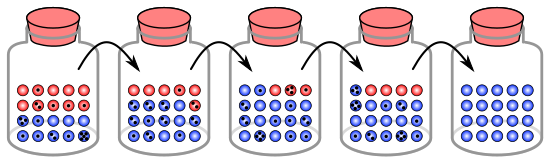
El proceso de deriva genética se puede ilustrar usando 20 canicas en un frasco para representar 20 organismos en una población. [9] Considere este frasco de canicas como la población inicial. La mitad de las canicas del frasco son rojas y la otra mitad son azules, y cada color corresponde a un alelo diferente de un gen de la población. En cada nueva generación, los organismos se reproducen al azar. Para representar esta reproducción, seleccione al azar una canica del frasco original y deposite una nueva canica del mismo color en un frasco nuevo. Esta es la "descendencia" del mármol original, lo que significa que el mármol original permanece en su frasco. Repita este proceso hasta que haya 20 canicas nuevas en el segundo frasco. El segundo frasco ahora contendrá 20 "descendientes", o canicas de varios colores. A menos que el segundo frasco contenga exactamente 10 canicas rojas y 10 canicas azules, se ha producido un cambio aleatorio en las frecuencias alélicas.
Si este proceso se repite varias veces, la cantidad de canicas rojas y azules recogidas en cada generación fluctúa. A veces, un frasco tiene más canicas rojas que su frasco "principal" y, a veces, más azules. Esta fluctuación es análoga a la deriva genética: un cambio en la frecuencia de los alelos de la población resultante de una variación aleatoria en la distribución de los alelos de una generación a la siguiente.
En cualquier generación, no se pueden elegir canicas de un color particular, lo que significa que no tienen descendencia. En este ejemplo, si no se selecciona ninguna canica roja, el frasco que representa la nueva generación solo contiene descendientes azules. Si esto sucede, el alelo rojo se habrá perdido permanentemente en la población, mientras que el alelo azul restante se habrá fijado: todas las generaciones futuras serán completamente azules. En poblaciones pequeñas, la fijación puede ocurrir en tan sólo unas pocas generaciones.
Los mecanismos de la deriva genética pueden ilustrarse con un ejemplo simplificado. Considere una colonia muy grande de bacterias aisladas en una gota de solución. Las bacterias son genéticamente idénticas excepto por un único gen con dos alelos etiquetados como A y B , que son alelos neutros, lo que significa que no afectan la capacidad de la bacteria para sobrevivir y reproducirse; Todas las bacterias de esta colonia tienen la misma probabilidad de sobrevivir y reproducirse. Supongamos que la mitad de las bacterias tiene el alelo A y la otra mitad tiene el alelo B. Por lo tanto, A y B tienen cada uno una frecuencia alélica de 1/2.
Luego, la gota de solución se encoge hasta que solo tiene suficiente alimento para sustentar cuatro bacterias. Todas las demás bacterias mueren sin reproducirse. Entre los cuatro que sobreviven, existen 16 combinaciones posibles para los alelos A y B
: (AAAA), (BAAA), (ABAA), (BBAA),
(AABA), (BABA), (ABBA), (BBBA),
( AAAB), (BAAB), (ABAB), (BBAB),
(AABB), (BABB), (ABBB), (BBBB).
Dado que todas las bacterias de la solución original tienen la misma probabilidad de sobrevivir cuando la solución se reduce, los cuatro supervivientes son una muestra aleatoria de la colonia original. La probabilidad de que cada uno de los cuatro supervivientes tenga un alelo determinado es 1/2, por lo que la probabilidad de que se produzca cualquier combinación de alelos particular cuando la solución se reduce es
(El tamaño de la población original es tan grande que el muestreo efectivamente se realiza con reemplazo). En otras palabras, cada una de las 16 combinaciones de alelos posibles tiene la misma probabilidad de ocurrir, con una probabilidad de 1/16.
Contando las combinaciones con el mismo número de A y B se obtiene la siguiente tabla:
Como se muestra en la tabla, el número total de combinaciones que tienen el mismo número de alelos A que de alelos B es seis, y la probabilidad de esta combinación es 6/16. El número total de otras combinaciones es diez, por lo que la probabilidad de que haya un número desigual de alelos A y B es 10/16. Por tanto, aunque la colonia original comenzó con un número igual de alelos A y B , es muy posible que el número de alelos en la población restante de cuatro miembros no sea igual. La situación de números iguales es en realidad menos probable que la de números desiguales. En el último caso, la deriva genética se ha producido porque las frecuencias alélicas de la población han cambiado debido al muestreo aleatorio. En este ejemplo, la población se redujo a solo cuatro supervivientes aleatorios, un fenómeno conocido como cuello de botella poblacional .
Las probabilidades del número de copias del alelo A (o B ) que sobreviven (dadas en la última columna de la tabla anterior) se pueden calcular directamente a partir de la distribución binomial , donde la probabilidad de "éxito" (probabilidad de que un alelo determinado esté presente ) es 1/2 (es decir, la probabilidad de que haya k copias de los alelos A (o B ) en la combinación) viene dada por:
donde n=4 es el número de bacterias supervivientes.
Se pueden diseñar modelos matemáticos de deriva genética utilizando procesos de ramificación o una ecuación de difusión que describa cambios en la frecuencia de los alelos en una población idealizada . [10]
Considere un gen con dos alelos , A o B. En la diploidía , las poblaciones formadas por N individuos tienen 2 N copias de cada gen. Un individuo puede tener dos copias del mismo alelo o dos alelos diferentes. A la frecuencia de un alelo se le asigna p y al otro q . El modelo de Wright-Fisher (llamado así en honor a Sewall Wright y Ronald Fisher ) supone que las generaciones no se superponen (por ejemplo, las plantas anuales tienen exactamente una generación por año) y que cada copia del gen que se encuentra en la nueva generación se extrae de forma independiente y aleatoria. de todas las copias del gen en la generación anterior. La fórmula para calcular la probabilidad de obtener k copias de un alelo que tuvo frecuencia p en la última generación es entonces [11] [12]
donde el símbolo " ! " significa la función factorial . Esta expresión también se puede formular utilizando el coeficiente binomial ,
El modelo de Moran supone generaciones superpuestas. En cada paso de tiempo, se elige un individuo para reproducirse y otro para morir. Entonces, en cada paso de tiempo, el número de copias de un alelo determinado puede aumentar en uno, disminuir en uno o permanecer igual. Esto significa que la matriz de transición es tridiagonal , lo que significa que las soluciones matemáticas son más fáciles para el modelo de Moran que para el modelo de Wright-Fisher. Por otro lado, las simulaciones por computadora suelen ser más fáciles de realizar utilizando el modelo de Wright-Fisher, porque es necesario calcular menos pasos de tiempo. En el modelo de Moran, se necesitan N pasos de tiempo para pasar una generación, donde N es el tamaño efectivo de la población . En el modelo de Wright-Fisher, sólo se necesita uno. [13]
En la práctica, los modelos de Moran y Wright-Fisher dan resultados cualitativamente similares, pero la deriva genética es dos veces más rápida en el modelo de Moran.
Si la varianza en el número de descendientes es mucho mayor que la dada por la distribución binomial supuesta por el modelo de Wright-Fisher, entonces, dada la misma velocidad general de deriva genética (la varianza del tamaño efectivo de la población), la deriva genética es una fuerza menos poderosa. en comparación con la selección. [14] Incluso para la misma varianza, si los momentos más altos de la distribución del número de descendientes exceden los de la distribución binomial, entonces nuevamente la fuerza de la deriva genética se debilita sustancialmente. [15]
Los cambios aleatorios en las frecuencias alélicas también pueden deberse a efectos distintos del error de muestreo , por ejemplo, cambios aleatorios en la presión de selección. [dieciséis]
Una fuente alternativa importante de estocasticidad , quizás más importante que la deriva genética, es el borrador genético . [17] El borrador genético es el efecto sobre un locus mediante la selección de loci vinculados . Las propiedades matemáticas de la tracción genética son diferentes de las de la deriva genética. [18] La dirección del cambio aleatorio en la frecuencia de los alelos se autocorrelaciona entre generaciones. [2]
El principio de Hardy-Weinberg establece que dentro de poblaciones suficientemente grandes, las frecuencias alélicas permanecen constantes de una generación a la siguiente, a menos que el equilibrio se vea alterado por migración , mutaciones genéticas o selección . [19]
Sin embargo, en poblaciones finitas, no se obtienen nuevos alelos a partir del muestreo aleatorio de alelos pasados a la siguiente generación, pero el muestreo puede provocar la desaparición de un alelo existente. Debido a que el muestreo aleatorio puede eliminar, pero no reemplazar, un alelo, y debido a que las disminuciones o aumentos aleatorios en la frecuencia de los alelos influyen en las distribuciones de alelos esperadas para la próxima generación, la deriva genética impulsa a una población hacia la uniformidad genética a lo largo del tiempo. Cuando un alelo alcanza una frecuencia de 1 (100%) se dice que está "fijado" en la población y cuando un alelo alcanza una frecuencia de 0 (0%) se pierde. Las poblaciones más pequeñas logran la fijación más rápidamente, mientras que en el límite de una población infinita, la fijación no se logra. Una vez que un alelo se fija, la deriva genética se detiene y la frecuencia del alelo no puede cambiar a menos que se introduzca un nuevo alelo en la población mediante mutación o flujo de genes . Por lo tanto, aunque la deriva genética es un proceso aleatorio y sin dirección, actúa para eliminar la variación genética con el tiempo. [20]

Suponiendo que la deriva genética es la única fuerza evolutiva que actúa sobre un alelo, después de t generaciones en muchas poblaciones replicadas, comenzando con las frecuencias alélicas de p y q , la variación en la frecuencia alélica entre esas poblaciones es
Suponiendo que la deriva genética es la única fuerza evolutiva que actúa sobre un alelo, en un momento dado la probabilidad de que un alelo finalmente quede fijo en la población es simplemente su frecuencia en la población en ese momento. [22] Por ejemplo, si la frecuencia p para el alelo A es del 75% y la frecuencia q para el alelo B es del 25%, entonces, dado un tiempo ilimitado, la probabilidad de que A finalmente se fije en la población es del 75% y la probabilidad de que B fijo es del 25%.
El número esperado de generaciones para que se produzca la fijación es proporcional al tamaño de la población, de modo que se predice que la fijación se producirá mucho más rápidamente en poblaciones más pequeñas. [23] Normalmente, para determinar estas probabilidades se utiliza el tamaño efectivo de la población, que es menor que la población total. La población efectiva ( N e ) tiene en cuenta factores como el nivel de endogamia , la etapa del ciclo de vida en la que la población es más pequeña y el hecho de que algunos genes neutros están genéticamente ligados a otros que están bajo selección. [14] El tamaño efectivo de la población puede no ser el mismo para todos los genes de la misma población. [24]
Una fórmula prospectiva utilizada para aproximar el tiempo esperado antes de que un alelo neutro se fije mediante deriva genética, según el modelo de Wright-Fisher, es
donde T es el número de generaciones, N e es el tamaño efectivo de la población y p es la frecuencia inicial para el alelo dado. El resultado es el número de generaciones que se espera que pasen antes de que se produzca la fijación de un alelo determinado en una población con un tamaño ( N e ) y una frecuencia alélica ( p ) determinados. [25]
El tiempo esperado para que el alelo neutro se pierda debido a la deriva genética se puede calcular como [11]
Cuando una mutación aparece sólo una vez en una población lo suficientemente grande como para que la frecuencia inicial sea insignificante, las fórmulas se pueden simplificar a [26]
para el número promedio de generaciones esperadas antes de la fijación de una mutación neutra, y
para el número promedio de generaciones esperadas antes de la pérdida de una mutación neutral en una población de tamaño real N. [27]
Las fórmulas anteriores se aplican a un alelo que ya está presente en una población y que no está sujeto a mutación ni a selección natural. Si un alelo se pierde por mutación con mucha más frecuencia de lo que se gana por mutación, entonces la mutación, así como la deriva, pueden influir en el tiempo transcurrido hasta la pérdida. Si el alelo propenso a la pérdida mutacional comienza como fijo en la población y se pierde por mutación a una tasa m por replicación, entonces el tiempo esperado en generaciones hasta su pérdida en una población haploide está dado por
¿Dónde está la constante de Euler ? [28] La primera aproximación representa el tiempo de espera hasta que aparece el primer mutante destinado a la pérdida, y la pérdida se produce entonces con relativa rapidez por deriva genética, lo que lleva tiempo1/metro≫ norte mi . La segunda aproximación representa el tiempo necesario para la pérdida determinista por acumulación de mutaciones. En ambos casos, el tiempo hasta la fijación está dominado por la mutación a través del término1/metroy se ve menos afectado por el tamaño efectivo de la población .
En las poblaciones naturales, la deriva genética y la selección natural no actúan de forma aislada; Ambos fenómenos están siempre en juego, junto con la mutación y la migración. La evolución neutral es producto tanto de la mutación como de la deriva, no sólo de la deriva. De manera similar, incluso cuando la selección supera la deriva genética, sólo puede actuar sobre la variación que proporciona la mutación.
Mientras que la selección natural tiene una dirección, que guía la evolución hacia adaptaciones hereditarias al entorno actual, la deriva genética no tiene dirección y se guía únicamente por las matemáticas del azar . [29] Como resultado, la deriva actúa sobre las frecuencias genotípicas dentro de una población sin tener en cuenta sus efectos fenotípicos. Por el contrario, la selección favorece la propagación de alelos cuyos efectos fenotípicos aumentan la supervivencia y/o la reproducción de sus portadores, reduce las frecuencias de los alelos que causan rasgos desfavorables e ignora aquellos que son neutrales. [30]
La ley de los grandes números predice que cuando el número absoluto de copias del alelo es pequeño (p. ej., en poblaciones pequeñas ), la magnitud de la deriva en las frecuencias alélicas por generación es mayor. La magnitud de la deriva es lo suficientemente grande como para abrumar la selección en cualquier frecuencia alélica cuando el coeficiente de selección es menor que 1 dividido por el tamaño efectivo de la población. Por lo tanto, la evolución no adaptativa resultante del producto de la mutación y la deriva genética se considera un mecanismo consecuente de cambio evolutivo principalmente dentro de poblaciones pequeñas y aisladas. [31] Las matemáticas de la deriva genética dependen del tamaño efectivo de la población, pero no está claro cómo se relaciona esto con el número real de individuos en una población. [17] El vínculo genético con otros genes que están bajo selección puede reducir el tamaño efectivo de la población experimentado por un alelo neutro. Con una tasa de recombinación más alta , el ligamiento disminuye y con él este efecto local sobre el tamaño efectivo de la población. [32] [33] Este efecto es visible en datos moleculares como una correlación entre la tasa de recombinación local y la diversidad genética , [34] y una correlación negativa entre la densidad genética y la diversidad en regiones de ADN no codificantes . [35] La estocasticidad asociada con el vínculo con otros genes que están bajo selección no es lo mismo que el error de muestreo y, a veces, se conoce como borrador genético para distinguirlo de la deriva genética. [17]
La baja frecuencia de los alelos hace que los alelos sean más vulnerables a ser eliminados por casualidad, incluso anulando la influencia de la selección natural. Por ejemplo, si bien las mutaciones desventajosas suelen eliminarse rápidamente dentro de la población, las nuevas mutaciones ventajosas son casi tan vulnerables a la pérdida por deriva genética como lo son las mutaciones neutrales. Hasta que la frecuencia alélica de la mutación ventajosa alcance un cierto umbral, la deriva genética no tendrá ningún efecto. [30]

Un cuello de botella poblacional se produce cuando una población se contrae a un tamaño significativamente menor en un corto período de tiempo debido a algún evento ambiental aleatorio. En un verdadero cuello de botella poblacional, las probabilidades de supervivencia de cualquier miembro de la población son puramente aleatorias y no mejoran con ninguna ventaja genética inherente particular. El cuello de botella puede dar lugar a cambios radicales en las frecuencias alélicas, completamente independientes de la selección. [36]
El impacto de un cuello de botella en la población puede ser sostenido, incluso cuando el cuello de botella es causado por un evento único como una catástrofe natural. Un ejemplo interesante de un cuello de botella que causa una distribución genética inusual es la proporción relativamente alta de individuos con daltonismo total de bastones ( acromatopsia ) en el atolón de Pingelap en Micronesia . [37] Después de un cuello de botella, la endogamia aumenta. Esto aumenta el daño causado por mutaciones nocivas recesivas, en un proceso conocido como depresión endogámica . Se seleccionan las peores de estas mutaciones, lo que lleva a la pérdida de otros alelos que están genéticamente vinculados a ellas, en un proceso de selección de fondo . [2] En el caso de mutaciones dañinas recesivas, esta selección puede verse potenciada como consecuencia del cuello de botella, debido a la purga genética . Esto conduce a una mayor pérdida de diversidad genética. Además, una reducción sostenida en el tamaño de la población aumenta la probabilidad de que se produzcan más fluctuaciones de alelos en las generaciones venideras.
Un cuello de botella puede reducir en gran medida la variación genética de una población, e incluso las adaptaciones beneficiosas pueden eliminarse permanentemente. [38] La pérdida de variación deja a la población superviviente vulnerable a cualquier nueva presión de selección, como enfermedades, cambios climáticos o cambios en la fuente de alimentos disponible, porque la adaptación en respuesta a los cambios ambientales requiere suficiente variación genética en la población para que la selección natural tenga lugar. lugar. [39] [40]
En el pasado reciente se han conocido muchos casos de cuello de botella demográfico. Antes de la llegada de los europeos , las praderas de América del Norte eran hábitat de millones de pollos de las praderas . Sólo en Illinois , su número se desplomó de unos 100 millones de aves en 1900 a unas 50 aves en la década de 1990. La disminución de la población se debió a la caza y la destrucción del hábitat, pero una consecuencia ha sido la pérdida de la mayor parte de la diversidad genética de la especie. Los análisis de ADN que comparan aves de mediados de siglo con aves de la década de 1990 documentan una fuerte disminución en la variación genética sólo en las últimas décadas. Actualmente, el pollo de las praderas está experimentando un bajo éxito reproductivo . [41]
Sin embargo, la pérdida genética causada por el cuello de botella y la deriva genética puede aumentar la aptitud, como en Ehrlichia . [42]
La caza excesiva también provocó un grave cuello de botella en la población del elefante marino del norte en el siglo XIX. La disminución resultante en la variación genética se puede deducir comparándola con la del elefante marino del sur , que no fue cazado tan agresivamente. [43]

El efecto fundador es un caso especial de cuello de botella poblacional, que ocurre cuando un pequeño grupo de una población se separa de la población original y forma una nueva. Se espera que la muestra aleatoria de alelos en la nueva colonia recién formada tergiverse enormemente la población original en al menos algunos aspectos. [44] Incluso es posible que el número de alelos de algunos genes en la población original sea mayor que el número de copias de genes en los fundadores, lo que hace imposible una representación completa. Cuando una colonia recién formada es pequeña, sus fundadores pueden afectar fuertemente la composición genética de la población en el futuro.
Un ejemplo bien documentado se encuentra en la migración Amish a Pensilvania en 1744. Dos miembros de la nueva colonia compartían el alelo recesivo del síndrome de Ellis-Van Creveld . Los miembros de la colonia y sus descendientes tienden a ser religiosos aislados y permanecen relativamente aislados. Como resultado de muchas generaciones de endogamia, el síndrome de Ellis-Van Creveld es ahora mucho más frecuente entre los Amish que entre la población general. [30] [45]
La diferencia en las frecuencias genéticas entre la población original y la colonia también puede provocar que los dos grupos diverjan significativamente a lo largo de muchas generaciones. A medida que aumenta la diferencia, o distancia genética , las dos poblaciones separadas pueden volverse distintas, tanto genética como fenéticamente , aunque no sólo la deriva genética sino también la selección natural, el flujo de genes y las mutaciones contribuyen a esta divergencia. Este potencial de cambios relativamente rápidos en la frecuencia genética de la colonia llevó a la mayoría de los científicos a considerar el efecto fundador (y, por extensión, la deriva genética) como una fuerza impulsora importante en la evolución de nuevas especies . Sewall Wright fue el primero en otorgar esta importancia a la deriva aleatoria y a las poblaciones pequeñas y recientemente aisladas con su teoría de la especiación del equilibrio cambiante . [46] Siguiendo a Wright, Ernst Mayr creó muchos modelos persuasivos para mostrar que la disminución de la variación genética y el pequeño tamaño de la población después del efecto fundador eran de importancia crítica para el desarrollo de nuevas especies. [47] Sin embargo, hoy en día hay mucho menos apoyo para esta opinión, ya que la hipótesis ha sido probada repetidamente a través de investigaciones experimentales y los resultados han sido, en el mejor de los casos, equívocos. [48]
El papel del azar en la evolución fue descrito por primera vez por Arend L. Hagedoorn y AC Hagedoorn-Vorstheuvel La Brand en 1921. [49] Destacaron que la supervivencia aleatoria juega un papel clave en la pérdida de variación de las poblaciones. Fisher (1922) respondió a esto con el primer tratamiento matemático, aunque ligeramente incorrecto, del "efecto Hagedoorn". [50] En particular, esperaba que muchas poblaciones naturales fueran demasiado grandes (un N ~ 10.000) para que los efectos de la deriva fueran sustanciales y pensó que la deriva tendría un efecto insignificante en el proceso evolutivo. El tratamiento matemático corregido y el término "deriva genética" fueron acuñados más tarde por uno de los fundadores de la genética de poblaciones , Sewall Wright . Su primer uso del término "deriva" fue en 1929, [51] aunque en ese momento lo usaba en el sentido de un proceso dirigido de cambio, o selección natural. La deriva aleatoria mediante un error de muestreo llegó a conocerse como el "efecto Sewall-Wright", aunque nunca se sintió del todo cómodo al ver que se le daba su nombre. Wright se refirió a todos los cambios en la frecuencia de los alelos como "deriva constante" (por ejemplo, selección) o "deriva aleatoria" (por ejemplo, error de muestreo). [52] "Deriva" llegó a ser adoptado como un término técnico exclusivamente en el sentido estocástico . [53] Hoy en día se suele definir aún más estrictamente, en términos de error de muestreo, [54] aunque esta definición estrecha no es universal. [55] [56] Wright escribió que la "restricción de la" deriva aleatoria "o incluso la" deriva "a un solo componente, los efectos de los accidentes de muestreo, tiende a generar confusión". [52] Sewall Wright consideró el proceso de deriva genética aleatoria mediante error de muestreo equivalente al de la endogamia, pero trabajos posteriores han demostrado que son distintos. [57]
En los primeros días de la síntesis evolutiva moderna , los científicos comenzaban a combinar la nueva ciencia de la genética de poblaciones con la teoría de la selección natural de Charles Darwin . Dentro de este marco, Wright se centró en los efectos de la endogamia en poblaciones pequeñas relativamente aisladas. Introdujo el concepto de un paisaje adaptativo en el que fenómenos como el cruzamiento y la deriva genética en poblaciones pequeñas podrían alejarlas de los picos adaptativos, lo que a su vez permite que la selección natural los empuje hacia nuevos picos adaptativos. [58] Wright pensó que las poblaciones más pequeñas eran más adecuadas para la selección natural porque "la endogamia era lo suficientemente intensa como para crear nuevos sistemas de interacción a través de la deriva aleatoria, pero no lo suficientemente intensa como para causar una fijación aleatoria no adaptativa de genes". [59]
Las opiniones de Wright sobre el papel de la deriva genética en el esquema evolutivo fueron controvertidas casi desde el principio. Uno de los críticos más ruidosos e influyentes fue su colega Ronald Fisher. Fisher admitió que la deriva genética jugó algún papel en la evolución, pero insignificante. Fisher ha sido acusado de malinterpretar los puntos de vista de Wright porque en sus críticas Fisher parecía argumentar que Wright había rechazado la selección casi por completo. Para Fisher, ver el proceso de evolución como una progresión larga, constante y adaptativa era la única manera de explicar la complejidad cada vez mayor a partir de formas más simples. Pero los debates han continuado entre los "gradualistas" y aquellos que se inclinan más hacia el modelo de evolución de Wright, donde la selección y la deriva juntas juegan un papel importante. [60]
En 1968, Motoo Kimura reavivó el debate con su teoría neutral de la evolución molecular , que afirma que la mayoría de los cambios genéticos son causados por la deriva genética que actúa sobre mutaciones neutrales. [5] [6]
El papel de la deriva genética mediante errores de muestreo en la evolución ha sido criticado por John H. Gillespie [61] y William B. Provine , quienes sostienen que la selección en sitios vinculados es una fuerza estocástica más importante.