La recombinación genética (también conocida como reorganización genética ) es el intercambio de material genético entre diferentes organismos que conduce a la producción de descendencia con combinaciones de rasgos que difieren de los encontrados en cualquiera de los progenitores. En eucariotas , la recombinación genética durante la meiosis puede conducir a un nuevo conjunto de información genética que puede transmitirse de padres a hijos. La mayor parte de la recombinación ocurre de forma natural y se puede clasificar en dos tipos: (1) recombinación intercromosómica , que ocurre a través de la distribución independiente de alelos cuyos loci están en cromosomas diferentes pero homólogos (orientación aleatoria de pares de cromosomas homólogos en la meiosis I); y (2) recombinación intracromosómica , que ocurre a través del entrecruzamiento. [1]
Durante la meiosis en eucariotas , la recombinación genética implica el apareamiento de cromosomas homólogos . Esto puede ir seguido de una transferencia de información entre los cromosomas. La transferencia de información puede ocurrir sin intercambio físico (una sección de material genético se copia de un cromosoma a otro, sin que se modifique el cromosoma donante) (véase la vía SDSA – Synthesis Dependent Strand Annealing en la Figura); o mediante la ruptura y la reunificación de las hebras de ADN , lo que forma nuevas moléculas de ADN (véase la vía DHJ en la Figura).
La recombinación también puede ocurrir durante la mitosis en eucariotas, donde normalmente involucra a los dos cromosomas hermanos formados después de la replicación cromosómica. En este caso, no se producen nuevas combinaciones de alelos ya que los cromosomas hermanos suelen ser idénticos. En la meiosis y la mitosis, la recombinación ocurre entre moléculas similares de ADN ( secuencias homólogas ). En la meiosis, los cromosomas homólogos no hermanos se aparean entre sí de modo que la recombinación ocurre característicamente entre homólogos no hermanos. Tanto en las células meióticas como en las mitóticas, la recombinación entre cromosomas homólogos es un mecanismo común utilizado en la reparación del ADN .
La conversión genética , el proceso mediante el cual las secuencias homólogas se vuelven idénticas, también se incluye dentro de la recombinación genética.
La recombinación genética y la reparación recombinatoria del ADN también ocurren en bacterias y arqueas , que utilizan la reproducción asexual .
La recombinación se puede inducir artificialmente en entornos de laboratorio ( in vitro ), produciendo ADN recombinante para fines que incluyen el desarrollo de vacunas .
La recombinación V(D)J en organismos con un sistema inmune adaptativo es un tipo de recombinación genética específica del sitio que ayuda a las células inmunes a diversificarse rápidamente para reconocer y adaptarse a nuevos patógenos .
Durante la meiosis, la sinapsis (el apareamiento de cromosomas homólogos) normalmente precede a la recombinación genética.
La recombinación genética está catalizada por muchas enzimas diferentes . Las recombinasas son enzimas clave que catalizan el paso de transferencia de cadena durante la recombinación. RecA , la principal recombinasa que se encuentra en Escherichia coli , es responsable de la reparación de las roturas de doble cadena del ADN (DSB). En la levadura y otros organismos eucariotas hay dos recombinasas necesarias para reparar las DSB. La proteína RAD51 es necesaria para la recombinación mitótica y meiótica , mientras que la proteína de reparación del ADN, DMC1 , es específica de la recombinación meiótica. En las arqueas, el ortólogo de la proteína bacteriana RecA es RadA.
Las bacterias experimentan regularmente una recombinación genética de tres maneras principales:
A veces, se transfiere una hebra de ADN a la célula diana, pero no se logra copiar a medida que la célula diana se divide. Esto se denomina transferencia abortada .

En los eucariotas , la recombinación durante la meiosis se ve facilitada por el entrecruzamiento cromosómico . El proceso de entrecruzamiento hace que los descendientes tengan combinaciones de genes diferentes a las de sus progenitores, y ocasionalmente puede producir nuevos alelos quiméricos . [ cita requerida ] La mezcla de genes provocada por la recombinación genética produce una mayor variación genética . También permite que los organismos que se reproducen sexualmente eviten el trinquete de Müller , en el que los genomas de una población asexual tienden a acumular más mutaciones perjudiciales con el tiempo que mutaciones beneficiosas o reversibles. [ cita requerida ]
El entrecruzamiento cromosómico implica la recombinación entre los cromosomas emparejados heredados de cada uno de los padres, que generalmente ocurre durante la meiosis . [ cita requerida ] Durante la profase I (etapa de paquiteno), las cuatro cromátidas disponibles están en formación apretada entre sí. [ cita requerida ] Mientras están en esta formación, los sitios homólogos en dos cromátidas pueden emparejarse estrechamente entre sí y pueden intercambiar información genética. [3]
Debido a que existe una pequeña probabilidad de recombinación en cualquier ubicación a lo largo de un cromosoma, la frecuencia de recombinación entre dos ubicaciones depende de la distancia que las separa. [ cita requerida ] Por lo tanto, para genes suficientemente distantes en el mismo cromosoma, la cantidad de cruce es lo suficientemente alta como para destruir la correlación entre alelos. [ cita requerida ]
El seguimiento del movimiento de los genes resultantes de los cruces ha resultado muy útil para los genetistas. Debido a que dos genes que están cerca uno del otro tienen menos probabilidades de separarse que los genes que están más alejados, los genetistas pueden deducir aproximadamente cuán separados están dos genes en un cromosoma si conocen la frecuencia de los cruces. [ cita requerida ] Los genetistas también pueden utilizar este método para inferir la presencia de ciertos genes. Los genes que normalmente permanecen juntos durante la recombinación se dice que están ligados . Un gen en un par ligado a veces se puede utilizar como marcador para deducir la presencia del otro gen. Esto se utiliza normalmente para detectar la presencia de un gen causante de enfermedades. [ 6 ]
La frecuencia de recombinación observada entre dos loci es el valor de entrecruzamiento . Es la frecuencia de entrecruzamiento entre dos loci genéticos ligados ( marcadores ), y depende de la distancia entre los loci genéticos observados. Para cualquier conjunto fijo de condiciones genéticas y ambientales, la recombinación en una región particular de una estructura de ligamiento ( cromosoma ) tiende a ser constante, y lo mismo es cierto para el valor de entrecruzamiento que se utiliza en la producción de mapas genéticos . [5] [7]
En la conversión genética, una sección de material genético se copia de un cromosoma a otro, sin que se modifique el cromosoma donante. La conversión genética ocurre con alta frecuencia en el sitio real del evento de recombinación durante la meiosis . Es un proceso por el cual una secuencia de ADN se copia de una hélice de ADN (que permanece inalterada) a otra hélice de ADN, cuya secuencia se altera. La conversión genética a menudo se ha estudiado en cruces de hongos [8] donde se pueden observar convenientemente los 4 productos de meiosis individuales. Los eventos de conversión genética se pueden distinguir como desviaciones en una meiosis individual del patrón normal de segregación 2:2 (por ejemplo, un patrón 3:1).
La recombinación puede ocurrir entre secuencias de ADN que no contienen homología de secuencia . Esto puede causar translocaciones cromosómicas , que a veces conducen al cáncer.
Las células B del sistema inmunitario realizan una recombinación genética, llamada cambio de clase de inmunoglobulina . Se trata de un mecanismo biológico que cambia un anticuerpo de una clase a otra, por ejemplo, de un isotipo llamado IgM a un isotipo llamado IgG .
En ingeniería genética , la recombinación también puede referirse a la recombinación artificial y deliberada de fragmentos dispares de ADN, a menudo de diferentes organismos, creando lo que se llama ADN recombinante . Un excelente ejemplo de este uso de la recombinación genética es la selección de genes , que se puede utilizar para agregar, eliminar o cambiar de otro modo los genes de un organismo. Esta técnica es importante para los investigadores biomédicos , ya que les permite estudiar los efectos de genes específicos. Las técnicas basadas en la recombinación genética también se aplican en la ingeniería de proteínas para desarrollar nuevas proteínas de interés biológico.
Los ejemplos incluyen la integración mediada por enzimas de restricción , el ensamblaje de Gibson y la clonación Golden Gate .
Los daños en el ADN causados por una variedad de agentes exógenos (por ejemplo, luz ultravioleta , rayos X , agentes de reticulación química ) pueden repararse mediante reparación recombinatoria homóloga (HRR). [9] [10] Estos hallazgos sugieren que los daños en el ADN que surgen de procesos naturales , como la exposición a especies reactivas de oxígeno que son subproductos del metabolismo normal, también son reparados por HRR. En los seres humanos, las deficiencias en los productos genéticos necesarios para HRR durante la meiosis probablemente causan infertilidad [11] En los seres humanos, las deficiencias en los productos genéticos necesarios para HRR, como BRCA1 y BRCA2 , aumentan el riesgo de cáncer (ver trastorno por deficiencia de reparación del ADN ).
En las bacterias, la transformación es un proceso de transferencia de genes que ocurre normalmente entre células individuales de la misma especie bacteriana. La transformación implica la integración del ADN del donante en el cromosoma receptor mediante recombinación. Este proceso parece ser una adaptación para reparar los daños del ADN en el cromosoma receptor mediante la HRR. [12] La transformación puede proporcionar un beneficio a las bacterias patógenas al permitir la reparación del daño del ADN, en particular los daños que ocurren en el entorno inflamatorio y oxidante asociado con la infección de un huésped.
Cuando dos o más virus, cada uno con daños genómicos letales, infectan la misma célula huésped, los genomas del virus a menudo pueden emparejarse entre sí y experimentar HRR para producir progenie viable. Este proceso, conocido como reactivación por multiplicidad, se ha estudiado en bacteriófagos lambda y T4 , [13] así como en varios virus patógenos. En el caso de los virus patógenos, la reactivación por multiplicidad puede ser un beneficio adaptativo para el virus ya que permite la reparación de los daños del ADN causados por la exposición al entorno oxidante producido durante la infección del huésped. [12] Véase también reordenamiento .
En la primera figura de este artículo se describe un modelo molecular del mecanismo de recombinación meiótica presentado por Anderson y Sekelsky [14] . Dos de las cuatro cromátidas presentes al principio de la meiosis (profase I) están emparejadas entre sí y pueden interactuar. La recombinación, en este modelo, se inicia mediante una rotura (o espacio) de doble cadena que se muestra en la molécula de ADN (cromátida) en la parte superior de la figura. Otros tipos de daño del ADN también pueden iniciar la recombinación. Por ejemplo, un enlace cruzado entre cadenas (causado por la exposición a un agente de enlace cruzado como la mitomicina C) puede ser reparado por la HRR.
Se producen dos tipos de productos recombinantes. En el lado derecho se indica un tipo "cruzado" (CO), en el que se intercambian las regiones flanqueantes de los cromosomas, y en el lado izquierdo, un tipo "no cruzado" (NCO), en el que no se intercambian las regiones flanqueantes. El tipo de recombinación CO implica la formación intermedia de dos "uniones de Holliday" indicadas en la parte inferior derecha de la figura por dos estructuras en forma de X en cada una de las cuales hay un intercambio de cadenas simples entre las dos cromátidas participantes. Esta vía se denomina en la figura vía DHJ (doble unión de Holliday).
Los recombinantes NCO (ilustrados a la izquierda de la figura) se producen mediante un proceso denominado "recocido de hebras dependiente de la síntesis" (SDSA). Los eventos de recombinación del tipo NCO/SDSA parecen ser más comunes que los del tipo CO/DHJ. [15] La vía NCO/SDSA contribuye poco a la variación genética, ya que los brazos de los cromosomas que flanquean el evento de recombinación permanecen en la configuración parental. Por lo tanto, las explicaciones de la función adaptativa de la meiosis que se centran exclusivamente en el entrecruzamiento son inadecuadas para explicar la mayoría de los eventos de recombinación.
La aquiasmia es el fenómeno en el que la recombinación autosómica está completamente ausente en uno de los sexos de una especie. La segregación cromosómica aquiasmática está bien documentada en los machos de Drosophila melanogaster . La "regla de Haldane-Huxley" establece que la aquiasmia suele ocurrir en el sexo heterogamético . [16]
La heteroquiasmia se produce cuando las tasas de recombinación difieren entre los sexos de una especie. [16] En los seres humanos, cada ovocito tiene en promedio 41,6 ± 11,3 recombinaciones, 1,63 veces más que los espermatozoides. Este patrón de dimorfismo sexual en la tasa de recombinación se ha observado en muchas especies. En los mamíferos, las hembras suelen tener tasas de recombinación más altas. [17]
Numerosos virus de ARN son capaces de recombinación genética cuando al menos dos genomas virales están presentes en la misma célula huésped. [18] [19] La recombinación es en gran medida responsable de la diversidad de virus de ARN y la evasión inmune. [20] La recombinación de ARN parece ser una fuerza impulsora importante en la determinación de la arquitectura del genoma y el curso de la evolución viral entre los picornaviridae ( (+)ssRNA ) (por ejemplo, el virus de la poliomielitis ). [21] En los retroviridae ((+)ssRNA)(por ejemplo, el VIH ), el daño en el genoma de ARN parece evitarse durante la transcripción inversa mediante el cambio de cadena, una forma de recombinación. [22] [23]
La recombinación también ocurre en reoviridae (dsRNA) (por ejemplo, reovirus), orthomyxoviridae ((-)ssRNA) (por ejemplo, virus de la influenza ) [23] y coronaviridae ((+)ssRNA) (por ejemplo, SARS ). [24] [25]
La recombinación en los virus de ARN parece ser una adaptación para hacer frente al daño del genoma. [18] El cambio entre cadenas de plantilla durante la replicación del genoma, conocido como recombinación por elección de copia, se propuso originalmente para explicar la correlación positiva de los eventos de recombinación en distancias cortas en organismos con un genoma de ADN (ver la primera figura, vía SDSA ). [26]
La recombinación puede ocurrir con poca frecuencia entre virus animales de la misma especie pero de linajes divergentes. Los virus recombinantes resultantes pueden causar a veces un brote de infección en humanos. [24]
Especialmente en los coronavirus, la recombinación también puede ocurrir incluso entre grupos evolutivos distantemente relacionados (subgéneros), debido a su mecanismo de transcripción característico, que involucra ARNm subgenómicos que se forman por cambio de plantilla. [27] [25]
Al replicar su genoma de (+)ssRNA , la ARN polimerasa dependiente de ARN (RdRp) del poliovirus es capaz de llevar a cabo la recombinación. La recombinación parece ocurrir por un mecanismo de elección de copia en el que la RdRp cambia las plantillas de (+)ssRNA durante la síntesis de la cadena negativa. [28] La recombinación por cambio de cadena de RdRp también ocurre en los carmovirus vegetales (+)ssRNA y tombusvirus . [29]
La recombinación parece ser una fuerza impulsora importante para determinar la variabilidad genética dentro de los coronavirus, así como la capacidad de las especies de coronavirus de saltar de un huésped a otro y, con poca frecuencia, para el surgimiento de nuevas especies, aunque el mecanismo de recombinación no está claro. [24]
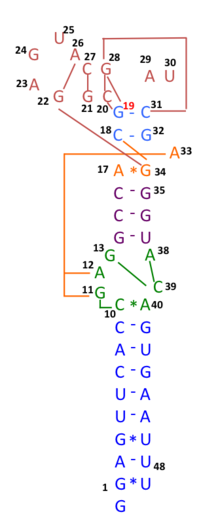
A principios de 2020, muchas secuencias genómicas de aislamientos australianos de SARS-CoV-2 tienen deleciones o mutaciones (29742G>A o 29742G>U; "G19A" o "G19U") en el s2m, lo que sugiere que la recombinación de ARN puede haber ocurrido en este elemento de ARN. 29742G("G19"), 29744G("G21") y 29751G("G28") se predijeron como puntos críticos de recombinación. [30] Durante los primeros meses de la pandemia de COVID-19, se sugirió que un evento de recombinación de este tipo había sido un paso crítico en la evolución de la capacidad del SARS-CoV-2 para infectar a los humanos. [31] El análisis de desequilibrio de ligamiento confirmó que la recombinación de ARN con la mutación 11083G > T también contribuyó al aumento de mutaciones entre la progenie viral. Los hallazgos indican que la mutación 11083G > T del SARS-CoV-2 se propagó durante la cuarentena a bordo del Diamond Princess y surgió a través de una recombinación de ARN de novo bajo presión de selección positiva. En tres pacientes del crucero Diamond Princess , dos mutaciones, 29736G > T y 29751G > T (G13 y G28) se localizaron en el motivo similar al tallo-bucle II (s2m) del coronavirus 3' del SARS-CoV-2. Aunque s2m se considera un motivo de ARN altamente conservado en la región no traducida 3' entre muchas especies de coronavirus, este resultado también sugiere que s2m del SARS-CoV-2 es un punto crítico de recombinación/mutación del ARN. [32]
Según observaciones preliminares, todo el motivo de unión al receptor del SARS-CoV-2 parecía haber sido introducido a través de la recombinación de coronavirus de pangolines . [33] Sin embargo, análisis más exhaustivos posteriores refutaron esta sugerencia y demostraron que el SARS-CoV-2 probablemente evolucionó únicamente dentro de los murciélagos y con poca o ninguna recombinación. [34] [35]
Nowak y Ohtsuki [36] observaron que el origen de la vida ( abiogénesis ) es también el origen de la evolución biológica . Señalaron que toda la vida conocida en la Tierra se basa en biopolímeros y propusieron que cualquier teoría sobre el origen de la vida debe involucrar polímeros biológicos que actúen como portadores de información y catalizadores. Lehman [37] argumentó que la recombinación fue un desarrollo evolutivo tan antiguo como los orígenes de la vida. Smail et al. [38] propusieron que en la Tierra primordial, la recombinación jugó un papel clave en la expansión de los polímeros informativos inicialmente cortos (se presume que son ARN ) que fueron los precursores de la vida.
![]() Este artículo incorpora material de dominio público de Science Primer. NCBI . Archivado desde el original el 8 de diciembre de 2009.
Este artículo incorpora material de dominio público de Science Primer. NCBI . Archivado desde el original el 8 de diciembre de 2009.