La conjugación bacteriana es la transferencia de material genético entre células bacterianas por contacto directo de célula a célula o por una conexión tipo puente entre dos células. [1] Esto tiene lugar a través de un pilus . [2] [ cita completa necesaria ] Es un modo de reproducción parasexual en bacterias.
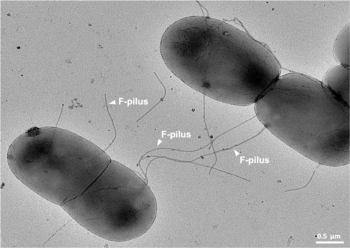
Es un mecanismo de transferencia horizontal de genes como lo son la transformación y la transducción, aunque estos otros dos mecanismos no implican contacto de célula a célula. [4]
La conjugación bacteriana clásica de E. coli suele considerarse el equivalente bacteriano de la reproducción sexual o el apareamiento , ya que implica el intercambio de material genético. Sin embargo, no se trata de una reproducción sexual, ya que no se produce ningún intercambio de gametos ni, de hecho, no se genera un nuevo organismo : en su lugar, se transforma un organismo existente. Durante la conjugación clásica de E. coli , la célula donante proporciona un elemento genético conjugativo o movilizable que, en la mayoría de los casos, es un plásmido o un transposón . [5] La mayoría de los plásmidos conjugativos tienen sistemas que garantizan que la célula receptora no contenga ya un elemento similar.
La información genética transferida suele ser beneficiosa para el receptor. Los beneficios pueden incluir resistencia a los antibióticos , tolerancia a los xenobióticos o la capacidad de utilizar nuevos metabolitos . [1] Otros elementos pueden ser perjudiciales y pueden considerarse parásitos bacterianos .
La conjugación en Escherichia coli por zigogénesis espontánea [6] y en Mycobacterium smegmatis por transferencia conjugal distributiva [7] [8] difieren de la conjugación clásica de E. coli mejor estudiada en que estos casos implican una mezcla sustancial de los genomas parentales .
El proceso fue descubierto por Joshua Lederberg y Edward Tatum [9] en 1946.

Diagrama de conjugación
El factor F es un episoma (un plásmido que puede integrarse en el cromosoma bacteriano por recombinación homóloga ) con una longitud de aproximadamente 100 kb . Lleva su propio origen de replicación , el oriV , y un origen de transferencia, u oriT . [5] Solo puede haber una copia del plásmido F en una bacteria dada, ya sea libre o integrada, y las bacterias que poseen una copia se denominan F-positivas o F-plus (denominadas F + ). Las células que carecen de plásmidos F se denominan F-negativas o F-minus (F − ) y, como tales, pueden funcionar como células receptoras. [ cita requerida ]
Entre otra información genética, el plásmido F lleva un locus tra y trb , que juntos tienen una longitud de aproximadamente 33 kb y constan de unos 40 genes . El locus tra incluye el gen de la pilina y los genes reguladores, que juntos forman pili en la superficie celular. El locus también incluye los genes de las proteínas que se adhieren a la superficie de las bacterias F − e inician la conjugación. Aunque existe cierto debate sobre el mecanismo exacto de la conjugación, parece que los pili son las estructuras a través de las cuales se produce el intercambio de ADN. Los pili F son extremadamente resistentes al estrés mecánico y termoquímico, lo que garantiza una conjugación exitosa en una variedad de entornos. [10] Varias proteínas codificadas en el locus tra o trb parecen abrir un canal entre las bacterias y se cree que la enzima traD, ubicada en la base del pilus, inicia la fusión de la membrana.
Cuando la conjugación se inicia mediante una señal, la enzima relaxasa crea una muesca en una de las hebras del plásmido conjugativo en el oriT . La relaxasa puede funcionar sola o en un complejo de más de una docena de proteínas conocidas colectivamente como relaxosoma . En el sistema del plásmido F, la enzima relaxasa se llama TraI y el relaxosoma consta de TraI, TraY, TraM y el factor huésped integrado IHF. La hebra mellada, o hebra T , se desenrolla a continuación de la hebra intacta y se transfiere a la célula receptora en una dirección del extremo 5' al extremo 3'. La hebra restante se replica independientemente de la acción conjugativa (replicación vegetativa que comienza en el oriV ) o en concierto con la conjugación (replicación conjugativa similar a la replicación de círculo rodante del fago lambda ). La replicación conjugativa puede requerir una segunda muesca antes de que pueda producirse una transferencia exitosa. Un informe reciente afirma haber inhibido la conjugación con sustancias químicas que imitan un paso intermedio de este segundo evento de mellado. [11]
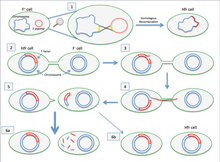
Si el plásmido F que se transfiere se ha integrado previamente en el genoma del donante (produciendo una cepa Hfr ["High Frequency of Recombination"]), parte del ADN cromosómico del donante también puede transferirse con el ADN del plásmido. [4] La cantidad de ADN cromosómico que se transfiere depende de cuánto tiempo permanezcan en contacto las dos bacterias conjugadas. En cepas comunes de laboratorio de E. coli, la transferencia de todo el cromosoma bacteriano tarda unos 100 minutos. El ADN transferido puede luego integrarse en el genoma del receptor mediante recombinación homóloga .
Un cultivo celular que contiene en su población células con plásmidos F no integrados normalmente también contiene algunas células que han integrado accidentalmente sus plásmidos. Son estas células las responsables de las transferencias de genes cromosómicos de baja frecuencia que ocurren en tales cultivos. Algunas cepas de bacterias con un plásmido F integrado pueden aislarse y cultivarse en cultivo puro. Debido a que estas cepas transfieren genes cromosómicos de manera muy eficiente, se las llama Hfr ( alta frecuencia de recombinación ) . El genoma de E. coli se cartografió originalmente mediante experimentos de apareamiento interrumpido en los que varias células Hfr en proceso de conjugación se separaron de los receptores después de menos de 100 minutos (inicialmente utilizando una licuadora Waring). Luego se investigaron los genes que se habían transferido.
Dado que la integración del plásmido F en el cromosoma de E. coli es un fenómeno espontáneo poco frecuente y que los numerosos genes que promueven la transferencia de ADN se encuentran en el genoma del plásmido y no en el genoma bacteriano, se ha argumentado que la transferencia conjugativa de genes bacterianos, tal como ocurre en el sistema Hfr de E. coli , no es una adaptación evolutiva del huésped bacteriano, ni es probable que sea ancestral al sexo eucariota. [13]
Cigogénesis espontánea en E. coli
Además de la conjugación bacteriana clásica descrita anteriormente para E. coli , se observa una forma de conjugación denominada cigogénesis espontánea (apareamiento Z para abreviar) en ciertas cepas de E. coli . [6] En el apareamiento Z hay una mezcla genética completa y se forman diploides inestables que eliminan células fenotípicamente haploides, de las cuales algunas muestran un fenotipo parental y algunas son recombinantes verdaderos .
La conjugación en Mycobacteria smegmatis , al igual que la conjugación en E. coli , requiere un contacto estable y prolongado entre una cepa donante y una receptora, es resistente a la DNasa y el ADN transferido se incorpora al cromosoma receptor mediante recombinación homóloga. Sin embargo, a diferencia de la conjugación Hfr de E. coli , la conjugación micobacteriana se basa en cromosomas en lugar de en plásmidos. [7] [8] Además, a diferencia de la conjugación Hfr de E. coli , en M. smegmatis todas las regiones del cromosoma se transfieren con eficiencias comparables. Las longitudes de los segmentos donantes varían ampliamente, pero tienen una longitud promedio de 44,2 kb. Dado que se transfiere una media de 13 tractos, el total promedio de ADN transferido por genoma es de 575 kb. [8] Este proceso se conoce como "transferencia conyugal distributiva". [7] [8] Gray et al. [7] encontraron una mezcla sustancial de los genomas parentales como resultado de la conjugación y consideraron que esta mezcla recordaba a la observada en los productos meióticos de la reproducción sexual.
Las arqueas hipertermófilas codifican pili estructuralmente similares a los pili conjugativos bacterianos. [14] Sin embargo, a diferencia de las bacterias, donde el aparato de conjugación típicamente media la transferencia de elementos genéticos móviles, como plásmidos o transposones, la maquinaria conjugativa de las arqueas hipertermófilas, llamada Ced (sistema crenarqueal para el intercambio de ADN) [15] y Ted (sistema termoproteal para el intercambio de ADN), [14] parece ser responsable de la transferencia de ADN celular entre miembros de la misma especie. Se ha sugerido que en estas arqueas la maquinaria de conjugación ha sido completamente domesticada para promover la reparación del ADN a través de la recombinación homóloga en lugar de la propagación de elementos genéticos móviles. [14] Además del pilus conjugativo similar a VirB2, los sistemas Ced y Ted incluyen componentes para el poro de apareamiento transmembrana similar a VirB6 y la ATPasa similar a VirB4. [14]

Las bacterias relacionadas con la fijación de nitrógeno Rhizobia son un caso interesante de conjugación entre reinos . [16] Por ejemplo, el plásmido inductor de tumores (Ti) de Agrobacterium y el plásmido inductor de tumores de raíz (Ri) de A. rhizogenes contienen genes que son capaces de transferirse a las células vegetales. La expresión de estos genes transforma eficazmente las células vegetales en fábricas productoras de opinas . Las bacterias utilizan las opinas como fuentes de nitrógeno y energía. Las células infectadas forman agallas de corona o tumores de raíz . Los plásmidos Ti y Ri son, por tanto, endosimbiontes de las bacterias, que a su vez son endosimbiontes (o parásitos) de la planta infectada. [ cita requerida ]
Los plásmidos Ti y Ri también pueden transferirse entre bacterias mediante un sistema (el operón tra o de transferencia ) que es diferente e independiente del sistema utilizado para la transferencia entre reinos (el operón vir o de virulencia ). Estas transferencias crean cepas virulentas a partir de cepas que antes no lo eran. [ cita requerida ]
La conjugación es un medio conveniente para transferir material genético a una variedad de objetivos. En los laboratorios, se han reportado transferencias exitosas de bacterias a levaduras, [17] plantas, células de mamíferos, [18] [19] diatomeas [20] y mitocondrias de mamíferos aisladas . [21] La conjugación tiene ventajas sobre otras formas de transferencia genética, incluyendo una mínima alteración de la envoltura celular del objetivo y la capacidad de transferir cantidades relativamente grandes de material genético (ver la discusión anterior sobre la transferencia de cromosomas de E. coli ). En ingeniería vegetal, la conjugación similar a Agrobacterium complementa otros vehículos estándar como el virus del mosaico del tabaco (TMV). Si bien el TMV es capaz de infectar a muchas familias de plantas, estas son principalmente dicotiledóneas herbáceas . La conjugación similar a Agrobacterium también se usa principalmente para dicotiledóneas, pero los receptores monocotiledóneos no son poco comunes. [ cita requerida ]