La transcripción es el proceso de copiar un segmento de ADN en ARN. Los segmentos de ADN transcritos en moléculas de ARN que pueden codificar proteínas producen ARN mensajero (ARNm). Otros segmentos de ADN se transcriben en moléculas de ARN llamadas ARN no codificantes (ARNnc).
Tanto el ADN como el ARN son ácidos nucleicos que utilizan pares de bases de nucleótidos como lenguaje complementario . Durante la transcripción, una secuencia de ADN es leída por una ARN polimerasa que produce una cadena de ARN complementaria y antiparalela llamada transcripción primaria .
En virología , el término transcripción se utiliza para referirse a la síntesis de ARN mensajero a partir de una molécula de ARN viral. El genoma de muchos virus de ARN [a] está compuesto de ARN de sentido negativo que actúa como plantilla para el ARN mensajero viral de sentido positivo, un paso necesario en la síntesis de proteínas virales necesarias para la replicación viral . Este proceso es catalizado por una ARN polimerasa dependiente de ARN viral . [1]
Una unidad de transcripción de ADN que codifica una proteína puede contener tanto una secuencia codificante , que se traducirá en la proteína, como secuencias reguladoras , que dirigen y regulan la síntesis de esa proteína. La secuencia reguladora anterior ( en sentido ascendente ) a la secuencia codificante se denomina cinco regiones no traducidas principales (5'UTR); la secuencia posterior ( en sentido descendente ) a la secuencia codificante se denomina tres regiones no traducidas principales (3'UTR). [2]
A diferencia de la replicación del ADN , la transcripción da como resultado un complemento de ARN que incluye el nucleótido uracilo (U) en todos los casos en los que habría aparecido timina (T) en un complemento de ADN. [3]
Sólo una de las dos cadenas de ADN sirve como plantilla para la transcripción. La cadena antisentido de ADN es leída por la ARN polimerasa desde el extremo 3' hasta el extremo 5' durante la transcripción (3' → 5'). El ARN complementario se crea en la dirección opuesta, en la dirección 5' → 3', coincidiendo con la secuencia de la cadena sentido excepto que cambia el uracilo por timina. Esta direccionalidad se debe a que la ARN polimerasa sólo puede añadir nucleótidos al extremo 3' de la cadena de ARNm en crecimiento. Este uso de sólo la cadena de ADN 3' → 5' elimina la necesidad de los fragmentos de Okazaki que se ven en la replicación del ADN. [2] Esto también elimina la necesidad de un cebador de ARN para iniciar la síntesis de ARN, como es el caso en la replicación del ADN.
La cadena de ADN que no es molde (con sentido) se denomina cadena codificante , porque su secuencia es la misma que la del ARN transcrito recién creado (excepto por la sustitución de uracilo por timina). Esta es la cadena que se utiliza por convención al presentar una secuencia de ADN. [4]
La transcripción tiene algunos mecanismos de corrección, pero son menos numerosos y menos eficaces que los controles para copiar el ADN. Como resultado, la transcripción tiene una fidelidad de copia menor que la replicación del ADN. [5]
La transcripción se divide en iniciación , escape del promotor , elongación y terminación . [6]

La preparación para la transcripción en mamíferos está regulada por muchos elementos cis-reguladores , incluyendo el promotor central y los elementos proximales al promotor que se encuentran cerca de los sitios de inicio de la transcripción de los genes. Los promotores centrales combinados con factores de transcripción generales son suficientes para dirigir la iniciación de la transcripción, pero generalmente tienen una actividad basal baja. [7] Otros módulos cis-reguladores importantes se localizan en regiones de ADN que están distantes de los sitios de inicio de la transcripción. Estos incluyen potenciadores , silenciadores , aislantes y elementos de anclaje. [8] Entre esta constelación de elementos, los potenciadores y sus factores de transcripción asociados tienen un papel principal en el inicio de la transcripción génica. [9] Un potenciador localizado en una región de ADN distante del promotor de un gen puede tener un efecto muy grande en la transcripción génica, con algunos genes experimentando una transcripción hasta 100 veces mayor debido a un potenciador activado. [10]
Los potenciadores son regiones del genoma que son elementos importantes de regulación de genes. Los potenciadores controlan programas de transcripción de genes específicos de cada tipo de célula, la mayoría de las veces mediante bucles a través de largas distancias para llegar a la proximidad física con los promotores de sus genes objetivo. [11] Si bien existen cientos de miles de regiones de ADN potenciadoras, [12] para un tipo particular de tejido solo los potenciadores específicos se acercan a los promotores que regulan. En un estudio de neuronas corticales cerebrales, se encontraron 24.937 bucles, que llevaban potenciadores a sus promotores objetivo. [10] Múltiples potenciadores, cada uno a menudo a decenas o cientos de miles de nucleótidos de distancia de sus genes objetivo, se unen a sus promotores de genes objetivo y pueden coordinarse entre sí para controlar la transcripción de su gen objetivo común. [11]
La ilustración esquemática de esta sección muestra un potenciador que forma un bucle para acercarse físicamente al promotor de un gen diana. El bucle está estabilizado por un dímero de una proteína conectora (por ejemplo, el dímero de CTCF o YY1 ), con un miembro del dímero anclado a su motivo de unión en el potenciador y el otro miembro anclado a su motivo de unión en el promotor (representado por los zigzags rojos en la ilustración). [13] Varios factores de transcripción específicos de la función celular (hay alrededor de 1.600 factores de transcripción en una célula humana [14] ) generalmente se unen a motivos específicos en un potenciador [15] y una pequeña combinación de estos factores de transcripción unidos al potenciador, cuando se acercan a un promotor mediante un bucle de ADN, gobiernan el nivel de transcripción del gen diana. El mediador (un complejo que generalmente consta de alrededor de 26 proteínas en una estructura interactuante) comunica señales reguladoras de los factores de transcripción unidos al ADN del potenciador directamente a la enzima ARN polimerasa II (pol II) unida al promotor. [16]
Los potenciadores, cuando están activos, generalmente se transcriben a partir de ambas cadenas de ADN con ARN polimerasas que actúan en dos direcciones diferentes, produciendo dos ARN potenciadores (eARN), como se ilustra en la Figura. [17] Un potenciador inactivo puede estar unido a un factor de transcripción inactivo. La fosforilación del factor de transcripción puede activarlo y ese factor de transcripción activado puede entonces activar el potenciador al que está unido (ver la pequeña estrella roja que representa la fosforilación del factor de transcripción unido al potenciador en la ilustración). [18] Un potenciador activado comienza la transcripción de su ARN antes de activar la transcripción del ARN mensajero de su gen diana. [19]

La regulación de la transcripción en aproximadamente el 60% de los promotores también está controlada por la metilación de las citosinas dentro de los dinucleótidos CpG (donde la citosina 5' es seguida por los sitios guanina o CpG 3' ). La 5-metilcitosina (5-mC) es una forma metilada de la base de ADN citosina (ver Figura). 5-mC es un marcador epigenético que se encuentra predominantemente dentro de los sitios CpG. Alrededor de 28 millones de dinucleótidos CpG ocurren en el genoma humano. [20] En la mayoría de los tejidos de los mamíferos, en promedio, entre el 70% y el 80% de las citosinas CpG están metiladas (formando 5-metilCpG o 5-mCpG). [21] Sin embargo, las citosinas no metiladas dentro de las secuencias 5'citosina-guanina 3' a menudo ocurren en grupos, llamados islas CpG , en promotores activos. Alrededor del 60% de las secuencias promotoras tienen una isla CpG, mientras que solo alrededor del 6% de las secuencias potenciadoras tienen una isla CpG. [22] Las islas CpG constituyen secuencias reguladoras, ya que si las islas CpG están metiladas en el promotor de un gen esto puede reducir o silenciar la transcripción genética. [23]
La metilación del ADN regula la transcripción génica a través de la interacción con proteínas de dominio de unión a metilo (MBD), como MeCP2, MBD1 y MBD2. Estas proteínas MBD se unen con mayor fuerza a islas CpG altamente metiladas . [24] Estas proteínas MBD tienen tanto un dominio de unión a metil-CpG como un dominio de represión de la transcripción. [24] Se unen al ADN metilado y guían o dirigen complejos proteicos con actividad de remodelación de cromatina y/o modificación de histonas a islas CpG metiladas. Las proteínas MBD generalmente reprimen la cromatina local, por ejemplo, catalizando la introducción de marcas de histonas represivas o creando un entorno de cromatina represivo general a través de la remodelación de nucleosomas y la reorganización de la cromatina. [24]

Como se señaló en la sección anterior, los factores de transcripción son proteínas que se unen a secuencias de ADN específicas para regular la expresión de un gen. La secuencia de unión de un factor de transcripción en el ADN suele tener una longitud de unos 10 u 11 nucleótidos. Como se resumió en 2009, Vaquerizas et al. indicaron que hay aproximadamente 1.400 factores de transcripción diferentes codificados en el genoma humano por genes que constituyen aproximadamente el 6% de todos los genes que codifican proteínas humanas. [25] Aproximadamente el 94% de los sitios de unión de factores de transcripción (TFBS) que están asociados con genes sensibles a señales se encuentran en potenciadores, mientras que solo alrededor del 6% de dichos TFBS se encuentran en promotores. [15]
La proteína EGR1 es un factor de transcripción particular que es importante para la regulación de la metilación de las islas CpG. Un sitio de unión del factor de transcripción EGR1 se encuentra frecuentemente en secuencias potenciadoras o promotoras. [26] Hay alrededor de 12.000 sitios de unión para EGR1 en el genoma de los mamíferos y aproximadamente la mitad de los sitios de unión de EGR1 se encuentran en promotores y la otra mitad en potenciadores. [26] La unión de EGR1 a su sitio de unión de ADN objetivo es insensible a la metilación de citosina en el ADN. [26]
Aunque sólo se detectan pequeñas cantidades de la proteína del factor de transcripción EGR1 en células que no están estimuladas, la traducción del gen EGR1 en proteína una hora después de la estimulación aumenta drásticamente. [27] La producción de proteínas del factor de transcripción EGR1, en varios tipos de células, puede ser estimulada por factores de crecimiento, neurotransmisores, hormonas, estrés y lesiones. [27] En el cerebro, cuando se activan las neuronas, las proteínas EGR1 se regulan positivamente y se unen a (reclutan) las enzimas TET1 preexistentes que se producen en grandes cantidades en las neuronas. Las enzimas TET pueden catalizar la desmetilación de la 5-metilcitosina. Cuando los factores de transcripción EGR1 llevan las enzimas TET1 a los sitios de unión de EGR1 en los promotores, las enzimas TET pueden desmetilar las islas CpG metiladas en esos promotores. Tras la desmetilación, estos promotores pueden iniciar la transcripción de sus genes diana. Cientos de genes en las neuronas se expresan de manera diferencial después de la activación neuronal a través del reclutamiento de TET1 por parte de EGR1 a secuencias reguladoras metiladas en sus promotores. [26]
La metilación de los promotores también se altera en respuesta a señales. Las tres metiltransferasas de ADN de mamíferos (DNMT1, DNMT3A y DNMT3B) catalizan la adición de grupos metilo a las citosinas del ADN. Mientras que DNMT1 es una metiltransferasa de mantenimiento, DNMT3A y DNMT3B pueden llevar a cabo nuevas metilaciones. También existen dos isoformas de proteína de empalme producidas a partir del gen DNMT3A : las proteínas metiltransferasas de ADN DNMT3A1 y DNMT3A2. [28]
La isoforma de empalme DNMT3A2 se comporta como el producto de un gen inmediato-temprano clásico y, por ejemplo, se produce de forma robusta y transitoria después de la activación neuronal. [29] El lugar donde la isoforma de la ADN metiltransferasa DNMT3A2 se une y agrega grupos metilo a las citosinas parece estar determinado por modificaciones postraduccionales de las histonas. [30] [31] [32]
Por otra parte, la activación neuronal provoca la degradación de DNMT3A1 acompañada de una metilación reducida de al menos un promotor objetivo evaluado. [33]
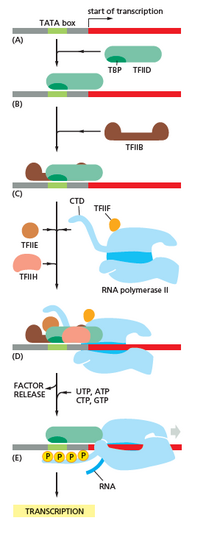
La transcripción comienza con la unión de la ARN polimerasa y uno o más factores de transcripción generales a una secuencia promotora de ADN para formar un complejo cerrado ARN polimerasa-promotor. En el complejo cerrado, el ADN promotor todavía es completamente bicatenario. [6]
La ARN polimerasa, con la ayuda de uno o más factores de transcripción generales, desenrolla aproximadamente 14 pares de bases de ADN para formar un complejo abierto ARN polimerasa-promotor. En el complejo abierto, el ADN promotor está parcialmente desenrollado y monocatenario. El ADN monocatenario expuesto se denomina "burbuja de transcripción". [6]
La ARN polimerasa, asistida por uno o más factores de transcripción generales, selecciona luego un sitio de inicio de la transcripción en la burbuja de transcripción, se une a un NTP iniciador y un NTP extensor (o un cebador de ARN corto y un NTP extensor) complementarios a la secuencia del sitio de inicio de la transcripción, y cataliza la formación de enlaces para producir un producto de ARN inicial. [6]
En las bacterias , la holoenzima de la ARN polimerasa consta de cinco subunidades: 2 subunidades α, 1 subunidad β, 1 subunidad β' y 1 subunidad ω. En las bacterias, hay un factor de transcripción de ARN general conocido como factor sigma . La enzima central de la ARN polimerasa se une al factor de transcripción general bacteriano (sigma) para formar la holoenzima de la ARN polimerasa y luego se une a un promotor. [6] (La ARN polimerasa se denomina holoenzima cuando la subunidad sigma está unida a la enzima central que consta de 2 subunidades α, 1 subunidad β y 1 subunidad β' solamente). A diferencia de los eucariotas, el nucleótido iniciador del ARNm bacteriano naciente no está cubierto con un nucleótido de guanina modificado. El nucleótido iniciador de las transcripciones bacterianas contiene un trifosfato 5′ (5′-PPP), que puede utilizarse para el mapeo de los sitios de inicio de la transcripción en todo el genoma. [35]
En arqueas y eucariotas , la ARN polimerasa contiene subunidades homólogas a cada una de las cinco subunidades de la ARN polimerasa en bacterias y también contiene subunidades adicionales. En arqueas y eucariotas, las funciones del factor de transcripción general bacteriano sigma son realizadas por múltiples factores de transcripción generales que trabajan juntos. [6] En arqueas, hay tres factores de transcripción generales: TBP , TFB y TFE . En eucariotas, en la transcripción dependiente de la ARN polimerasa II , hay seis factores de transcripción generales: TFIIA , TFIIB (un ortólogo de TFB arqueal), TFIID (un factor multisubunidad en el que la subunidad clave, TBP , es un ortólogo de TBP arqueal), TFIIE (un ortólogo de TFE arqueal), TFIIF y TFIIH . El TFIID es el primer componente que se une al ADN debido a la unión de TBP, mientras que TFIIH es el último componente en ser reclutado. En arqueas y eucariotas, el complejo cerrado promotor-ARN polimerasa se suele denominar " complejo de preiniciación ". [36]
La iniciación de la transcripción está regulada por proteínas adicionales, conocidas como activadores y represores , y, en algunos casos, coactivadores o correpresores asociados , que modulan la formación y la función del complejo de iniciación de la transcripción. [6]
Después de sintetizar el primer enlace, la ARN polimerasa debe escapar del promotor. Durante este tiempo, existe una tendencia a liberar la transcripción de ARN y producir transcripciones truncadas. Esto se denomina iniciación abortiva y es común tanto para eucariotas como para procariotas. [37] La iniciación abortiva continúa ocurriendo hasta que se sintetiza un producto de ARN de una longitud umbral de aproximadamente 10 nucleótidos, momento en el que se produce el escape del promotor y se forma un complejo de elongación de transcripción. [ cita requerida ]
Mecanísticamente, el escape del promotor ocurre a través del aplastamiento del ADN , lo que proporciona la energía necesaria para romper las interacciones entre la holoenzima ARN polimerasa y el promotor. [38]
En las bacterias, históricamente se creía que el factor sigma se liberaba definitivamente después de que se despejaba el promotor. Esta teoría se conocía como el modelo de liberación obligada. Sin embargo, datos posteriores demostraron que tras la depuración del promotor y después de ella, el factor sigma se libera según un modelo estocástico conocido como el modelo de liberación estocástica . [39]
En eucariotas, en un promotor dependiente de la ARN polimerasa II, tras la eliminación del promotor, TFIIH fosforila la serina 5 en el dominio carboxiterminal de la ARN polimerasa II, lo que conduce al reclutamiento de la enzima de protección (CE). [40] [41] Aún no se conoce el mecanismo exacto de cómo la CE induce la eliminación del promotor en eucariotas.

Una hebra del ADN, la hebra molde (o hebra no codificante), se utiliza como plantilla para la síntesis de ARN. A medida que avanza la transcripción, la ARN polimerasa atraviesa la hebra molde y utiliza la complementariedad de apareamiento de bases con la plantilla de ADN para crear una copia de ARN (que se alarga durante el recorrido). Aunque la ARN polimerasa atraviesa la hebra molde de 3' → 5', la hebra codificante (no molde) y el ARN recién formado también se pueden utilizar como puntos de referencia, por lo que la transcripción puede describirse como que ocurre de 5' → 3'. Esto produce una molécula de ARN de 5' → 3', una copia exacta de la hebra codificante (excepto que las timinas se reemplazan con uracilos y los nucleótidos están compuestos de un azúcar ribosa (5 carbonos), mientras que el ADN tiene desoxirribosa (un átomo de oxígeno menos) en su estructura de azúcar-fosfato). [3]
La transcripción del ARNm puede implicar múltiples polimerasas de ARN en una única plantilla de ADN y múltiples rondas de transcripción (amplificación de un ARNm particular), por lo que se pueden producir rápidamente muchas moléculas de ARNm a partir de una única copia de un gen. [ cita requerida ] Las tasas de elongación características en procariotas y eucariotas son de aproximadamente 10 a 100 nts/seg. [42] En eucariotas, sin embargo, los nucleosomas actúan como barreras importantes para la transcripción de polimerasas durante la elongación de la transcripción. [43] [44] En estos organismos, la pausa inducida por los nucleosomas puede ser regulada por factores de elongación de la transcripción como TFIIS. [44]
La elongación también implica un mecanismo de corrección de errores que puede reemplazar bases incorporadas incorrectamente. En los eucariotas, esto puede corresponderse con pausas breves durante la transcripción que permiten que se unan los factores de edición de ARN apropiados. Estas pausas pueden ser intrínsecas a la ARN polimerasa o deberse a la estructura de la cromatina. [ cita requerida ]
Las roturas de doble cadena en regiones de ADN transcritas activamente se reparan mediante recombinación homóloga durante las fases S y G2 del ciclo celular . [45] [46] Dado que la transcripción mejora la accesibilidad del ADN a sustancias químicas exógenas y metabolitos internos que pueden causar lesiones recombinogénicas, la recombinación homóloga de una secuencia de ADN particular puede ser fuertemente estimulada por la transcripción. [47]
Las bacterias utilizan dos estrategias diferentes para la terminación de la transcripción: la terminación independiente de Rho y la terminación dependiente de Rho. En la terminación de la transcripción independiente de Rho , la transcripción del ARN se detiene cuando la molécula de ARN recién sintetizada forma un bucle de horquilla rico en GC seguido de una serie de Us. Cuando se forma la horquilla, la tensión mecánica rompe los enlaces débiles rU-dA, que ahora llenan el híbrido ADN-ARN. Esto extrae la transcripción poli-U del sitio activo de la ARN polimerasa, terminando la transcripción. En la terminación dependiente de Rho, Rho , un factor proteico, desestabiliza la interacción entre la plantilla y el ARNm, liberando así el ARNm recién sintetizado del complejo de elongación. [48]
La terminación de la transcripción en eucariotas es menos conocida que en bacterias, pero implica la escisión de la nueva transcripción seguida de la adición independiente de la plantilla de adeninas en su nuevo extremo 3', en un proceso llamado poliadenilación . [49]
Más allá de la terminación por una secuencia terminadora (que es parte de un gen ), la transcripción también puede necesitar ser terminada cuando encuentra condiciones tales como daño del ADN o una horquilla de replicación activa . En bacterias, la ATPasa Mfd puede eliminar una ARN polimerasa bloqueada en una lesión abriendo su pinza. También recluta maquinaria de reparación por escisión de nucleótidos para reparar la lesión. Se propone que Mfd también resuelva conflictos entre la replicación y la transcripción del ADN. [50] En eucariotas, la ATPasa TTF2 ayuda a suprimir la acción de RNAP I y II durante la mitosis , previniendo errores en la segregación cromosómica. [51] En arqueas, se propone que la ATPasa Eta juega un papel similar. [52]
La ARN polimerasa juega un papel muy importante en todos los pasos, incluidos los cambios postranscripcionales en el ARN.
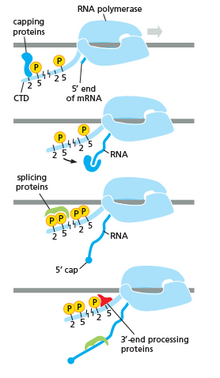
Como se muestra en la imagen de la derecha se evidencia que el CTD (C Terminal Domain) es una cola que cambia de forma; esta cola será utilizada como portadora de splicing, capping y poliadenilación , como se muestra en la imagen de la izquierda. [53]
Los inhibidores de la transcripción se pueden utilizar como antibióticos contra, por ejemplo, bacterias patógenas ( antibacterianos ) y hongos ( antifúngicos ). Un ejemplo de un antibacteriano de este tipo es la rifampicina , que inhibe la transcripción bacteriana de ADN en ARNm inhibiendo la ARN polimerasa dependiente de ADN mediante la unión a su subunidad beta, mientras que la 8-hidroxiquinolina es un inhibidor de la transcripción antifúngico. [54] Los efectos de la metilación de histonas también pueden funcionar para inhibir la acción de la transcripción. Productos naturales bioactivos potentes como la triptolida que inhiben la transcripción de mamíferos a través de la inhibición de la subunidad XPB del factor de transcripción general TFIIH se ha informado recientemente como un conjugado de glucosa para dirigirse a las células cancerosas hipóxicas con una mayor producción de transportador de glucosa. [55]
En los vertebrados, la mayoría de los promotores de genes contienen una isla CpG con numerosos sitios CpG . [56] Cuando muchos de los sitios CpG del promotor de un gen están metilados , el gen se inhibe (silencia). [57] Los cánceres colorrectales suelen tener de 3 a 6 mutaciones impulsoras y de 33 a 66 mutaciones autoestopistas o pasajeras. [58] Sin embargo, la inhibición transcripcional (silenciamiento) puede ser más importante que la mutación en la causa de la progresión al cáncer. Por ejemplo, en los cánceres colorrectales, alrededor de 600 a 800 genes son inhibidos transcripcionalmente por la metilación de la isla CpG (ver regulación de la transcripción en el cáncer ). La represión transcripcional en el cáncer también puede ocurrir por otros mecanismos epigenéticos , como la producción alterada de microARN . [59] En el cáncer de mama, la represión transcripcional de BRCA1 puede ocurrir con mayor frecuencia por una sobreproducción de microARN-182 que por hipermetilación del promotor BRCA1 (ver Baja expresión de BRCA1 en cánceres de mama y ovario ). [ cita requerida ]
Las unidades de transcripción activas se agrupan en el núcleo, en sitios discretos llamados fábricas de transcripción o eucromatina . Dichos sitios se pueden visualizar permitiendo que las polimerasas activas extiendan sus transcripciones en precursores marcados (Br-UTP o Br-U) y marcando inmunológicamente el ARN naciente marcado. Las fábricas de transcripción también se pueden localizar utilizando hibridación in situ con fluorescencia o marcadas con anticuerpos dirigidos contra las polimerasas. Hay ~10.000 fábricas en el nucleoplasma de una célula HeLa , entre las que se encuentran ~8.000 fábricas de polimerasa II y ~2.000 fábricas de polimerasa III. Cada fábrica de polimerasa II contiene ~8 polimerasas. Como la mayoría de las unidades de transcripción activas están asociadas con una sola polimerasa, cada fábrica suele contener ~8 unidades de transcripción diferentes. Estas unidades pueden estar asociadas a través de promotores y/o potenciadores, con bucles que forman una "nube" alrededor del factor. [60]
François Jacob y Jacques Monod fueron los primeros en plantear la hipótesis de que una molécula que permite que el material genético se convierta en proteína . Severo Ochoa ganó el Premio Nobel de Fisiología o Medicina en 1959 por desarrollar un proceso para sintetizar ARN in vitro con polinucleótido fosforilasa , que fue útil para descifrar el código genético . La síntesis de ARN por ARN polimerasa fue establecida in vitro por varios laboratorios en 1965; sin embargo, el ARN sintetizado por estas enzimas tenía propiedades que sugerían la existencia de un factor adicional necesario para terminar la transcripción correctamente. [ cita requerida ]
Roger D. Kornberg ganó el Premio Nobel de Química en 2006 "por sus estudios de la base molecular de la transcripción eucariota ". [61]

La transcripción se puede medir y detectar de diversas maneras: [ cita requerida ]

Algunos virus (como el VIH , causante del sida ) tienen la capacidad de transcribir el ARN en ADN. El VIH tiene un genoma de ARN que se transcribe de forma inversa en ADN. El ADN resultante se puede fusionar con el genoma de ADN de la célula huésped. La enzima principal responsable de la síntesis de ADN a partir de una plantilla de ARN se denomina transcriptasa inversa . [64]
En el caso del VIH, la transcriptasa inversa es responsable de sintetizar una cadena complementaria de ADN (ADNc) al genoma de ARN viral. La enzima ribonucleasa H luego digiere la cadena de ARN, y la transcriptasa inversa sintetiza una cadena complementaria de ADN para formar una estructura de ADN de doble hélice (ADNc). El ADNc se integra en el genoma de la célula huésped por la enzima integrasa , que hace que la célula huésped genere proteínas virales que se reensamblan en nuevas partículas virales. En el VIH, posteriormente a esto, la célula huésped sufre muerte celular programada, o apoptosis , de células T. [65] Sin embargo, en otros retrovirus, la célula huésped permanece intacta mientras el virus brota fuera de la célula. [ cita requerida ]
Algunas células eucariotas contienen una enzima con actividad de transcripción inversa llamada telomerasa . La telomerasa transporta una plantilla de ARN a partir de la cual sintetiza un telómero , una secuencia repetitiva de ADN, hasta el extremo de los cromosomas lineales. Es importante porque cada vez que se duplica un cromosoma lineal, se acorta. Con el telómero en los extremos de los cromosomas, el acortamiento elimina parte de la secuencia repetida no esencial, en lugar de la secuencia de ADN que codifica la proteína más alejada del extremo del cromosoma.
La telomerasa se activa a menudo en las células cancerosas para permitir que dupliquen sus genomas indefinidamente sin perder una importante secuencia de ADN que codifica proteínas. La activación de la telomerasa podría ser parte del proceso que permite que las células cancerosas se vuelvan inmortales. Se ha demostrado que el factor de inmortalización del cáncer a través del alargamiento de los telómeros debido a la telomerasa ocurre en el 90% de todos los tumores cancerígenos in vivo, y el 10% restante utiliza una ruta alternativa de mantenimiento de los telómeros llamada ALT o alargamiento alternativo de los telómeros. [66]