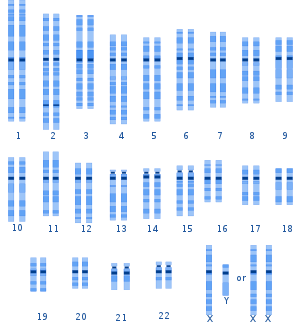

La variación genética humana son las diferencias genéticas dentro y entre poblaciones . Puede haber múltiples variantes de un gen determinado en la población humana ( alelos ), una situación llamada polimorfismo .
No existen dos seres humanos genéticamente idénticos. Incluso los gemelos monocigóticos (que se desarrollan a partir de un cigoto) presentan diferencias genéticas poco frecuentes debido a mutaciones que ocurren durante el desarrollo y a variaciones en el número de copias de los genes . [1] Las diferencias entre individuos, incluso entre individuos estrechamente relacionados, son la clave de técnicas como la huella genética .
El genoma humano tiene una longitud total de aproximadamente 3.200 millones de pares de bases (pb) en 46 cromosomas de ADN, así como un poco menos de 17.000 pb de ADN en las mitocondrias celulares . En 2015, la diferencia típica entre el genoma de un individuo y el genoma de referencia se estimó en 20 millones de pares de bases (o el 0,6% del total). [2] En 2017, había un total de 324 millones de variantes conocidas a partir de genomas humanos secuenciados . [3]
Comparativamente hablando, los humanos somos una especie genéticamente homogénea. Aunque se encuentra una pequeña cantidad de variantes genéticas con mayor frecuencia en ciertas regiones geográficas o en personas con ascendencia de esas regiones, esta variación representa una pequeña porción (~15%) de la variabilidad del genoma humano. La mayor parte de la variación existe dentro de los miembros de cada población humana. A modo de comparación, los macacos rhesus muestran una diversidad de secuencias de ADN 2,5 veces mayor en comparación con los humanos. [4] Estas tasas difieren según las macromoléculas que se estén analizando. Los chimpancés tienen más variación genética que los humanos cuando examinan el ADN nuclear, pero los humanos tienen más variación genética cuando lo examinan a nivel de proteínas. [5]
La falta de discontinuidades en las distancias genéticas entre las poblaciones humanas, la ausencia de ramas discretas en la especie humana y la sorprendente homogeneidad de los seres humanos a nivel mundial implican que no existe una base científica para inferir razas o subespecies en los humanos, y para la mayoría de los rasgos , hay mucha más variación dentro de las poblaciones que entre ellas. [6] [ 7] [8] [9] [10] [11] [12] [13] A pesar de esto, los estudios genéticos modernos han encontrado diferencias genéticas promedio sustanciales entre las poblaciones humanas en rasgos como el color de la piel, las dimensiones corporales, la digestión de la lactosa y el almidón, las adaptaciones a grandes altitudes, la respuesta a los fármacos, los receptores del gusto y la predisposición a desarrollar enfermedades particulares. [14] [12] La mayor diversidad se encuentra dentro y entre las poblaciones de África , [15] y disminuye gradualmente con el aumento de la distancia desde el continente africano, en consonancia con la teoría de los orígenes humanos de Fuera de África . [15]
El estudio de la variación genética humana tiene importancia evolutiva y aplicaciones médicas. Puede ayudar a los científicos a reconstruir y comprender los patrones de migración humana del pasado. En medicina, el estudio de la variación genética humana puede ser importante porque algunos alelos causantes de enfermedades se dan con mayor frecuencia en ciertos grupos de población. Por ejemplo, la mutación que causa la anemia de células falciformes se encuentra con mayor frecuencia en personas con ascendencia de ciertas poblaciones del África subsahariana, el sur de Europa, Arabia e India, debido a la presión evolutiva de los mosquitos portadores de malaria en estas regiones.
Nuevos hallazgos muestran que cada ser humano tiene en promedio 60 nuevas mutaciones en comparación con sus padres. [16] [17]
Las causas de las diferencias entre individuos incluyen la distribución independiente , el intercambio de genes (entrecruzamiento y recombinación) durante la reproducción (a través de la meiosis ) y varios eventos mutacionales .
Existen al menos tres razones por las que existe variación genética entre poblaciones. La selección natural puede conferir una ventaja adaptativa a los individuos en un entorno específico si un alelo proporciona una ventaja competitiva. Los alelos bajo selección probablemente se presenten solo en aquellas regiones geográficas donde confieren una ventaja. Un segundo proceso importante es la deriva genética , que es el efecto de los cambios aleatorios en el acervo genético, en condiciones en las que la mayoría de las mutaciones son neutrales (es decir, no parecen tener ningún efecto selectivo positivo o negativo en el organismo). Finalmente, las pequeñas poblaciones migrantes tienen diferencias estadísticas -llamadas el efecto fundador- con respecto a las poblaciones generales de donde se originaron; cuando estos migrantes se establecen en nuevas áreas, su población descendiente generalmente difiere de su población de origen: predominan genes diferentes y es menos diversa genéticamente.
En los seres humanos, la causa principal es la deriva genética . [18] Los efectos fundadores seriales y el pequeño tamaño de la población en el pasado (que aumenta la probabilidad de deriva genética) pueden haber tenido una influencia importante en las diferencias neutrales entre las poblaciones. [ cita requerida ] La segunda causa principal de variación genética se debe al alto grado de neutralidad de la mayoría de las mutaciones . Un pequeño, pero significativo número de genes parece haber sufrido una selección natural reciente, y estas presiones selectivas a veces son específicas de una región. [19] [20]
La variación genética entre los seres humanos se produce en muchas escalas, desde alteraciones importantes en el cariotipo humano hasta cambios de un solo nucleótido . [21] Las anomalías cromosómicas se detectan en 1 de cada 160 nacimientos humanos vivos. Aparte de los trastornos de los cromosomas sexuales , la mayoría de los casos de aneuploidía resultan en la muerte del feto en desarrollo ( aborto espontáneo ); los cromosomas autosómicos adicionales más comunes entre los nacidos vivos son el 21 , el 18 y el 13. [22]
La diversidad de nucleótidos es la proporción promedio de nucleótidos que difieren entre dos individuos. En 2004, se estimó que la diversidad de nucleótidos humanos era del 0,1% [23] al 0,4% de los pares de bases . [24] En 2015, el Proyecto 1000 Genomas , que secuenció a mil individuos de 26 poblaciones humanas, descubrió que "un genoma [individual] típico difiere del genoma humano de referencia en 4,1 millones a 5,0 millones de sitios... afectando a 20 millones de bases de secuencia"; la última cifra corresponde al 0,6% del número total de pares de bases. [2] Casi todos (>99,9%) de estos sitios son pequeñas diferencias, ya sean polimorfismos de un solo nucleótido o breves inserciones o deleciones ( indels ) en la secuencia genética, pero las variaciones estructurales representan un mayor número de pares de bases que los SNP y los indels. [2] [25]
En 2017 [actualizar], la base de datos de polimorfismos de nucleótido único ( dbSNP ), que enumera los SNP y otras variantes, enumeró 324 millones de variantes encontradas en genomas humanos secuenciados. [3]

Un polimorfismo de un solo nucleótido (SNP) es una diferencia en un solo nucleótido entre miembros de una especie que se produce en al menos el 1% de la población. Los 2.504 individuos caracterizados por el Proyecto de los 1000 Genomas tenían 84,7 millones de SNP entre ellos. [2] Los SNP son el tipo más común de variación de secuencia, y en 1998 se estimó que representaban el 90% de todas las variantes de secuencia. [26] Otras variaciones de secuencia son los intercambios, deleciones e inserciones de una sola base. [27] Los SNP se producen en promedio cada 100 a 300 bases [28] y, por lo tanto, son la principal fuente de heterogeneidad.
Un SNP funcional o no sinónimo es aquel que afecta a algún factor, como el empalme de genes o el ARN mensajero , y por lo tanto causa una diferencia fenotípica entre los miembros de la especie. Entre el 3% y el 5% de los SNP humanos son funcionales (véase el Proyecto Internacional HapMap ). Los SNP neutros o sinónimos siguen siendo útiles como marcadores genéticos en estudios de asociación de todo el genoma , debido a su gran número y a la herencia estable a lo largo de las generaciones. [26]
Un SNP codificante es aquel que se produce dentro de un gen. Hay 105 SNP de referencia humana que dan lugar a codones de terminación prematuros en 103 genes. Esto corresponde al 0,5 % de los SNP codificantes. Se producen debido a la duplicación segmentaria en el genoma. Estos SNP dan lugar a la pérdida de proteínas, pero todos estos alelos de SNP son comunes y no se purifican en la selección negativa . [29]
La variación estructural es la variación en la estructura de los cromosomas de un organismo . Las variaciones estructurales, como la variación en el número de copias y las deleciones , inversiones , inserciones y duplicaciones , explican mucha más variación genética humana que la diversidad de un solo nucleótido. Esto se concluyó en 2007 a partir del análisis de las secuencias completas diploides de los genomas de dos humanos: Craig Venter y James D. Watson . Esto se sumó a las dos secuencias haploides que eran amalgamas de secuencias de muchos individuos, publicadas por el Proyecto Genoma Humano y Celera Genomics respectivamente. [30]
Según el Proyecto 1000 Genomas, un ser humano típico tiene entre 2100 y 2500 variaciones estructurales, que incluyen aproximadamente 1000 grandes deleciones, 160 variantes de número de copias, 915 inserciones Alu , 128 inserciones L1 , 51 inserciones SVA, 4 NUMT y 10 inversiones. [2]
Una variación en el número de copias (CNV) es una diferencia en el genoma debido a la eliminación o duplicación de grandes regiones de ADN en algún cromosoma. Se estima que el 0,4% de los genomas de humanos no emparentados difieren con respecto al número de copias. Cuando se incluye la variación en el número de copias, se estima que la variación genética entre humanos es de al menos el 0,5% (99,5% de similitud). [31] [32] [33] [34] Las variaciones en el número de copias se heredan, pero también pueden surgir durante el desarrollo. [35] [36] [37] [38]
Pratas et al. [40] han construido un mapa visual con las regiones con alta variación genómica del conjunto de referencia humano-moderno en relación con un neandertal de 50k [39].
La variación epigenética es la variación en las etiquetas químicas que se adhieren al ADN y afectan la forma en que se leen los genes. Las etiquetas, "llamadas marcas epigenéticas, actúan como interruptores que controlan cómo se pueden leer los genes". [41] En algunos alelos, el estado epigenético del ADN y el fenotipo asociado pueden heredarse a lo largo de generaciones de individuos . [42]
La variabilidad genética es una medida de la tendencia de los genotipos individuales de una población a variar (volverse diferentes) entre sí. La variabilidad es diferente de la diversidad genética , que es la cantidad de variación observada en una población particular. La variabilidad de un rasgo es cuánto tiende a variar ese rasgo en respuesta a influencias ambientales y genéticas .
En biología , una clina es un continuo de especies , poblaciones, variedades o formas de organismos que exhiben diferencias fenotípicas y/o genéticas graduales a lo largo de un área geográfica, típicamente como resultado de la heterogeneidad ambiental. [43] [44] [45] En el estudio científico de la variación genética humana, una clina genética puede definirse rigurosamente y someterse a métricas cuantitativas.
En el estudio de la evolución molecular , un haplogrupo es un grupo de haplotipos similares que comparten un ancestro común con una mutación de polimorfismo de un solo nucleótido (SNP). El estudio de los haplogrupos proporciona información sobre los orígenes ancestrales que se remontan a miles de años. [46]
Los haplogrupos humanos que se estudian con más frecuencia son los haplogrupos del cromosoma Y (ADN-Y) y los haplogrupos del ADN mitocondrial (ADNmt) , ambos de los cuales se pueden utilizar para definir poblaciones genéticas. El ADN-Y se transmite únicamente a lo largo de la línea patrilineal , de padre a hijo, mientras que el ADNmt se transmite a lo largo de la línea matrilineal , de madre a hija o hijo. El ADN-Y y el ADNmt pueden cambiar por mutación aleatoria en cada generación.
Una repetición en tándem de número variable (VNTR) es la variación de longitud de una repetición en tándem . Una repetición en tándem es la repetición adyacente de una secuencia corta de nucleótidos . Las repeticiones en tándem existen en muchos cromosomas y su longitud varía entre individuos. Cada variante actúa como un alelo heredado , por lo que se utilizan para la identificación personal o parental. Su análisis es útil en la investigación genética y biológica, la ciencia forense y la huella genética .
Las repeticiones en tándem cortas (de unos 5 pares de bases) se denominan microsatélites , mientras que las más largas se denominan minisatélites .


El paradigma del origen africano reciente de los humanos modernos presupone la dispersión de poblaciones no africanas de humanos anatómicamente modernos después de hace 70.000 años. La dispersión dentro de África ocurrió significativamente antes, al menos hace 130.000 años. La teoría del "origen de África" se origina en el siglo XIX, como una sugerencia tentativa en El origen del hombre de Charles Darwin , [47] pero siguió siendo especulativa hasta la década de 1980, cuando fue apoyada por el estudio del ADN mitocondrial actual, combinado con evidencia de la antropología física de especímenes arcaicos .
Según un estudio de 2000 sobre la variación de la secuencia del cromosoma Y, [48] los cromosomas Y humanos tienen su origen en África, y los descendientes del linaje derivado abandonaron África y finalmente fueron reemplazados por cromosomas Y humanos arcaicos en Eurasia. El estudio también muestra que una minoría de las poblaciones contemporáneas en África Oriental y los Khoisan son descendientes de los linajes paternos más ancestrales de humanos anatómicamente modernos que abandonaron África hace entre 35.000 y 89.000 años. [48] Otra evidencia que apoya la teoría es que las variaciones en las medidas del cráneo disminuyen con la distancia desde África al mismo ritmo que disminuye la diversidad genética. La diversidad genética humana disminuye en las poblaciones nativas con la distancia migratoria desde África, y se cree que esto se debe a los cuellos de botella durante la migración humana, que son eventos que reducen temporalmente el tamaño de la población. [49] [50]
Un estudio de agrupamiento genético de 2009, que genotipó 1327 marcadores polimórficos en varias poblaciones africanas, identificó seis grupos ancestrales. El agrupamiento se correspondía estrechamente con la etnicidad, la cultura y el idioma. [51] Un estudio de secuenciación del genoma completo de 2018 de las poblaciones del mundo observó agrupaciones similares entre las poblaciones de África. En K=9, los componentes ancestrales distintos definieron las poblaciones de habla afroasiática que habitan el norte de África y el noreste de África ; las poblaciones de habla nilo-sahariana en el noreste de África y el este de África ; las poblaciones ari en el noreste de África; las poblaciones de habla niger-congo en África centro-occidental, África occidental , África oriental y África meridional ; las poblaciones pigmeas en África central ; y las poblaciones khoisan en África meridional. [52]
En mayo de 2023, los científicos informaron, basándose en estudios genéticos, de una vía de evolución humana más compleja de lo que se creía hasta ahora. Según los estudios, los humanos evolucionaron a partir de diferentes lugares y épocas de África, en lugar de hacerlo a partir de un único lugar y período de tiempo. [53] [54]
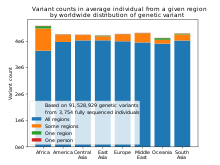
Debido a la ascendencia común de todos los seres humanos, solo una pequeña cantidad de variantes tienen grandes diferencias de frecuencia entre poblaciones. Sin embargo, algunas variantes raras en la población humana mundial son mucho más frecuentes en al menos una población (más del 5%). [55]

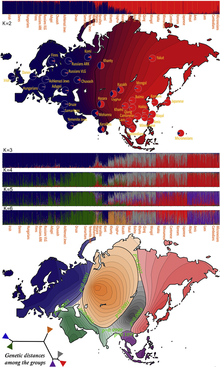
Se suele suponer que los primeros humanos abandonaron África y, por lo tanto, debieron atravesar un cuello de botella poblacional antes de su divergencia entre África y Eurasia hace unos 100.000 años (aproximadamente 3.000 generaciones). La rápida expansión de una población que antes era pequeña tiene dos efectos importantes en la distribución de la variación genética. En primer lugar, el llamado efecto fundador se produce cuando las poblaciones fundadoras aportan solo un subconjunto de la variación genética de su población ancestral. En segundo lugar, a medida que los fundadores se separan geográficamente más, la probabilidad de que dos individuos de diferentes poblaciones fundadoras se apareen se hace menor. El efecto de este apareamiento selectivo es reducir el flujo genético entre grupos geográficos y aumentar la distancia genética entre grupos. [ cita requerida ]
La expansión de los humanos desde África afectó la distribución de la variación genética de otras dos maneras. En primer lugar, las poblaciones más pequeñas (las fundadoras) experimentan una mayor deriva genética debido al aumento de las fluctuaciones en los polimorfismos neutros. En segundo lugar, los nuevos polimorfismos que surgían en un grupo tenían menos probabilidades de transmitirse a otros grupos, ya que el flujo genético estaba restringido. [ cita requerida ]
Las poblaciones de África tienden a tener menores niveles de desequilibrio de ligamiento que las poblaciones fuera de África, en parte debido al mayor tamaño de las poblaciones humanas en África a lo largo de la historia humana y en parte porque el número de humanos modernos que abandonaron África para colonizar el resto del mundo parece haber sido relativamente bajo. [57] En contraste, las poblaciones que han experimentado reducciones de tamaño dramáticas o expansiones rápidas en el pasado y las poblaciones formadas por la mezcla de grupos ancestrales previamente separados pueden tener niveles inusualmente altos de desequilibrio de ligamiento [57]

La distribución de variantes genéticas dentro y entre poblaciones humanas es imposible de describir sucintamente debido a la dificultad de definir una "población", la naturaleza clinal de la variación y la heterogeneidad a lo largo del genoma (Long y Kittles 2003). Sin embargo, en general, un promedio del 85% de la variación genética existe dentro de las poblaciones locales, ~7% entre poblaciones locales dentro del mismo continente y ~8% de la variación ocurre entre grandes grupos que viven en diferentes continentes. [58] [59] La reciente teoría del origen africano de los humanos predeciría que en África existe mucha más diversidad que en otros lugares y que la diversidad debería disminuir cuanto más lejos de África se muestrea una población.
El África subsahariana tiene la mayor diversidad genética humana y se ha demostrado que lo mismo es cierto para la variación fenotípica en la forma del cráneo. [49] [60] El fenotipo está conectado al genotipo a través de la expresión genética . La diversidad genética disminuye suavemente con la distancia migratoria desde esa región, que muchos científicos creen que es el origen de los humanos modernos, y esa disminución se refleja en una disminución de la variación fenotípica. Las medidas del cráneo son un ejemplo de un atributo físico cuya variación dentro de la población disminuye con la distancia desde África.
La distribución de muchos rasgos físicos se asemeja a la distribución de la variación genética dentro y entre poblaciones humanas ( American Association of Physical Anthropologists 1996; Keita y Kittles 1997). Por ejemplo, aproximadamente el 90% de la variación en las formas de la cabeza humana ocurre dentro de grupos continentales y aproximadamente el 10% entre grupos, con una mayor variabilidad en la forma de la cabeza entre individuos con ancestros africanos recientes (Relethford 2002).
Una excepción destacada a la distribución común de las características físicas dentro y entre los grupos es el color de la piel . Aproximadamente el 10% de la variación en el color de la piel ocurre dentro de los grupos, y ~90% ocurre entre grupos (Relethford 2002). Esta distribución del color de la piel y su patrón geográfico (las personas cuyos antepasados vivieron predominantemente cerca del ecuador tienen la piel más oscura que aquellos cuyos antepasados vivieron predominantemente en latitudes más altas) indican que este atributo ha estado bajo una fuerte presión selectiva . La piel más oscura parece ser fuertemente seleccionada en las regiones ecuatoriales para prevenir las quemaduras solares, el cáncer de piel, la fotólisis del folato y el daño a las glándulas sudoríparas. [61]
La comprensión de cómo la diversidad genética en la población humana afecta a los distintos niveles de expresión génica es un área activa de investigación. Si bien los estudios anteriores se centraron en la relación entre la variación del ADN y la expresión del ARN, los esfuerzos más recientes están caracterizando el control genético de varios aspectos de la expresión génica, incluidos los estados de la cromatina, [62] la traducción, [63] y los niveles de proteína. [64] Un estudio publicado en 2007 encontró que el 25% de los genes mostraban diferentes niveles de expresión génica entre las poblaciones de ascendencia europea y asiática. [65] [66] [67] [68] [69] Se pensaba que la causa principal de esta diferencia en la expresión génica eran los SNP en las regiones reguladoras de genes del ADN. Otro estudio publicado en 2007 encontró que aproximadamente el 83% de los genes se expresaban en diferentes niveles entre individuos y alrededor del 17% entre las poblaciones de ascendencia europea y africana. [70] [71]
El genetista de poblaciones Sewall Wright desarrolló el índice de fijación (a menudo abreviado como F ST ) como una forma de medir las diferencias genéticas entre poblaciones. Esta estadística se utiliza a menudo en taxonomía para comparar las diferencias entre dos poblaciones dadas midiendo las diferencias genéticas entre poblaciones para genes individuales, o para muchos genes simultáneamente. [72] A menudo se afirma que el índice de fijación para los humanos es de aproximadamente 0,15. Esto se traduce en un estimado del 85% de la variación medida en la población humana general se encuentra dentro de los individuos de la misma población, y aproximadamente el 15% de la variación ocurre entre poblaciones. Estas estimaciones implican que dos individuos de diferentes poblaciones pueden ser más similares entre sí que cualquiera de ellos con un miembro de su propio grupo. [73] [74] "La historia evolutiva compartida de los humanos vivos ha dado como resultado un alto parentesco entre todas las personas vivas, como lo indica, por ejemplo, el índice de fijación muy bajo (F ST ) entre las poblaciones humanas vivas". Richard Lewontin , quien afirmó estas proporciones, concluyó que ni la "raza" ni las "subespecies" eran formas apropiadas o útiles para describir las poblaciones humanas. [58]
El propio Wright creía que los valores >0,25 representan una variación genética muy grande y que un F ST de 0,15-0,25 representaba una gran variación. Sin embargo, alrededor del 5% de la variación humana se produce entre poblaciones dentro de los continentes, por lo que en algunos estudios se han encontrado valores de F ST entre grupos continentales de humanos (o razas) de tan solo 0,1 (o posiblemente menores), lo que sugiere niveles más moderados de variación genética. [72] Graves (1996) ha rebatido que el F ST no debería utilizarse como marcador del estado de subespecie, ya que la estadística se utiliza para medir el grado de diferenciación entre poblaciones, [72] aunque véase también Wright (1978). [75]
Jeffrey Long y Rick Kittles ofrecen una extensa crítica de la aplicación de la F ST a las poblaciones humanas en su artículo de 2003 "La diversidad genética humana y la inexistencia de razas biológicas". Encuentran que la cifra del 85% es engañosa porque implica que todas las poblaciones humanas contienen en promedio el 85% de toda la diversidad genética. Argumentan que el modelo estadístico subyacente supone incorrectamente historias de variación iguales e independientes para cada gran población humana. Un enfoque más realista es entender que algunos grupos humanos son parentales de otros grupos y que estos grupos representan grupos parafiléticos para sus grupos de descendencia. Por ejemplo, según la reciente teoría del origen africano, la población humana en África es parafilética para todos los demás grupos humanos porque representa el grupo ancestral del que derivan todas las poblaciones no africanas, pero más que eso, los grupos no africanos solo derivan de una pequeña muestra no representativa de esta población africana. Esto significa que todos los grupos no africanos están más estrechamente relacionados entre sí y con algunos grupos africanos (probablemente los africanos orientales) que con otros, y además que la migración fuera de África representó un cuello de botella genético , ya que gran parte de la diversidad que existía en África no fue trasladada fuera de África por los grupos emigrantes. Bajo este escenario, las poblaciones humanas no tienen cantidades iguales de variabilidad local, sino más bien cantidades disminuidas de diversidad cuanto más lejos de África vive cualquier población. Long y Kittles encuentran que, en lugar de que el 85% de la diversidad genética humana exista en todas las poblaciones humanas, aproximadamente el 100% de la diversidad humana existe en una sola población africana, mientras que solo alrededor del 70% de la diversidad genética humana existe en una población derivada de Nueva Guinea. Long y Kittles argumentaron que esto todavía produce una población humana global que es genéticamente homogénea en comparación con otras poblaciones de mamíferos. [76]
Los humanos anatómicamente modernos se cruzaron con los neandertales durante el Paleolítico Medio . En mayo de 2010, el Proyecto Genoma Neandertal presentó evidencia genética de que se produjo mestizaje y de que una pequeña pero significativa porción, alrededor del 2-4%, de mezcla neandertal está presente en el ADN de los euroasiáticos y oceánicos modernos, y casi ausente en las poblaciones del África subsahariana. [77] [78]
Entre el 4% y el 6% del genoma de los melanesios (representados por los habitantes de Papúa Nueva Guinea y de las islas de Bougainville) parece derivar de los denisovanos , un homínido hasta entonces desconocido que está más estrechamente relacionado con los neandertales que con los sapiens. Es posible que se introdujera durante la migración temprana de los ancestros de los melanesios al sudeste asiático. Esta historia de interacción sugiere que los denisovanos alguna vez se extendieron ampliamente por el este de Asia. [79]
De este modo, los melanesios surgen como una de las poblaciones con mayor mezcla arcaica, con una proporción de denisovanos y neandertales relacionada de aproximadamente el 8 %. [79]
En un estudio publicado en 2013, Jeffrey Wall de la Universidad de California estudió datos completos del genoma y encontró tasas más altas de introgresión en asiáticos en comparación con los europeos. [80] Hammer et al. probaron la hipótesis de que los genomas africanos contemporáneos tienen firmas de flujo genético con ancestros humanos arcaicos y encontraron evidencia de mezcla arcaica en los genomas de algunos grupos africanos, lo que sugiere que cantidades modestas de flujo genético se extendieron a lo largo del tiempo y el espacio durante la evolución de los humanos anatómicamente modernos. [81]
Un estudio publicado en 2020 encontró que las poblaciones yoruba y mende de África occidental derivan entre el 2% y el 19% de su genoma de una población de homínidos arcaicos aún no identificada que probablemente divergió antes de la división de los humanos modernos y los ancestros de los neandertales y los denisovanos, [82] lo que potencialmente convierte a estos grupos en las poblaciones humanas más arcaicas mezcladas identificadas hasta ahora.

Los nuevos datos sobre la variación genética humana han reavivado el debate sobre una posible base biológica para la categorización de los seres humanos en razas. La mayor parte de la controversia gira en torno a la cuestión de cómo interpretar los datos genéticos y si las conclusiones basadas en ellos son sólidas. Algunos investigadores sostienen que la raza con la que uno se identifica puede utilizarse como indicador de ascendencia geográfica para determinados riesgos de salud y medicamentos .
Aunque las diferencias genéticas entre los grupos humanos son relativamente pequeñas, estas diferencias en ciertos genes como duffy , ABCC11 , SLC24A5 , llamados marcadores informativos de ascendencia (AIM), pueden usarse para ubicar de manera confiable a muchos individuos dentro de agrupaciones amplias basadas en la geografía. Por ejemplo, los análisis informáticos de cientos de loci polimórficos muestreados en poblaciones distribuidas globalmente han revelado la existencia de agrupaciones genéticas que están aproximadamente asociadas con grupos que históricamente han ocupado grandes regiones continentales y subcontinentales (Rosenberg et al. 2002; Bamshad et al. 2003).
Algunos comentaristas han argumentado que estos patrones de variación proporcionan una justificación biológica para el uso de categorías raciales tradicionales. Argumentan que las agrupaciones continentales corresponden aproximadamente con la división de los seres humanos en africanos subsaharianos ; europeos , asiáticos occidentales , asiáticos centrales , asiáticos meridionales y africanos del norte ; asiáticos orientales , asiáticos sudorientales , polinesios y nativos americanos ; y otros habitantes de Oceanía (melanesios, micronesios y aborígenes australianos) (Risch et al. 2002). Otros observadores no están de acuerdo, diciendo que los mismos datos socavan las nociones tradicionales de grupos raciales (King y Motulsky 2002; Calafell 2003; Tishkoff y Kidd 2004 [24] ). Señalan, por ejemplo, que las principales poblaciones consideradas razas o subgrupos dentro de las razas no necesariamente forman sus propias agrupaciones.
Las categorías raciales también se ven socavadas por los hallazgos de que las variantes genéticas que se limitan a una región tienden a ser raras dentro de esa región, las variantes que son comunes dentro de una región tienden a ser compartidas en todo el mundo y la mayoría de las diferencias entre individuos, ya sea que provengan de la misma región o de diferentes regiones, se deben a variantes globales. [85] No se han encontrado variantes genéticas que estén fijadas dentro de un continente o una región importante y no se encuentren en ningún otro lugar. [86]
Además, como la variación genética humana es clinal, muchos individuos se afilian a dos o más grupos continentales. Por lo tanto, la "ascendencia biogeográfica" de base genética asignada a una persona determinada generalmente estará ampliamente distribuida y estará acompañada de incertidumbres considerables (Pfaff et al. 2004).
En muchas partes del mundo, los grupos se han mezclado de tal manera que muchos individuos tienen ancestros relativamente recientes provenientes de regiones muy distantes. Aunque los análisis genéticos de un gran número de loci pueden producir estimaciones del porcentaje de ancestros de una persona que provienen de varias poblaciones continentales (Shriver et al. 2003; Bamshad et al. 2004), estas estimaciones pueden suponer una falsa distinción de las poblaciones parentales, ya que los grupos humanos han intercambiado parejas desde escalas locales a escalas continentales a lo largo de la historia (Cavalli-Sforza et al. 1994; Hoerder 2002). Incluso con un gran número de marcadores, la información para estimar las proporciones de mezcla de individuos o grupos es limitada, y las estimaciones normalmente tendrán intervalos de confianza amplios (Pfaff et al. 2004).
Los datos genéticos pueden utilizarse para inferir la estructura de una población y asignar individuos a grupos que a menudo se corresponden con su ascendencia geográfica autoidentificada. Jorde y Wooding (2004) afirmaron que "el análisis de muchos loci ahora produce estimaciones razonablemente precisas de similitud genética entre individuos, en lugar de poblaciones. La agrupación de individuos está correlacionada con el origen geográfico o la ascendencia". [23] Sin embargo, la identificación por origen geográfico puede fallar rápidamente cuando se considera la ascendencia histórica compartida entre individuos en el pasado. [87]
En 2009 se publicó un análisis de datos de SNP autosómicos del Proyecto Internacional HapMap (Fase II) y muestras del Panel de Diversidad del Genoma Humano del CEPH. El estudio de 53 poblaciones tomadas de los datos de HapMap y CEPH (1138 individuos no relacionados) sugirió que la selección natural puede dar forma al genoma humano mucho más lentamente de lo que se pensaba anteriormente, con factores como la migración dentro y entre continentes que influyen más fuertemente en la distribución de variaciones genéticas. [88] Un estudio similar publicado en 2010 encontró evidencia sólida en todo el genoma de selección debido a cambios en la ecorregión, la dieta y la subsistencia, particularmente en relación con las ecorregiones polares, con la búsqueda de alimento y con una dieta rica en raíces y tubérculos. [89] En un estudio de 2016, el análisis de componentes principales de datos de todo el genoma fue capaz de recuperar objetivos previamente conocidos para la selección positiva (sin una definición previa de poblaciones), así como una serie de nuevos genes candidatos. [90]
Los antropólogos forenses pueden evaluar la ascendencia de los restos óseos mediante el análisis de la morfología esquelética, así como utilizando marcadores genéticos y químicos, cuando sea posible. [91] Si bien estas evaluaciones nunca son seguras, se ha estimado que la precisión de los análisis de morfología esquelética para determinar la verdadera ascendencia es del 90%. [92]

El flujo genético entre dos poblaciones reduce la distancia genética media entre ellas; sólo las poblaciones humanas totalmente aisladas no experimentan flujo genético y la mayoría de las poblaciones tienen un flujo genético continuo con otras poblaciones vecinas, lo que crea la distribución clinal observada para la mayor parte de la variación genética. Cuando el flujo genético tiene lugar entre poblaciones genéticamente bien diferenciadas, el resultado se denomina "mezcla genética".
El mapeo de mezclas es una técnica utilizada para estudiar cómo las variantes genéticas causan diferencias en las tasas de enfermedades entre poblaciones. [93] Las poblaciones de mezclas recientes que rastrean su ascendencia a múltiples continentes son muy adecuadas para identificar genes para rasgos y enfermedades que difieren en prevalencia entre poblaciones parentales. Las poblaciones afroamericanas han sido el foco de numerosos estudios de mapeo genético y de mezclas poblacionales, incluidos estudios de rasgos genéticos complejos como el recuento de glóbulos blancos, el índice de masa corporal, el cáncer de próstata y la enfermedad renal. [94]
Se realizó un análisis de la variación fenotípica y genética, incluyendo el color de la piel y el estatus socioeconómico, en la población de Cabo Verde, que tiene una historia bien documentada de contacto entre europeos y africanos. Los estudios mostraron que el patrón de mezcla en esta población ha estado sesgado por el sexo (que implica principalmente apareamientos entre hombres europeos y mujeres africanas) y existe una interacción significativa entre el estatus socioeconómico y el color de la piel, independientemente de la ascendencia. [95] Otro estudio muestra un mayor riesgo de complicaciones de la enfermedad de injerto contra huésped después del trasplante debido a variantes genéticas en el antígeno leucocitario humano (HLA) y proteínas no HLA. [96]
Las diferencias en las frecuencias de los alelos contribuyen a las diferencias de grupo en la incidencia de algunas enfermedades monogénicas , y pueden contribuir a las diferencias en la incidencia de algunas enfermedades comunes. [97] Para las enfermedades monogénicas, la frecuencia de los alelos causales generalmente se correlaciona mejor con la ascendencia, ya sea familiar (por ejemplo, el síndrome de Ellis-Van Creveld entre los Amish de Pensilvania ), étnica ( enfermedad de Tay-Sachs entre las poblaciones judías asquenazíes ) o geográfica (hemoglobinopatías entre personas con antepasados que vivieron en regiones palúdicas). En la medida en que la ascendencia se corresponda con grupos o subgrupos raciales o étnicos, la incidencia de enfermedades monogénicas puede diferir entre grupos categorizados por raza o etnia, y los profesionales de la salud generalmente tienen en cuenta estos patrones al realizar diagnósticos. [98]
Incluso en el caso de enfermedades comunes en las que intervienen numerosas variantes genéticas y factores ambientales, los investigadores señalan pruebas que sugieren la participación de alelos distribuidos de forma diferencial con efectos pequeños a moderados. Entre los ejemplos citados con frecuencia se incluyen la hipertensión (Douglas et al. 1996), la diabetes (Gower et al. 2003), la obesidad (Fernandez et al. 2003) y el cáncer de próstata (Platz et al. 2000). Sin embargo, en ninguno de estos casos se ha demostrado que la variación alélica en un gen de susceptibilidad explique una fracción significativa de la diferencia en la prevalencia de la enfermedad entre grupos, y el papel de los factores genéticos en la generación de estas diferencias sigue siendo incierto (Mountain y Risch 2004).
Por otra parte, otras variaciones son beneficiosas para el ser humano, ya que previenen ciertas enfermedades y aumentan la posibilidad de adaptarse al medio ambiente. Por ejemplo, la mutación en el gen CCR5 que protege contra el SIDA . El gen CCR5 está ausente en la superficie de la célula debido a la mutación. Sin el gen CCR5 en la superficie, no hay nada a lo que los virus del VIH puedan agarrarse y unirse. Por lo tanto, la mutación en el gen CCR5 disminuye la posibilidad de que un individuo tenga riesgo de contraer SIDA. La mutación en CCR5 también es bastante común en ciertas áreas, con más del 14% de la población portadora de la mutación en Europa y alrededor del 6-10% en Asia y el norte de África . [99]

Aparte de las mutaciones, muchos genes que pueden haber ayudado a los humanos en la antigüedad afectan a los humanos de hoy. Por ejemplo, se sospecha que los genes que permiten a los humanos procesar los alimentos de manera más eficiente son los que hacen que las personas sean susceptibles a la obesidad y la diabetes en la actualidad. [100]
Neil Risch, de la Universidad de Stanford, ha propuesto que la autoidentificación racial/étnica podría ser un medio válido de categorización en los EE. UU. para consideraciones de salud pública y políticas. [101] [97] Un artículo de 2002 del grupo de Noah Rosenberg hace una afirmación similar: "La estructura de las poblaciones humanas es relevante en varios contextos epidemiológicos. Como resultado de la variación en las frecuencias de los factores de riesgo genéticos y no genéticos, las tasas de enfermedad y de fenotipos como la respuesta adversa a los medicamentos varían entre las poblaciones. Además, la información sobre la población de origen de un paciente podría proporcionar a los profesionales de la salud información sobre el riesgo cuando se desconocen las causas directas de la enfermedad". [102] Sin embargo, en 2018 Noah Rosenberg publicó un estudio que argumentaba en contra de las ideas genéticamente esencialistas de las disparidades de salud entre las poblaciones, afirmando que las variantes ambientales son una causa más probable [103]
Los proyectos del genoma humano son iniciativas científicas que determinan o estudian la estructura del genoma humano . El Proyecto Genoma Humano fue un proyecto histórico sobre el genoma.
{{cite book}}: Mantenimiento de CS1: otros ( enlace ){{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace ){{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace )Según estos criterios, se encontró que el 1,6% de los SNP de Perlegen exhibían la arquitectura genética de la selección.
En cada gran región del mundo, los mamíferos vivos están estrechamente relacionados con las especies extintas de la misma región. Por lo tanto, es probable que África estuviera habitada anteriormente por simios extintos estrechamente relacionados con el gorila y el chimpancé; y como estas dos especies son ahora los aliados más cercanos del hombre, es algo más probable que nuestros primeros progenitores vivieran en el continente africano que en cualquier otro lugar. Pero es inútil especular sobre este tema, ya que un simio casi tan grande como un hombre, a saber, el Dryopithecus de Lartet, que estaba estrechamente relacionado con el antropomorfo Hylobates, existió en Europa durante el período del Mioceno superior; y desde un período tan remoto, la Tierra ciertamente ha experimentado muchas grandes revoluciones, y ha habido tiempo suficiente para la migración en la mayor escala.
Incorporamos datos geográficos en un análisis de agrupamiento bayesiano, asumiendo que no hay mezcla (software TESS) (25) y distinguimos seis grupos dentro de África continental (Fig. 5A). El grupo geográficamente más extendido (naranja) se extiende desde el extremo occidental de África (los mandinga) a través de África central hasta los hablantes bantú de Sudáfrica (los venda y los xhosa) y corresponde a la distribución de la familia de lenguas nigerino-kordofanianas, posiblemente reflejando la propagación de las poblaciones de habla bantú desde cerca de las tierras altas de Nigeria/Camerún a través del este y sur de África en los últimos 5000 a 3000 años (26,27). Otro grupo inferido incluye las poblaciones pigmeas y SAK (verde), con una distribución geográfica no contigua en África central y sudoriental, consistente con la ESTRUCTURA (Fig. 3) y los análisis filogenéticos (Fig. 1). Otro grupo geográficamente contiguo se extiende a través del norte de África (azul) hasta Malí (los dogones), Etiopía y el norte de Kenia. Con la excepción de los dogones, estas poblaciones hablan una lengua afroasiática. Las poblaciones de habla chádica y nilo-sahariana de Nigeria, Camerún y Chad central, así como varias poblaciones de habla nilo-sahariana del sur de Sudán, constituyen otro grupo (rojo). Los hablantes nilo-saharianos y cusíticos de Sudán, Kenia y Tanzania, así como algunos de los hablantes bantúes de Kenia, Tanzania y Ruanda (hutu/tutsi), constituyen otro grupo (púrpura), lo que refleja evidencia lingüística de flujo genético entre estas poblaciones durante los últimos ~5000 años (28,29). Finalmente, los hadza son los únicos constituyentes de un sexto grupo (amarillo), en consonancia con su estructura genética distintiva identificada por PCA y STRUCTURE.
{{cite book}}: |journal=ignorado ( ayuda )