La historia genética de Italia incluye información sobre la formación, etnogénesis y otra información específica del ADN sobre los habitantes de Italia. Los italianos modernos descienden en su mayoría de los pueblos antiguos de Italia , incluidos los hablantes indoeuropeos ( romanos y otros latinos , faliscos , picentos , umbros , samnitas , oscos , sículos y vénetos adriáticos , así como magnogriegos , galos cisalpinos y yapigios ilirios ) y hablantes preindoeuropeos ( etruscos , ligures , réticos, camunios , sicanos , pueblos nurágicos , así como colonos de Fenicia y Cartago ). Otros grupos migraron a Italia como resultado del Imperio Romano, cuando la península itálica atrajo a personas de las diversas regiones del imperio (África del Norte, Asia Occidental y el resto de Europa), [2] y durante la Edad Media con la llegada de ostrogodos , longobardos , sarracenos y normandos , entre otros. Con base en el análisis de ADN, existe evidencia de una subestructura genética regional y de continuidad dentro de la Italia moderna que se remonta a la antigüedad. [3] [4] [5] [6]
En sus proporciones de mezcla, los italianos son similares a otros europeos del sur, y es que tienen principalmente ascendencia de agricultores europeos tempranos del Neolítico , junto con cantidades más pequeñas, pero aún significativas, de cazadores-recolectores occidentales del Mesolítico , pastores esteparios de la Edad del Bronce ( hablantes indoeuropeos ) y ascendencia iraní/ relacionada con el Cáucaso del Calcolítico o la Edad del Bronce . [4] [7] [8] [9]
También hay mezcla de la Edad del Bronce/Hierro con Asia occidental y Oriente Medio en Italia, con una incidencia mucho menor en el norte de Italia en comparación con Italia central y el sur de Italia. [10] [8] La mezcla del norte de África también se encuentra en el sur de Italia y las islas principales, con la mayor incidencia en Sicilia y Cerdeña . [10] [8] [4]
En general, se encontró que las muestras latinas del Lacio en la Edad del Hierro y el período republicano romano temprano se agrupaban genéticamente más cerca de los italianos del norte y centro modernos (cuatro de seis eran más cercanas a los italianos del norte y centro, mientras que las otras dos eran más cercanas a los italianos del sur). [11] El análisis de ADN demuestra que la colonización griega antigua tuvo un efecto duradero significativo en el paisaje genético local del sur de Italia y Sicilia ( Magna Grecia ), y que la gente moderna de esa región tenía una mezcla griega significativa. [12] [13] En general, se encontró que la diferenciación genética entre los latinos, los etruscos y la población protovillanovana precedente de Italia era insignificante. [14] En 2019, el análisis de ADNa de fósiles romanos detectó un cambio sustancial en la ascendencia genética hacia la ascendencia de Europa central y septentrional en los habitantes de la ciudad de Roma en la antigüedad tardía y la era medieval. Los autores vinculan tentativamente el origen de esta ascendencia con los visigodos y los lombardos . Anteriormente, la mayoría de los ciudadanos de la era imperial se agrupaban con pueblos del centro y este del Mediterráneo, como los italianos del centro y sur, los griegos, los chipriotas y los malteses, y en cierta medida, los pueblos del Levante y del Cercano Oriente. Esto fue causado por la inmigración directa y el contacto con las diásporas griegas, fenicias y púnicas. [2] [15] Un análisis de 2020 de los haplogrupos maternos de muestras antiguas y modernas indica una similitud genética sustancial y una continuidad entre los habitantes modernos de Umbría en el centro de Italia y los antiguos habitantes de la región pertenecientes a la cultura umbría de habla itálica . [6]
Múltiples estudios de ADN confirmaron que la variación genética en Italia es clinal , y va desde el Mediterráneo oriental hasta el occidental. Los sardos son la excepción como valores genéticos atípicos en Italia y, de hecho, en Europa, como resultado de su ascendencia predominantemente neolítica , preindoeuropea y nurágica no itálica . [16] [7] [8] [9] Como reflejo de la historia de Europa y de la cuenca mediterránea más amplia, se ha descubierto que las poblaciones italianas están formadas principalmente por los mismos componentes ancestrales, aunque en diferentes proporciones, de los asentamientos mesolíticos , neolíticos y de la Edad del Bronce de Europa. [10] [17] [18]
La brecha genética entre los italianos del norte y del sur está llena por un grupo intermedio de italianos centrales , creando una clina continua de variación que refleja la geografía. [19] Las únicas excepciones son algunas poblaciones minoritarias (principalmente minorías eslovenas de la región de Friuli-Venecia Julia ) que se agrupan con los centroeuropeos de habla eslava en Eslovenia , [20] así como los sardos, que se diferencian claramente de las poblaciones de Italia continental y Sicilia . [17] [4] Un estudio sobre algunas comunidades lingüísticas y aisladas que residen en Italia reveló que su diversidad genética a distancias cortas (0-200 km) e intermedias (700-800 km) era mayor que la observada en todo el continente europeo. [5]
La distancia genética entre los italianos del norte y del sur, aunque grande para una sola nacionalidad europea, es similar a la que existe entre los alemanes del norte y del sur . [21] Los italianos del norte y del sur comenzaron a divergir ya en la Edad del Hielo Tardío , y parecen encapsular a menor escala la clina de diversidad genética observable en toda Europa. [22]

Los humanos modernos aparecieron durante el Paleolítico superior . En la cueva de Fumane se descubrieron ejemplares de la época del Auriñaciense que datan de hace unos 34.000 años. Durante el período Magdaleniense , los primeros humanos procedentes de los Pirineos poblaron Cerdeña . [23]
Durante el Neolítico , la agricultura fue introducida por los pueblos procedentes del este y se construyeron los primeros poblados. Las armas se volvieron más sofisticadas y se produjeron los primeros objetos de arcilla. A finales del Neolítico se extendió el uso del cobre y se construyeron poblados sobre pilotes cerca de lagos. En Cerdeña, Sicilia y una parte de la Italia continental se extendió la cultura campaniforme procedente de Europa occidental y central. Sicilia también sufrió las influencias del Egeo en el período micénico.
Durante la Edad del Bronce Final, en el centro y norte de Italia apareció la cultura protovillano-villano de los campos de urnas , caracterizada por el rito de la cremación de los cadáveres, originario de Europa Central. Comenzó a extenderse el uso del hierro. [24] En Cerdeña floreció la civilización nurágica .
En los albores de la Edad del Hierro, gran parte de Italia estaba habitada por tribus itálicas como los latinos , sabinos , samnitas y umbros . Los territorios del noroeste y los alpinos estaban poblados principalmente por hablantes preindoeuropeos como los etruscos , ligures , camunios y retios ; mientras que las tribus yapigias , posiblemente de origen ilirio , poblaban Apulia .
A partir del siglo VIII a. C., los colonos griegos se asentaron en la costa sur de Italia y fundaron ciudades, formando lo que más tarde se llamaría Magna Grecia . Casi al mismo tiempo, los colonos fenicios se asentaron en el lado occidental de Sicilia. Durante el mismo período, la civilización etrusca se desarrolló en la costa del sur de la Toscana y el norte del Lacio . En el siglo IV a. C., los galos se asentaron en el norte de Italia y en partes de Italia central . [25] Con la caída del Imperio romano de Occidente, diferentes poblaciones de origen germánico invadieron Italia, siendo las más significativas los lombardos , [26] seguidos cinco siglos después por los normandos en Sicilia .

Se han realizado numerosos estudios genéticos de los etruscos y los toscanos modernos en comparación con otras poblaciones, algunos de los cuales indican el origen local europeo de los etruscos y otros apoyan un origen en otro lugar. En general, las pruebas directas del ADN etrusco antiguo han apoyado un origen local profundo, mientras que las pruebas de muestras modernas como sustituto de los etruscos son más bien poco concluyentes e inconsistentes. [27] [28]
El estudio de ADNmt de gran tamaño de 2013 indica, basándose en el ADN heredado por vía materna de 30 muestras de huesos tomadas de tumbas que datan del siglo VIII al siglo I a. C. de Toscana y Lacio , que los etruscos eran una población nativa. [29] [30] El estudio extrajo y tipificó la región hipervariable del ADN mitocondrial de 14 individuos enterrados en dos necrópolis etruscas, analizándolos junto con el ADNmt etrusco analizado previamente, otro ADNmt europeo antiguo, muestras modernas y medievales de Toscana y 4.910 individuos modernos de la cuenca mediterránea . Las secuencias de ADN antiguas (30 etruscos, 27 toscanos medievales) y modernas (370 toscanos) fueron sometidas a varios millones de ejecuciones de simulación por computadora, lo que demuestra que los etruscos pueden considerarse ancestrales de los medievales y, especialmente en las subpoblaciones de Casentino y Volterra , de los toscanos modernos; En cambio, se demostró que las poblaciones modernas de Murlo y Florencia no continuaban con la población medieval. Al considerar más a fondo dos muestras de Anatolia (35 y 123 individuos), se estimó que los vínculos genéticos entre Toscana y Anatolia se remontan al menos a hace 5.000 años, y el "momento de separación más probable entre Toscana y Anatolia occidental se sitúa alrededor de hace 7.600 años", lo que sugiere firmemente que la cultura etrusca se desarrolló localmente, y no como consecuencia inmediata de la inmigración desde las costas del Mediterráneo oriental. Según el estudio, el ADNmt etrusco antiguo es el más cercano entre las poblaciones europeas modernas y no está particularmente cerca de las poblaciones de Anatolia u otras poblaciones del Mediterráneo oriental. Entre las poblaciones antiguas basadas en el ADNmt, se descubrió que los antiguos etruscos eran los más cercanos a los agricultores neolíticos LBK de Europa central . [29] [30]
Este resultado está en gran medida en línea con los resultados previos de ADNmt de 2004 (en un estudio más pequeño también basado en ADN antiguo), y contradictorio con los resultados de 2007 (basados en ADN moderno). El estudio de 2004 se basó en ADN mitocondrial (ADNmt) de 80 muestras de hueso, reducidas a 28 muestras de hueso en la fase de análisis, tomadas de tumbas que datan del siglo VII al siglo III a. C. de Veneto , Toscana , Lacio y Campania . [31] Este estudio encontró que el ADN antiguo extraído de los restos etruscos tenía algunas afinidades con las poblaciones europeas modernas, incluidos los alemanes , los ingleses de Cornualles y los toscanos en Italia. Además, las muestras etruscas posiblemente revelaron más herencia genética del Mediterráneo oriental y meridional de la que contienen las muestras italianas modernas. El estudio se vio empañado por las preocupaciones de que las secuencias de ADNmt de las muestras arqueológicas representaban ADN severamente dañado o contaminado; [32] sin embargo, la investigación posterior mostró que las muestras pasaron las pruebas más estrictas de degradación del ADN disponibles. [33]
Un estudio de ADNmt, publicado en 2018 en la revista American Journal of Physical Anthropology , comparó muestras antiguas y modernas de Toscana, de la Prehistoria , la época etrusca, la época romana , el Renacimiento y la actualidad, y concluyó que los etruscos aparecen como una población local, intermedia entre las muestras prehistóricas y las otras, situándose en la red temporal entre el Eneolítico y la época romana. [34]
Un estudio genético de 2019 publicado en la revista Science analizó los restos de once individuos de la Edad del Hierro de las áreas alrededor de Roma, de los cuales cuatro eran individuos etruscos, uno enterrado en Veio Grotta Gramiccia del período Villanovano (900-800 a. C.) y tres enterrados en la Necrópolis de La Mattonara cerca de Civitavecchia del período orientalizante (700-600 a. C.). El estudio concluyó que los etruscos (900-600 a. C.) y los latinos (900-500 a. C.) del Lacio Vetus eran genéticamente similares., [35] Se encontró que las diferencias genéticas entre los etruscos y los latinos examinados eran insignificantes. [36] Los individuos etruscos y los latinos contemporáneos se distinguían de las poblaciones precedentes de Italia por la presencia de un 30,7% de ascendencia esteparia . [37] Su ADN era una mezcla de dos tercios de ascendencia de la Edad del Cobre ( EEF + WHG ; etruscos ~66–72%, latinos ~62–75%) y un tercio de ascendencia relacionada con la estepa (etruscos ~27–33%, latinos ~24–37%) (con el componente EEF principalmente derivado de migrantes de la era neolítica a Europa desde Anatolia y los WHG siendo cazadores-recolectores locales de Europa occidental, con ambos componentes, junto con el de la estepa, encontrándose en prácticamente todas las poblaciones europeas). [35] La única muestra de ADN-Y extraída pertenecía al haplogrupo J-M12 (J2b-L283) , encontrado en un individuo datado entre 700 y 600 a. C., y portaba exactamente el alelo derivado de M314 también encontrado en un individuo de la Edad del Bronce Medio de Croacia (1631-1531 calAEC). Mientras que las cuatro muestras de ADNmt extraídas pertenecían a los haplogrupos U5a1 , H , T2b32 , K1a4 . [38] Por lo tanto, los etruscos también tenían ascendencia relacionada con la estepa a pesar de hablar una lengua preindoeuropea.
Un estudio de 2021 del Instituto Max Planck , las Universidades de Tubinga , Florencia y Harvard , publicado en la revista Science Advances , analizó el cromosoma Y , el ADN mitocondrial y el ADN autosómico de 82 muestras antiguas de Etruria (Toscana y Lacio) y el sur de Italia (Basilicata) que abarcan desde el 800 a. C. hasta el 1000 d. C., incluidos 48 individuos de la Edad del Hierro. El estudio confirmó que en las muestras de individuos etruscos de Toscana y Lacio el componente ancestral estepario estaba presente en los mismos porcentajes encontrados en las muestras analizadas previamente de latinos de la Edad del Hierro, y añadió que en el ADN de los etruscos estaba completamente ausente, una señal de mezcla reciente con Anatolia o el Mediterráneo oriental. El estudio concluyó que los etruscos eran autóctonos y tenían un perfil genético similar al de sus vecinos latinos de la Edad del Hierro temprana. Tanto los etruscos como los latinos pertenecían firmemente al clúster europeo: el 75% de las muestras de individuos masculinos etruscos se encontró que pertenecían al haplogrupo R1b , especialmente R1b-P312 y su derivado R1b-L2 cuyo ancestro directo es R1b-U152. En cuanto a los haplogrupos de ADN mitocondrial, el más prevalente fue en gran medida el H, seguido del J y el T. Los datos de marcadores uniparentales y los datos de ADN autosómico de muestras de individuos etruscos de la Edad del Hierro sugieren que Etruria recibió migraciones con un gran componente ancestral de estepa durante el segundo milenio a. C. , relacionadas con la difusión de las lenguas indoeuropeas , comenzando con la cultura del vaso campaniforme , y que estas migraciones se fusionaron con poblaciones del estrato preindoeuropeo más antiguo presente desde al menos el Neolítico, pero fue la lengua de esta última la que sobrevivió, una situación similar a lo que sucedió en la región vasca del norte de España . El estudio concluyó también que las muestras analizadas muestran que los etruscos mantuvieron inalterado su perfil genético durante casi 1000 años, lo que indica la escasa presencia en Etruria de extranjeros, y que un cambio demográfico en Etruria se produjo solo a partir del período imperial romano , en el que se produce la mezcla en la población local de componentes ancestrales procedentes del mar Mediterráneo oriental. El análisis de muestras de individuos que vivieron en el período imperial romano y los de la Edad Media también sugieren que el paisaje genético de la actual Italia central se formó en gran parte hace unos 1000 años después de las invasiones bárbaras , y que la llegada de los lombardos germánicosEn Italia contribuyeron a la formación del acervo genético de la población moderna de Toscana y el norte del Lacio. [39]
En 2024, se estudiaron 6 individuos de restos etruscos de Tarquinia , Lacio , que datan del siglo IX al VII a. C., y confirmaron los hallazgos anteriores. El modelo de mezcla mostró que tenían un 84-92% de ascendencia campaniforme italiana y un 8-26% adicional de ascendencia yamna samara (relacionada con la estepa), pero con un individuo más similar a las poblaciones de la Edad del Hierro de Escandinavia y el noroeste de Europa. Los dos individuos masculinos estudiados para el cromosoma Y pertenecían al linaje J2b/J-M12, y los cinco haplogrupos mitocondriales estudiados eran típicos de la Europa posneolítica. Los rasgos fenotípicos mostraban ojos azules, cabello castaño claro/oscuro y tonos de piel pálidos a intermedios. [40]
En cambio, un estudio de ADNmt de 2007 ya había sugerido un origen en Oriente Próximo . [41] Achilli et al . (2007) encontraron en una muestra moderna de 86 individuos de Murlo, una pequeña ciudad del sur de la Toscana, una frecuencia inusualmente alta (17,5%) de supuestos haplogrupos de ADNmt de Oriente Próximo, mientras que otras poblaciones toscanas no muestran la misma característica sorprendente. Basándose en este resultado, Achilli concluyó que "sus datos apoyan el escenario de un aporte genético post-neolítico desde Oriente Próximo a la población actual de la Toscana, un escenario que concuerda con el origen lidio de los etruscos". Esta investigación ha sido muy criticada por arqueólogos, etruscólogos y clasicistas. [42] En ausencia de cualquier evidencia de datación, no hay un vínculo directo entre este aporte genético encontrado en Murlo y los etruscos. Además, no hay evidencia de que estos haplogrupos de ADNmt encontrados en Murlo puedan ser una prueba de un origen oriental de los etruscos, ya que algunos de estos haplogrupos de ADNmt se han encontrado en otros estudios ya en el Neolítico y el Eneolítico en Italia y Alemania. [34] Todos los haplogrupos de ADNmt encontrados en la muestra moderna de Murlo y clasificados por Achilli et al. como de origen del Cercano Oriente están en realidad muy extendidos en muestras modernas de otras áreas de Italia y Europa sin ningún vínculo con los etruscos. [43]
Un reciente estudio de Y-ADN de 2018 sobre una muestra moderna de 113 individuos de Volterra , una ciudad de origen etrusco, Grugni et al. mantiene abiertas todas las posibilidades, aunque el escenario autóctono es el más respaldado por los números, y concluye que "la presencia de J2a-M67* (2,7%) sugiere contactos por mar con pueblos anatolios, el hallazgo del linaje centroeuropeo G2a-L497 (7,1%) con una frecuencia considerable apoyaría más bien un origen noreuropeo de los etruscos, mientras que la alta incidencia de linajes europeos R1b (R1b 49,8%, R1b-U152 24,5%) no puede descartar el escenario de un proceso autóctono de formación de la civilización etrusca a partir de la sociedad villanoviana precedente, como lo sugirió Dionisio de Halicarnaso". [44] En Italia, el Y-ADN J2a-M67*, que aún no se ha encontrado en muestras etruscas, está más extendido en la costa del mar Adriático entre Marcas y Abruzos , y no en aquellos donde alguna vez vivieron los etruscos, y en el estudio tiene su pico en el lado jónico de Calabria . [45] [46] En 2014, se encontró que una muestra de la cultura Kyjatice de la Edad del Bronce tardía en Hungría era J2a1-M67, [47] se encontraron un par de J2a1b en muestras del Neolítico tardío de la cultura LBK en Austria , [48] se encontró un J2a1a en una muestra de la cultura Sopot del Neolítico medio de Croacia , [48] se encontró un J2a en una muestra de la cultura Lengyel del Neolítico tardío de Hungría . [49] En 2019, en un estudio de Stanford publicado en Science, se descubrió que dos muestras antiguas del asentamiento neolítico de Ripabianca di Monterado en la provincia de Ancona , en la región de Marche en Italia, eran Y-DNA J-L26 y J-M304. [35] En 2021, se descubrió que dos muestras más antiguas del asentamiento calcolítico de Grotta La Sassa, en la provincia de Latina en el sur de Lazio , eran Y-DNA J2a7-Z2397. [50] Por lo tanto, es probable que Y-DNA J2a-M67 esté en Italia desde el Neolítico y no puede ser la prueba de contactos recientes con Anatolia.
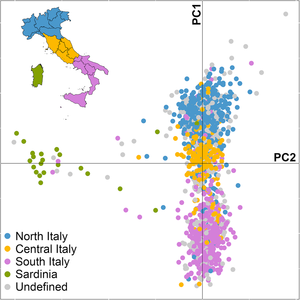
Estudios recientes sobre la estructura poblacional de los italianos actuales han demostrado que en Italia existe una clina norte-sur para los linajes del cromosoma Y y los loci autosómicos, con una clara diferenciación de los italianos peninsulares de los sardos, y que los toscanos modernos son la población de Italia central más cercana genéticamente a los habitantes del norte de Italia . [51] Un estudio de 2019, basado en el ADN autosómico de 1616 individuos de las 20 regiones administrativas italianas, concluye que los toscanos se unen al grupo del norte de Italia, cerca de los habitantes de Liguria y Emilia-Romaña . [52] Un estudio de 2013, basado en marcadores uniparentales de 884 individuos no relacionados de 23 localidades italianas, había demostrado que la estructura observada para los linajes paternos en Italia continental y Sicilia sugiere un trasfondo genético compartido entre personas de Toscana y el norte de Italia por un lado, y personas del sur de Italia y la costa adriática por el otro lado. Los haplogrupos de ADN-Y más frecuentes en el grupo representado por poblaciones del noroeste de Italia, incluida Toscana y la mayor parte de la llanura Padana, son cuatro linajes R1b (R-U152*, R-M269*, R-P312* y R-L2*). [46]
En el volumen colectivo Etruscology publicado en 2017, el arqueólogo británico Phil Perkins ofrece un análisis del estado de los estudios de ADN y escribe que "ninguno de los estudios de ADN realizados hasta la fecha prueba de manera concluyente que los etruscos fueran una población intrusiva en Italia que se originó en el Mediterráneo oriental o Anatolia" y "hay indicios de que la evidencia del ADN puede respaldar la teoría de que los etruscos son autóctonos en el centro de Italia". [28]
En su libro Breve historia de la humanidad publicado en 2021, el genetista alemán Johannes Krause , codirector del Instituto Max Planck de Antropología Evolutiva de Jena , concluye que es probable que la lengua etrusca (así como el vasco , el paleosardo y el minoico ) "se desarrollara en el continente en el curso de la Revolución Neolítica ". [53]
Muchos italianos , especialmente en el norte de Italia y en el centro de Italia, pertenecen al haplogrupo R1b , común en Europa occidental y central. La frecuencia más alta de R1b se encuentra en Garfagnana (76,2%) en Toscana y en los valles de Bérgamo (80,8%) en Lombardía . [54] [55] Este porcentaje disminuye en el sur de Italia en Calabria (33,2%). [54] Por otro lado, el 39% de los sardos pertenecen al haplogrupo europeo mesolítico I2a1a . [56] [57]
Un estudio de la Università Cattolica del Sacro Cuore concluyó que, si bien la colonización griega dejó una contribución genética poco significativa, el análisis de datos de 12 sitios de la península italiana respaldó un modelo de difusión démica masculina y una mezcla neolítica con habitantes mesolíticos . [58] Los resultados respaldaron una distribución de la variación genética a lo largo de un eje norte-sur y respaldaron la difusión démica. Las muestras del sur de Italia se agruparon con muestras del sudeste y centro-sur de Europa, y los grupos del norte con Europa occidental. [58] [59]
Un estudio de 2004 realizado por Semino et al. mostró que los italianos de las regiones del centro-norte tenían alrededor del 26,9% de J2; los apulios, calabreses y sicilianos tenían el 29,1%, el 21,5% y el 16,7% de J2 respectivamente; los sardos tenían el 9,7% de J2. [60]
Un estudio genético de 2018, centrado en los linajes del cromosoma Y y de los haplogrupos, su diversidad y su distribución tomando unos 817 sujetos representativos, da crédito a la tradicional división norte-sur de la población, al concluir que debido a las migraciones neolíticas, los italianos del sur "muestran una mayor similitud con las poblaciones de Oriente Medio y del sur de los Balcanes que las del norte; por el contrario, las muestras del norte son genéticamente más cercanas a los grupos del noroeste de Europa y del norte de los Balcanes". La posición de Volterra en la Toscana central mantiene abierto el debate sobre los orígenes de los etruscos , aunque los números están fuertemente a favor de la tesis autóctona: la baja presencia de J2a-M67* (2,7%) sugiere contactos por mar con pueblos de Anatolia; la presencia del linaje centroeuropeo G2a-L497 (7,1%) con una frecuencia considerable apoyaría más bien un origen centroeuropeo de los etruscos; y finalmente, la alta incidencia de linajes europeos R1b (R1b 50% aprox., R1b-U152 24,5%) —especialmente del haplogrupo R1b-U152— podría sugerir un origen autóctono debido a un proceso de formación de la civilización etrusca a partir de la cultura villanoviana precedente , siguiendo las teorías de Dionisio de Halicarnaso , [54] como ya apoyan la arqueología, la antropología y la lingüística. [61] [62] [63] [64] En 2019, en un estudio de Stanford publicado en Science, se encontró que dos muestras antiguas del asentamiento neolítico de Ripabianca di Monterado en la provincia de Ancona , en la región de Marche en Italia, eran Y-DNA J-L26 y J-M304. [2] Por lo tanto, el Y-DNA J2a-M67, posterior al Y-DNA J-L26 y J-M304, probablemente se encuentra en Italia desde el Neolítico y no puede ser una prueba de contactos recientes con Anatolia.
En dos pueblos de Lacio y Abruzos ( Capadocia y Vallepietra ), el I1 es el ADN-Y más común, registrado en niveles del 35% y 28%. [65] En Sicilia , las migraciones posteriores de los vándalos y sarracenos solo han afectado ligeramente a la composición étnica del pueblo siciliano. Sin embargo, se estima que el legado genético específicamente griego es del 37% en Sicilia. [12]
La conquista normanda del sur de Italia provocó la creación del Reino normando de Sicilia en 1130, con Palermo como capital, 70 años después de la invasión normanda inicial y 40 después de la conquista de la última ciudad, Noto en 1091, y duraría hasta 1198. Hoy en día es en Sicilia central y occidental, donde el ADN-Y normando es común, con un 15% a 20% de los linajes pertenecientes al haplogrupo I , este porcentaje cae al 8% en la parte oriental de la isla. La contribución masculina norteafricana a Sicilia se estimó entre el 0% y el 7,5%. [66] [13] [67] En general, las contribuciones paternas estimadas del sur de los Balcanes y de Europa occidental en Sicilia son de alrededor del 63% y el 26% respectivamente. [67]
Un estudio genético de 2015 de seis pequeños pueblos de montaña en el este de Lacio y una comunidad de montaña en el cercano oeste de Abruzzo encontró algunas similitudes genéticas entre estas comunidades y las poblaciones del Cercano Oriente , principalmente en el acervo genético masculino. El haplogrupo Y Q , común en Asia occidental y Asia central , también se encontró entre esta población de muestra, lo que sugiere que en el pasado podría haber albergado un asentamiento de Anatolia. [65] Además, es de alrededor del 0,6% en Italia continental , pero aumenta al 2,5% (6/236) en Sicilia , donde alcanza el 16,7% (3/18) en la región de Mazara del Vallo , seguido del 7,1% (2/28) en Ragusa , el 3,6% en Sciacca , [12] y el 3,7% en Belvedere Marittimo . [68]
En Italia, como en el resto de Europa, la mayoría de los linajes de ADNmt pertenecen al haplogrupo H. Varios estudios independientes concluyen que el haplogrupo H probablemente evolucionó en Asia occidental hace unos 25.000 años. Fue llevado a Europa por migraciones hace unos 20-25.000 años y se extendió con la población del suroeste del continente. [69] [70] Su llegada fue aproximadamente contemporánea con el surgimiento de la cultura gravetiense . La propagación de los subclados H1, H3 y el haplogrupo hermano V reflejan una segunda expansión intraeuropea desde la región franco-cantábrica después del último máximo glacial , hace unos 13.000 años. [69] [71]
Los linajes del haplogrupo L africano son relativamente poco frecuentes (menos del 1%) en toda Italia, con excepción del Lacio , Volterra , Basilicata y Sicilia , donde se han encontrado frecuencias entre el 2 y el 3%. [72]
Un estudio de 2012 realizado por Brisighelli et al. afirmó que un análisis de marcadores informativos ancestrales "como el realizado en el presente estudio indicó que Italia muestra un componente africano subsahariano muy menor que, sin embargo, es ligeramente superior al de la Europa no mediterránea". Al hablar de los ADNmt africanos, el estudio afirma que estos indican que una proporción significativa de estos linajes podrían haber llegado a Italia hace más de 10.000 años; por lo tanto, su presencia en Italia no data necesariamente de la época del Imperio Romano, la trata de esclavos del Atlántico o la migración moderna". [18] Estos ADNmt de Brisighelli et al. se informaron con los resultados dados como "Los haplotipos de ADN mitocondrial de origen africano están representados principalmente por los haplogrupos M1 (0,3%), U6 (0,8%) y L (1,2%) para las 583 muestras analizadas. [18] Los haplogrupos M1 y U6 pueden considerarse de origen norteafricano y, por lo tanto, podrían utilizarse para señalar el aporte histórico africano documentado. El haplogrupo M1 se observó solo en dos portadores de Trapani (Sicilia occidental), mientras que el U6 se observó solo en Lucera, Apulia meridional, y otro en la punta de la península (Calabria). [18]
Un estudio de 2013 realizado por Alessio Boattini et al. no encontró ningún haplogrupo africano L en toda Italia de 865 muestras. Los porcentajes de los haplogrupos bereberes M1 y U6 fueron 0,46% y 0,35%, respectivamente. [73]
Un estudio de 2014 realizado por Stefania Sarno et al. no encontró ningún haplogrupo africano L y M1 en el sur de Italia continental de 115 muestras. Solo se encontraron dos U6 bereberes de 115 muestras, una de Lecce y otra de Cosenza. [67]
En estudios genéticos se ha observado una estrecha similitud genética entre los ashkenazíes y los italianos, posiblemente debido al hecho de que los judíos ashkenazíes tienen una importante mezcla europea (30-60%), gran parte de ella del sur de Europa, gran parte de la cual vino de Italia cuando los hombres judíos de la diáspora de origen del Medio Oriente emigraron a Roma y encontraron esposas entre las mujeres locales que luego se convirtieron al judaísmo . [74] [75] [76] [77] [78] [79] [80] Más específicamente, los judíos ashkenazíes podrían modelarse como 50% levantinos y 50% europeos, con una mezcla media estimada del sur de Europa del 37,5%. La mayor parte (30,5%) parece derivar de una fuente italiana. [81] [82]
Un estudio de 2010 sobre la genealogía judía descubrió que, con respecto a los grupos europeos no judíos, las poblaciones más estrechamente relacionadas con los judíos asquenazíes son los italianos actuales, seguidos por los franceses y los sardos. [83] [84]
Estudios recientes han demostrado que Italia jugó un papel importante en la recuperación de la " Europa Occidental " al final de la Última Edad Glacial . El estudio que se centró en el haplogrupo mitocondrial U5b3 descubrió que este linaje femenino se había originado de hecho en Italia y hace unos 10.000 años se expandió desde la Península hacia Provenza y los Balcanes . En Provenza, probablemente hace entre 9.000 y 7.000 años, dio origen al subclado del haplogrupo U5b3a1. Este subclado U5b3a1 llegó más tarde desde Provenza a la isla de Cerdeña a través de comerciantes de obsidiana, porque se estima que el 80% de la obsidiana que se encuentra en Francia proviene de Monte Arci en Cerdeña, lo que refleja la estrecha relación que alguna vez existió entre estas dos regiones. Todavía alrededor del 4% de la población femenina de Cerdeña pertenece a este haplotipo. [85]
Un estudio de ADNmt, publicado en 2018 en la revista American Journal of Physical Anthropology , comparó muestras antiguas y modernas de Toscana, de la Prehistoria , la época etrusca, la época romana , el Renacimiento y la actualidad, y concluyó que los etruscos aparecen como una población local, intermedia entre las muestras prehistóricas y las otras, situándose en la red temporal entre el Eneolítico y la época romana. [86]
Un análisis de 2020 de haplogrupos maternos de muestras antiguas y modernas en la región de Umbría, en el centro de Italia, encuentra una similitud genética sustancial entre los umbros modernos y los habitantes prerromanos de la zona, y evidencia de una continuidad genética sustancial en la región desde la época prerromana hasta el presente. Se encontró que tanto los umbros modernos como los antiguos tenían altas tasas de haplogrupos de ADNmt U4 y U5a, y una sobrerrepresentación de J (aproximadamente un 30%). El estudio también encontró que "las continuidades genéticas locales están atestiguadas además por seis ramas terminales (H1e1, J1c3, J2b1, U2e2a, U8b1b1 y K1a4a)" también compartidas por los umbros antiguos y modernos. [6]

En 1994, un estudio del SNP indicó que los habitantes del centro y sur de Italia estaban más cerca de los pueblos de Grecia y otros países mediterráneos. [88]
Wade et al. (2008) determinaron que Italia es una de las dos últimas islas genéticas que quedan en Europa, la otra es Finlandia . Esto se debe en parte a la presencia de la cadena montañosa alpina que, a lo largo de los siglos, ha impedido grandes flujos migratorios. [89]
Estudios recientes de todo el genoma han sido capaces de detectar y cuantificar la mezcla como nunca antes. Li et al. (2008), utilizando más de 600.000 SNP autosómicos, identificaron siete grupos de población global, incluidos los de Europa, Oriente Medio y Asia central y meridional. Todas las muestras italianas pertenecen al grupo centro-occidental con influencias menores que datan del período Neolítico. [90]
López Herráez et al. (2009) tipificaron las mismas muestras en cerca de un millón de SNP y las analizaron en un contexto euroasiático occidental, identificando una serie de subgrupos. Esta vez, todas las muestras europeas muestran alguna mezcla menor. Entre los italianos, Toscana todavía tiene la mayor cantidad, y Cerdeña también tiene un poco, pero también Lombardía (Bérgamo), que está incluso más al norte. [91]
Un estudio de 2011 realizado por Moorjani et al. descubrió que muchos europeos del sur han heredado entre un 1 y un 3 % de ascendencia subsahariana, aunque los porcentajes fueron más bajos (entre un 0,2 y un 2,1 %) cuando se volvió a analizar con el modelo estadístico 'STRUCTURE'. También se calculó una fecha de mezcla promedio de alrededor de 55 generaciones/1100 años atrás, "coherente con el flujo genético del norte de África al final del Imperio Romano y las migraciones árabes posteriores" [92].
Un estudio de 2012 realizado por Di Gaetano et al. utilizó 1.014 italianos de una amplia cobertura geográfica y demostró que la población actual de Cerdeña puede diferenciarse claramente genéticamente de la Italia continental y Sicilia, y que es detectable un cierto grado de diferenciación genética dentro de la población actual de la península itálica.
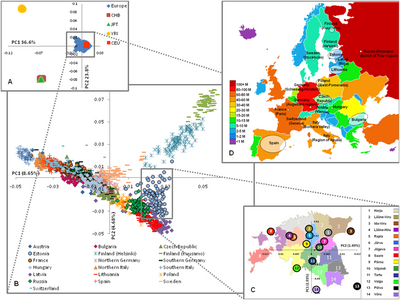
Al utilizar el software ADMIXTURE, los autores obtuvieron en K = 4 el error de validación cruzada más bajo. Los individuos CEU de HapMap mostraron una ascendencia media de Europa del Norte (NE) del 83%. Se observa un patrón similar en las poblaciones francesa, del norte de Italia y del centro de Italia con una ascendencia NE del 70%, 56% y 52% respectivamente. Según el gráfico de PCA, también en el análisis ADMIXTURE hay diferencias relativamente pequeñas en la ascendencia entre los italianos del norte y los del centro, mientras que los italianos del sur mostraron una proporción media de mezcla NE inferior (44%) que el norte y el centro de Italia, y una ascendencia caucásica superior del 28%. Las muestras sardas muestran un patrón de carmesí común a las otras poblaciones europeas, pero con una frecuencia más alta (70%). La proporción promedio de mezcla de ascendencia del norte de Europa dentro de la población sarda actual es del 14,3%, y algunos individuos muestran una ascendencia del norte de Europa muy baja (menos del 5% en 36 individuos de 268, lo que representa el 13% de la muestra). [17]
Un estudio de 2013 realizado por Peristera Paschou et al. confirma que el mar Mediterráneo ha actuado como una fuerte barrera al flujo genético a través del aislamiento geográfico tras los asentamientos iniciales. Las muestras de Italia (norte), Toscana, Sicilia y Cerdeña son las más cercanas a otros europeos meridionales de Iberia, los Balcanes y Grecia, que a su vez son los más cercanos a los migrantes neolíticos que difundieron la agricultura por toda Europa, representados aquí por la muestra capadocia de Anatolia. Pero no ha habido ninguna mezcla significativa desde Oriente Medio o el norte de África en Italia y el resto del sur de Europa desde entonces. [93]
Los análisis de ADN antiguo revelan que Ötzi, el hombre de hielo, se agrupa con los europeos meridionales modernos y más cercanos a los italianos (los puntos naranjas con la "S" de Europa en los gráficos de abajo), especialmente aquellos de la isla de Cerdeña. Otros italianos se alejan hacia el sudeste y el centro de Europa en consonancia con la geografía y algún flujo genético post-neolítico de esas áreas (por ejemplo, itálicos, griegos, etruscos, celtas), pero a pesar de eso y de siglos de historia, siguen siendo muy similares a su ancestro prehistórico. [94]
En un estudio de 2013 realizado por Botigué et al. (2013) se aplicó un algoritmo de agrupamiento no supervisado, ADMIXTURE, para estimar la compartición basada en alelos entre africanos y europeos. En cuanto a los italianos, la ascendencia norteafricana no supera el 2% de sus genomas. En promedio, el 1% de la ascendencia judía se encuentra en la población de HapMap toscana y suizos italianos, así como en griegos y chipriotas. Contrariamente a observaciones anteriores, la ascendencia subsahariana se detecta en <1% en Europa, con la excepción de las Islas Canarias. [67]
Haak et al. (2015) realizaron un estudio de todo el genoma de 94 esqueletos antiguos de Europa y Rusia. El estudio sostiene que los pastores esteparios de la Edad de Bronce de la cultura Yamna difundieron lenguas indoeuropeas en Europa. Las pruebas autosómicas indican que el pueblo Yamnaya fue el resultado de la mezcla entre dos poblaciones de cazadores-recolectores diferentes: cazadores-recolectores orientales de la estepa rusa y cazadores-recolectores del Cáucaso o iraníes calcolíticos (que son muy similares). Wolfgang Haak estimó una contribución ancestral del 27% de los Yamnaya en el ADN de los toscanos modernos, una contribución ancestral del 25% de los Yamnaya en el ADN de los italianos del norte modernos de Bérgamo , excluyendo a los sardos (7%) y, en menor medida, a los sicilianos (12%). [95]
Un estudio de 2016 de Sazzini et al. confirma los resultados de estudios previos de Di Gaetano et al. (2012) y Fiorito et al. (2015), pero tiene una cobertura geográfica mucho mejor de las muestras, ya que se analizaron 737 individuos de 20 lugares en 15 regiones diferentes. El estudio también incluye por primera vez una prueba de mezcla formal que modela la ascendencia de los italianos al inferir eventos de mezcla utilizando todas las muestras de Eurasia occidental. Los resultados son muy interesantes a la luz de la evidencia de ADN antiguo que ha surgido en los últimos años:
Además del patrón descrito en el texto principal, la muestra SARD parecía haber jugado un papel importante como fuente de mezcla para la mayoría de las poblaciones examinadas, especialmente las italianas, en lugar de como receptora de procesos migratorios. De hecho, las puntuaciones f3 más significativas para los tríos que incluían SARD indicaron que los italianos peninsulares eran resultados plausibles de la mezcla entre SARD y poblaciones de Irán , el Cáucaso y Rusia . Este escenario podría interpretarse como una prueba más de que los sardos conservan altas proporciones de un supuesto trasfondo genómico ancestral que estaba considerablemente extendido en toda Europa al menos hasta el Neolítico y que posteriormente se ha borrado o enmascarado en la mayoría de las poblaciones europeas actuales. [19]
Sarno et al. (2017) se concentran en el impacto genético provocado por las migraciones históricas alrededor del Mediterráneo en el sur de Italia y Sicilia, y concluyen que los "resultados demuestran que la variabilidad genética de las poblaciones actuales del sur de Italia se caracteriza por una continuidad genética compartida, que se extiende a grandes porciones de las costas centrales y orientales del Mediterráneo", al tiempo que muestran que "el sur de Italia parece más similar a las islas de habla griega del mar Mediterráneo, llegando tan al este como Chipre , que a las muestras de la Grecia continental, lo que sugiere un posible vínculo ancestral que podría haber sobrevivido en una forma menos mezclada en las islas", también precisa cómo "además de un componente predominante similar al Neolítico , nuestros análisis revelan impactos significativos de ancestros relacionados con el Cáucaso post-Neolítico y el Levante ". [13] Un artículo de noticias asociado con la Sociedad Max Planck , que revisa los resultados, aunque comienza afirmando que "las poblaciones a lo largo de la costa mediterránea oriental comparten una herencia genética que trasciende la nacionalidad", también señala cómo este estudio es interesante en los debates sobre la difusión de la familia de lenguas indoeuropeas en Europa, ya que, si bien muestra la influencia del Cáucaso , no hay ningún marcador genético asociado con la estepa póntico-caspia , "una señal genética muy característica bien representada en Europa central y oriental del norte, que estudios previos asociaron con la introducción de lenguas indoeuropeas en el continente". [13]
Raveane et al. (2019) descubrieron en un estudio de todo el genoma de los italianos actuales una contribución de los cazadores-recolectores del Cáucaso de la Edad del Bronce de Anatolia del tercer milenio, predominantemente en el sur de Italia. Además, los patrones de variación regional mostraron una estructura geográfica en el sur de Italia, el norte de Italia y Cerdeña, en línea con estudios anteriores. Se observó una estructura aún más detallada entre los grupos subregionales, causada por la geografía y la distancia, y una mezcla histórica posiblemente asociada con eventos al final del Imperio Romano y durante períodos posteriores. [4]
Antonio et al. (2019) estudiaron poblaciones históricas de varios períodos de tiempo en el Lacio y Roma. Encontraron que, a pesar de las diferencias lingüísticas, los latinos y los etruscos no mostraban diferencias genéticas significativas. Su ADN autosómico era una mezcla en proporciones similares de cazadores-recolectores occidentales (Mesolítico), agricultores europeos primitivos (Neolítico) y pastores esteparios occidentales (Edad del Bronce). [2]
Un estudio de 2022 de todo el genoma de más de 700 individuos del área del sur del Mediterráneo (102 del sur de Italia), combinado con ADN antiguo de áreas vecinas, encontró altas afinidades de los italianos del sudeste con los peloponesios orientales modernos , y una afinidad más cercana de los genomas de la antigua Grecia con los de regiones específicas del sur de Italia que con los genomas de la Grecia moderna. El estudio también descubrió fuentes genéticas comunes compartidas entre el sur de Italia y el Peloponeso, que pueden modelarse como una mezcla de ascendencias del Neolítico de Anatolia y del Calcolítico iraní. [96]
Monnereau et al (2024) analizaron los entierros en el sitio de Segesta para investigar las interacciones entre las comunidades musulmana y cristiana durante la Edad Media en Sicilia. Los resultados biomoleculares e isotópicos sugieren que los cristianos se mantuvieron genéticamente distintos de la comunidad musulmana en Segesta, aunque siguieron una dieta sustancialmente similar. Con base en estos resultados, los autores sugieren que las dos comunidades en Segesta podrían haber seguido reglas de endogamia. [97]
Curiosamente, aunque los individuos de la Edad del Hierro fueron muestreados tanto de contextos etruscos (n = 3) como latinos (n = 6), no detectamos ninguna diferencia significativa entre los dos grupos con estadísticas f4 en forma de f4(RMPR_Etruscan, RMPR_Latin; población de prueba, Onge), lo que sugiere orígenes compartidos o un amplio intercambio genético entre ellos. ... En los períodos medieval y moderno temprano (n = 28 individuos), observamos un cambio de ascendencia hacia Europa central y septentrional en PCA (Fig. 3E), así como un aumento adicional en el grupo europeo (C7) y la pérdida de los grupos del Cercano Oriente y Mediterráneo oriental (C4 y C5) en ChromoPainter (Fig. 4C). La población medieval está centrada aproximadamente en los italianos centrales de la actualidad (Fig. 3F). Puede modelarse como una combinación bidireccional de la población tardoantigua de Roma y una población donante europea, con fuentes potenciales que incluyen muchas poblaciones antiguas y modernas del centro y norte de Europa: lombardos de Hungría, sajones de Inglaterra y vikingos de Suecia, entre otros (tabla S26).
Se estima que la contribución genética de los cromosomas griegos al acervo genético siciliano es de alrededor del 37%, mientras que la contribución de las poblaciones del norte de África se estima en alrededor del 6%.
Curiosamente, aunque se tomaron muestras de individuos de la Edad del Hierro tanto de contextos etruscos (n = 3) como latinos (n = 6), no detectamos ninguna diferencia significativa entre los dos grupos con estadísticas f4 en forma de f4(RMPR_Etruscan, RMPR_Latin; población de prueba, Onge), lo que sugiere orígenes compartidos o un intercambio genético extenso entre ellos.
De hecho, mientras que la presencia de J2a-M67* sugiere contactos por mar con pueblos de Anatolia, de acuerdo con la hipótesis de Heródoto de una fuente anatolia externa de etruscos, el hallazgo del linaje centroeuropeo G2a-L497 con una frecuencia considerable apoyaría más bien un origen de los etruscos en el norte de Europa. Por otra parte, la alta incidencia de linajes europeos R1b no puede descartar el escenario de un proceso autóctono de formación de la civilización etrusca a partir de la sociedad villanoviana precedente, como sugirió por primera vez Dionisio de Halicarnaso; un análisis detallado del haplogrupo R1b-U152 podría resultar muy informativo a este respecto.
La Grotta La Sassa (ID del Catastro Nacional de Cuevas: LA 2001) fue descubierta en 2015 durante un estudio de las cuevas naturales de los Montes Ausoni realizado por dos grupos espeleológicos: Gruppo Grotte Castelli Romani y Speleo Club Roma. (. ..) En La Sassa, los dos machos LSC002/004 y LSC011 tienen un haplotipo Ychr idéntico (J2a-M410/J2a7-Z2397; Tabla 1; Datos S1B y S1F)
que el vasco, el paleosardo, el minoico y el etrusco se desarrollaran en el continente durante la revolución neolítica. Lamentablemente, nunca se conocerá la verdadera diversidad de las lenguas que alguna vez existieron en Europa.
4/138=2,9% en Lacio; 3/114=2,6% en Volterra; 2/92=2,2% en Basilicata y 3/154=2,0% en Sicilia