El citoesqueleto procariota es el nombre colectivo de todos los filamentos estructurales en procariotas . Alguna vez se pensó que las células procariotas no poseían citoesqueletos , pero los avances en la tecnología de visualización y la determinación de la estructura llevaron al descubrimiento de filamentos en estas células a principios de la década de 1990. [2] No solo se han encontrado análogos de todas las principales proteínas citoesqueléticas en eucariotas en procariotas, sino que también se han descubierto proteínas citoesqueléticas sin homólogos eucariotas conocidos. [3] [4] [5] [6] Los elementos del citoesqueleto desempeñan funciones esenciales en la división celular , la protección, la determinación de la forma y la determinación de la polaridad en varios procariotas. [7] [8]
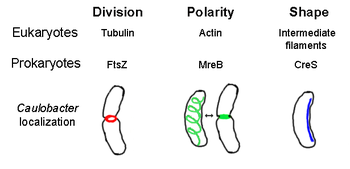
FtsZ, el primer elemento citoesquelético procariota identificado, forma una estructura de anillo filamentoso ubicada en el medio de la célula llamada anillo Z que se contrae durante la división celular , similar al anillo contráctil de actina-miosina en eucariotas. [2] El anillo Z es una estructura altamente dinámica que consta de numerosos haces de protofilamentos que se extienden y encogen, aunque el mecanismo detrás de la contracción del anillo Z y el número de protofilamentos involucrados no están claros. [1] FtsZ actúa como una proteína organizadora y es necesaria para la división celular. Es el primer componente del septo durante la citocinesis y recluta todas las demás proteínas de división celular conocidas al sitio de división. [9]
A pesar de esta similitud funcional con la actina , FtsZ es homóloga a la tubulina eucariota . Aunque la comparación de las estructuras primarias de FtsZ y tubulina revela una relación débil, sus estructuras tridimensionales son notablemente similares. Además, al igual que la tubulina, FtsZ monomérico está unido a GTP y polimeriza con otros monómeros de FtsZ con la hidrólisis de GTP en un mecanismo similar a la dimerización de la tubulina . [10] Dado que FtsZ es esencial para la división celular en bacterias, esta proteína es un objetivo para el diseño de nuevos antibióticos . [11] Actualmente existen varios modelos y mecanismos que regulan la formación del anillo Z, pero estos mecanismos dependen de la especie. Varias especies con forma de bastón, incluidas Escherichia coli y Caulobacter crescentus , utilizan uno o más inhibidores del ensamblaje de FtsZ que forman un gradiente bipolar en la célula, mejorando la polimerización de FtsZ en el centro celular. [12] Uno de estos sistemas formadores de gradientes consiste en proteínas MinCDE (ver a continuación).
MreB es una proteína bacteriana que se cree que es homóloga de la actina eucariota . MreB y la actina tienen una débil coincidencia en su estructura primaria , pero son muy similares en términos de estructura tridimensional y polimerización de filamentos.
Casi todas las bacterias no esféricas dependen de MreB para determinar su forma. MreB se ensambla en una red helicoidal de estructuras filamentosas justo debajo de la membrana citoplasmática , cubriendo toda la longitud de la célula. [13] MreB determina la forma de la célula al mediar la posición y la actividad de las enzimas que sintetizan peptidoglicano y al actuar como un filamento rígido debajo de la membrana celular que ejerce presión hacia afuera para esculpir y reforzar la célula. [1] MreB se condensa a partir de su red helicoidal normal y forma un anillo apretado en el tabique en Caulobacter crescentus justo antes de la división celular, un mecanismo que se cree que ayuda a localizar su tabique descentrado. [14] MreB también es importante para la determinación de la polaridad en bacterias polares, ya que es responsable del posicionamiento correcto de al menos cuatro proteínas polares diferentes en C. crescentus . [14]
ParM es un elemento del citoesqueleto que posee una estructura similar a la actina , aunque se comporta funcionalmente como la tubulina . Además, polimeriza bidireccionalmente y exhibe inestabilidad dinámica , que son ambos comportamientos característicos de la polimerización de la tubulina. [4] [15] Forma un sistema con ParR y parC que es responsable de la separación del plásmido R1 . ParM se fija a ParR, una proteína de unión al ADN que se une específicamente a 10 repeticiones directas en la región parC en el plásmido R1. Esta unión ocurre en ambos extremos del filamento de ParM. Luego, este filamento se extiende, separando los plásmidos. [16] El sistema es análogo a la segregación de cromosomas eucariotas, ya que ParM actúa como la tubulina eucariota en el huso mitótico , ParR actúa como el complejo cinetocoro y parC actúa como el centrómero del cromosoma . [17]
La segregación del plásmido F ocurre en un sistema similar donde SopA actúa como filamento citoesquelético y SopB se une a la secuencia sopC en el plásmido F, como el cinetocoro y el centrómero respectivamente. [17] Últimamente se ha encontrado un homólogo de ParM similar a la actina en una bacteria grampositiva Bacillus thuringiensis , que se ensambla en una estructura similar a un microtúbulo y está involucrado en la segregación del plásmido . [18]
La crenactina es un homólogo de actina exclusivo del reino arqueológico Thermoproteota (anteriormente Crenarchaeota) que se ha encontrado en los órdenes Thermoproteales y Candidatus Korarchaeum . [19] En el momento de su descubrimiento en 2009, tiene la mayor similitud de secuencia con actinas eucariotas de cualquier homólogo de actina conocido. [20] La crenactina ha sido bien caracterizada en Pyryobaculum calidifontis ( A3MWN5 ) y se ha demostrado que tiene una alta especificidad para ATP y GTP. [19] Las especies que contienen crenactina tienen forma de bastón o aguja. En P. calidifontis , se ha demostrado que la crenactina forma estructuras helicoidales que se extienden a lo largo de la célula, lo que sugiere un papel para la crenactina en la determinación de la forma similar al de MreB en otros procariotas. [19] [21]
Un sistema de actina aún más cercano al eucariota se encuentra en el superfilo propuesto de Asgardarchaeota . Utilizan versiones primitivas de profilina , gelsolina y cofilina para regular el citoesqueleto. [22]
La crescentina (codificada por el gen creS ) es un análogo de los filamentos intermedios eucariotas (FI). A diferencia de las otras relaciones análogas analizadas aquí, la crescentina tiene una homología primaria bastante grande con las proteínas FI además de una similitud tridimensional: la secuencia de creS tiene una coincidencia de identidad del 25% y una similitud del 40% con la citoqueratina 19 y una coincidencia de identidad del 24% y una similitud del 40% con la lámina nuclear A. Además, los filamentos de crescentina tienen aproximadamente 10 nm de diámetro y, por lo tanto, se encuentran dentro del rango de diámetro de los FI eucariotas (8-15 nm). [23] La crescentina forma un filamento continuo de polo a polo a lo largo del lado cóncavo interno de la bacteria en forma de medialuna Caulobacter crescentus . Tanto MreB como crescentina son necesarios para que C. crescentus exista en su forma característica; se cree que MreB moldea la célula en forma de bastón y la crescentina dobla esta forma en una medialuna. [1]
El sistema MinCDE es un sistema de filamentos que posiciona correctamente el septo en el medio de la célula en Escherichia coli . Según Shih et al., MinC inhibe la formación del septo al prohibir la polimerización del anillo Z. MinC, MinD y MinE forman una estructura helicoidal que se enrolla alrededor de la célula y se une a la membrana por MinD. La hélice MinCDE ocupa un polo y termina en una estructura filamentosa llamada anillo E hecho de MinE en el borde más medio de la zona polar. A partir de esta configuración, el anillo E se contraerá y se moverá hacia ese polo, desmontando la hélice MinCDE a medida que avanza. Al mismo tiempo, los fragmentos desmontados se volverán a ensamblar en el extremo polar opuesto, reformando la bobina MinCDE en el polo opuesto mientras se descompone la hélice MinCDE actual. Este proceso luego se repite, con la hélice MinCDE oscilando de polo a polo. Esta oscilación ocurre repetidamente durante el ciclo celular, manteniendo así a MinC (y su efecto inhibidor del tabique) en una concentración promedio en el tiempo más baja en el centro de la célula que en los extremos de la célula. [24]
El comportamiento dinámico de las proteínas Min se ha reconstituido in vitro utilizando una bicapa lipídica artificial como imitador de la membrana celular. MinE y MinD se autoorganizaron en ondas proteicas paralelas y espirales mediante un mecanismo de reacción-difusión. [25]
La bactofilina ( InterPro : IPR007607 ) es un elemento citoesquelético β-helicoidal que forma filamentos en todas las células de la proteobacteria en forma de bastón Myxococcus xanthus . [26] La proteína bactofilina, BacM, es necesaria para el mantenimiento adecuado de la forma celular y la integridad de la pared celular. Las células de M. xanthus que carecen de BacM tienen una morfología deformada caracterizada por un cuerpo celular doblado, y los mutantes bacM tienen una resistencia reducida a los antibióticos dirigidos a la pared celular bacteriana. La proteína BacM de M. xanthus se escinde de su forma de longitud completa para permitir la polimerización. Las bactofilinas se han implicado en la regulación de la forma celular en otras bacterias, incluida la curvatura de las células de Proteus mirabilis , [27] la formación del tallo por Caulobacter crescentus , [28] y la forma helicoidal de Helicobacter pylori . [29]