Caulobacter crescentus es una bacteria oligotrófica gramnegativa ampliamente distribuida en lagos y arroyos de agua dulce. El taxón se conoce más apropiadamente como Caulobacter vibrioides (Henrici y Johnson 1935). [1]
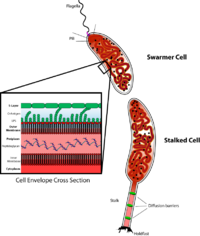
C. crescentus es un organismo modelo importante para estudiar la regulación del ciclo celular , la división celular asimétrica y la diferenciación celular . Las células hijas de Caulobacter tienen dos formas muy diferentes. Una hija es una célula "enjambre" móvil que tiene un solo flagelo en un polo celular que proporciona motilidad de natación para la quimiotaxis . La otra hija, llamada célula "acechada", tiene una estructura de tallo tubular que sobresale de un polo que tiene un material de sujeción adhesivo en su extremo, con el que la célula acechada puede adherirse a las superficies. Las células enjambre se diferencian en células acechadas después de un corto período de motilidad. La replicación cromosómica y la división celular solo ocurren en la etapa de célula acechada.
C. crescentus debe su nombre a su forma de medialuna , causada por la proteína crescentina . Es un organismo interesante para estudiar porque habita en ambientes acuáticos pobres en nutrientes. Su capacidad para prosperar en niveles bajos de nutrientes se ve facilitada por su ciclo de desarrollo dimórfico. La célula enjambre tiene un flagelo que sobresale de un solo polo y es incapaz de iniciar la replicación del ADN a menos que se diferencie en una célula pedunculada. El proceso de diferenciación incluye una transición morfológica caracterizada por la expulsión de su flagelo y el crecimiento de un tallo en el mismo polo. Las células pedunculadas pueden alargarse y replicar su ADN mientras desarrollan un flagelo en el polo opuesto, dando lugar a una célula predivisional. Aunque todavía se está investigando la función precisa de los tallos, es probable que estos estén involucrados en la absorción de nutrientes en condiciones de nutrientes limitados. [2] Su uso como modelo se originó con la bióloga del desarrollo Lucy Shapiro . [3] [4]
En el laboratorio, los investigadores distinguen entre la cepa CB15 de C. crescentus (la cepa aislada originalmente de un lago de agua dulce) y la NA1000 (la cepa experimental primaria). En la cepa NA1000, que se derivó de la CB15 en la década de 1970, [5] las células pedunculadas y predivisionales se pueden separar físicamente en el laboratorio de las nuevas células enjambre, mientras que los tipos de células de la cepa CB15 no se pueden separar físicamente. Las células enjambre aisladas se pueden cultivar entonces como un cultivo celular sincronizado. El estudio detallado del desarrollo molecular de estas células a medida que progresan a través del ciclo celular ha permitido a los investigadores comprender la regulación del ciclo celular de Caulobacter en gran detalle. Debido a esta capacidad de estar físicamente sincronizada, la cepa NA1000 se ha convertido en la cepa experimental predominante de Caulobacter en todo el mundo. Posteriormente, se han acumulado diferencias fenotípicas adicionales entre las dos cepas debido a las presiones selectivas sobre la cepa NA1000 en el entorno de laboratorio. La base genética de las diferencias fenotípicas entre las dos cepas resulta de polimorfismos codificantes, reguladores y de inserción/deleción en cinco loci cromosómicos. [6] C. crescentus es sinónimo de Caulobacter vibrioides . [1]
El genoma de Caulobacter CB15 tiene 4.016.942 pares de bases en un único cromosoma circular que codifica 3.767 genes. [7] El genoma contiene múltiples grupos de genes que codifican proteínas esenciales para la supervivencia en un hábitat pobre en nutrientes. Se incluyen aquellos involucrados en la quimiotaxis, la función del canal de la membrana externa, la degradación de compuestos de anillos aromáticos y la descomposición de fuentes de carbono derivadas de plantas, además de muchos factores sigma de función extracitoplasmática, que proporcionan al organismo la capacidad de responder a una amplia gama de fluctuaciones ambientales. En 2010, se secuenció la cepa Caulobacter NA1000 y se identificaron todas las diferencias con la cepa "de tipo salvaje" CB15. [6]
La etapa de célula pedunculada de Caulobacter proporciona una ventaja de aptitud al anclar la célula a superficies para formar biopelículas o explotar fuentes de nutrientes. En general, las especies bacterianas que se dividen más rápido serán las más efectivas para explotar recursos y ocupar nichos ecológicos de manera efectiva. Sin embargo, Caulobacter tiene la etapa de célula enjambre que da como resultado un crecimiento poblacional más lento. Se cree que la célula enjambre proporciona dispersión celular, de modo que el organismo busca constantemente nuevos entornos. Esto puede ser particularmente útil en entornos con una limitación severa de nutrientes cuando los escasos recursos disponibles pueden agotarse muy rápidamente. Muchas, tal vez la mayoría, de las células hijas enjambre no encontrarán un entorno productivo, pero la etapa de dispersión obligada debe aumentar la aptitud reproductiva de la especie en su conjunto.
El sistema regulador del ciclo celular de Caulobacter controla muchos subsistemas modulares que organizan la progresión del crecimiento y la reproducción celular. Un sistema de control construido utilizando circuitos lógicos bioquímicos y genéticos organiza el momento de iniciación de cada uno de estos subsistemas. La característica central de la regulación del ciclo celular es un circuito genético cíclico (un motor del ciclo celular) que se centra en las interacciones sucesivas de cinco proteínas reguladoras maestras: DnaA, GcrA, CtrA, SciP y CcrM, cuyas funciones fueron descifradas por los laboratorios de Lucy Shapiro y Harley McAdams . [8] [9] [10] Estas cinco proteínas controlan directamente el momento de la expresión de más de 200 genes. Las cinco proteínas reguladoras maestras se sintetizan y luego se eliminan de la célula una tras otra a lo largo del ciclo celular. Varias vías de señalización celular adicionales también son esenciales para el funcionamiento adecuado de este motor del ciclo celular. La función principal de estas vías de señalización es asegurar la producción y eliminación confiables de la proteína CtrA de la célula en los momentos correctos del ciclo celular.
Una característica esencial del ciclo celular de Caulobacter es que el cromosoma se replica una sola vez por ciclo celular, a diferencia del ciclo celular de E. coli , donde puede haber rondas superpuestas de replicación cromosómica en curso simultáneamente. Los papeles opuestos de las proteínas DnaA y CtrA de Caulobacter son esenciales para el control estricto de la replicación cromosómica de Caulobacter . [11] La proteína DnaA actúa en el origen de la replicación para iniciar la replicación del cromosoma. La proteína CtrA, por el contrario, actúa para bloquear el inicio de la replicación, por lo que debe eliminarse de la célula antes de que pueda comenzar la replicación cromosómica. Múltiples vías reguladoras adicionales integrales para la regulación del ciclo celular que involucran tanto las vías de señalización de fosfo como el control regulado de la proteólisis de proteínas [12] actúan para asegurar que DnaA y CtrA estén presentes en la célula exactamente cuando se necesitan.
Cada proceso activado por las proteínas del motor del ciclo celular implica una cascada de muchas reacciones. La cascada de subsistemas más larga es la replicación del ADN. En las células de Caulobacter , la replicación del cromosoma implica alrededor de 2 millones de reacciones de síntesis de ADN para cada brazo del cromosoma durante 40 a 80 minutos, según las condiciones. Si bien el tiempo promedio para cada reacción de síntesis individual se puede estimar a partir del tiempo total promedio observado para replicar el cromosoma, el tiempo de reacción real para cada reacción varía ampliamente en torno a la velocidad promedio. Esto conduce a una variación significativa e inevitable de célula a célula en el tiempo para completar la replicación del cromosoma. Existe una variación aleatoria similar en las velocidades de progresión de todas las demás cascadas de reacciones de subsistemas. El efecto neto es que el tiempo para completar el ciclo celular varía ampliamente entre las células de una población, incluso cuando todas crecen en condiciones ambientales idénticas. La regulación del ciclo celular incluye señales de retroalimentación que marcan el ritmo de la progresión del motor del ciclo celular para que coincida con el progreso de los eventos a nivel del subsistema regulador en cada célula en particular. Esta organización del sistema de control, con un controlador (el motor del ciclo celular) que impulsa un sistema complejo, con modulación mediante señales de retroalimentación del sistema controlado, crea un sistema de control de circuito cerrado.
La tasa de progresión del ciclo celular se ajusta aún más mediante señales adicionales que surgen de sensores celulares que monitorean las condiciones ambientales (por ejemplo, los niveles de nutrientes y el nivel de oxígeno) o el estado interno de la célula (por ejemplo, la presencia de daño en el ADN). [13]
El circuito de control que dirige y regula la progresión del ciclo celular de Caulobacter implica que toda la célula funcione como un sistema integrado. El circuito de control monitorea el entorno y el estado interno de la célula, incluida la topología celular, a medida que orquesta la activación de los subsistemas del ciclo celular y la división celular asimétrica de Caulobacter crescentus . Las proteínas del sistema de control del ciclo celular de Caulobacter y su organización interna se conservan conjuntamente en muchas especies de alfaproteobacterias, pero existen grandes diferencias en la funcionalidad del aparato regulador y la conectividad periférica con otros subsistemas celulares de una especie a otra. [14] [15] El sistema de control del ciclo celular de Caulobacter ha sido exquisitamente optimizado por selección evolutiva como un sistema total para un funcionamiento robusto frente al ruido estocástico interno y la incertidumbre ambiental.
El sistema de control de la célula bacteriana tiene una organización jerárquica. [16] El subsistema de señalización y control interactúa con el entorno por medio de módulos sensoriales ubicados en gran parte en la superficie celular. La lógica de la red genética responde a las señales recibidas del entorno y de los sensores de estado interno de la célula para adaptar la célula a las condiciones actuales. Una función principal del control de nivel superior es asegurar que las operaciones involucradas en el ciclo celular ocurran en el orden temporal apropiado. En Caulobacter , esto se logra mediante el circuito regulador genético compuesto por cinco reguladores maestros y una red de señalización fosfo asociada. La red de señalización fosfo monitorea el estado de progresión del ciclo celular y juega un papel esencial en el logro de la división celular asimétrica. El sistema de control del ciclo celular administra el tiempo y el lugar del inicio de la replicación cromosómica y la citocinesis , así como el desarrollo de orgánulos polares . Subyacentes a todas estas operaciones están los mecanismos para la producción de proteínas y componentes estructurales y la producción de energía. Los subsistemas metabólicos y catabólicos de “mantenimiento” proporcionan la energía y las materias primas moleculares para la síntesis de proteínas, la construcción de la pared celular y otras operaciones de la célula. Las funciones de mantenimiento están acopladas bidireccionalmente al sistema de control del ciclo celular. Sin embargo, pueden adaptarse, de manera algo independiente de la lógica de control del ciclo celular, a los cambios en la composición y los niveles de las fuentes de nutrientes disponibles.
Las proteínas del sistema de control del ciclo celular de Caulobacter están ampliamente co-conservadas en las alfa-proteobacterias, pero la función última de este sistema regulador varía ampliamente en diferentes especies. Estos cambios evolutivos reflejan enormes diferencias entre las especies individuales en cuanto a estrategias de adaptación y nichos ecológicos. Por ejemplo, Agrobacterium tumefaciens es un patógeno vegetal, Brucella abortus es un patógeno animal y Sinorhizobium meliloti es una bacteria del suelo que invade y se convierte en simbionte en los nódulos de las raíces de las plantas que fijan el nitrógeno, pero la mayoría de las proteínas del control del ciclo celular de Caulobacter también se encuentran en estas especies. El acoplamiento específico entre los componentes proteicos de la red de control del ciclo celular y la lectura descendente del circuito difiere de una especie a otra. El patrón es que la funcionalidad interna del circuito de la red se conserva, pero el acoplamiento en los "bordes" del aparato regulador con las proteínas que controlan funciones celulares específicas difiere ampliamente entre las diferentes especies.

Caulobacter crescentus es miembro de un grupo de bacterias que poseen la estructura del tallo, una extensión tubular del cuerpo celular. Sin embargo, la posición del tallo no se conserva necesariamente en el polo del cuerpo celular en diferentes especies estrechamente relacionadas. Específicamente, la investigación ha demostrado que no solo la posición del tallo puede cambiar, sino que también puede variar el número en el género estrechamente relacionado Asticcacaulis . [17] [18] Se ha demostrado que SpmX, una proteína localizada polarmente en Caulobacter crescentus, manipula la posición del tallo en estas especies de Asticcacaulis . [17] Presumiblemente, lo hace mediante una ganancia de función después de la expansión de la proteína de alrededor de 400 aminoácidos en Caulobacter crescentus a más de 800 aminoácidos en las especies de Asticcacaulis .
Caulobacter fue la primera bacteria asimétrica en la que se demostró que envejecía. La senescencia reproductiva se midió como la disminución del número de progenie producida con el tiempo. [19] [20] Sobre la base de estudios de evolución experimental en C. crescentus , Ackermann et al. [19] sugirieron que el envejecimiento es probablemente una propiedad fundamental de todos los organismos celulares. Desde entonces se ha descrito un fenómeno similar en la bacteria Escherichia coli , que da lugar a células hijas morfológicamente similares. [21]
En C. crescentus , la polaridad celular es fácilmente aparente por el ensamblaje de orgánulos polares y por la polarización del plano de división, lo que resulta en la generación de progenie pedunculada que es más larga que la progenie enjambre. La formación de nuevos polos celulares en la división implica que la polaridad celular debe restablecerse en la progenie pedunculada y revertirse en la progenie enjambre. [22]
El ciclo de vida de C. crescentus está gobernado por reguladores como TipN , una proteína del ciclo celular. Los datos de la Universidad de Yale sugieren firmemente un modelo en el que TipN regula la orientación del eje de polaridad al proporcionar una señal posicional del ciclo celular anterior. En este modelo, TipN especifica el sitio de la división más reciente al identificar el nuevo polo. La célula utiliza esta información posicional como una fuente de asimetría intracelular para establecer y mantener la orientación del eje de polaridad, que es crucial para la morfogénesis y la división polares. El reclutamiento de TipN a los polos nacientes al final del ciclo de división redefine la identidad de los polos y restablece la polaridad correcta en ambas células hijas futuras (con una inversión de polaridad en la célula enjambre). [22] La síntesis y eliminación reguladas por el ciclo celular de estas estructuras localizadas polarmente han proporcionado un rico campo de juego para la identificación de proteínas de referencia importantes para su localización adecuada. [23] TipN tiene dos regiones transmembrana en la región N-terminal y un gran dominio de hélice superenrollada en la C-terminal . Los homólogos de TipN están presentes en otras alfa-proteobacterias. TipN se localiza en el nuevo polo en ambas células hijas después de la división y se relocaliza en el sitio de división celular en la célula predivisional tardía. Por lo tanto, ambas células hijas tienen TipN en el nuevo polo después de la división. [23]
La proteína TipN es esencial para la colocación adecuada del flagelo. [24] Los mutantes que carecen de TipN cometen graves errores en el desarrollo. En lugar de producir un solo flagelo en el polo celular correcto, la célula produce múltiples flagelos en varias ubicaciones, incluso en el tallo. [22]

El desarrollo celular implica el trabajo conjunto de muchas de estas proteínas. La figura 1 muestra cómo TipN interactúa con otras dos proteínas polares: el marcador flagelar PodJ y el marcador del tallo DivJ. [25]
{{cite journal}}: CS1 maint: nombres numéricos: lista de autores ( enlace )