
Bioinformática ( / ˌ b aɪ . oʊ ˌ ɪ n f ər ˈ m æ t ɪ k s / ) es uncampointerdisciplinariola cienciaque desarrolla métodos yherramientas de softwarepara comprenderdatosbiológicosla biología,la química,la física,la informática,la programación informática,la ingeniería de la información,las matemáticasyla estadísticapara analizar e interpretardatos biológicos. El proceso posterior de análisis e interpretación de datos a menudo se conoce comobiología computacional, aunque la distinción entre los dos términos a menudo es discutida.
Las técnicas computacionales, estadísticas y de programación informática se han utilizado para los análisis de simulación por ordenador de consultas biológicas. Incluyen "canalizaciones" de análisis específicas reutilizadas, en particular en el campo de la genómica , como la identificación de genes y polimorfismos de un solo nucleótido ( SNP ). Estas canalizaciones se utilizan para comprender mejor la base genética de las enfermedades, las adaptaciones únicas, las propiedades deseables (especialmente en especies agrícolas) o las diferencias entre poblaciones. La bioinformática también incluye la proteómica , que intenta comprender los principios organizativos dentro de las secuencias de ácidos nucleicos y proteínas . [1]
El procesamiento de imágenes y señales permite extraer resultados útiles de grandes cantidades de datos sin procesar. En el campo de la genética, ayuda a secuenciar y anotar genomas y sus mutaciones observadas . La bioinformática incluye la minería de texto de literatura biológica y el desarrollo de ontologías biológicas y genéticas para organizar y consultar datos biológicos. También desempeña un papel en el análisis de la expresión y regulación de genes y proteínas. Las herramientas bioinformáticas ayudan a comparar, analizar e interpretar datos genéticos y genómicos y, de manera más general, a comprender los aspectos evolutivos de la biología molecular. A un nivel más integrador, ayuda a analizar y catalogar las vías y redes biológicas que son una parte importante de la biología de sistemas . En biología estructural , ayuda en la simulación y modelado de ADN, [2] ARN, [2] [3] proteínas [4] así como interacciones biomoleculares. [5] [6] [7] [8]
La primera definición del término bioinformática fue acuñada por Paulien Hogeweg y Ben Hesper en 1970, para referirse al estudio de los procesos de información en sistemas bióticos. [9] [10] [11] [12] [13] Esta definición colocó a la bioinformática como un campo paralelo a la bioquímica (el estudio de los procesos químicos en sistemas biológicos). [10]
La bioinformática y la biología computacional implican el análisis de datos biológicos, en particular secuencias de ADN, ARN y proteínas. El campo de la bioinformática experimentó un crecimiento explosivo a partir de mediados de la década de 1990, impulsado en gran medida por el Proyecto Genoma Humano y por los rápidos avances en la tecnología de secuenciación de ADN. [ cita requerida ]
El análisis de datos biológicos para producir información significativa implica escribir y ejecutar programas de software que utilizan algoritmos de teoría de grafos , inteligencia artificial , computación blanda , minería de datos , procesamiento de imágenes y simulación por computadora . Los algoritmos, a su vez, dependen de fundamentos teóricos como las matemáticas discretas , la teoría del control , la teoría de sistemas , la teoría de la información y la estadística . [ cita requerida ]


Se ha producido un enorme avance en velocidad y reducción de costes desde la finalización del Proyecto Genoma Humano: algunos laboratorios son capaces de secuenciar más de 100.000 billones de bases cada año, y se puede secuenciar un genoma completo por 1.000 dólares o menos. [14]
Las computadoras se volvieron esenciales en la biología molecular cuando las secuencias de proteínas estuvieron disponibles después de que Frederick Sanger determinara la secuencia de la insulina a principios de la década de 1950. [15] [16] Comparar múltiples secuencias manualmente resultó ser poco práctico. Margaret Oakley Dayhoff , una pionera en el campo, [17] compiló una de las primeras bases de datos de secuencias de proteínas, publicada inicialmente como libros [18] así como métodos de alineación de secuencias y evolución molecular . [19] Otro contribuyente temprano a la bioinformática fue Elvin A. Kabat , quien fue pionero en el análisis de secuencias biológicas en 1970 con sus volúmenes completos de secuencias de anticuerpos publicados en línea con Tai Te Wu entre 1980 y 1991. [20]
En la década de 1970, se aplicaron nuevas técnicas de secuenciación de ADN a los bacteriófagos MS2 y øX174, y luego se analizaron las secuencias de nucleótidos extendidas con algoritmos estadísticos e informativos. Estos estudios demostraron que las características bien conocidas, como los segmentos codificantes y el código de tripletes, se revelan en análisis estadísticos sencillos y fueron la prueba del concepto de que la bioinformática sería reveladora. [21] [22]
Para estudiar cómo se alteran las actividades celulares normales en diferentes estados patológicos, es necesario combinar datos biológicos en bruto para formar una imagen completa de estas actividades. Por lo tanto , el campo de la bioinformática ha evolucionado de tal manera que la tarea más urgente ahora implica el análisis e interpretación de varios tipos de datos. Esto también incluye secuencias de nucleótidos y aminoácidos , dominios proteicos y estructuras proteicas . [23]
Las subdisciplinas importantes dentro de la bioinformática y la biología computacional incluyen:
El objetivo principal de la bioinformática es aumentar la comprensión de los procesos biológicos. Lo que la distingue de otros enfoques es su enfoque en el desarrollo y la aplicación de técnicas computacionalmente intensivas para lograr este objetivo. Los ejemplos incluyen: reconocimiento de patrones , minería de datos , algoritmos de aprendizaje automático y visualización . Los principales esfuerzos de investigación en el campo incluyen la alineación de secuencias , el hallazgo de genes , el ensamblaje del genoma , el diseño de fármacos , el descubrimiento de fármacos , la alineación de la estructura de proteínas , la predicción de la estructura de proteínas , la predicción de la expresión genética y las interacciones proteína-proteína , los estudios de asociación de todo el genoma , el modelado de la evolución y la división celular/mitosis.
La bioinformática implica la creación y el avance de bases de datos, algoritmos, técnicas computacionales y estadísticas y teoría para resolver problemas formales y prácticos que surgen de la gestión y el análisis de datos biológicos.
En las últimas décadas, los rápidos avances en las tecnologías de investigación genómica y molecular y los avances en las tecnologías de la información se han combinado para producir una enorme cantidad de información relacionada con la biología molecular. La bioinformática es el nombre que se le da a estos enfoques matemáticos e informáticos que se utilizan para comprender mejor los procesos biológicos.
Las actividades comunes en bioinformática incluyen el mapeo y análisis de secuencias de ADN y proteínas, la alineación de secuencias de ADN y proteínas para compararlas y la creación y visualización de modelos tridimensionales de estructuras de proteínas.
Desde que se secuenció el bacteriófago Phage Φ-X174 en 1977, [24] las secuencias de ADN de miles de organismos han sido decodificadas y almacenadas en bases de datos. Esta información de secuencia se analiza para determinar genes que codifican proteínas , genes de ARN, secuencias reguladoras, motivos estructurales y secuencias repetitivas. Una comparación de genes dentro de una especie o entre diferentes especies puede mostrar similitudes entre funciones proteicas o relaciones entre especies (el uso de sistemática molecular para construir árboles filogenéticos ). Con la creciente cantidad de datos, hace mucho tiempo que se volvió poco práctico analizar secuencias de ADN manualmente. Los programas informáticos como BLAST se utilizan rutinariamente para buscar secuencias: a partir de 2008, de más de 260.000 organismos, que contienen más de 190 mil millones de nucleótidos . [25]
Antes de poder analizar las secuencias, se obtienen de un banco de datos, como GenBank. La secuenciación de ADN sigue siendo un problema nada trivial, ya que los datos en bruto pueden ser ruidosos o verse afectados por señales débiles. Se han desarrollado algoritmos para la determinación de bases para los diversos enfoques experimentales de la secuenciación de ADN.
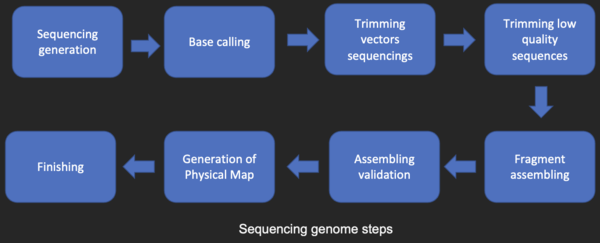
La mayoría de las técnicas de secuenciación de ADN producen fragmentos cortos de secuencia que deben ensamblarse para obtener secuencias completas de genes o genomas. La técnica de secuenciación shotgun (utilizada por el Instituto de Investigación Genómica (TIGR) para secuenciar el primer genoma bacteriano, Haemophilus influenzae ) [26] genera las secuencias de muchos miles de pequeños fragmentos de ADN (que van desde 35 a 900 nucleótidos de longitud, dependiendo de la tecnología de secuenciación). Los extremos de estos fragmentos se superponen y, cuando se alinean correctamente mediante un programa de ensamblaje de genomas, se pueden utilizar para reconstruir el genoma completo. La secuenciación shotgun produce datos de secuencia rápidamente, pero la tarea de ensamblar los fragmentos puede ser bastante complicada para genomas más grandes. Para un genoma tan grande como el genoma humano , puede llevar muchos días de tiempo de CPU en computadoras multiprocesador con gran memoria ensamblar los fragmentos, y el ensamblaje resultante generalmente contiene numerosos espacios que deben llenarse más tarde. La secuenciación por escopeta es el método de elección para prácticamente todos los genomas secuenciados (en lugar de los métodos de terminación de cadena o de degradación química), y los algoritmos de ensamblaje del genoma son un área crítica de la investigación bioinformática.
En genómica , la anotación se refiere al proceso de marcar las regiones de inicio y fin de los genes y otras características biológicas en una secuencia de ADN secuenciada. Muchos genomas son demasiado grandes para ser anotados a mano. Como la tasa de secuenciación excede la tasa de anotación del genoma, la anotación del genoma se ha convertido en el nuevo cuello de botella en bioinformática [ ¿cuándo? ] .
La anotación del genoma se puede clasificar en tres niveles: nivel de nucleótido , nivel de proteína y nivel de proceso.
El descubrimiento de genes es un aspecto fundamental de la anotación a nivel de nucleótidos. En el caso de genomas complejos, puede ser eficaz una combinación de predicción de genes ab initio y comparación de secuencias con bases de datos de secuencias expresadas y otros organismos. La anotación a nivel de nucleótidos también permite la integración de la secuencia del genoma con otros mapas genéticos y físicos del genoma.
El objetivo principal de la anotación a nivel de proteínas es asignar una función a los productos proteicos del genoma. Para este tipo de anotación se utilizan bases de datos de secuencias proteicas y dominios y motivos funcionales. Aproximadamente la mitad de las proteínas predichas en una nueva secuencia del genoma no suelen tener una función evidente.
El objetivo de la anotación a nivel de proceso es comprender la función de los genes y sus productos en el contexto de la fisiología celular y de los organismos. Un obstáculo de la anotación a nivel de proceso ha sido la inconsistencia de los términos utilizados por los diferentes sistemas modelo. El Consorcio de Ontología Génica está ayudando a resolver este problema. [27]
La primera descripción de un sistema de anotación integral fue publicada en 1995 [26] por el Instituto de Investigación Genómica , que realizó la primera secuenciación y análisis completos del genoma de un organismo de vida libre (no simbiótico ), la bacteria Haemophilus influenzae . [26] El sistema identifica los genes que codifican todas las proteínas, ARN de transferencia, ARN ribosómicos, para realizar asignaciones funcionales iniciales. El programa GeneMark entrenado para encontrar genes codificadores de proteínas en Haemophilus influenzae cambia y mejora constantemente.
Siguiendo los objetivos que el Proyecto Genoma Humano dejó pendientes tras su cierre en 2003, el Instituto Nacional de Investigación del Genoma Humano desarrolló el proyecto ENCODE , una recopilación colaborativa de datos de los elementos funcionales del genoma humano que utiliza tecnologías de secuenciación de ADN de última generación y matrices de teselación genómica, tecnologías capaces de generar automáticamente grandes cantidades de datos a un coste por base drásticamente reducido, pero con la misma precisión (error de llamada de base) y fidelidad (error de ensamblaje).
Si bien la anotación del genoma se basa principalmente en la similitud de secuencias (y, por lo tanto, en la homología ), se pueden utilizar otras propiedades de las secuencias para predecir la función de los genes. De hecho, la mayoría de los métodos de predicción de la función de los genes se centran en las secuencias de proteínas , ya que son más informativas y más ricas en características. Por ejemplo, la distribución de aminoácidos hidrofóbicos predice segmentos transmembrana en proteínas. Sin embargo, la predicción de la función de las proteínas también puede utilizar información externa, como datos de expresión de genes (o proteínas), estructura de proteínas o interacciones proteína-proteína . [28]
La biología evolutiva es el estudio del origen y la descendencia de las especies , así como de sus cambios a lo largo del tiempo. La informática ha ayudado a los biólogos evolutivos al permitirles a los investigadores:
Los trabajos futuros intentarán reconstruir el ahora más complejo árbol de la vida . [ ¿según quién? ]
El núcleo del análisis comparativo del genoma es el establecimiento de la correspondencia entre genes ( análisis de ortología ) u otras características genómicas en diferentes organismos. Los mapas intergenómicos se realizan para rastrear los procesos evolutivos responsables de la divergencia de dos genomas. Una multitud de eventos evolutivos que actúan en varios niveles organizativos dan forma a la evolución del genoma. En el nivel más bajo, las mutaciones puntuales afectan a nucleótidos individuales. En un nivel superior, grandes segmentos cromosómicos experimentan duplicación, transferencia lateral, inversión, transposición, deleción e inserción. [30] Los genomas completos están involucrados en procesos de hibridación, poliploidización y endosimbiosis que conducen a una rápida especiación. La complejidad de la evolución del genoma plantea muchos desafíos apasionantes a los desarrolladores de modelos y algoritmos matemáticos, que recurren a un espectro de técnicas algorítmicas, estadísticas y matemáticas, que van desde algoritmos exactos, heurísticos , de parámetros fijos y de aproximación para problemas basados en modelos de parsimonia hasta algoritmos de Monte Carlo de cadena de Markov para el análisis bayesiano de problemas basados en modelos probabilísticos.
Muchos de estos estudios se basan en la detección de homología de secuencias para asignar secuencias a familias de proteínas . [31]
La pangenómica es un concepto introducido en 2005 por Tettelin y Medini. El pangenoma es el repertorio completo de genes de un grupo taxonómico monofilético particular . Aunque inicialmente se aplicó a cepas estrechamente relacionadas de una especie, se puede aplicar a un contexto más amplio como género, filo, etc. Se divide en dos partes: el genoma central, un conjunto de genes comunes a todos los genomas en estudio (a menudo genes de mantenimiento vitales para la supervivencia), y el genoma prescindible/flexible: un conjunto de genes que no están presentes en todos los genomas en estudio excepto en uno o algunos. Se puede utilizar una herramienta bioinformática BPGA para caracterizar el pangenoma de especies bacterianas. [32]
A partir de 2013, la existencia de tecnología de secuenciación de última generación de alto rendimiento y eficiente permite la identificación de la causa de muchos trastornos humanos diferentes. Se ha observado la herencia mendeliana simple en más de 3000 trastornos que se han identificado en la base de datos Online Mendelian Inheritance in Man , pero las enfermedades complejas son más difíciles. Los estudios de asociación han encontrado muchas regiones genéticas individuales que individualmente están débilmente asociadas con enfermedades complejas (como la infertilidad , [33] el cáncer de mama [34] y la enfermedad de Alzheimer [35] ), en lugar de una única causa. [36] [37] Actualmente existen muchos desafíos para el uso de genes para el diagnóstico y el tratamiento, como el hecho de que no sabemos qué genes son importantes o qué tan estables son las opciones que proporciona un algoritmo. [38]
Los estudios de asociación de todo el genoma han identificado con éxito miles de variantes genéticas comunes para enfermedades y rasgos complejos; sin embargo, estas variantes comunes solo explican una pequeña fracción de la heredabilidad. [39] Las variantes raras pueden explicar parte de la heredabilidad faltante . [40] Los estudios de secuenciación del genoma completo a gran escala han secuenciado rápidamente millones de genomas completos, y dichos estudios han identificado cientos de millones de variantes raras . [41] Las anotaciones funcionales predicen el efecto o la función de una variante genética y ayudan a priorizar las variantes funcionales raras, y la incorporación de estas anotaciones puede aumentar eficazmente el poder del análisis de asociación genética de variantes raras de los estudios de secuenciación del genoma completo. [42] Se han desarrollado algunas herramientas para proporcionar un análisis de asociación de variantes raras todo en uno para datos de secuenciación del genoma completo, incluida la integración de datos de genotipo y sus anotaciones funcionales, análisis de asociación, resumen de resultados y visualización. [43] [44] El metanálisis de estudios de secuenciación del genoma completo proporciona una solución atractiva al problema de recolectar muestras de gran tamaño para descubrir variantes raras asociadas con fenotipos complejos. [45]
En el cáncer , los genomas de las células afectadas se reorganizan de formas complejas o impredecibles. Además de los conjuntos de polimorfismos de un solo nucleótido que identifican mutaciones puntuales que causan cáncer, se pueden utilizar microarreglos de oligonucleótidos para identificar ganancias y pérdidas cromosómicas (lo que se denomina hibridación genómica comparativa ). Estos métodos de detección generan terabytes de datos por experimento. A menudo se descubre que los datos contienen una variabilidad considerable, o ruido , y por lo tanto se están desarrollando métodos de análisis de puntos de cambio y modelos de Markov ocultos para inferir cambios reales en el número de copias . [ cita requerida ]
Se pueden utilizar dos principios importantes para identificar el cáncer mediante mutaciones en el exoma . En primer lugar, el cáncer es una enfermedad de mutaciones somáticas acumuladas en los genes. En segundo lugar, el cáncer contiene mutaciones impulsoras que deben distinguirse de las pasajeras. [46]
Las mejoras en la bioinformática podrían permitir clasificar los tipos de cáncer mediante el análisis de las mutaciones que provocan el cáncer en el genoma. Además, en el futuro podría ser posible hacer un seguimiento de los pacientes a medida que progresa la enfermedad con la secuenciación de muestras de cáncer. Otro tipo de datos que requiere un desarrollo informático novedoso es el análisis de las lesiones que se detectan como recurrentes en muchos tumores. [47]
La expresión de muchos genes se puede determinar midiendo los niveles de ARNm con múltiples técnicas, incluyendo microarrays , secuenciación de etiquetas de secuencia de ADNc expresado (EST), secuenciación de etiquetas de análisis serial de expresión génica (SAGE), secuenciación de firmas masivamente paralelas (MPSS), RNA-Seq , también conocida como "Secuenciación Shotgun del Transcriptoma Completo" (WTSS), o varias aplicaciones de hibridación in situ multiplexada. Todas estas técnicas son extremadamente propensas al ruido y/o sujetas a sesgo en la medición biológica, y un área de investigación importante en biología computacional implica el desarrollo de herramientas estadísticas para separar la señal del ruido en estudios de expresión génica de alto rendimiento. [48] Dichos estudios se utilizan a menudo para determinar los genes implicados en un trastorno: uno podría comparar datos de microarrays de células epiteliales cancerosas con datos de células no cancerosas para determinar las transcripciones que están reguladas al alza y a la baja en una población particular de células cancerosas.

Los microarrays de proteínas y la espectrometría de masas (MS) de alto rendimiento (HT) pueden proporcionar una instantánea de las proteínas presentes en una muestra biológica. El primer enfoque enfrenta problemas similares a los de los microarrays dirigidos al ARNm, el segundo implica el problema de hacer coincidir grandes cantidades de datos de masa con las masas predichas a partir de bases de datos de secuencias de proteínas y el complicado análisis estadístico de las muestras cuando se detectan múltiples péptidos incompletos de cada proteína. La localización de proteínas celulares en un contexto tisular se puede lograr a través de la proteómica de afinidad mostrada como datos espaciales basados en inmunohistoquímica y microarrays de tejido . [49]
La regulación genética es un proceso complejo en el que una señal, como una señal extracelular como una hormona , eventualmente conduce a un aumento o disminución en la actividad de una o más proteínas . Se han aplicado técnicas de bioinformática para explorar varios pasos en este proceso.
Por ejemplo, la expresión génica puede ser regulada por elementos cercanos en el genoma. El análisis del promotor implica la identificación y el estudio de motivos de secuencia en el ADN que rodean la región codificante de proteínas de un gen. Estos motivos influyen en el grado en que esa región se transcribe en ARNm. Los elementos potenciadores alejados del promotor también pueden regular la expresión génica, a través de interacciones tridimensionales en bucle. Estas interacciones pueden determinarse mediante el análisis bioinformático de experimentos de captura de conformación cromosómica .
Los datos de expresión se pueden utilizar para inferir la regulación genética: se pueden comparar datos de microarrays de una amplia variedad de estados de un organismo para formar hipótesis sobre los genes implicados en cada estado. En un organismo unicelular, se pueden comparar las etapas del ciclo celular , junto con varias condiciones de estrés (choque térmico, inanición, etc.). Los algoritmos de agrupamiento se pueden aplicar a los datos de expresión para determinar qué genes se coexpresan. Por ejemplo, se pueden buscar elementos reguladores sobrerrepresentados en las regiones ascendentes (promotores) de los genes coexpresados . Algunos ejemplos de algoritmos de agrupamiento aplicados en el agrupamiento de genes son el agrupamiento de k-medias , los mapas autoorganizados (SOM), el agrupamiento jerárquico y los métodos de agrupamiento por consenso .
Se han desarrollado varios enfoques para analizar la ubicación de orgánulos, genes, proteínas y otros componentes dentro de las células. Se ha ideado una categoría de ontología génica , componente celular , para capturar la localización subcelular en muchas bases de datos biológicas .
Las imágenes microscópicas permiten localizar orgánulos y moléculas que pueden ser fuente de anomalías en las enfermedades.
Encontrar la ubicación de las proteínas nos permite predecir lo que hacen. Esto se llama predicción de la función de las proteínas . Por ejemplo, si una proteína se encuentra en el núcleo , puede estar involucrada en la regulación o empalme de genes . Por el contrario, si una proteína se encuentra en las mitocondrias , puede estar involucrada en la respiración u otros procesos metabólicos . Hay recursos de predicción de la localización subcelular de proteínas bien desarrollados disponibles, incluidas bases de datos de ubicación subcelular de proteínas y herramientas de predicción. [50] [51]
Los datos de experimentos de captura de conformación cromosómica de alto rendimiento , como Hi-C (experimento) y ChIA-PET , pueden proporcionar información sobre la estructura tridimensional y la organización nuclear de la cromatina . Los desafíos bioinformáticos en este campo incluyen la partición del genoma en dominios, como los dominios de asociación topológica (TAD), que se organizan juntos en el espacio tridimensional. [52]

La determinación de la estructura de las proteínas es una aplicación importante de la bioinformática. La Evaluación crítica de la predicción de la estructura de las proteínas (CASP) es una competición abierta en la que grupos de investigación de todo el mundo presentan modelos proteicos para evaluar modelos proteicos desconocidos. [53] [54]
La secuencia lineal de aminoácidos de una proteína se denomina estructura primaria . La estructura primaria se puede determinar fácilmente a partir de la secuencia de codones del gen de ADN que la codifica. En la mayoría de las proteínas, la estructura primaria determina de forma única la estructura tridimensional de una proteína en su entorno nativo. Una excepción es la proteína mal plegada implicada en la encefalopatía espongiforme bovina . Esta estructura está vinculada a la función de la proteína. La información estructural adicional incluye la estructura secundaria , terciaria y cuaternaria . Una solución general viable para la predicción de la función de una proteína sigue siendo un problema abierto. La mayoría de los esfuerzos hasta ahora se han dirigido hacia heurísticas que funcionan la mayor parte del tiempo. [ cita requerida ]
En la rama genómica de la bioinformática, la homología se utiliza para predecir la función de un gen: si la secuencia del gen A , cuya función se conoce, es homóloga a la secuencia del gen B, cuya función se desconoce, se podría inferir que B puede compartir la función de A. En la bioinformática estructural, la homología se utiliza para determinar qué partes de una proteína son importantes en la formación de la estructura y la interacción con otras proteínas. El modelado de homología se utiliza para predecir la estructura de una proteína desconocida a partir de proteínas homólogas existentes.
Un ejemplo de esto es la hemoglobina en humanos y la hemoglobina en legumbres ( leghemoglobina ), que son parientes lejanos de la misma superfamilia de proteínas . Ambas cumplen la misma función de transportar oxígeno en el organismo. Aunque ambas proteínas tienen secuencias de aminoácidos completamente diferentes, sus estructuras proteínicas son prácticamente idénticas, lo que refleja sus funciones casi idénticas y su ancestro compartido. [55]
Otras técnicas para predecir la estructura de las proteínas incluyen el enhebrado de proteínas y el modelado basado en la física de novo (desde cero).
Otro aspecto de la bioinformática estructural incluye el uso de estructuras proteínicas para modelos de detección virtual , como los modelos de relación cuantitativa estructura-actividad y los modelos proteoquimiométricos (PCM). Además, la estructura cristalina de una proteína se puede utilizar en la simulación, por ejemplo, de estudios de unión de ligandos y estudios de mutagénesis in silico .
Un software basado en algoritmos de aprendizaje profundo de 2021 llamado AlphaFold , desarrollado por DeepMind de Google , supera ampliamente a todos los demás métodos de software de predicción [56] [ ¿cómo? ] y ha publicado estructuras predichas para cientos de millones de proteínas en la base de datos de estructura de proteínas AlphaFold. [57]
El análisis de redes busca comprender las relaciones dentro de las redes biológicas, como las redes metabólicas o de interacción proteína-proteína . Si bien las redes biológicas pueden construirse a partir de un solo tipo de molécula o entidad (como los genes), la biología de redes a menudo intenta integrar muchos tipos de datos diferentes, como proteínas, moléculas pequeñas, datos de expresión genética y otros, que están todos conectados física, funcionalmente o de ambas maneras.
La biología de sistemas implica el uso de simulaciones por computadora de subsistemas celulares (como las redes de metabolitos y enzimas que comprenden el metabolismo , las vías de transducción de señales y las redes de regulación genética ) para analizar y visualizar las conexiones complejas de estos procesos celulares. La vida artificial o evolución virtual intenta comprender los procesos evolutivos a través de la simulación por computadora de formas de vida simples (artificiales).

Se han determinado decenas de miles de estructuras proteínicas tridimensionales mediante cristalografía de rayos X y espectroscopia de resonancia magnética nuclear de proteínas (RMN de proteínas), y una cuestión central en bioinformática estructural es si es práctico predecir posibles interacciones proteína-proteína basándose únicamente en estas formas 3D, sin realizar experimentos de interacción proteína-proteína . Se han desarrollado diversos métodos para abordar el problema del acoplamiento proteína-proteína , aunque parece que todavía queda mucho trabajo por hacer en este campo.
Otras interacciones que se encuentran en este campo incluyen las interacciones proteína-ligando (incluido el fármaco) y proteína-péptido. La simulación dinámica molecular del movimiento de los átomos alrededor de enlaces rotatorios es el principio fundamental detrás de los algoritmos computacionales , denominados algoritmos de acoplamiento, para estudiar las interacciones moleculares .
La informática de la biodiversidad se ocupa de la recopilación y el análisis de datos sobre biodiversidad , como bases de datos taxonómicas o datos sobre el microbioma . Entre los ejemplos de estos análisis se incluyen la filogenética , el modelado de nichos , el mapeo de la riqueza de especies , los códigos de barras de ADN o las herramientas de identificación de especies . Un área en crecimiento también es la macroecología , es decir, el estudio de cómo la biodiversidad está conectada con la ecología y el impacto humano, como el cambio climático .
La enorme cantidad de literatura publicada hace que sea prácticamente imposible que las personas lean todos los artículos, lo que da lugar a subcampos de investigación desarticulados. El análisis de la literatura tiene como objetivo emplear la lingüística computacional y estadística para explotar esta creciente biblioteca de recursos textuales. Por ejemplo:
El área de investigación se nutre de la estadística y la lingüística computacional .
Las tecnologías computacionales se utilizan para automatizar el procesamiento, la cuantificación y el análisis de grandes cantidades de imágenes biomédicas con un alto contenido de información . Los sistemas de análisis de imágenes modernos pueden mejorar la precisión , la objetividad o la velocidad del observador . El análisis de imágenes es importante tanto para el diagnóstico como para la investigación. Algunos ejemplos son:
Las técnicas computacionales se utilizan para analizar datos de células individuales de alto rendimiento y baja medición, como los obtenidos mediante citometría de flujo . Estos métodos generalmente implican la búsqueda de poblaciones de células que sean relevantes para un estado patológico o una condición experimental en particular.
Las ontologías biológicas son grafos acíclicos dirigidos de vocabularios controlados . Crean categorías para conceptos y descripciones biológicas de modo que puedan analizarse fácilmente con computadoras. Cuando se categorizan de esta manera, es posible obtener un valor agregado a partir de un análisis holístico e integrado. [ cita requerida ]
La OBO Foundry fue un esfuerzo por estandarizar ciertas ontologías. Una de las más difundidas es la ontología génica , que describe la función de los genes. También existen ontologías que describen fenotipos.
Las bases de datos son esenciales para la investigación y las aplicaciones de la bioinformática. Existen bases de datos para muchos tipos de información diferentes, incluidas secuencias de ADN y proteínas, estructuras moleculares, fenotipos y biodiversidad. Las bases de datos pueden contener tanto datos empíricos (obtenidos directamente de experimentos) como datos previstos (obtenidos del análisis de datos existentes). Pueden ser específicos de un organismo, una vía o una molécula de interés en particular. Alternativamente, pueden incorporar datos compilados de varias otras bases de datos. Las bases de datos pueden tener diferentes formatos, mecanismos de acceso y ser públicas o privadas.
A continuación se enumeran algunas de las bases de datos más utilizadas:
Las herramientas de software para bioinformática incluyen herramientas de línea de comandos sencillas, programas gráficos más complejos y servicios web independientes. Son creadas por empresas de bioinformática o instituciones públicas.
Desde la década de 1980 han existido y seguido creciendo muchas herramientas de software libre y de código abierto . [59] La combinación de una necesidad continua de nuevos algoritmos para el análisis de tipos emergentes de lecturas biológicas, el potencial para experimentos in silico innovadores y bases de código abierto disponibles libremente han creado oportunidades para que los grupos de investigación contribuyan tanto a la bioinformática como a la bioinformática, independientemente de la financiación . Las herramientas de código abierto a menudo actúan como incubadoras de ideas o complementos respaldados por la comunidad en aplicaciones comerciales. También pueden proporcionar estándares de facto y modelos de objetos compartidos para ayudar con el desafío de la integración de la bioinformación.
El software de bioinformática de código abierto incluye Bioconductor , BioPerl , Biopython , BioJava , BioJS , BioRuby , Bioclipse , EMBOSS , .NET Bio, Orange con su complemento de bioinformática, Apache Taverna , UGENE y GenoCAD .
La fundación sin fines de lucro Open Bioinformatics Foundation [59] y la conferencia anual Bioinformatics Open Source Conference promueven el software bioinformático de código abierto. [60]
Se han desarrollado interfaces basadas en SOAP y REST para permitir que los equipos cliente utilicen algoritmos, datos y recursos informáticos de servidores ubicados en otras partes del mundo. La principal ventaja es que los usuarios finales no tienen que lidiar con gastos generales de mantenimiento de software y bases de datos.
Los servicios básicos de bioinformática están clasificados por el EBI en tres categorías: SSS (servicios de búsqueda de secuencias), MSA (alineación de secuencias múltiples) y BSA (análisis de secuencias biológicas). [61] La disponibilidad de estos recursos bioinformáticos orientados a servicios demuestra la aplicabilidad de las soluciones bioinformáticas basadas en la web, y varían desde una colección de herramientas independientes con un formato de datos común bajo una única interfaz basada en la web, hasta sistemas de gestión de flujo de trabajo bioinformático integradores, distribuidos y extensibles .
Un sistema de gestión de flujo de trabajo de bioinformática es una forma especializada de un sistema de gestión de flujo de trabajo diseñado específicamente para componer y ejecutar una serie de pasos computacionales o de manipulación de datos, o un flujo de trabajo, en una aplicación de bioinformática. Dichos sistemas están diseñados para
Algunas de las plataformas que ofrecen este servicio: Galaxy , Kepler , Taverna , UGENE , Anduril , HIVE .
En 2014, la Administración de Alimentos y Medicamentos de los Estados Unidos patrocinó una conferencia celebrada en el campus Bethesda de los Institutos Nacionales de Salud para discutir la reproducibilidad en bioinformática. [62] Durante los siguientes tres años, un consorcio de partes interesadas se reunió regularmente para discutir lo que se convertiría en el paradigma BioCompute. [63] Estas partes interesadas incluyeron representantes del gobierno, la industria y entidades académicas. Los líderes de la sesión representaron numerosas ramas de la FDA y los institutos y centros del NIH, entidades sin fines de lucro, incluido el Proyecto Varioma Humano y la Federación Europea de Informática Médica , e instituciones de investigación como Stanford , el Centro del Genoma de Nueva York y la Universidad George Washington .
Se decidió que el paradigma BioCompute adoptaría la forma de "cuadernos de laboratorio" digitales que permiten la reproducibilidad, replicación, revisión y reutilización de los protocolos bioinformáticos. Se propuso esto para permitir una mayor continuidad dentro de un grupo de investigación a lo largo del flujo normal de personal, al tiempo que se fomentaba el intercambio de ideas entre grupos. La FDA de los EE. UU. financió este trabajo para que la información sobre los proyectos fuera más transparente y accesible para su personal regulador. [64]
En 2016, el grupo se volvió a reunir en el NIH en Bethesda y discutió el potencial de un objeto BioCompute , una instancia del paradigma BioCompute. Este trabajo se copió como un documento de "uso de prueba estándar" y un artículo preimpreso cargado en bioRxiv. El objeto BioCompute permite que el registro JSON se comparta entre empleados, colaboradores y reguladores. [65] [66]
La bioinformática no solo se enseña en forma de maestrías presenciales en muchas universidades. La naturaleza computacional de la bioinformática la lleva al aprendizaje asistido por computadora y en línea . [67] [68] Las plataformas de software diseñadas para enseñar conceptos y métodos de bioinformática incluyen Rosalind y cursos en línea ofrecidos a través del Portal de capacitación del Instituto Suizo de Bioinformática . Los Talleres de Bioinformática Canadiense proporcionan videos y diapositivas de talleres de capacitación en su sitio web bajo una licencia Creative Commons . El proyecto 4273π o proyecto 4273pi [69] también ofrece materiales educativos de código abierto de forma gratuita. El curso se ejecuta en computadoras Raspberry Pi de bajo costo y se ha utilizado para enseñar a adultos y alumnos de escuelas. [70] [71] 4273 es desarrollado activamente por un consorcio de académicos y personal de investigación que han realizado bioinformática de nivel de investigación utilizando computadoras Raspberry Pi y el sistema operativo 4273π. [72] [73]
Las plataformas MOOC también ofrecen certificaciones en línea en bioinformática y disciplinas relacionadas, incluida la Especialización en Bioinformática de Coursera en la Universidad de California en San Diego , la Especialización en Ciencia de Datos Genómicos de la Universidad Johns Hopkins y la XSeries de Análisis de Datos para Ciencias de la Vida de EdX en la Universidad de Harvard .
Existen varios congresos importantes que se ocupan de la bioinformática. Algunos de los ejemplos más destacados son Intelligent Systems for Molecular Biology (ISMB), European Conference on Computational Biology (ECCB) y Research in Computational Molecular Biology (RECOMB).