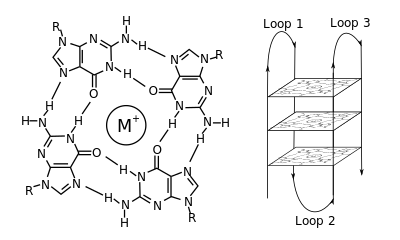
En biología molecular , las estructuras secundarias G-quadruplex (G4) se forman en ácidos nucleicos por secuencias ricas en guanina . [2] Tienen forma helicoidal y contienen tétradas de guanina que pueden formarse a partir de una, [3] dos [4] o cuatro hebras. [5] Las formas unimoleculares a menudo ocurren naturalmente cerca de los extremos de los cromosomas, mejor conocidos como regiones teloméricas , y en regiones reguladoras de la transcripción de múltiples genes, tanto en microbios [6] [7] como en vertebrados [8] [7] incluidos los oncogenes en humanos. [9] Cuatro bases de guanina pueden asociarse a través de enlaces de hidrógeno de Hoogsteen para formar una estructura plana cuadrada llamada tétrada de guanina (G-tetrad o G-quartet), y dos o más tétradas de guanina (de G-tracts, corridas continuas de guanina) pueden apilarse una sobre otra para formar un G-quadruplex.
La colocación y unión para formar G-quadruplexes no es aleatoria y sirve para propósitos funcionales muy inusuales. La estructura quadruplex se estabiliza aún más por la presencia de un catión , especialmente potasio , que se encuentra en un canal central entre cada par de tétradas. [3] Pueden estar formados por ADN , ARN , LNA y PNA , y pueden ser intramoleculares , bimoleculares o tetramoleculares. [10] Dependiendo de la dirección de las hebras o partes de una hebra que forman las tétradas, las estructuras pueden describirse como paralelas o antiparalelas . Las estructuras G-quadruplex pueden predecirse computacionalmente a partir de motivos de secuencia de ADN o ARN, [11] [12] pero sus estructuras reales pueden variar bastante dentro y entre los motivos, que pueden sumar más de 100.000 por genoma. Sus actividades en procesos genéticos básicos son un área activa de investigación en la investigación de telómeros, regulación genética y genómica funcional. [13] [14]
La identificación de estructuras con una alta asociación de guanina se hizo evidente a principios de la década de 1960, a través de la identificación de sustancias similares a geles asociadas con guaninas. [15] Más específicamente, esta investigación detalló las estructuras de ADN de cuatro cadenas con una alta asociación de guaninas, que luego se identificaron en regiones teloméricas eucariotas del ADN en la década de 1980. [16] La importancia de descubrir la estructura del G-cuadrúplex se describió a través de la declaración: "Si los G-cuadrúplex se forman tan fácilmente in vitro , la naturaleza habrá encontrado una manera de usarlos in vivo " - Aaron Klug , Premio Nobel de Química (1982). El interés en la función in vivo de los G-quadruplexes surgió después de que un análisis a gran escala del genoma completo mostrara la prevalencia de secuencias formadoras potenciales de G-quadruplex (pG4) dentro de los promotores de genes de humanos, chimpancés, ratones y ratas, presentado en la Primera Reunión Internacional de G-quadruplex celebrada en abril de 2007 en Louisville, Kentucky. [7] En 2006, se informó de la prevalencia de G-quadruplexes dentro de los promotores de genes de varios genomas bacterianos, lo que predijo la regulación génica mediada por G-quadruplex. [6] Con la abundancia de G-quadruplexes in vivo , estas estructuras desempeñan un papel biológicamente relevante a través de interacciones con las regiones promotoras de oncogenes y las regiones teloméricas de las cadenas de ADN. La investigación actual consiste en identificar la función biológica de estas estructuras G-quadruplex para oncogenes específicos y descubrir tratamientos terapéuticos eficaces para el cáncer basados en interacciones con G-quadruplexes. La evidencia temprana de la formación de G-cuadruplex in vivo en células se estableció al aislarlos de las células, [17] y más tarde mediante la observación de que se podían identificar helicasas de ADN específicas donde pequeñas moléculas específicas para estas estructuras de ADN se acumulaban en las células. [18]
La longitud de las secuencias de ácidos nucleicos implicadas en la formación de tétradas determina cómo se pliega el cuádruplex. Las secuencias cortas, que consisten en una sola serie contigua de tres o más bases de guanina, requieren cuatro hebras individuales para formar un cuádruplex. Un cuádruplex de este tipo se describe como tetramolecular, lo que refleja el requisito de cuatro hebras separadas. El término ADN G4 se reservó originalmente para estas estructuras tetramoleculares que podrían desempeñar un papel en la meiosis . [5] Sin embargo, como se usa actualmente en biología molecular, el término G4 puede significar G-cuádruplex de cualquier molecularidad. Las secuencias más largas, que contienen dos series contiguas de tres o más bases de guanina, donde las regiones de guanina están separadas por una o más bases, solo requieren dos de estas secuencias para proporcionar suficientes bases de guanina para formar un cuádruplex. Estas estructuras, formadas a partir de dos hebras separadas ricas en G, se denominan cuádruplex bimoleculares. Finalmente, las secuencias que contienen cuatro cadenas distintas de bases de guanina pueden formar estructuras cuádruplex estables por sí mismas, y un cuádruplex formado enteramente a partir de una sola cadena se denomina cuádruplex intramolecular. [19]
Dependiendo de cómo se organizan las series individuales de bases de guanina en un cuádruplex bimolecular o intramolecular, un cuádruplex puede adoptar una de varias topologías con configuraciones de bucle variables. [20] Si todas las hebras de ADN proceden en la misma dirección, el cuádruplex se denomina paralelo. Para los cuádruplex intramoleculares, esto significa que cualquier región de bucle presente debe ser del tipo hélice, posicionada a los lados del cuádruplex. Si una o más de las series de bases de guanina tiene una dirección 5'-3' opuesta a las otras series de bases de guanina, se dice que el cuádruplex ha adoptado una topología antiparalela. Los bucles que unen series de bases de guanina en cuádruplex antiparalelos intramoleculares son diagonales, uniendo dos series diagonalmente opuestas de bases de guanina, o bucles de tipo lateral (en el borde), uniendo dos series adyacentes de pares de bases de guanina.
En los cuádruplex formados a partir de ADN bicatenario, también se han discutido posibles topologías entre cadenas [21] . [22] Los cuádruplex entre cadenas contienen guaninas que se originan en ambas cadenas de dsADN.
Tras la secuenciación del genoma humano , se descubrieron muchas secuencias ricas en guanina que tenían el potencial de formar cuádruplexes. [23] Dependiendo del tipo de célula y del ciclo celular, factores mediadores como las proteínas de unión al ADN en la cromatina , compuesta de ADN fuertemente enrollado alrededor de las proteínas histonas , y otras condiciones ambientales y tensiones afectan la formación dinámica de cuádruplexes. Por ejemplo, las evaluaciones cuantitativas de la termodinámica del hacinamiento molecular indican que el g-cuádruplex antiparalelo se estabiliza por el hacinamiento molecular. [24] Este efecto parece estar mediado por la alteración de la hidratación del ADN y su efecto en la unión de pares de bases de Hoogsteen . [25] Estos cuádruplexes parecían ocurrir fácilmente en los extremos del cromosoma . Además, la propensión a la formación de g-cuádruplex durante la transcripción en secuencias de ARN con el potencial de formar estructuras de horquilla o G-cuádruplex mutuamente excluyentes depende en gran medida de la posición de la secuencia formadora de horquilla. [26]
Dado que las enzimas reparadoras reconocerían de forma natural los extremos de los cromosomas lineales como ADN dañado y los procesarían como tales, lo que tendría efectos nocivos para la célula, se necesita una señalización clara y una regulación estricta en los extremos de los cromosomas lineales. Los telómeros funcionan para proporcionar esta señalización. Los telómeros, ricos en guanina y con una propensión a formar g-quadruplex, están ubicados en los extremos terminales de los cromosomas y ayudan a mantener la integridad del genoma al proteger estos extremos terminales vulnerables de la inestabilidad.
Estas regiones teloméricas se caracterizan por largas regiones de repeticiones CCCTAA:TTAGGG de doble cadena. Las repeticiones terminan con una protuberancia en el extremo 3' de entre 10 y 50 repeticiones TTAGGG de cadena sencilla. La enzima telomerasa, un complejo heterodímero de ribonucleoproteínas, añade repeticiones TTAGGG en el extremo 3' de las cadenas de ADN. En estas protuberancias del extremo 3', el saliente rico en G puede formar estructuras secundarias como los G-quadruplex si el saliente es más largo que cuatro repeticiones TTAGGG. La presencia de estas estructuras impide la elongación de los telómeros por el complejo de telomerasa. [27]
Se ha demostrado que las repeticiones teloméricas en una variedad de organismos forman estas estructuras cuádruplex in vitro , y posteriormente también se ha demostrado que se forman in vivo . [28] [29] La repetición telomérica humana (que es la misma para todos los vertebrados ) consta de muchas repeticiones de la secuencia (TTAGGG), y los cuádruplex formados por esta estructura pueden tener estructuras similares a perlas de 5 nm a 8 nm de tamaño y han sido bien estudiados por RMN , TEM y determinación de la estructura cristalina de rayos X. [30] Se ha demostrado que la formación de estos cuádruplex en los telómeros disminuye la actividad de la enzima telomerasa , que es responsable de mantener la longitud de los telómeros y está involucrada en alrededor del 85% de todos los cánceres . Este es un objetivo activo del descubrimiento de fármacos, incluida la telomestatina .
Los cuadrúplex están presentes en lugares distintos del telómero . El análisis de genomas humanos, de chimpancés, de ratones y de ratas mostró una enorme cantidad de secuencias formadoras potenciales de G-cuadrúplex (pG4) en regiones no teloméricas. Una gran cantidad de los G-cuadrúplex no teloméricos se encontraron dentro de los promotores de genes y se conservaron en todas las especies. [6] [7] De manera similar, se encontró una gran cantidad de G-cuadrúplex en E. coli y cientos de otros genomas microbianos. Aquí también, como en los vertebrados, los G-cuadrúplex se enriquecieron dentro de los promotores de genes. [6] Además, se encontró un locus G-cuadrúplex conservado durante más de mil millones de años en plantas y algas, en el gen que codifica la subunidad grande de la ARN polimerasa II. [31] Aunque estos estudios predijeron la regulación genética mediada por G-cuadrúplex, es poco probable que todos los pG4 se formaran in vivo. El protooncogen c-myc forma un cuádruplex en una región hipersensible a la nucleasa crítica para la actividad genética. [32] [33] Otros genes que han demostrado formar G-cuádruplex en sus regiones promotoras incluyen el gen de la β-globina de pollo , la ubiquitina -ligasa humana RFP2 y los protooncogenes c-kit , bcl-2 , VEGF , H-ras y N-ras . [34] [35] [36]
Se han realizado estudios de todo el genoma basados en una regla de plegamiento cuádruplex, que han identificado 376.000 supuestas secuencias cuádruplex (PQS) en el genoma humano , aunque probablemente no todas se formen in vivo . [37] Estudios similares han identificado supuestos G-quadruplexes en procariotas , concretamente la bacteria E. coli . [38] Hay varios modelos posibles de cómo los cuádruplex podrían influir en la actividad genética, ya sea por regulación positiva o negativa . A continuación se muestra un modelo, con la formación de G-quadruplex en o cerca de un promotor que bloquea la transcripción del gen y, por lo tanto, lo desactiva. En otro modelo, el cuádruplex formado en la cadena de ADN no codificante ayuda a mantener una conformación abierta de la cadena de ADN codificante y mejora la expresión del gen respectivo.
Se ha sugerido que la formación de cuádruplex desempeña un papel en el cambio de cadena pesada de inmunoglobulina . [5] A medida que las células han desarrollado mecanismos para resolver (es decir, desenrollar) los cuádruplex que se forman, la formación de cuádruplex puede ser potencialmente dañina para una célula; las helicasas WRN y la proteína del síndrome de Bloom tienen una alta afinidad para resolver los G-cuádruplex de ADN. [39] La helicasa DEAH/RHA, DHX36 , también se ha identificado como una resolvasa G-cuádruplex clave. [40] [41] En 2009, se descubrió que una proteína supresora de metástasis NM23H2 (también conocida como NME2) interactuaba directamente con el G-cuádruplex en el promotor del gen c-myc y regulaba transcripcionalmente c-myc. [42] [43] Más recientemente, se informó que NM23H2 interactúa con G-quadruplex en el promotor del gen de la telomerasa humana (hTERT) y regula la expresión de hTERT [44] En 2019, se demostró que el factor de unión a telómeros 2 (TRF2 o TERF2) se une a miles de G-quadruplex no teloméricos en el genoma humano mediante TRF2 ChIP-seq. [45] Hay muchos estudios que implican a los cuadrúplex en la regulación transcripcional tanto positiva como negativa, incluida la regulación epigenética de genes como hTERT. [44] También se ha informado que la función de los G-quadruplex permite la recombinación programada de genes pesados de inmunoglobulina y el sistema de variación antigénica de pilina de la Neisseria patógena . [46] Los roles de la estructura del cuadrúplex en el control de la traducción no están tan bien explorados. La visualización directa de las estructuras de G-quadruplex en células humanas [47], así como la estructura de cocristal de una helicasa de ARN unida a un G-quadruplex [48], han proporcionado importantes confirmaciones de su relevancia para la biología celular. Los posibles papeles positivos y negativos de los cuádruplex en la replicación y función de los telómeros siguen siendo controvertidos. Los T-loops y los G-quadruplex se describen como las dos estructuras terciarias del ADN que protegen los extremos de los telómeros y regulan su longitud. [49]
Muchos de los procesos reguladores del genoma se han vinculado a la formación de estructuras G-quadruplex, atribuibles al enorme papel que desempeña en la reparación del ADN de los sitios apurínicos/apirimidínicos también conocidos como sitios AP. [50] Se ha desarrollado una nueva técnica para mapear los sitios AP conocida como AP-seq que utiliza una sonda reactiva a aldehído (ARP) marcada con biotina para marcar ciertas regiones del genoma donde la ocurrencia de daño del sitio AP ha sido significativa. [51] Otro método de secuenciación de mapeo de todo el genoma conocido como secuenciación ChIP , se utilizó para mapear tanto el daño en los sitios AP como la enzima responsable de su reparación, la endonucleasa AP 1 (APE1). Ambos métodos de secuenciación de mapeo de todo el genoma, la secuenciación ChIP y la ARP, han indicado que la ocurrencia de daño en el sitio AP no es aleatoria. El daño del sitio AP también fue más frecuente en ciertas regiones del genoma que contienen marcadores promotores y potenciadores activos específicos, algunos de los cuales estaban vinculados a regiones responsables del adenocarcinoma de pulmón y el cáncer de colon. [52] Se encontró que el daño del sitio AP era predominante en las regiones PQS del genoma, donde la formación de estructuras G-quadruplex está regulada y promovida por el proceso de reparación del ADN, reparación por escisión de bases (BER). [52] Se ha demostrado que los procesos de reparación por escisión de bases en las células se reducen con el envejecimiento a medida que sus componentes en las mitocondrias comienzan a declinar, lo que puede conducir a la formación de muchas enfermedades como la enfermedad de Alzheimer (EA). [53] Se dice que estas estructuras G-quadruplex se forman en las regiones promotoras del ADN a través de la superhelicidad, que favorece el desenrollado de la estructura de doble hélice del ADN y, a su vez, enrolla las hebras para formar estructuras G-quadruplex en regiones ricas en guanina. [54] La vía BER se señala cuando indica un daño oxidativo en la base del ADN, donde estructuras como la 8-oxoguanina-ADN glicosilasa 1 (OGG1), APE1 y G-quadruplex juegan un papel enorme en su reparación. Estas enzimas participan en la BER para reparar ciertas lesiones del ADN como la 7,8-dihidro-8-oxoguanina (8-oxoG), que se forma bajo estrés oxidativo a bases de guanina. [55]
Las bases de guanina (G) en G-quadruplex tienen el potencial redox más bajo, lo que hace que sea más susceptible a la formación de 8-oxoguanina (8-oxoG), un daño endógeno de la base del ADN oxidado en el genoma. Debido a que la guanina tiene un potencial de reducción de electrones más bajo que las otras bases de nucleótidos, [56] 8-oxo-2'-desoxiguanosina (8-oxo-dG), es un producto principal conocido de la oxidación del ADN. Su concentración se utiliza como una medida del estrés oxidativo dentro de una célula. [57] Cuando el ADN sufre daño oxidativo, un posible cambio estructural en la guanina, después de la radiación ionizante, da lugar a una forma enólica, 8-OH-Gua. Este producto oxidativo se forma a través de un cambio tautomérico de la guanina dañada original, 8-oxo-Gua, y representa un daño del ADN que causa cambios en la estructura. Esta forma permite que la enzima de reparación por escisión de bases (BER) OGG1 se una y elimine el daño oxidativo con la ayuda de APE1, lo que da como resultado un sitio AP. [55] [53] Además, un sitio AP es una ubicación en el ADN que no tiene una base de purina o pirimidina debido al daño del ADN, son el tipo más frecuente de daño endógeno del ADN en las células. Los sitios AP se pueden generar espontáneamente o después de la escisión de bases modificadas, como 8-OH-Gua. [51] La generación de un sitio AP permite la fusión del ADN dúplex para desenmascarar el PQS, adoptando un [53] pliegue G-cuadrúplex. Con el uso de análisis de secuenciación ChIP de todo el genoma , ensayos basados en células y análisis bioquímicos in vitro, se ha establecido una conexión entre los sitios AP derivados de bases de ADN oxidadas y la formación del G-cuadrúplex. [52]
Contribución de la oxidación del ADN a las enfermedades
Además, la concentración de 8-oxo-dG es un biomarcador conocido de estrés oxidativo dentro de una célula, y una cantidad excesiva de estrés oxidativo se ha relacionado con la carcinogénesis y otras enfermedades. [58] Cuando se produce, 8-oxo-dG, tiene la capacidad de inactivar OGG1, evitando así la reparación del daño del ADN causado por la oxidación de guanina. [52] La posible inactivación permite que los daños del ADN no reparados se acumulen en células no replicantes, como el músculo, y también pueden causar envejecimiento. [57] Además, el daño oxidativo del ADN como 8-oxo-dG contribuye a la carcinogénesis a través de la modulación de la expresión genética o la inducción de mutaciones. [57] Con la condición de que 8-oxo-dG sea reparado por BER, partes de la proteína reparadora se quedan atrás, lo que puede conducir a alteraciones epigenéticas o la modulación de la expresión genética. [59] Tras la inserción de 8-oxo-dG en el gen de la timidina quinasa de humanos, se determinó que si el 8-oxo-dG no se controla y no se repara mediante BER, puede provocar mutaciones frecuentes y, eventualmente, carcinogénesis. [52] [53]
La endonucleasa AP 1 (APE1) es una enzima responsable de la promoción y la formación de estructuras G-quadruplex. APE1 se encarga principalmente de reparar el daño causado a los sitios AP a través de la vía BER. APE1 se considera muy crucial ya que se sabe que el daño al sitio AP es el tipo más recurrente de daño endógeno al ADN. [59] La oxidación de ciertas bases de purina, como la guanina, forma nucleótidos oxidados que perjudican la función del ADN al desemparejar los nucleótidos en las secuencias. [57] Esto es más común en secuencias PQS que forman estructuras oxidadas, como la 8-oxoguanina . Una vez que la célula es consciente del estrés oxidativo y el daño, recluta a OGG1 al sitio, cuya función principal es iniciar la vía BER. [52] OGG1 lo hace escindiendo la base oxidada y creando así un sitio AP, principalmente a través del proceso de superhelicidad negativa. [54] Este sitio AP luego envía señales a las células para que activen la unión de APE1, que se une a la región dúplex abierta. [58] La unión de APE1 luego juega un papel importante al estabilizar la formación de estructuras G-quadruplex en esa región. Esto promueve la formación de estructuras G-quadruplex por el plegamiento del soporte. [60] Este proceso de bucle trae cuatro bases en estrecha proximidad que se mantendrán juntas por el apareamiento de bases de Hoogsteen. Después de esta etapa, el APE1 se acetila por múltiples residuos de lisina en la cromatina, formando APE1 acetilado (AcAPE1). [60] AcAPE1 es muy crucial para la vía BER, ya que actúa como un coactivador o correpresor transcripcional, que funciona para cargar factores de transcripción (TF) en el sitio del daño, lo que le permite regular la expresión génica. [61] AcAPE1 también es muy importante ya que permite que APE1 se una durante períodos de tiempo más largos al retrasar su disociación de la secuencia, lo que permite que el proceso de reparación sea más eficiente. [62] La desacetilación de AcAPE1 es la fuerza impulsora detrás de la carga de estos TF, donde APE1 se disocia de las estructuras G-quadruplex. [63] Cuando un estudio reguló negativamente la presencia de APE1 y AcAPE1 en la célula, se inhibió la formación de estructuras G-quadruplex, lo que demuestra la importancia de APE1 para la formación de estas estructuras. Sin embargo, no todas las estructuras G-quadruplex requieren APE1 para su formación, de hecho algunas de ellas formaron estructuras G-quadruplex mayores en su ausencia. [52] Por lo tanto, podemos concluir que APE1 tiene dos funciones importantes en la regulación del genoma: estabilizar la formación de estructuras g-quadruplex y cargar los factores de transcripción en el sitio AP.
Las secuencias formadoras de G-cuadruplex son frecuentes en las células eucariotas , especialmente en los telómeros, las cadenas 5` no traducidas y los puntos calientes de translocación. Los G-cuadruplex pueden inhibir la función celular normal y, en las células sanas, la helicasa los desenrolla fácilmente . Sin embargo, en las células cancerosas que tienen la helicasa mutada, estos complejos no se pueden desenrollar y provocan un daño potencial a la célula. Esto provoca la replicación de células dañadas y cancerosas. Para los avances terapéuticos, la estabilización de los G-cuadruplex de las células cancerosas puede inhibir el crecimiento y la replicación celular, lo que conduce a la muerte de la célula . [64]
Junto con la asociación de G-quadruplexes en regiones teloméricas del ADN, se han identificado estructuras G-quadruplex en varias regiones promotoras de protooncogenes humanos. Las estructuras más presentes en las regiones promotoras de estos oncogenes tienden a ser estructuras de ADN G-quadruplex de cadena paralela. [65] Algunos de estos oncogenes incluyen c-KIT, PDGF-A, c-Myc y VEGF, lo que demuestra la importancia de esta estructura secundaria en el crecimiento y desarrollo del cáncer. Si bien la formación de la estructura G-quadruplex varía en cierta medida para las diferentes regiones promotoras de los oncogenes, se ha encontrado la estabilización constante de estas estructuras en el desarrollo del cáncer. [66] La investigación terapéutica actual se centra activamente en apuntar a esta estabilización de las estructuras G-quadruplex para detener el crecimiento y la división celular descontrolados.
Una región particular del gen, la vía c-myc, desempeña un papel integral en la regulación de un producto proteico, c-Myc. Con este producto, la proteína c-Myc funciona en los procesos de apoptosis y crecimiento o desarrollo celular y como un control transcripcional en la transcriptasa inversa de la telomerasa humana . [67] Se demostró que la interacción del promotor c-Myc G-quadruplex con NM23H2 regula c-Myc en células cancerosas en 2009 [42].
La regulación de c-myc a través de la transcriptasa inversa de la telomerasa humana (hTERT) también está regulada directamente a través del promotor G-quadruplex por interacción con el factor de transcripción NM23H2, donde las modificaciones epigenéticas dependían de la asociación NM23H2-G-quadruplex. [44] Recientemente, se informó que la regulación epigenética de hTERT está mediada por la interacción del promotor G-quadruplex de hTERT con el factor telomérico TRF2. [68]
Otra vía genética se relaciona con el gen VEGF, el factor de crecimiento endotelial vascular, que sigue estando implicado en el proceso de angiogénesis o la formación de nuevos vasos sanguíneos. La formación de una estructura G-quadruplex intramolecular se ha demostrado mediante estudios sobre el tracto de polipurina de la región promotora del gen VEGF. A través de investigaciones recientes sobre el papel de la función G-quadruplex in vivo, se ha demostrado que la estabilización de las estructuras G-quadruplex regula la transcripción del gen VEGF, con inhibición de los factores de transcripción en esta vía. Las estructuras G-quadruplex intramoleculares se forman principalmente a través de la abundante secuencia de guanina en la región promotora de esta vía específica. [69] El gen CDKN1A (también conocido como p21) del inhibidor de la quinasa del punto de control del ciclo celular dependiente de ciclina alberga el promotor G-quadruplex. La interacción de este G-cuadrúplex con TRF2 (también conocido como TERF2) resultó en una regulación epigenética de p21, que se probó utilizando el ligando de unión al G-cuadrúplex 360A. [70]
El factor inducible por hipoxia 1ɑ, HIF-1ɑ, sigue involucrado en la señalización del cáncer a través de su unión al elemento de respuesta a la hipoxia, HRE, en presencia de hipoxia para iniciar el proceso de angiogénesis . A través de investigaciones recientes sobre esta vía genética específica, la región de polipurina y polipirimidina permite la transcripción de este gen específico y la formación de una estructura G-quadruplex intramolecular. Sin embargo, se necesita más investigación para determinar si la formación de G-quadruplex regula la expresión de este gen de manera positiva o negativa. [71]
El oncogén c-kit se relaciona con una vía que codifica una RTK, que se ha demostrado que tiene niveles elevados de expresión en ciertos tipos de cáncer. La rica secuencia de guanina de esta región promotora ha demostrado la capacidad de formar una variedad de cuádruplex. La investigación actual sobre esta vía se centra en descubrir la función biológica de esta formación de cuádruplex específica en la vía c-kit, mientras que esta secuencia de cuádruplex se ha observado en varias especies. [36]
El oncogén RET funciona en la transcripción de la quinasa , que ha sido abundante en ciertos tipos de cáncer. La secuencia rica en guanina en la región promotora de esta vía exuda una necesidad para la transcripción de base de este receptor de tirosina quinasa. En ciertos tipos de cáncer, la proteína RET ha mostrado niveles aumentados de expresión. La investigación sobre esta vía sugirió la formación de un G-quadruplex en la región promotora y un objetivo aplicable para tratamientos terapéuticos. [72]
Otra vía oncogénica que involucra al PDGF-A, el factor de crecimiento derivado de plaquetas, involucra el proceso de cicatrización de heridas y funciona como factor de crecimiento mitogénico para las células. Los altos niveles de expresión del PDGF se han asociado con un mayor crecimiento celular y cáncer. La presencia de una secuencia rica en guanina en la región promotora del PDGF-A ha mostrado la capacidad de formar estructuras G-quadruplex paralelas intramoleculares y sigue sugiriendo que desempeña un papel en la regulación transcripcional del PDGF-A. Sin embargo, la investigación también ha identificado la presencia de estructuras G-quadruplex dentro de esta región debido a la interacción de TMPyP4 con esta secuencia promotora. [73]
Los telómeros están generalmente formados por G-quadruplex y siguen siendo objetivos importantes para la investigación y los descubrimientos terapéuticos. Estos complejos tienen una alta afinidad por los anillos de porfirina , lo que los convierte en agentes anticancerígenos eficaces. Sin embargo, el uso de TMPyP4 ha sido limitado debido a su falta de selectividad hacia los telómeros de las células cancerosas y el ADN bicatenario normal (dsADN). Para abordar esta cuestión, se sintetizó un análogo de TMPyP4 conocido como 5Me que se dirige únicamente al ADN G-quadruplex que inhibe el crecimiento del cáncer de forma más eficaz que TMPyP4. [74]
El diseño y desarrollo de ligandos sigue siendo un campo importante de investigación en reactivos terapéuticos debido a la abundancia de G-quadruplexes y sus múltiples diferencias conformacionales. Un tipo de ligando que involucra un derivado de quindolina, SYUIQ-05, utiliza la estabilización de G-quadruplexes en regiones promotoras para inhibir la producción tanto del producto proteico c-Myc como de la transcriptasa inversa de la telomerasa humana (hTERT). Esta vía principal de apuntar a esta región da como resultado la falta de elongación de la telomerasa, lo que lleva a la detención del desarrollo celular. Sigue siendo necesaria una mayor investigación para el descubrimiento de un único gen diana para minimizar la reactividad no deseada con una actividad antitumoral más eficiente. [67]
Una forma de inducir o estabilizar la formación de G-quadruplex es introducir una molécula que pueda unirse a la estructura de G-quadruplex. Una serie de ligandos , que pueden ser tanto moléculas pequeñas como proteínas , pueden unirse al G-quadruplex. Estos ligandos pueden ser naturales o sintéticos. Este se ha convertido en un campo de investigación cada vez más amplio en genética, bioquímica y farmacología.
Se ha demostrado que las porfirinas catiónicas se unen de forma intercalada con los G-cuadruplex, así como con la molécula telomestatina .
La unión de ligandos a los G-quadruplexes es vital para las actividades contra el cáncer porque los G-quadruplexes se encuentran típicamente en puntos calientes de translocación. MM41, un ligando que se une selectivamente a un cuádruplex en el promotor BCL-2 , tiene una forma con un núcleo central y 4 cadenas laterales que se ramifican estéricamente. La forma del ligando es vital porque coincide estrechamente con el cuádruplex que tiene cuartetos apilados y los bucles de ácidos nucleicos que lo mantienen unido. Cuando está unido, el cromóforo central de MM41 está situado en la parte superior del G-cuarteto terminal 3' y las cadenas laterales del ligando se asocian a los bucles del cuádruplex. El cuarteto y el cromóforo están unidos con un enlace π-π mientras que las cadenas laterales y los bucles no están unidos pero están muy cerca. Lo que hace que esta unión sea fuerte es la fluidez en la posición de los bucles para asociarse mejor con las cadenas laterales del ligando. [75]
TMPyP4, una porfirina catiónica, es un ligando de unión a G4 más conocido que ayuda a reprimir c-Myc. La forma en que TMPyP4 se une a G4 es similar a MM41, con el anillo apilándose sobre el cuarteto G externo y las cadenas laterales asociándose a los bucles de G4. [76]
Al diseñar ligandos para que se unan a los G-cuadrúplex, los ligandos tienen una mayor afinidad por los G-cuadrúplex plegados en paralelo. Se ha descubierto que los ligandos con cadenas laterales más pequeñas se unen mejor al cuádruplex porque los ligandos más pequeños tienen una densidad electrónica más concentrada . Además, los enlaces de hidrógeno de los ligandos con cadenas laterales más pequeñas son más cortos y, por lo tanto, más fuertes. Los ligandos con cadenas laterales móviles, aquellos que pueden girar alrededor de su cromóforo central, se asocian más fuertemente a los G-cuadrúplex porque la conformación de los bucles G4 y las cadenas laterales del ligando pueden alinearse. [77]
Identificar y predecir secuencias que tienen la capacidad de formar cuádruplexes es una herramienta importante para comprender mejor su papel. Generalmente, se utiliza una coincidencia de patrones simple para buscar posibles secuencias formadoras de cuádruplex intracatenarios: d(G 3+ N 1-7 G 3+ N 1-7 G 3+ N 1-7 G 3+ ), donde N es cualquier base de nucleótido (incluida la guanina ). [78] Esta regla ha sido ampliamente utilizada en algoritmos en línea . Aunque la regla identifica efectivamente los sitios de formación de G-cuádruplex, también identifica un subconjunto de las repeticiones espejo de homopurina imperfectas capaces de formación de triplex [79] y formación de i-motif de cadena C. [80] Además, estas secuencias también tienen la capacidad de formar estructuras deslizadas y plegadas que son intermediarios implícitos en la formación de estructuras de ADN cuádruplex [4] y triplex [81] . En un estudio [82] se descubrió que el número observado por par de bases (es decir, la frecuencia) de estos motivos ha aumentado rápidamente en los eumetazoos para los que se dispone de secuencias genómicas completas. Esto sugiere que las secuencias pueden estar bajo selección positiva posibilitada por la evolución de sistemas capaces de suprimir la formación de estructuras no B.
Más recientemente, se desarrollaron cajas de herramientas avanzadas basadas en la web para identificar secuencias formadoras de G-cuadruplex, incluida una versión de acceso abierto y fácil de usar de G4Hunter basada en un enfoque de ventana deslizante [83] o G4RNA Screener basado en un algoritmo de aprendizaje automático. [84]
Se han desarrollado varios métodos experimentales para identificar los G-cuadruplex. Estos métodos pueden definirse en dos clases: métodos biofísicos y bioquímicos. [85]
Se emplearon técnicas bioquímicas para investigar la formación de un G-quadruplex en un contexto de secuencia más larga. En el ensayo de detención de la polimerasa de ADN, la formación de un G-quadruplex en una plantilla de ADN puede actuar como un obstáculo y provocar el estancamiento de la polimerasa, lo que detiene la extensión del cebador. [86] El ensayo de escisión con dimetilsulfato (DMS) seguido de piperidina se basa en el hecho de que la formación de un G-quadruplex prohibirá la metilación de la guanina N7 causada por el DMS, lo que conduce a un patrón de protección observado en la región del G-quadruplex de ADN después de la escisión con piperidina. [87]
La topología de la estructura del G-cuadrúplex se puede determinar monitoreando las señales de dicroísmo circular (CD) positivas o negativas a longitudes de onda específicas. [88] Los G-cuadrúplex paralelos tienen señales de CD negativas y positivas a 240 y 262 nm, respectivamente, mientras que los G-cuadrúplex antiparalelos colocan estas señales a 262 y 295 nm, respectivamente. Para verificar la formación del G-cuadrúplex, también se deben realizar los experimentos de CD en condiciones de estabilización del G-cuadrúplex no estabilizadoras (Li+) y estabilizadoras del G-cuadrúplex (como K+ o con ligandos del G-cuadrúplex), y escanear hacia la región UV lejana (180–230 nm). Del mismo modo, la termoestabilidad de la estructura del G-cuadrúplex se puede identificar observando la señal UV a 295 nm. [89] Tras la fusión del G-quadruplex, la absorbancia UV a 295 nm disminuye, lo que produce un cambio hipocrómico que es una característica distintiva de la estructura del G-quadruplex. Otro enfoque para la detección de G-quadruplexes incluye métodos basados en nanoporos . En primer lugar, se demostró que los nanoporos biológicos pueden detectar G-quadruplexes basándose en la exclusión por tamaño y la interacción específica del G-quadruplex y la nanocavidad proteica. [90] El nuevo enfoque combina nanoporos de estado sólido y nanotecnología de ADN para la detección sin etiquetas de G-quadruplexes, para su mapeo en dsADN y para monitorear la formación de G-quadruplex. [91]
Los G-quadruplex se han implicado en trastornos neurológicos a través de dos mecanismos principales. El primero es a través de expansiones de repeticiones G dentro de genes que conducen a la formación de estructuras G-quadruplex que causan enfermedades directamente, como es el caso del gen C9orf72 y la esclerosis lateral amiotrófica (ELA) o la demencia frontotemporal (DFT). El segundo mecanismo es a través de mutaciones que afectan la expresión de proteínas de unión a G-quadruplex, como se ve en el gen 1 del retraso mental del cromosoma X frágil (FMR1) y el síndrome del cromosoma X frágil . [92]
El gen C9orf72 codifica la proteína C9orf72 que se encuentra en todo el cerebro, en el citoplasma neuronal y en las terminales presinápticas . [93] Las mutaciones del gen C9orf72 se han relacionado con el desarrollo de FTD y ELA. [94] Estas dos enfermedades tienen una relación causal con las repeticiones GGGGCC (G 4 C 2 ) dentro del primer intrón del gen C9orf72. Los individuos normales suelen tener alrededor de 2 a 8 repeticiones G 4 C 2 , pero los individuos con FTD o ELA tienen de 500 a varios miles de repeticiones G 4 C 2 . [95] [96] Se ha demostrado que el ARN transcrito de estas repeticiones forma G-cuadruplexes estables, y la evidencia muestra que las repeticiones G 4 C 2 en el ADN también tienen la capacidad de formar estructuras G-cuadruplex paralelas-antiparalelas mixtas. [97] [98] Se ha demostrado que estas transcripciones de ARN que contienen repeticiones G 4 C 2 se unen y separan una amplia variedad de proteínas, incluida la nucleolina . La nucleolina está involucrada en la síntesis y maduración de los ribosomas dentro del núcleo, y la separación de la nucleolina por las transcripciones de ARN mutadas perjudica la función nucleolar y la síntesis de ARN ribosómico. [99]
La proteína de retraso mental del cromosoma X frágil (FMRP) es una proteína ampliamente expresada codificada por el gen FMR1 que se une a las estructuras secundarias G-quadruplex en las neuronas y está involucrada en la plasticidad sináptica . [100] La FMRP actúa como un regulador negativo de la traducción, y su unión estabiliza las estructuras G-quadruplex en las transcripciones de ARNm, inhibiendo la elongación del ribosoma del ARNm en la dendrita de la neurona y controlando el momento de la expresión de la transcripción. [101] [102] Las mutaciones de este gen pueden causar el desarrollo del síndrome del cromosoma X frágil, autismo y otros trastornos neurológicos. [103] Específicamente, el síndrome del cromosoma X frágil es causado por un aumento de 50 a más de 200 repeticiones CGG dentro del exón 13 del gen FMR1. Esta expansión de repeticiones promueve la metilación del ADN y otras modificaciones epigenéticas de la heterocromatina de FMR1 que previenen la transcripción del gen, lo que lleva a niveles patológicos bajos de FMRP. [104] [105]
Las intervenciones mediadas por antisentido y los ligandos de moléculas pequeñas son estrategias comunes que se utilizan para atacar enfermedades neurológicas vinculadas a repeticiones de expansión de G-quadruplex. Por lo tanto, estas técnicas son especialmente ventajosas para atacar enfermedades neurológicas que tienen un mecanismo de ganancia de función, que es cuando el producto génico alterado tiene una nueva función o nueva expresión de un gen; esto se ha detectado en el C9orf72 (marco de lectura abierto 72 del cromosoma 9). [106]
La terapia antisentido es el proceso mediante el cual se utilizan cadenas sintetizadas de ácidos nucleicos para unirse de forma directa y específica al ARNm producido por un determinado gen, lo que lo inactivará. Los oligonucleótidos antisentido (ASO) se utilizan habitualmente para dirigirse al ARN C9orf72 de la región de repetición de expansión GGGGCC del G-quadruplex, lo que ha reducido la toxicidad en modelos celulares de C9orf72. [107] [108] [109] Los ASO se han utilizado anteriormente para restaurar fenotipos normales en otras enfermedades neurológicas que tienen mecanismos de ganancia de función, la única diferencia es que se utilizó en ausencia de regiones de repetición de expansión del G-quadruplex. [110] [111] [112] [113]
La estrategia de señuelo G-quadruplex es otro enfoque prometedor para atacar a las células cancerosas mediante la explotación de las características estructurales únicas del G-quadruplex. La estrategia implica el diseño de oligonucleótidos sintéticos que imitan la estructura del G-quadruplex y compiten con los G-quadruplex endógenos para unirse a los factores de transcripción. Estos señuelos suelen estar compuestos por una secuencia rica en G que puede formar una estructura estable de G-quadruplex y una región de enlace corta que se puede modificar para optimizar sus propiedades. [114] Cuando se introduce en células cancerosas, el señuelo puede interceptar factores de transcripción asociados y unirse a ellos, lo que conduce a la regulación de la expresión génica. Se ha demostrado con éxito que los señuelos inhiben KRAS oncogénico en ratones SCID, lo que conduce a una reducción del crecimiento del tumor y un aumento del tiempo de supervivencia medio. [115]
Otra técnica comúnmente utilizada es la utilización de ligandos de moléculas pequeñas . Estos se pueden utilizar para dirigirse a las regiones G-quadruplex que causan trastornos neurológicos. Existen aproximadamente 1.000 ligandos G-quadruplex diferentes en los que pueden interactuar a través de sus anillos aromáticos ; esto permite que los ligandos de moléculas pequeñas se apilen en las tétradas terminales planas dentro de las regiones G-quadruplex. Una desventaja de usar ligandos de moléculas pequeñas como técnica terapéutica es que la especificidad es difícil de manejar debido a la variabilidad de los G-quadruplex en sus secuencias primarias, orientación, estabilidad termodinámica y estequiometría de la cadena de ácido nucleico. Hasta ahora, [ ¿cuándo? ] ningún ligando de molécula pequeña ha podido ser perfectamente específico para una sola secuencia G-quadruplex. [116] [117] Sin embargo, una porfirina catiónica conocida como TMPyP4 es capaz de unirse a la región de repetición GGGGCC C9orf72, lo que hace que la región de repetición G-quadruplex se despliegue y pierda sus interacciones con las proteínas, lo que hace que pierda su funcionalidad. [118] Los ligandos de moléculas pequeñas, compuestos principalmente de plomo, también pueden dirigirse a las regiones de repetición GGGGCC y, en última instancia, disminuyeron tanto la traducción no ATG asociada a la repetición como los focos de ARN en células neuronales derivadas de pacientes con esclerosis lateral amiotrófica (ELA). Esto proporciona evidencia de que los ligandos de moléculas pequeñas son un proceso eficaz y eficiente para dirigirse a las regiones GGGGCC, y que la especificidad para la unión de ligandos de moléculas pequeñas es un objetivo factible para la comunidad científica.
Los complejos metálicos tienen una serie de características que los hacen especialmente adecuados como aglutinantes de ADN G4 y, por lo tanto, como posibles fármacos. Si bien el metal desempeña en gran medida un papel estructural en la mayoría de los aglutinantes G4, también hay ejemplos en los que interactúa directamente con los G4 mediante interacciones electrostáticas o coordinación directa con nucleobases. [119]
{{cite book}}: Mantenimiento de CS1: falta la ubicación del editor ( enlace ){{cite book}}: |journal=ignorado ( ayuda ){{cite book}}: |journal=ignorado ( ayuda )