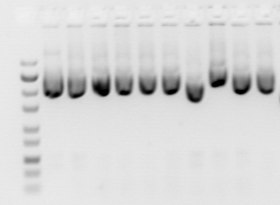
Electroforesis de ácidos nucleicos en gel
Las moléculas de ácido nucleico que se van a analizar se colocan sobre un medio viscoso, el gel, donde un campo eléctrico induce a los ácidos nucleicos (que están cargados negativamente debido a su esqueleto de azúcar-fosfato) a migrar hacia el ánodo (que está cargado positivamente porque se trata de una célula electrolítica en lugar de galvánica).
Sin embargo, el voltaje no es el único factor que determina la electroforesis de los ácidos nucleicos.
En otros casos, como las muestras amplificadas por PCR, las enzimas presentes en la muestra que podrían afectar a la separación de las moléculas se eliminan por diversos medios antes del análisis.
[2] El gel tamiza el ADN en función del tamaño de la molécula de ADN, por lo que las moléculas más pequeñas se desplazan más rápidamente.
Estas bandas menores pueden ser ADN mellado (forma circular abierta) y la forma circular cerrada relajada que normalmente se desplazan más lentamente que el ADN superenrollado, y la forma monocatenaria (que a veces puede aparecer dependiendo de los métodos de preparación) puede desplazarse por delante del ADN superenrollado.
Sin embargo, la velocidad a la que se mueven las distintas formas puede cambiar si se utilizan diferentes condiciones de electroforesis, por ejemplo, el ADN lineal puede moverse más rápido o más lento que el ADN superenrollado dependiendo de las condiciones,[6] y la movilidad del ADN circular más grande puede verse más afectada que la del ADN lineal por el tamaño del poro del gel.
Los geles de bajo porcentaje (0,1-0,2%) son frágiles y pueden romperse.
[11] Sin embargo, el modelo de Ogston no funciona con moléculas grandes, ya que los poros son mucho más pequeños que el tamaño de la molécula.
Sin embargo, en la práctica no se observa una alineación paralela perfecta de la cadena con el campo, ya que eso significaría la misma movilidad para las moléculas largas y cortas.
[14] El modelo de reptación sesgada también se ha utilizado para explicar la movilidad del ADN en PFGE.
Cuando se cambia el campo, las moléculas más grandes tardan más en reorientarse, por lo que es posible discriminar entre las cadenas largas que no pueden alcanzar su velocidad de estado estacionario de las cortas que viajan la mayor parte del tiempo a velocidad estacionaria.
Fluoresce bajo la luz UV cuando se intercala en el surco principal del ADN (o ARN).
Al pasar ADN por un gel tratado con EtBr y visualizarlo con luz UV, cualquier banda que contenga más de ~20 nanogramos (ng) de ADN se hace claramente visible.
El EtBr es un mutágeno conocido,[19] y existen alternativas más seguras, como el GelRed, producido por Biotium, que se une al surco menor.
[20] SYBR Green I es otra tinción de dsADN, producida por Invitrogen.
Dado que el ADN teñido con EtBr no es visible a la luz natural, los científicos mezclan el ADN con tampones de carga cargados negativamente antes de añadir la mezcla al gel.
Los tampones de carga son útiles porque son visibles a la luz natural (a diferencia de la luz UV para el ADN teñido con EtBr) y se sedimentan conjuntamente con el ADN (lo que significa que se mueven a la misma velocidad que el ADN de una longitud determinada).
Otros marcadores de progreso utilizados con menos frecuencia son el Rojo Cresol y el Naranja G, que corren a unos 125 pb y 50 pb, respectivamente.
A continuación, el gel puede fotografiarse normalmente con una cámara digital o polaroid.
Aunque el ácido nucleico teñido presenta una fluorescencia rojo-anaranjada, las imágenes suelen mostrarse en blanco y negro (véanse las figuras).