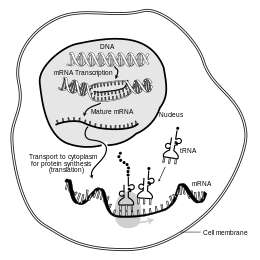
En biología molecular , el ácido ribonucleico mensajero ( ARNm ) es una molécula de ARN monocatenaria que corresponde a la secuencia genética de un gen , y es leída por un ribosoma en el proceso de síntesis de una proteína .
El ARNm se crea durante el proceso de transcripción , donde una enzima ( ARN polimerasa ) convierte el gen en ARNm de transcripción primaria (también conocido como pre-ARNm ). Este pre-ARNm generalmente todavía contiene intrones , regiones que no codificarán la secuencia final de aminoácidos . Estos se eliminan en el proceso de empalme del ARN , dejando solo los exones , regiones que codificarán la proteína. Esta secuencia de exones constituye ARNm maduro . Luego, el ribosoma lee el ARNm maduro y el ribosoma crea la proteína utilizando aminoácidos transportados por el ARN de transferencia (ARNt). Este proceso se conoce como traducción . Todos estos procesos forman parte del dogma central de la biología molecular , que describe el flujo de información genética en un sistema biológico.
Al igual que en el ADN , la información genética en el ARNm está contenida en la secuencia de nucleótidos , que están ordenados en codones que constan de tres ribonucleótidos cada uno. Cada codón codifica un aminoácido específico , excepto los codones de parada , que finalizan la síntesis de proteínas. La traducción de codones en aminoácidos requiere otros dos tipos de ARN: ARN de transferencia, que reconoce el codón y proporciona el aminoácido correspondiente, y ARN ribosomal (ARNr), el componente central de la maquinaria de fabricación de proteínas del ribosoma.
El concepto de ARNm fue desarrollado por Sydney Brenner y Francis Crick en 1960 durante una conversación con François Jacob . En 1961, el ARNm fue identificado y descrito de forma independiente por un equipo formado por Brenner, Jacob y Matthew Meselson , y otro equipo dirigido por James Watson . Mientras analizaban los datos en preparación para su publicación, Jacob y Jacques Monod acuñaron el nombre "ARN mensajero".
La breve existencia de una molécula de ARNm comienza con la transcripción y finalmente termina con la degradación. Durante su vida, una molécula de ARNm también puede procesarse, editarse y transportarse antes de la traducción. Las moléculas de ARNm eucariotas a menudo requieren un procesamiento y transporte extensos, mientras que las moléculas de ARNm procarióticas no. Una molécula de ARNm eucariota y las proteínas que la rodean se denominan en conjunto RNP mensajera .
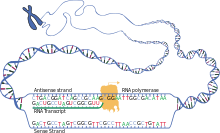
La transcripción es cuando el ARN se copia del ADN. Durante la transcripción, la ARN polimerasa produce una copia de un gen del ADN al ARNm según sea necesario. Este proceso difiere ligeramente en eucariotas y procariotas. Una diferencia notable es que la ARN polimerasa procariótica se asocia con enzimas procesadoras de ADN durante la transcripción para que el procesamiento pueda continuar durante la transcripción. Por lo tanto, esto hace que la nueva cadena de ARNm se vuelva bicatenaria al producir una cadena complementaria conocida como cadena de ARNt, que cuando se combinan no pueden formar estructuras a partir del emparejamiento de bases. Además, el molde para el ARNm es la cadena complementaria del ARNt, cuya secuencia es idéntica a la secuencia del anticodón a la que se une el ADN. El producto de vida corta, sin procesar o parcialmente procesado se denomina ARNm precursor o pre-ARNm ; una vez completamente procesado, se denomina ARNm maduro .

El procesamiento del ARNm difiere mucho entre eucariotas , bacterias y arqueas . El ARNm no eucariota, en esencia, madura tras la transcripción y no requiere procesamiento, excepto en casos raros. [1] El pre-ARNm eucariota, sin embargo, requiere varios pasos de procesamiento antes de su transporte al citoplasma y su traducción por el ribosoma.
El procesamiento extensivo del pre-ARNm eucariótico que conduce al ARNm maduro es el empalme del ARN , un mecanismo mediante el cual se eliminan intrones o outrones (regiones no codificantes) y se unen exones (regiones codificantes).
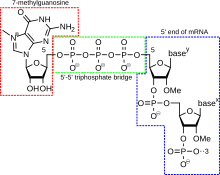
Una tapa 5' (también denominada tapa de ARN, tapa de ARN 7-metilguanosina o tapa de ARN m 7 G) es un nucleótido de guanina modificado que se ha añadido al extremo "frontal" o 5' de un ARN mensajero eucariota en breve. después del inicio de la transcripción. La tapa 5' consta de un residuo terminal de 7-metilguanosina que está unido mediante un enlace 5'-5'-trifosfato al primer nucleótido transcrito. Su presencia es fundamental para el reconocimiento por parte del ribosoma y la protección contra las RNasas .
La adición de mayúsculas está acoplada a la transcripción y ocurre cotranscripcionalmente, de modo que cada una influye en la otra. Poco después del inicio de la transcripción, el extremo 5' del ARNm que se está sintetizando está unido por un complejo de síntesis de cápsulas asociado con la ARN polimerasa . Este complejo enzimático cataliza las reacciones químicas necesarias para la protección del ARNm. La síntesis se produce como una reacción bioquímica de varios pasos .
En algunos casos, se editará un ARNm , cambiando la composición de nucleótidos de ese ARNm. Un ejemplo en humanos es el ARNm de la apolipoproteína B , que se edita en algunos tejidos, pero no en otros. La edición crea un codón de parada temprano que, tras la traducción, produce una proteína más corta.

La poliadenilación es el enlace covalente de un resto poliadenililo a una molécula de ARN mensajero. En los organismos eucariotas, la mayoría de las moléculas de ARN mensajero (ARNm) están poliadeniladas en el extremo 3', pero estudios recientes han demostrado que también son comunes tramos cortos de uridina (oligouridilación). [2] La cola poli(A) y la proteína unida a ella ayudan a proteger el ARNm de la degradación por exonucleasas. La poliadenilación también es importante para la terminación de la transcripción, la exportación del ARNm desde el núcleo y la traducción. El ARNm también se puede poliadenilar en organismos procarióticos, donde las colas poli(A) actúan para facilitar, en lugar de impedir, la degradación exonucleolítica.
La poliadenilación ocurre durante y/o inmediatamente después de la transcripción de ADN a ARN. Una vez terminada la transcripción, la cadena de ARNm se escinde mediante la acción de un complejo de endonucleasa asociado con la ARN polimerasa. Una vez escindido el ARNm, se añaden alrededor de 250 residuos de adenosina al extremo 3' libre del lugar de escisión. Esta reacción está catalizada por la poliadenilato polimerasa . Al igual que en el empalme alternativo , puede haber más de una variante de poliadenilación de un ARNm.
También se producen mutaciones en el sitio de poliadenilación. El transcrito de ARN primario de un gen se escinde en el sitio de adición de poli-A y se añaden entre 100 y 200 A al extremo 3' del ARN. Si este sitio se altera, se formará una construcción de ARNm anormalmente larga e inestable.
Otra diferencia entre eucariotas y procariotas es el transporte de ARNm. Debido a que la transcripción y la traducción en eucariotas están separadas por compartimentos, los ARNm de eucariotas deben exportarse desde el núcleo al citoplasma , un proceso que puede estar regulado por diferentes vías de señalización. [3] Los ARNm maduros se reconocen por sus modificaciones procesadas y luego se exportan a través del poro nuclear uniéndose a las proteínas de unión a cápsulas CBP20 y CBP80, [4] así como al complejo de transcripción/exportación (TREX). [5] [6] Se han identificado múltiples vías de exportación de ARNm en eucariotas. [7]
En células espacialmente complejas, algunos ARNm se transportan a destinos subcelulares particulares. En las neuronas maduras , ciertos ARNm se transportan desde el soma a las dendritas . Un sitio de traducción del ARNm se encuentra en los polirribosomas localizados selectivamente debajo de las sinapsis. [8] El ARNm de Arc/Arg3.1 es inducido por la actividad sináptica y se localiza selectivamente cerca de las sinapsis activas basándose en señales generadas por los receptores NMDA . [9] Otros ARNm también se mueven hacia las dendritas en respuesta a estímulos externos, como el ARNm de β-actina . [10] Para la exportación desde el núcleo, el ARNm de actina se asocia con ZBP1 [11] y posteriormente con la subunidad 40S . El complejo está unido por una proteína motora y se transporta al lugar objetivo ( extensión de neuritas ) a lo largo del citoesqueleto . Finalmente, Src fosforila ZBP1 para que se inicie la traducción. [12] En las neuronas en desarrollo, los ARNm también se transportan a los axones en crecimiento y especialmente a los conos de crecimiento. Muchos ARNm están marcados con los llamados "códigos postales", que apuntan a su transporte a una ubicación específica. [13] [14] Los ARNm también pueden transferirse entre células de mamíferos a través de estructuras llamadas nanotubos tunelizadores . [15] [16]
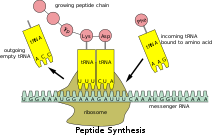
Debido a que el ARNm procariótico no necesita ser procesado ni transportado, la traducción por parte del ribosoma puede comenzar inmediatamente después del final de la transcripción. Por tanto, se puede decir que la traducción procariótica está acoplada a la transcripción y se produce de forma cotranscripcional .
El ribosoma puede traducir el ARNm eucariota que ha sido procesado y transportado al citoplasma (es decir, ARNm maduro). La traducción puede ocurrir en los ribosomas que flotan libremente en el citoplasma o dirigida al retículo endoplasmático por la partícula de reconocimiento de señales . Por lo tanto, a diferencia de los procariotas, la traducción en eucariotas no está directamente acoplada a la transcripción. Incluso es posible en algunos contextos que los niveles reducidos de ARNm vayan acompañados de niveles elevados de proteína, como se ha observado para los niveles de ARNm/proteína de EEF1A1 en el cáncer de mama . [17] [ se necesita fuente no primaria ]

Las regiones codificantes están compuestas por codones , que son decodificados y traducidos en proteínas por el ribosoma; en eucariotas normalmente en uno y en procariotas normalmente en varios. Las regiones codificantes comienzan con el codón de inicio y terminan con un codón de parada . En general, el codón de inicio es un triplete AUG y el codón de parada es UAG ("ámbar"), UAA ("ocre") o UGA ("ópalo"). Las regiones codificantes tienden a estabilizarse mediante pares de bases internos; esto impide la degradación. [18] [19] Además de codificar proteínas, porciones de regiones codificantes pueden servir como secuencias reguladoras en el pre-ARNm como potenciadores del empalme exónico o silenciadores del empalme exónico .

Las regiones no traducidas (UTR) son secciones del ARNm antes del codón de inicio y después del codón de parada que no están traducidas, denominadas región cinco principales no traducidas (5 'UTR) y región tres principales no traducidas (3' UTR), respectivamente. Estas regiones se transcriben con la región codificante y, por tanto, son exónicas ya que están presentes en el ARNm maduro. Se han atribuido varias funciones en la expresión génica a las regiones no traducidas, incluida la estabilidad del ARNm, la localización del ARNm y la eficiencia traduccional . La capacidad de una UTR para realizar estas funciones depende de la secuencia de la UTR y puede diferir entre los ARNm. Las variantes genéticas en 3'UTR también se han implicado en la susceptibilidad a enfermedades debido al cambio en la estructura del ARN y la traducción de proteínas. [20]
La estabilidad de los ARNm puede estar controlada por la UTR 5' y/o la UTR 3' debido a la afinidad variable por las enzimas que degradan el ARN llamadas ribonucleasas y por las proteínas auxiliares que pueden promover o inhibir la degradación del ARN. (Ver también elemento de estabilidad rico en C ).
La eficiencia de la traducción, incluida a veces la inhibición completa de la traducción, puede controlarse mediante UTR. Las proteínas que se unen a la UTR 3' o 5' pueden afectar la traducción al influir en la capacidad del ribosoma para unirse al ARNm. Los microARN unidos a la UTR 3' también pueden afectar la eficiencia traduccional o la estabilidad del ARNm.
Se cree que la localización citoplásmica del ARNm es una función de la UTR 3'. Las proteínas que se necesitan en una región particular de la célula también pueden traducirse allí; en tal caso, la UTR 3' puede contener secuencias que permiten localizar el transcrito en esta región para su traducción.
Algunos de los elementos contenidos en regiones no traducidas forman una estructura secundaria característica cuando se transcriben a ARN. Estos elementos estructurales del ARNm participan en la regulación del ARNm. Algunos, como el elemento SECIS , son objetivos de unión de proteínas. Una clase de elemento de ARNm, los ribointerruptores , se unen directamente a moléculas pequeñas, cambiando su pliegue para modificar los niveles de transcripción o traducción. En estos casos, el ARNm se regula a sí mismo.
La cola 3' poli(A) es una secuencia larga de nucleótidos de adenina (a menudo varios cientos) añadidos al extremo 3' del pre-ARNm. Esta cola promueve la exportación desde el núcleo y la traducción, y protege el ARNm de la degradación.
Se dice que una molécula de ARNm es monocistrónica cuando contiene la información genética para traducir una sola cadena proteica (polipéptido). Este es el caso de la mayoría de los ARNm eucariotas . [21] [22] Por otro lado, el ARNm policistrónico lleva varios marcos de lectura abiertos (ORF), cada uno de los cuales se traduce en un polipéptido. Estos polipéptidos suelen tener una función relacionada (a menudo son las subunidades que componen una proteína compleja final) y su secuencia codificante está agrupada y regulada en una región reguladora, que contiene un promotor y un operador . La mayor parte del ARNm que se encuentra en bacterias y arqueas es policistrónico, [21] al igual que el genoma mitocondrial humano. [23] El ARNm dicistrónico o bicistrónico codifica sólo dos proteínas .

En eucariotas, las moléculas de ARNm forman estructuras circulares debido a una interacción entre eIF4E y la proteína de unión poli(A) , que se unen a eIF4G , formando un puente ARNm-proteína-ARNm. [24] Se cree que la circularización promueve el ciclo de los ribosomas en el ARNm, lo que conduce a una traducción eficiente en el tiempo, y también puede funcionar para garantizar que solo se traduzca el ARNm intacto (el ARNm parcialmente degradado característicamente no tiene tapa m7G ni cola poli-A). [25]
Existen otros mecanismos de circularización, particularmente en el ARNm del virus. El ARNm del poliovirus utiliza una sección de hoja de trébol hacia su extremo 5' para unirse a PCBP2, que se une a la proteína de unión poli(A) , formando el conocido círculo ARNm-proteína-ARNm. El virus del enano amarillo de la cebada se une entre segmentos de ARNm en su extremo 5' y 3' (llamados bucles de tallo besantes), circularizando el ARNm sin ninguna proteína involucrada.
Los genomas de los virus de ARN (cuyas cadenas + se traducen como ARNm) también suelen estar circularizados. [26] Durante la replicación del genoma, la circularización actúa para mejorar las velocidades de replicación del genoma, ciclando la ARN polimerasa viral dependiente de ARN de manera muy similar a como se supone que cicla el ribosoma.
Diferentes ARNm dentro de la misma célula tienen vidas distintas (estabilidades). En las células bacterianas, los ARNm individuales pueden sobrevivir desde segundos hasta más de una hora. Sin embargo, la vida útil promedio es de entre 1 y 3 minutos, lo que hace que el ARNm bacteriano sea mucho menos estable que el ARNm eucariota. [27] En las células de mamíferos, la vida útil del ARNm varía desde varios minutos hasta días. [28] Cuanto mayor sea la estabilidad de un ARNm, más proteínas se podrán producir a partir de ese ARNm. La vida útil limitada del ARNm permite que una célula altere rápidamente la síntesis de proteínas en respuesta a sus necesidades cambiantes. Existen muchos mecanismos que conducen a la destrucción de un ARNm, algunos de los cuales se describen a continuación.

En general, en los procariotas la vida útil del ARNm es mucho más corta que en los eucariotas. Los procariotas degradan los mensajes mediante el uso de una combinación de ribonucleasas, incluidas endonucleasas , 3' exonucleasas y 5' exonucleasas. En algunos casos, pequeñas moléculas de ARN (ARNs) de decenas a cientos de nucleótidos de largo pueden estimular la degradación de ARNm específicos mediante el emparejamiento de bases con secuencias complementarias y facilitando la escisión de la ribonucleasa por la ARNasa III . Recientemente se ha demostrado que las bacterias también tienen una especie de tapa 5' formada por un trifosfato en el extremo 5' . [29] La eliminación de dos de los fosfatos deja un monofosfato 5', lo que provoca que la exonucleasa RNasa J destruya el mensaje, que degrada 5' a 3'.
Dentro de las células eucariotas, existe un equilibrio entre los procesos de traducción y la descomposición del ARNm. Los mensajes que se traducen activamente están unidos por ribosomas , los factores de iniciación eucarióticos eIF-4E y eIF-4G , y la proteína de unión a poli(A) . eIF-4E y eIF-4G bloquean la enzima decapante ( DCP2 ) y la proteína de unión a poli(A) bloquea el complejo exosoma , protegiendo los extremos del mensaje. El equilibrio entre traducción y desintegración se refleja en el tamaño y la abundancia de estructuras citoplasmáticas conocidas como cuerpos P. [30] La cola poli(A) del ARNm se acorta mediante exonucleasas especializadas que se dirigen a ARN mensajeros específicos mediante una combinación de secuencias reguladoras cis en el ARN y proteínas de unión a ARN que actúan en trans. Se cree que la eliminación de la cola de poli(A) altera la estructura circular del mensaje y desestabiliza el complejo de unión de la tapa . Luego, el mensaje está sujeto a degradación por el complejo exosoma o por el complejo decapante . De esta manera, los mensajes traduccionalmente inactivos se pueden destruir rápidamente, mientras que los mensajes activos permanecen intactos. El mecanismo por el cual se detiene la traducción y el mensaje pasa a complejos de descomposición no se comprende en detalle.
La presencia de elementos ricos en AU en algunos ARNm de mamíferos tiende a desestabilizar esas transcripciones mediante la acción de proteínas celulares que se unen a estas secuencias y estimulan la eliminación de la cola poli(A) . Se cree que la pérdida de la cola poli(A) promueve la degradación del ARNm al facilitar el ataque tanto del complejo exosomal [31] como del complejo de decapado . [32] La rápida degradación del ARNm a través de elementos ricos en AU es un mecanismo crítico para prevenir la sobreproducción de citoquinas potentes como el factor de necrosis tumoral (TNF) y el factor estimulante de colonias de granulocitos y macrófagos (GM-CSF). [33] Los elementos ricos en AU también regulan la biosíntesis de factores de transcripción protooncogénicos como c-Jun y c-Fos . [34]
Los mensajes eucariotas están sujetos a vigilancia mediante desintegración mediada por sin sentido (NMD), que comprueba la presencia de codones de parada prematuros (codones sin sentido) en el mensaje. Estos pueden surgir a través de un empalme incompleto, recombinación V(D)J en el sistema inmunológico adaptativo , mutaciones en el ADN, errores de transcripción, escaneo con fugas por parte del ribosoma que causa un cambio de marco y otras causas. "La detección de un codón de parada prematuro desencadena la degradación del ARNm mediante descapsulación 5', eliminación de la cola poli(A) 3' o escisión endonucleolítica ". [35]
En los metazoos , los pequeños ARN de interferencia (ARNip) procesados por Dicer se incorporan a un complejo conocido como complejo silenciador inducido por ARN o RISC. Este complejo contiene una endonucleasa que escinde mensajes perfectamente complementarios a los que se une el ARNip. Los fragmentos de ARNm resultantes son luego destruidos por exonucleasas . El ARNip se utiliza habitualmente en laboratorios para bloquear la función de genes en cultivos celulares. Se cree que forma parte del sistema inmunológico innato como defensa contra los virus de ARN bicatenario. [36]
Los microARN (miARN) son ARN pequeños que normalmente son parcialmente complementarios a las secuencias de los ARN mensajeros de metazoos. [37] [38] La unión de un miARN a un mensaje puede reprimir la traducción de ese mensaje y acelerar la eliminación de la cola poli(A), acelerando así la degradación del ARNm. El mecanismo de acción de los miARN es objeto de investigación activa. [39] [40]
Hay otras formas mediante las cuales se pueden degradar los mensajes, incluida la descomposición continua y el silenciamiento mediante ARN que interactúa con Piwi (piRNA), entre otras.
La administración de una secuencia de ARN mensajero modificada con nucleósidos puede hacer que una célula produzca una proteína, que a su vez podría tratar directamente una enfermedad o podría funcionar como una vacuna ; De manera más indirecta, la proteína podría impulsar a una célula madre endógena a diferenciarse de la manera deseada. [41] [42]
Los principales desafíos de la terapia con ARN se centran en entregar el ARN a las células apropiadas. [43] Los desafíos incluyen el hecho de que las secuencias de ARN desnudas se degradan naturalmente después de la preparación; pueden hacer que el sistema inmunológico del cuerpo los ataque como a un invasor; y son impermeables a la membrana celular . [42] Una vez dentro de la célula, deben abandonar el mecanismo de transporte de la célula para actuar dentro del citoplasma , que alberga los ribosomas necesarios . [41]
Para superar estos desafíos, el ARNm como tratamiento se propuso por primera vez en 1989 "después del desarrollo de una técnica de transfección in vitro de amplia aplicación". [44] En la década de 1990, se desarrollaron vacunas de ARNm para el cáncer personalizado, basándose en ARNm modificado sin nucleósidos. Las terapias basadas en ARNm continúan investigándose como método de tratamiento o terapia tanto para el cáncer como para enfermedades inflamatorias autoinmunes, metabólicas y respiratorias. Las terapias de edición genética como CRISPR también pueden beneficiarse del uso de ARNm para inducir a las células a producir la proteína Cas deseada . [45]
Desde la década de 2010, las vacunas de ARN y otras terapias de ARN se consideran "una nueva clase de medicamentos". [46] Las primeras vacunas basadas en ARNm recibieron autorización restringida y fueron implementadas en todo el mundo durante la pandemia de COVID-19 mediante la vacuna Pfizer-BioNTech COVID-19 y Moderna , por ejemplo. [47] El Premio Nobel de Fisiología o Medicina 2023 fue otorgado a Katalin Karikó y Drew Weissman por el desarrollo de vacunas de ARNm eficaces contra la COVID-19. [48] [49] [50]
Varios estudios de biología molecular durante la década de 1950 indicaron que el ARN desempeñaba algún tipo de papel en la síntesis de proteínas, pero ese papel no se entendía claramente. Por ejemplo, en uno de los primeros informes, Jacques Monod y su equipo demostraron que la síntesis de ARN era necesaria para la síntesis de proteínas, específicamente durante la producción de la enzima β-galactosidasa en la bacteria E. coli . [51] Arthur Pardee también encontró una acumulación de ARN similar en 1954 . [52] En 1953, Alfred Hershey , June Dixon y Martha Chase describieron cierto ADN que contenía citosina (lo que indica que era ARN) que desapareció rápidamente después de su síntesis en E. coli . [53] En retrospectiva, esta puede haber sido una de las primeras observaciones de la existencia de ARNm, pero en ese momento no se reconoció como tal. [54]
La idea del ARNm fue concebida por primera vez por Sydney Brenner y Francis Crick el 15 de abril de 1960 en el King's College de Cambridge , mientras François Jacob les hablaba de un experimento reciente realizado por Arthur Pardee , él mismo y Monod (el llamado experimento PaJaMo). que no probó la existencia del ARNm pero sugirió la posibilidad de su existencia). Con el apoyo de Crick, Brenner y Jacob se dispusieron inmediatamente a probar esta nueva hipótesis y se pusieron en contacto con Matthew Meselson del Instituto de Tecnología de California para solicitar ayuda. Durante el verano de 1960, Brenner, Jacob y Meselson llevaron a cabo un experimento en el laboratorio de Meselson en Caltech que fue el primero en demostrar la existencia del ARNm. Ese otoño, Jacob y Monod acuñaron el nombre "ARN mensajero" y desarrollaron el primer marco teórico para explicar su función. [54]
En febrero de 1961, James Watson reveló que su grupo de investigación con sede en Harvard había estado detrás de ellos con una serie de experimentos cuyos resultados apuntaban aproximadamente en la misma dirección. Brenner y los demás aceptaron la solicitud de Watson de retrasar la publicación de los resultados de su investigación. Como resultado, los artículos de Brenner y Watson se publicaron simultáneamente en el mismo número de Nature en mayo de 1961, mientras que ese mismo mes, Jacob y Monod publicaron su marco teórico para el ARNm en el Journal of Molecular Biology . [54]