
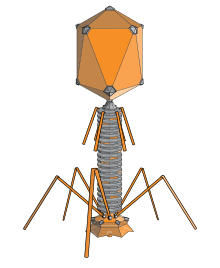

Un bacteriófago ( / bækˈtɪərioʊfeɪdʒ / ) , también conocido informalmente como fago ( / ˈfeɪdʒ / ), es un virus que infecta y se replica dentro de bacterias y arqueas . El término se deriva de "bacteria" y del griego φαγεῖν ( phagein ) , que significa "devorar". Los bacteriófagos están compuestos de proteínas que encapsulan un genoma de ADN o ARN , y pueden tener estructuras simples o elaboradas. Sus genomas pueden codificar tan solo cuatro genes (por ejemplo, MS2 ) y hasta cientos de genes . Los fagos se replican dentro de la bacteria luego de la inyección de su genoma en su citoplasma .
Los bacteriófagos se encuentran entre las entidades más comunes y diversas de la biosfera . [2] Los bacteriófagos son virus ubicuos, que se encuentran dondequiera que existan bacterias. Se estima que hay más de 10 31 bacteriófagos en el planeta, más que cualquier otro organismo en la Tierra, incluidas las bacterias, combinados. [3] Los virus son la entidad biológica más abundante en la columna de agua de los océanos del mundo, y el segundo componente más grande de la biomasa después de los procariotas , [4] donde se han encontrado hasta 9x10 8 viriones por mililitro en tapetes microbianos en la superficie, [5] y hasta el 70% de las bacterias marinas pueden estar infectadas por bacteriófagos. [6]
Los bacteriófagos se han utilizado desde la década de 1920 como alternativa a los antibióticos en la ex Unión Soviética y Europa Central, así como en Francia. [7] [8] Se consideran una posible terapia contra cepas de muchas bacterias resistentes a múltiples fármacos (véase terapia con fagos ). [9] [10] [11] [12]
Se sabe que los bacteriófagos interactúan con el sistema inmunológico tanto indirectamente a través de la expresión bacteriana de proteínas codificadas por fagos como directamente al influir en la inmunidad innata y la eliminación bacteriana. [13] Las interacciones fago-huésped se están convirtiendo en áreas de investigación cada vez más importantes. [14]
Los bacteriófagos son abundantes en la biosfera y tienen diferentes genomas y estilos de vida. El Comité Internacional de Taxonomía de Virus (ICTV) clasifica los fagos según su morfología y su ácido nucleico.


Se ha sugerido que los miembros de Picobirnaviridae infectan bacterias, pero no mamíferos. [16]
También hay muchos géneros no asignados de la clase Leviviricetes : Chimpavirus , Hohglivirus , Mahrahvirus , Meihzavirus , Nicedsevirus , Sculuvirus , Skrubnovirus , Tetipavirus y Winunavirus que contienen genomas de ARNmc lineales [17] y el género no asignado Lilyvirus del orden Caudovirales que contiene un genoma de ADNmc lineal.

En 1896, Ernest Hanbury Hankin informó que algo en las aguas de los ríos Ganges y Yamuna en la India tenía una marcada acción antibacteriana contra el cólera y podía pasar a través de un filtro de porcelana muy fino. [18] En 1915, el bacteriólogo británico Frederick Twort , superintendente de la Brown Institution de Londres, descubrió un pequeño agente que infectaba y mataba bacterias. Creía que el agente debía ser uno de los siguientes:
La investigación de Twort se vio interrumpida por el inicio de la Primera Guerra Mundial , así como por la falta de financiación y el descubrimiento de antibióticos.
El 3 de septiembre de 1917 , el microbiólogo francocanadiense Félix d'Hérelle , que trabajaba en el Instituto Pasteur de París , anunció que había descubierto "un microbio invisible, antagonista del bacilo de la disentería ". Para d'Hérelle, no había ninguna duda sobre la naturaleza de su descubrimiento: "En un instante lo comprendí: lo que causaba mis manchas claras era de hecho un microbio invisible... un virus parásito de las bacterias". [20] D'Hérelle llamó al virus bacteriófago, un devorador de bacterias (del griego phagein , que significa "devorar"). También registró un dramático relato de un hombre que sufría de disentería y que recuperó la salud gracias a los bacteriófagos. [21] Fue d'Hérelle quien realizó muchas investigaciones sobre los bacteriófagos e introdujo el concepto de terapia con fagos . [22] En 1919, en París, Francia, d'Hérelle realizó la primera aplicación clínica de un bacteriófago; el primer uso informado en los Estados Unidos fue en 1922. [23]
En 1969, Max Delbrück , Alfred Hershey y Salvador Luria recibieron el Premio Nobel de Fisiología o Medicina por sus descubrimientos sobre la replicación de los virus y su estructura genética. [24] En concreto, el trabajo de Hershey, como colaborador del experimento Hershey-Chase en 1952, proporcionó pruebas convincentes de que el ADN, no las proteínas, era el material genético de la vida. Delbrück y Luria llevaron a cabo el experimento Luria-Delbrück que demostró estadísticamente que las mutaciones en las bacterias se producen de forma aleatoria y, por tanto, siguen principios darwinianos en lugar de lamarckianos .

Se descubrió que los fagos eran agentes antibacterianos y se utilizaron en la ex República Soviética de Georgia (allí fue pionero Giorgi Eliava con la ayuda del codescubridor de los bacteriófagos, Félix d'Hérelle ) durante las décadas de 1920 y 1930 para tratar infecciones bacterianas.
D'Herelle "aprendió rápidamente que los bacteriófagos se encuentran dondequiera que prosperen las bacterias: en las alcantarillas, en los ríos que recogen los desechos de las tuberías y en las heces de los pacientes convalecientes". [25]
Su uso fue generalizado, incluido el tratamiento de soldados del Ejército Rojo . [26] Sin embargo, su uso generalizado en Occidente fue abandonado por varias razones:
El uso de fagos ha continuado desde el final de la Guerra Fría en Rusia, [28] Georgia y otras partes de Europa central y oriental. El primer ensayo clínico regulado, aleatorizado y doble ciego se informó en el Journal of Wound Care en junio de 2009, que evaluó la seguridad y eficacia de un cóctel de bacteriófagos para tratar úlceras venosas infectadas de la pierna en pacientes humanos. [29] La FDA aprobó el estudio como un ensayo clínico de fase I. Los resultados del estudio demostraron la seguridad de la aplicación terapéutica de bacteriófagos, pero no mostraron eficacia. Los autores explicaron que el uso de ciertos productos químicos que forman parte del cuidado estándar de las heridas (por ejemplo, lactoferrina o plata) puede haber interferido con la viabilidad de los bacteriófagos. [29] Poco después, otro ensayo clínico controlado en Europa occidental (tratamiento de infecciones de oído causadas por Pseudomonas aeruginosa ) fue publicado en la revista Clinical Otolaryngology en agosto de 2009. [30] El estudio concluye que las preparaciones de bacteriófagos eran seguras y efectivas para el tratamiento de infecciones de oído crónicas en humanos. Además, ha habido numerosos ensayos clínicos experimentales en animales y otros que evalúan la eficacia de los bacteriófagos para varias enfermedades, como quemaduras y heridas infectadas e infecciones pulmonares asociadas a la fibrosis quística, entre otras. [30] Por otro lado, se ha demostrado que los fagos de Inoviridae complican las biopelículas involucradas en la neumonía y la fibrosis quística y protegen a las bacterias de los medicamentos destinados a erradicar la enfermedad, promoviendo así la infección persistente. [31]
Mientras tanto, los investigadores de bacteriófagos han estado desarrollando virus modificados para superar la resistencia a los antibióticos y modificando los genes de los fagos responsables de codificar las enzimas que degradan la matriz del biofilm, las proteínas estructurales de los fagos y las enzimas responsables de la lisis de la pared celular bacteriana. [5] [6] [7] Ha habido resultados que muestran que los fagos T4 que son pequeños en tamaño y de cola corta pueden ser útiles para detectar E. coli en el cuerpo humano. [32]
Se evaluó la eficacia terapéutica de un cóctel de fagos en un modelo de ratón con infección nasal por A. baumannii resistente a múltiples fármacos (MDR) . Los ratones tratados con el cóctel de fagos mostraron una tasa de supervivencia 2,3 veces mayor en comparación con los no tratados a los siete días posteriores a la infección. [33]
En 2017, un paciente diabético de 68 años con pancreatitis necrotizante complicada por un pseudoquiste infectado con cepas de A. baumannii MDR estaba siendo tratado con un cóctel de azitromicina, rifampicina y colistina durante 4 meses sin resultados y con una salud general en rápido deterioro.
Como ya se había empezado a hablar de la inutilidad clínica de un tratamiento posterior, se presentó una solicitud de autorización de un nuevo fármaco en investigación de emergencia (eIND, por sus siglas en inglés) como último esfuerzo para, al menos, obtener datos médicos valiosos de la situación, y se aprobó, por lo que se lo sometió a una terapia con fagos utilizando un cóctel inyectado por vía percutánea (PC) que contenía nueve fagos diferentes que se habían identificado como eficaces contra la cepa de infección primaria mediante técnicas rápidas de aislamiento y prueba (un proceso que llevó menos de un día). Esto resultó eficaz durante un período muy breve, aunque el paciente no respondió y su salud siguió empeorando; pronto se recogieron aislamientos de una cepa de A. baumannii del drenaje del quiste que mostraba resistencia a este cóctel, y se añadió un segundo cóctel que se probó como eficaz contra esta nueva cepa, esta vez mediante inyección intravenosa (IV), ya que había quedado claro que la infección era más generalizada de lo que se pensaba originalmente. [34]
Una vez que comenzó a administrar la combinación de la terapia intravenosa y la terapia de PC, la trayectoria clínica descendente del paciente se revirtió y en dos días había despertado del coma y se había vuelto receptivo. Cuando su sistema inmunológico comenzó a funcionar, tuvieron que retirarle temporalmente el cóctel porque su fiebre estaba subiendo a más de 104 °F (40 °C), pero después de dos días se le volvieron a administrar los cócteles de fagos a niveles que podía tolerar. El cóctel original de tres antibióticos fue reemplazado por minociclina después de que se descubrió que la cepa bacteriana no era resistente a esto y rápidamente recuperó la lucidez total, aunque no fue dado de alta del hospital hasta aproximadamente 145 días después de que comenzara la terapia con fagos. Hacia el final de la terapia se descubrió que las bacterias se habían vuelto resistentes a ambos cócteles de fagos originales, pero se continuaron porque parecían estar impidiendo que se desarrollara resistencia a la minociclina en las muestras bacterianas recolectadas, por lo que estaban teniendo un efecto sinérgico útil. [34]
Los fagos se han utilizado cada vez más para proteger los productos alimenticios y prevenir las bacterias que los deterioran . [35] Desde 2006, la Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) y el Departamento de Agricultura de los Estados Unidos (USDA) han aprobado varios productos bacteriófagos. LMP-102 (Intralytix) fue aprobado para el tratamiento de productos cárnicos y avícolas listos para comer (RTE). Ese mismo año, la FDA aprobó LISTEX (desarrollado y producido por Micreos ) utilizando bacteriófagos en queso para matar las bacterias Listeria monocytogenes , con el fin de darles el estado de generalmente reconocidos como seguros (GRAS). [36] En julio de 2007, los mismos bacteriófagos fueron aprobados para su uso en todos los productos alimenticios. [37] En 2011, el USDA confirmó que LISTEX es un auxiliar de procesamiento de etiqueta limpia y está incluido en el USDA. [38] La investigación en el campo de la seguridad alimentaria continúa para ver si los fagos líticos son una opción viable para controlar otros patógenos transmitidos por alimentos en varios productos alimenticios. [39]
Los bacteriófagos, incluidos los específicos de Escherichia coli , se han empleado como indicadores de contaminación fecal en fuentes de agua. Debido a sus características estructurales y biológicas compartidas, los colifagos pueden servir como indicadores de contaminación fecal viral y de la presencia de virus patógenos como rotavirus, norovirus y VHA. Las investigaciones realizadas en sistemas de tratamiento de aguas residuales han revelado disparidades significativas en el comportamiento de los colifagos en comparación con los coliformes fecales, lo que demuestra una correlación clara con la recuperación de virus patógenos al final del tratamiento. Al establecer un umbral de descarga seguro, los estudios han determinado que las descargas por debajo de 3000 PFU/100 mL se consideran seguras en términos de limitar la liberación de virus patógenos. [40]
En 2011, la FDA autorizó el primer producto basado en bacteriófagos para uso diagnóstico in vitro. [41] La prueba KeyPath MRSA/MSSA Blood Culture Test utiliza un cóctel de bacteriófagos para detectar Staphylococcus aureus en hemocultivos positivos y determinar la resistencia o susceptibilidad a la meticilina . La prueba arroja resultados en aproximadamente cinco horas, en comparación con los dos o tres días que tardan los métodos estándar de identificación microbiana y prueba de susceptibilidad. Fue la primera prueba acelerada de susceptibilidad a antibióticos aprobada por la FDA. [42]
Las agencias gubernamentales en Occidente han estado buscando ayuda durante varios años en Georgia y la ex Unión Soviética para explotar fagos para contrarrestar armas biológicas y toxinas, como el ántrax y el botulismo . [43] Los desarrollos continúan entre los grupos de investigación en los EE. UU. Otros usos incluyen la aplicación de aerosoles en horticultura para proteger las plantas y los productos vegetales de la descomposición y la propagación de enfermedades bacterianas. Otras aplicaciones para bacteriófagos son como biocidas para superficies ambientales, por ejemplo, en hospitales, y como tratamientos preventivos para catéteres y dispositivos médicos antes de su uso en entornos clínicos. Ahora existe la tecnología para aplicar fagos a superficies secas, por ejemplo, uniformes, cortinas o incluso suturas para cirugía. Los ensayos clínicos informados en Clinical Otolaryngology [30] muestran éxito en el tratamiento veterinario de perros domésticos con otitis .
El método de detección e identificación de bacterias mediante cascadas de iones desencadenadas por fagos (SEPTIC) utiliza la emisión de iones y su dinámica durante la infección del fago y ofrece alta especificidad y velocidad para la detección. [44]
La visualización de fagos es un uso diferente de los fagos que implica una biblioteca de fagos con un péptido variable unido a una proteína de superficie. Cada genoma de fago codifica la variante de la proteína que se muestra en su superficie (de ahí el nombre), lo que proporciona un vínculo entre la variante del péptido y su gen codificador. Los fagos variantes de la biblioteca pueden seleccionarse a través de su afinidad de unión a una molécula inmovilizada (por ejemplo, la toxina del botulismo) para neutralizarla. Los fagos seleccionados y unidos pueden multiplicarse reinfectando una cepa bacteriana susceptible, lo que les permite recuperar los péptidos codificados en ellos para un estudio posterior. [45]
Las proteínas de fagos a menudo tienen actividad antimicrobiana y pueden servir como líderes para los peptidomiméticos , es decir, medicamentos que imitan a los péptidos. [46] La tecnología fago-ligando hace uso de proteínas de fagos para diversas aplicaciones, como la unión de bacterias y componentes bacterianos (por ejemplo, endotoxina ) y la lisis de bacterias. [47]
Los bacteriófagos son organismos modelo importantes para estudiar los principios de la evolución y la ecología . [48]
Los bacteriófagos presentes en el ambiente pueden provocar que el queso no fermente. Para evitarlo, se pueden utilizar cultivos iniciadores de cepas mixtas y regímenes de rotación de cultivos. [49] Se ha estudiado la ingeniería genética de microbios de cultivo, especialmente Lactococcus lactis y Streptococcus thermophilus , para el análisis genético y la modificación con el fin de mejorar la resistencia a los fagos . Esto se ha centrado especialmente en modificaciones cromosómicas recombinantes y de plásmidos . [50] [35]
Algunas investigaciones se han centrado en el potencial de los bacteriófagos como antimicrobianos contra patógenos transmitidos por los alimentos y la formación de biopelículas en la industria láctea. Como la propagación de la resistencia a los antibióticos es una preocupación principal en la industria láctea, los fagos pueden servir como una alternativa prometedora. [51]

El ciclo de vida de los bacteriófagos tiende a ser un ciclo lítico o un ciclo lisogénico . Además, algunos fagos presentan comportamientos pseudolisogénicos. [13]
Con fagos líticos como el fago T4 , las células bacterianas se abren (se liza) y se destruyen después de la replicación inmediata del virión. Tan pronto como se destruye la célula, la progenie del fago puede encontrar nuevos huéspedes para infectar. [13] Los fagos líticos son más adecuados para la terapia con fagos . Algunos fagos líticos experimentan un fenómeno conocido como inhibición de la lisis, donde la progenie del fago completa no se lisará inmediatamente fuera de la célula si las concentraciones de fagos extracelulares son altas. Este mecanismo no es idéntico al del fago templado que entra en estado latente y generalmente es temporal. [52]
Por el contrario, el ciclo lisogénico no produce una lisis inmediata de la célula huésped. Los fagos capaces de experimentar lisogenia se conocen como fagos templados . Su genoma viral se integrará con el ADN del huésped y se replicará junto con él, de forma relativamente inofensiva, o incluso puede establecerse como un plásmido . El virus permanece inactivo hasta que las condiciones del huésped se deterioran, tal vez debido al agotamiento de nutrientes, luego, los fagos endógenos (conocidos como profagos ) se activan. En este punto, inician el ciclo reproductivo, lo que resulta en la lisis de la célula huésped. A medida que el ciclo lisogénico permite que la célula huésped continúe sobreviviendo y reproduciéndose, el virus se replica en toda la descendencia de la célula. Un ejemplo de un bacteriófago conocido por seguir el ciclo lisogénico y el ciclo lítico es el fago lambda de E. coli. [53]
A veces, los profagos pueden proporcionar beneficios a la bacteria huésped mientras están inactivos al agregar nuevas funciones al genoma bacteriano , en un fenómeno llamado conversión lisogénica . Algunos ejemplos son la conversión de cepas inofensivas de Corynebacterium diphtheriae o Vibrio cholerae por bacteriófagos en cepas altamente virulentas que causan difteria o cólera , respectivamente. [54] [55] Se han propuesto estrategias para combatir ciertas infecciones bacterianas apuntando a estos profagos que codifican toxinas. [56]

Las células bacterianas están protegidas por una pared celular de polisacáridos , que son factores de virulencia importantes que protegen a las células bacterianas contra las defensas inmunes del huésped y los antibióticos . [57] Para entrar en una célula huésped, los bacteriófagos se unen a receptores específicos en la superficie de las bacterias, incluidos lipopolisacáridos , ácidos teicoicos , proteínas o incluso flagelos . Esta especificidad significa que un bacteriófago puede infectar solo ciertas bacterias que tienen receptores a los que puede unirse, lo que a su vez determina el rango de huésped del fago. Las enzimas que degradan polisacáridos son proteínas asociadas a viriones que degradan enzimáticamente la capa externa capsular de sus huéspedes en el paso inicial de un proceso de infección de fagos estrictamente programado. [ cita requerida ] Las condiciones de crecimiento del huésped también influyen en la capacidad del fago para unirse e invadirlos. [58] Como los viriones de los fagos no se mueven de forma independiente, deben depender de encuentros aleatorios con los receptores correctos cuando están en solución, como la sangre, la circulación linfática, la irrigación, el agua del suelo, etc. [ cita requerida ]
Los bacteriófagos miovirus utilizan un movimiento similar al de una jeringa hipodérmica para inyectar su material genético en la célula. Después de entrar en contacto con el receptor apropiado, las fibras de la cola se flexionan para acercar la placa base a la superficie de la célula. Esto se conoce como unión reversible. Una vez que se han unido por completo, se inicia la unión irreversible y la cola se contrae, posiblemente con la ayuda del ATP presente en la cola, [6] inyectando material genético a través de la membrana bacteriana. [59] La inyección se lleva a cabo mediante una especie de movimiento de flexión del eje yendo hacia un lado, contrayéndose más cerca de la célula y empujando hacia arriba. Los podovirus carecen de una vaina de cola alargada como la de un miovirus, por lo que, en su lugar, utilizan sus pequeñas fibras de cola similares a dientes de forma enzimática para degradar una parte de la membrana celular antes de insertar su material genético.
En cuestión de minutos, los ribosomas bacterianos comienzan a traducir el ARNm viral en proteína. Para los fagos basados en ARN, la replicasa de ARN se sintetiza al principio del proceso. Las proteínas modifican la ARN polimerasa bacteriana para que transcriba preferentemente el ARNm viral. La síntesis normal de proteínas y ácidos nucleicos del huésped se ve interrumpida, y se ve obligado a fabricar productos virales en su lugar. Estos productos pasan a formar parte de nuevos viriones dentro de la célula, proteínas auxiliares que contribuyen al ensamblaje de nuevos viriones o proteínas involucradas en la lisis celular . En 1972, Walter Fiers ( Universidad de Ghent , Bélgica ) fue el primero en establecer la secuencia de nucleótidos completa de un gen y, en 1976, del genoma viral del bacteriófago MS2 . [60] Algunos bacteriófagos dsADN codifican proteínas ribosómicas, que se cree que modulan la traducción de proteínas durante la infección del fago. [61]
En el caso del fago T4 , la construcción de nuevas partículas virales implica la asistencia de proteínas auxiliares que actúan catalíticamente durante la morfogénesis del fago . [62] Las placas base se ensamblan primero, y las colas se construyen sobre ellas después. Las cápsides de la cabeza, construidas por separado, se ensamblarán espontáneamente con las colas. Durante el ensamblaje del virión del fago T4 , las proteínas morfogenéticas codificadas por los genes del fago interactúan entre sí en una secuencia característica. Mantener un equilibrio apropiado en las cantidades de cada una de estas proteínas producidas durante la infección viral parece ser crítico para la morfogénesis normal del fago T4 . [63] El ADN se empaqueta eficientemente dentro de las cabezas. [64] Todo el proceso lleva unos 15 minutos.
Los primeros estudios del bacteriófago T4 (1962-1964) proporcionaron una oportunidad para comprender prácticamente todos los genes que son esenciales para el crecimiento del bacteriófago en condiciones de laboratorio. [65] [66] Estos estudios fueron posibles gracias a la disponibilidad de dos clases de mutantes letales condicionales . [67] Una clase de estos mutantes se denominó mutantes ámbar . [67] La otra clase de mutantes letales condicionales se denominó mutantes sensibles a la temperatura . [68] Los estudios de estas dos clases de mutantes condujeron a un conocimiento considerable de las funciones e interacciones de las proteínas empleadas en la maquinaria de replicación , reparación y recombinación del ADN , y sobre cómo se ensamblan los virus a partir de componentes de proteínas y ácidos nucleicos ( morfogénesis molecular ).
Los fagos pueden liberarse por lisis celular, por extrusión o, en algunos casos, por gemación. La lisis, por parte de los fagos con cola, se logra mediante una enzima llamada endolisina , que ataca y descompone el peptidoglicano de la pared celular . Un tipo de fago completamente diferente, el fago filamentoso , hace que la célula huésped secrete continuamente nuevas partículas de virus. Los viriones liberados se describen como libres y, a menos que sean defectuosos, son capaces de infectar una nueva bacteria. La gemación está asociada con ciertos fagos de Mycoplasma . A diferencia de la liberación de viriones, los fagos que muestran un ciclo lisogénico no matan al huésped y, en cambio, se convierten en residentes a largo plazo como profagos . [69]
Una investigación realizada en 2017 reveló que el bacteriófago Φ3T produce una proteína viral corta que envía señales a otros bacteriófagos para que permanezcan inactivos en lugar de matar a la bacteria huésped. Arbitrium es el nombre que le dieron a esta proteína los investigadores que la descubrieron. [70] [71]
Dados los millones de fagos diferentes que existen en el medio ambiente, los genomas de los fagos se presentan en una variedad de formas y tamaños. Los fagos de ARN como MS2 tienen los genomas más pequeños, con solo unas pocas kilobases. Sin embargo, algunos fagos de ADN como T4 pueden tener genomas grandes con cientos de genes; el tamaño y la forma de la cápside varían junto con el tamaño del genoma. [72] Los genomas de bacteriófagos más grandes alcanzan un tamaño de 735 kb. [73]

Los genomas de los bacteriófagos pueden ser altamente mosaicos , es decir, el genoma de muchas especies de fagos parece estar compuesto de numerosos módulos individuales. Estos módulos pueden encontrarse en otras especies de fagos en diferentes disposiciones. Los micobacteriófagos , bacteriófagos con huéspedes micobacterianos , han proporcionado excelentes ejemplos de este mosaicismo. En estos micobacteriófagos, la variedad genética puede ser el resultado de instancias repetidas de recombinación específica del sitio y recombinación ilegítima (el resultado de la adquisición del genoma del fago de secuencias genéticas del huésped bacteriano). [75] Los mecanismos evolutivos que dan forma a los genomas de los virus bacterianos varían entre diferentes familias y dependen del tipo de ácido nucleico, las características de la estructura del virión, así como el modo del ciclo de vida viral. [76]
Algunos fagos marinos de roseobacter contienen desoxiuridina (dU) en lugar de desoxitimidina (dT) en su ADN genómico. Hay algunas pruebas de que este componente inusual es un mecanismo para evadir los mecanismos de defensa bacterianos, como las endonucleasas de restricción y los sistemas CRISPR/Cas que evolucionaron para reconocer y escindir secuencias dentro de los fagos invasores, inactivándolos así. Se sabe desde hace mucho tiempo que otros fagos utilizan nucleótidos inusuales. En 1963, Takahashi y Marmur identificaron un fago de Bacillus que tiene dU sustituyendo a dT en su genoma [77] y en 1977, Kirnos et al. identificaron un cianófago que contiene 2-aminoadenina (Z) en lugar de adenina (A). [78]
El campo de la biología de sistemas investiga las redes complejas de interacciones dentro de un organismo, generalmente utilizando herramientas computacionales y modelos. [79] Por ejemplo, un genoma de fago que ingresa a una célula huésped bacteriana puede expresar cientos de proteínas de fago que afectarán la expresión de numerosos genes del huésped o el metabolismo del huésped . Todas estas interacciones complejas se pueden describir y simular en modelos informáticos. [79]
Por ejemplo, la infección de Pseudomonas aeruginosa por el fago templado PaP3 modificó la expresión del 38% (2160/5633) de los genes de su hospedador. Muchos de estos efectos son probablemente indirectos, por lo que el desafío es identificar las interacciones directas entre las bacterias y el fago. [80]
Se han hecho varios intentos para mapear las interacciones proteína-proteína entre los fagos y su huésped. Por ejemplo, se encontró que el bacteriófago lambda interactuaba con su huésped, E. coli , mediante docenas de interacciones. Una vez más, la importancia de muchas de estas interacciones sigue sin estar clara, pero estos estudios sugieren que es muy probable que haya varias interacciones clave y muchas interacciones indirectas cuyo papel sigue sin caracterizarse. [81]
Los bacteriófagos son una amenaza importante para las bacterias y los procariotas han desarrollado numerosos mecanismos para bloquear la infección o la replicación de bacteriófagos dentro de las células huésped. El sistema CRISPR es uno de esos mecanismos, al igual que los retrones y el sistema antitoxina codificado por ellos. [82] Se sabe que el sistema de defensa Thoeris implementa una estrategia única para la resistencia antifágica bacteriana a través de la degradación de NAD+ . [83]
Los fagos templados son bacteriófagos que integran su material genético en el huésped como episomas extracromosómicos o como un profago durante un ciclo lisogénico . [84] [85] [86] Algunos fagos templados pueden conferir ventajas de aptitud a su huésped de numerosas maneras, incluyendo dando resistencia a los antibióticos a través de la transferencia o introducción de genes de resistencia a los antibióticos (ARGs), [85] [87] protegiendo a los huéspedes de la fagocitosis, [88] [89] protegiendo a los huéspedes de la infección secundaria a través de la exclusión de la superinfección, [90] [91] [92] mejorando la patogenicidad del huésped, [84] [93] o mejorando el metabolismo o crecimiento bacteriano. [94] [95] [96] [97] La simbiosis bacteriófago-huésped puede beneficiar a las bacterias al proporcionar ventajas selectivas mientras se replica pasivamente el genoma del fago. [98]
La metagenómica ha permitido la detección de bacteriófagos en el agua, algo que antes no era posible. [99]
Además, los bacteriófagos se han utilizado en el rastreo y modelado hidrológico de sistemas fluviales , especialmente donde se producen interacciones entre aguas superficiales y subterráneas . El uso de fagos se prefiere al marcador de colorante más convencional porque se absorben significativamente menos al pasar a través de aguas subterráneas y se detectan fácilmente en concentraciones muy bajas. [100] El agua no contaminada puede contener aproximadamente 2×10 8 bacteriófagos por ml. [101]
Se cree que los bacteriófagos contribuyen ampliamente a la transferencia horizontal de genes en entornos naturales, principalmente a través de la transducción , pero también a través de la transformación . [102] Los estudios basados en la metagenómica también han revelado que los viromas de una variedad de entornos albergan genes de resistencia a los antibióticos, incluidos aquellos que podrían conferir resistencia a múltiples fármacos . [103]
Hallazgos recientes han mapeado el arsenal complejo e interrelacionado de herramientas de defensa antifagos en bacterias ambientales. [104]
Aunque los fagos no infectan a los humanos, existen innumerables partículas de fagos en el cuerpo humano, dado el extenso microbioma humano . La población de fagos de una persona se ha denominado fagoma humano , que incluye el "fagoma intestinal sano" (HGP) y el "fagoma humano enfermo" (DHP). [105] Se ha estimado que el fagoma activo de un humano sano (es decir, que se replica activamente en lugar de un profago integrado que no se replica ) comprende de docenas a miles de virus diferentes. [106] Hay evidencia de que los bacteriófagos y las bacterias interactúan en el microbioma intestinal humano tanto de forma antagónica como beneficiosa. [107]
Estudios preliminares han indicado que los bacteriófagos comunes se encuentran en el 62% de los individuos sanos en promedio, mientras que su prevalencia se redujo en un 42% y un 54% en promedio en pacientes con colitis ulcerosa (CU) y enfermedad de Crohn (EC). [105] La abundancia de fagos también puede disminuir en los ancianos. [107]
Los fagos más comunes en el intestino humano, que se encuentran en todo el mundo, son los crAssphages . Los crAssphages se transmiten de madre a hijo poco después del nacimiento, y hay algunas pruebas que sugieren que pueden transmitirse localmente. Cada persona desarrolla sus propios grupos de crAssphage únicos. Los fagos similares a los crAss también pueden estar presentes en primates además de en humanos. [107]
Entre los innumerables fagos, sólo unos pocos han sido estudiados en detalle, incluidos algunos fagos de importancia histórica que se descubrieron en los primeros tiempos de la genética microbiana. Estos, especialmente el fago T, ayudaron a descubrir principios importantes de la estructura y la función de los genes.
{{cite journal}}: CS1 maint: bot: estado de URL original desconocido ( enlace )