En biología , la traducción es el proceso en las células vivas en el que se producen proteínas utilizando moléculas de ARN como plantillas. La proteína generada es una secuencia de aminoácidos . Esta secuencia está determinada por la secuencia de nucleótidos en el ARN. Los nucleótidos se consideran de tres en tres. Cada uno de estos tripletes da como resultado la adición de un aminoácido específico a la proteína que se está generando. La correspondencia entre el triplete de nucleótidos y el aminoácido se denomina código genético . La traducción la realiza un gran complejo de ARN y proteínas funcionales llamados ribosomas . El proceso completo se denomina expresión génica .
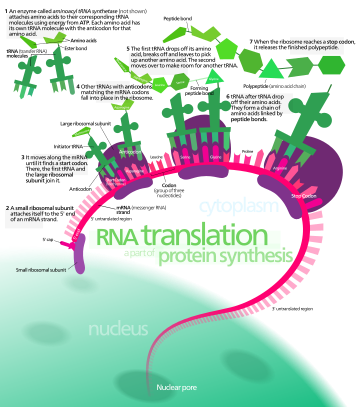
En la traducción, el ARN mensajero (ARNm) se decodifica en un ribosoma, fuera del núcleo, para producir una cadena de aminoácidos específica, o polipéptido . El polipéptido luego se pliega en una proteína activa y realiza sus funciones en la célula. El ribosoma facilita la decodificación al inducir la unión de secuencias anticodón del ARN de transferencia complementario (ARNt) a los codones del ARNm . Los ARNt transportan aminoácidos específicos que se encadenan entre sí para formar un polipéptido a medida que el ARNm pasa a través de ellos y es "leído" por el ribosoma.
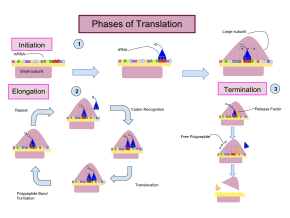
La traducción se desarrolla en tres fases:
En los procariotas (bacterias y arqueas), la traducción ocurre en el citosol, donde las subunidades grande y pequeña del ribosoma se unen al ARNm. En los eucariotas , la traducción ocurre en el citoplasma o a través de la membrana del retículo endoplasmático en un proceso llamado translocación co-traduccional . En la translocación co-traduccional, todo el complejo ribosoma/ARNm se une a la membrana externa del retículo endoplasmático rugoso (RE), y la nueva proteína se sintetiza y se libera en el RE; el polipéptido recién creado puede almacenarse dentro del RE para su futuro transporte en vesículas y secreción fuera de la célula, o secretarse inmediatamente.
Muchos tipos de ARN transcrito, como el ARNt, el ARN ribosómico y el ARN nuclear pequeño, no experimentan una traducción a proteínas.
Varios antibióticos actúan inhibiendo la traducción. Entre ellos se encuentran la anisomicina , la cicloheximida , el cloranfenicol , la tetraciclina , la estreptomicina , la eritromicina y la puromicina . Los ribosomas procariotas tienen una estructura diferente a la de los ribosomas eucariotas, por lo que los antibióticos pueden dirigirse específicamente a las infecciones bacterianas sin causar daño a las células del huésped eucariota.
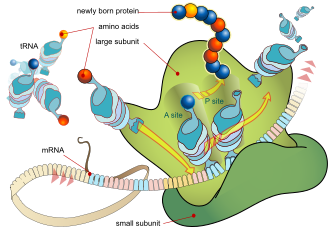
El proceso básico de producción de proteínas es la adición de un aminoácido a la vez al final de una proteína. Esta operación la realiza un ribosoma . [1] Un ribosoma se compone de dos subunidades, una subunidad pequeña y una subunidad grande. Estas subunidades se unen antes de la traducción del ARNm en una proteína para proporcionar una ubicación para que se lleve a cabo la traducción y se produzca un polipéptido. [2] La elección del tipo de aminoácido a agregar está determinada por una molécula de ARN mensajero (ARNm). Cada aminoácido agregado se combina con una subsecuencia de tres nucleótidos del ARNm. Para cada triplete posible, se acepta el aminoácido correspondiente. Los aminoácidos sucesivos agregados a la cadena se combinan con tripletes de nucleótidos sucesivos en el ARNm. De esta manera, la secuencia de nucleótidos en la cadena de ARNm molde determina la secuencia de aminoácidos en la cadena de aminoácidos generada. [3] La adición de un aminoácido ocurre en el extremo C del péptido; Por lo tanto, se dice que la traducción está dirigida de amina a carboxilo. [4]
El ARNm lleva la información genética codificada como una secuencia de ribonucleótidos desde los cromosomas hasta los ribosomas. Los ribonucleótidos son "leídos" por la maquinaria de traducción en una secuencia de tripletes de nucleótidos llamados codones. Cada uno de esos tripletes codifica un aminoácido específico . [ cita requerida ]
Las moléculas del ribosoma traducen este código a una secuencia específica de aminoácidos. El ribosoma es una estructura de múltiples subunidades que contiene ARN ribosómico (ARNr) y proteínas. Es la "fábrica" donde los aminoácidos se ensamblan para formar proteínas.
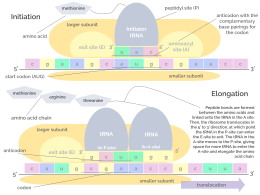
Los ARN de transferencia (ARNt) son pequeñas cadenas de ARN no codificantes (74-93 nucleótidos) que transportan aminoácidos al ribosoma. El repertorio de genes de ARNt varía ampliamente entre especies: algunas bacterias tienen entre 20 y 30 genes, mientras que los eucariotas complejos pueden tener miles. [5] Los ARNt tienen un sitio para la unión de aminoácidos y un sitio llamado anticodón. El anticodón es un triplete de ARN complementario al triplete de ARNm que codifica su aminoácido de carga .
Las aminoacil-ARNt sintetasas ( enzimas ) catalizan la unión entre ARNt específicos y los aminoácidos que requieren sus secuencias anticodón. El producto de esta reacción es un aminoacil-ARNt . El aminoácido se une por su grupo carboxilo al 3' OH del ARNt mediante un enlace éster . Cuando el ARNt tiene un aminoácido unido a él, el ARNt se denomina "cargado". En las bacterias, este aminoacil-ARNt es transportado al ribosoma por EF-Tu , donde los codones del ARNm se emparejan mediante el apareamiento de bases complementarias con anticodones específicos del ARNt. Las aminoacil-ARNt sintetasas que emparejan incorrectamente los ARNt con los aminoácidos incorrectos pueden producir aminoacil-ARNt mal cargados, lo que puede dar lugar a aminoácidos inapropiados en la posición respectiva en la proteína. Esta "traducción errónea" [6] del código genético ocurre naturalmente en niveles bajos en la mayoría de los organismos, pero ciertos entornos celulares causan un aumento en la decodificación permisiva del ARNm, a veces en beneficio de la célula.
El ribosoma tiene dos sitios de unión para el ARNt. Son el sitio aminoacilo (abreviado A) y el sitio peptidilo/sitio de salida (abreviado P/E). Con respecto al ARNm, los tres sitios están orientados de 5' a 3' EPA, porque los ribosomas se mueven hacia el extremo 3' del ARNm. El sitio A une el ARNt entrante con el codón complementario en el ARNm. El sitio P/E mantiene el ARNt con la cadena polipeptídica en crecimiento. Cuando un aminoacilo-ARNt se une inicialmente a su codón correspondiente en el ARNm, está en el sitio A. Luego, se forma un enlace peptídico entre el aminoácido del ARNt en el sitio A y el aminoácido del ARNt cargado en el sitio P/E. La cadena polipeptídica en crecimiento se transfiere al ARNt en el sitio A. Se produce la translocación, moviendo el ARNt al sitio P/E, ahora sin un aminoácido; El ARNt que estaba en el sitio A, ahora cargado con la cadena polipeptídica, se mueve al sitio P/E y el ARNt no cargado sale, y otro aminoacil-ARNt ingresa al sitio A para repetir el proceso. [7]
Después de que el nuevo aminoácido se añade a la cadena, y después de que el ARNt se libera del ribosoma al citosol, la energía proporcionada por la hidrólisis de un GTP unido a la translocasa EF-G (en bacterias ) y a/eEF-2 (en eucariotas y arqueas ) mueve el ribosoma un codón hacia abajo hacia el extremo 3' . La energía requerida para la traducción de proteínas es significativa. Para una proteína que contiene n aminoácidos, el número de enlaces de fosfato de alta energía necesarios para traducirla es 4 n -1. [8] La tasa de traducción varía; es significativamente mayor en células procariotas (hasta 17-21 residuos de aminoácidos por segundo) que en células eucariotas (hasta 6-9 residuos de aminoácidos por segundo). [9]
La iniciación implica la unión de la subunidad pequeña del ribosoma al extremo 5' del ARNm con la ayuda de factores de iniciación (IF). En las bacterias y una minoría de arqueas, la iniciación de la síntesis de proteínas implica el reconocimiento de una secuencia de iniciación rica en purinas en el ARNm llamada secuencia Shine-Dalgarno . La secuencia Shine-Dalgarno se une a una secuencia complementaria rica en pirimidinas en el extremo 3' de la parte 16S del ARNr de la subunidad ribosómica 30S. La unión de estas secuencias complementarias garantiza que la subunidad ribosómica 30S esté unida al ARNm y esté alineada de tal manera que el codón de iniciación se coloque en la porción 30S del sitio P. Una vez que el ARNm y la subunidad 30S están unidos correctamente, un factor de iniciación lleva el complejo ARNt-aminoácido iniciador, f-Met -ARNt, al sitio P 30S. La fase de iniciación se completa una vez que una subunidad 50S se une a la subunidad 30S, formando un ribosoma 70S activo. [10] La terminación del polipéptido ocurre cuando el sitio A del ribosoma está ocupado por un codón de terminación (UAA, UAG o UGA) en el ARNm, creando la estructura primaria de una proteína. El ARNt generalmente no puede reconocer ni unirse a los codones de terminación. En cambio, el codón de terminación induce la unión de una proteína de factor de liberación [11] (RF1 y RF2) que impulsa el desmontaje de todo el complejo ribosoma/ARNm por la hidrólisis de la cadena polipeptídica del centro de la peptidil transferasa [1] del ribosoma. [12] Los fármacos o motivos de secuencia especiales en el ARNm pueden cambiar la estructura ribosómica de modo que los ARNt casi cognados se unan al codón de terminación en lugar de a los factores de liberación. En tales casos de "lectura transcripcional", la traducción continúa hasta que el ribosoma encuentra el siguiente codón de terminación. [13]
Aunque los ribosomas suelen considerarse máquinas precisas y procesivas, el proceso de traducción está sujeto a errores que pueden conducir a la síntesis de proteínas erróneas o al abandono prematuro de la traducción, ya sea porque un ARNt se acopla a un codón incorrecto o porque un ARNt se acopla al aminoácido incorrecto. [14] Se ha estimado que la tasa de error en la síntesis de proteínas es de entre 1 en 10 5 y 1 en 10 3 aminoácidos mal incorporados, dependiendo de las condiciones experimentales. [15] En cambio, se ha estimado que la tasa de abandono prematuro de la traducción es del orden de magnitud de 10 −4 eventos por codón traducido. [16] [17]
El proceso de traducción está altamente regulado tanto en organismos eucariotas como procariotas. La regulación de la traducción puede afectar la tasa global de síntesis de proteínas, que está estrechamente relacionada con el estado metabólico y proliferativo de una célula.
Para profundizar en este intrincado proceso, los científicos suelen utilizar una técnica conocida como perfil ribosómico. [18] Este método permite a los investigadores tomar una instantánea del translatoma, mostrando qué partes del ARNm están siendo traducidas en proteínas por los ribosomas en un momento dado. El perfil ribosómico proporciona información valiosa sobre la dinámica de la traducción, revelando la compleja interacción entre la secuencia genética, la estructura del ARNm y la regulación de la traducción. Por ejemplo, la investigación que utiliza este método ha revelado que las diferencias genéticas y su posterior expresión como ARNm también pueden afectar la tasa de traducción de una manera específica del ARN. [19]
Ampliando este concepto, un desarrollo más reciente es el perfilado de ribosomas de células individuales, una técnica que nos permite estudiar el proceso de traducción a nivel de células individuales. [20] Esto es particularmente significativo ya que las células, incluso las del mismo tipo, pueden exhibir una variabilidad considerable en su síntesis de proteínas. El perfilado de ribosomas de células individuales tiene el potencial de arrojar luz sobre la naturaleza heterogénea de las células, lo que conduce a una comprensión más matizada de cómo la regulación de la traducción puede afectar el comportamiento celular, el estado metabólico y la capacidad de respuesta a diversos estímulos o condiciones.
El control de la traducción es fundamental para el desarrollo y la supervivencia del cáncer . Las células cancerosas deben regular con frecuencia la fase de traducción de la expresión génica, aunque no se entiende completamente por qué la traducción se dirige a pasos como la transcripción. Si bien las células cancerosas a menudo tienen factores de traducción genéticamente alterados, es mucho más común que las células cancerosas modifiquen los niveles de factores de traducción existentes. [21] Varias vías de señalización oncogénica importantes, incluidas las vías RAS–MAPK , PI3K/AKT/mTOR , MYC y WNT–β-catenina , en última instancia reprograman el genoma a través de la traducción. [22] Las células cancerosas también controlan la traducción para adaptarse al estrés celular. Durante el estrés, la célula traduce ARNm que pueden mitigar el estrés y promover la supervivencia. Un ejemplo de esto es la expresión de AMPK en varios cánceres; su activación desencadena una cascada que en última instancia puede permitir que el cáncer escape a la apoptosis (muerte celular programada) desencadenada por la privación de nutrición. Las futuras terapias contra el cáncer pueden implicar la interrupción de la maquinaria de traducción de la célula para contrarrestar los efectos posteriores del cáncer. [21]
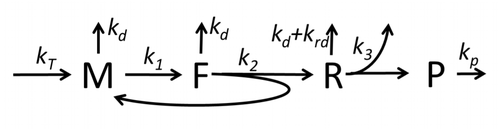
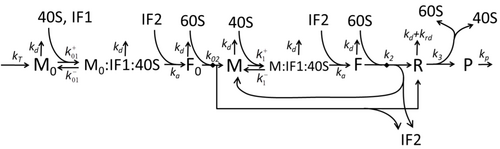
La descripción del proceso de transcripción-traducción, mencionando sólo los procesos "elementales" más básicos, consta de:
El proceso de construcción de aminoácidos para crear proteínas en la traducción es un tema de varios modelos físicos durante mucho tiempo comenzando con los primeros modelos cinéticos detallados como [24] u otros que tienen en cuenta los aspectos estocásticos de la traducción y utilizan simulaciones por computadora. Muchos modelos basados en cinética química de síntesis de proteínas se han desarrollado y analizado en las últimas cuatro décadas. [25] [26] Más allá de la cinética química, varios formalismos de modelado como el Proceso de Exclusión Simple Totalmente Asimétrico , [26] Redes Booleanas Probabilísticas , Redes de Petri y álgebra max-plus se han aplicado para modelar la cinética detallada de la síntesis de proteínas o algunas de sus etapas. Se ha desarrollado un modelo básico de síntesis de proteínas que tiene en cuenta los ocho procesos "elementales", [23] siguiendo el paradigma de que " los modelos útiles son simples y extensibles". [27] El modelo más simple M0 está representado por el mecanismo cinético de reacción (Figura M0). Se generalizó para incluir la unión de 40S, 60S y factores de iniciación (IF) (Figura M1'). Se amplió aún más para incluir el efecto del microARN en la síntesis de proteínas. [28] La mayoría de los modelos en esta jerarquía se pueden resolver analíticamente. Estas soluciones se utilizaron para extraer "firmas cinéticas" de diferentes mecanismos específicos de regulación de la síntesis.
También es posible traducir a mano (para secuencias cortas) o por computadora (después de programarla adecuadamente, ver la sección a continuación); esto permite a los biólogos y químicos dibujar la estructura química de la proteína codificada en papel.
En primer lugar, convierta cada base de ADN molde en su complemento de ARN (observe que el complemento de A ahora es U), como se muestra a continuación. Observe que la cadena molde del ADN es aquella contra la que se polimeriza el ARN; la otra cadena de ADN sería la misma que la del ARN, pero con timina en lugar de uracilo.
ADN -> ARN A -> U T -> A C -> G G -> C A=T-> A=U
Luego, divide el ARN en tripletes (grupos de tres bases). Ten en cuenta que hay tres "ventanas" de traducción, o marcos de lectura , según dónde comiences a leer el código. Por último, utiliza la tabla de Código genético para traducir lo anterior a una fórmula estructural como la que se utiliza en química.
Esto dará la estructura primaria de la proteína. Sin embargo, las proteínas tienden a plegarse , dependiendo en parte de los segmentos hidrofílicos e hidrofóbicos a lo largo de la cadena. A menudo, todavía se puede adivinar la estructura secundaria , pero la estructura terciaria adecuada suele ser muy difícil de determinar.
Mientras que otros aspectos como la estructura 3D, llamada estructura terciaria , de la proteína sólo se pueden predecir utilizando algoritmos sofisticados , la secuencia de aminoácidos, llamada estructura primaria, se puede determinar únicamente a partir de la secuencia de ácidos nucleicos con la ayuda de una tabla de traducción .
Es posible que este enfoque no proporcione la composición correcta de aminoácidos de la proteína, en particular si se incorporan a la proteína aminoácidos no convencionales como la selenocisteína , que está codificada por un codón de terminación convencional en combinación con una horquilla corriente abajo (secuencia de inserción de selenocisteína o SECIS).
Existen muchos programas informáticos capaces de traducir una secuencia de ADN/ARN en una secuencia de proteínas. Normalmente, esto se lleva a cabo utilizando el Código Genético Estándar, sin embargo, pocos programas pueden manejar todos los casos "especiales", como el uso de codones de iniciación alternativos que son biológicamente significativos. Por ejemplo, el raro codón de inicio alternativo CTG codifica la metionina cuando se utiliza como codón de inicio y la leucina en todas las demás posiciones.
Ejemplo: Tabla de traducción condensada del Código Genético Estándar (de la página web de Taxonomía del NCBI). [29]
AAs = FFLLSSSSYY**CC*WLLLLPPPPHHQQRRRRIIIMTTTTNNKKSSRRVVVVAAAADDEEGGGG Comienza = ---M---------------M---------------M---------------------------- Base1 = TTTTTTTTTTTTTTTTCCCCCCCCCCCCCCAAAAAAAAAAAAAAAAGGGGGGGGGGGGGGGG Base2 = TTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGGTTTTCCCCAAAAGGGG Base3 = TCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAGTCAG
La fila "Inicios" indica tres codones de inicio: UUG, CUG y el muy común AUG. También indica el primer residuo de aminoácido cuando se interpreta como inicio: en este caso, es todo metionina.
Incluso cuando se trabaja con secuencias eucariotas comunes, como el genoma de la levadura , a menudo se desea poder utilizar tablas de traducción alternativas, en concreto para la traducción de los genes mitocondriales. En la actualidad, el Grupo de Taxonomía del NCBI define las siguientes tablas de traducción para la traducción de las secuencias en GenBank : [29]
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace )