La genética evolutiva humana estudia las diferencias entre genomas humanos , el pasado evolutivo que dio origen al genoma humano y sus efectos actuales. Las diferencias entre genomas tienen implicaciones y aplicaciones antropológicas , médicas , históricas y forenses . Los datos genéticos pueden proporcionar información importante sobre la evolución humana .
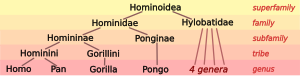
Los biólogos clasifican a los humanos , junto con solo unas pocas otras especies , como grandes simios (especies de la familia Hominidae ). Los Hominidae vivos incluyen dos especies distintas de chimpancés (el bonobo , Pan paniscus , y el chimpancé , Pan troglodytes ), dos especies de gorilas (el gorila occidental , Gorilla gorilla , y el gorila oriental , Gorilla graueri ), y dos especies de orangutanes (el orangután de Borneo , Pongo pygmaeus , y el orangután de Sumatra , Pongo abelii ). Los grandes simios con la familia Hylobatidae de gibones forman la superfamilia Hominoidea de simios .
Los simios, a su vez, pertenecen al orden de los primates (>400 especies), junto con los monos del Viejo Mundo , los monos del Nuevo Mundo y otros. Los datos tanto del ADN mitocondrial (ADNmt) como del ADN nuclear (ADNn) indican que los primates pertenecen al grupo de los Euarchontoglires , junto con Rodentia , Lagomorpha , Dermoptera y Scandentia . [1] Esto está respaldado además por los elementos nucleares cortos intercalados (SINE) similares a Alu que se han encontrado solo en miembros de los Euarchontoglires. [2]
Un árbol filogenético suele derivarse de secuencias de ADN o proteínas de poblaciones. A menudo, se utilizan secuencias de ADN mitocondrial o del cromosoma Y para estudiar la demografía humana antigua. Estas fuentes de ADN de un solo locus no se recombinan y casi siempre se heredan de un solo progenitor, con una única excepción conocida en el ADNmt. [3] Los individuos de regiones geográficas más cercanas suelen ser más similares que los individuos de regiones más lejanas. La distancia en un árbol filogenético se puede utilizar de forma aproximada para indicar:
La separación de los humanos de sus parientes más cercanos, los simios africanos no humanos (chimpancés y gorilas), ha sido objeto de estudios exhaustivos durante más de un siglo. Se han abordado cinco cuestiones principales:
Como se ha comentado antes, las distintas partes del genoma muestran divergencias de secuencias diferentes entre los distintos homínidos . También se ha demostrado que la divergencia de secuencias entre el ADN de los humanos y el de los chimpancés varía considerablemente. Por ejemplo, la divergencia de secuencias varía entre el 0% y el 2,66% entre las regiones genómicas no codificantes y no repetitivas de los humanos y los chimpancés. [8] El porcentaje de nucleótidos en el genoma humano (hg38) que tenían coincidencias exactas uno a uno con el genoma del chimpancé (pantro6) fue del 84,38%. Además, los árboles genéticos, generados mediante análisis comparativos de segmentos de ADN, no siempre encajan en el árbol de especies. Resumiendo:
El tiempo de divergencia de los humanos respecto de otros simios es de gran interés. Uno de los primeros estudios moleculares, publicado en 1967, midió las distancias inmunológicas (DI) entre diferentes primates. [10] Básicamente, el estudio midió la fuerza de la respuesta inmunológica que un antígeno de una especie (albúmina humana) induce en el sistema inmunológico de otra especie (humano, chimpancé, gorila y monos del Viejo Mundo ). Las especies estrechamente relacionadas deberían tener antígenos similares y, por lo tanto, una respuesta inmunológica más débil a los antígenos de las demás. La respuesta inmunológica de una especie a sus propios antígenos (por ejemplo, de humano a humano) se estableció en 1.
La identificación entre humanos y gorilas se determinó en 1,09, y entre humanos y chimpancés en 1,14. Sin embargo, la distancia a seis monos del Viejo Mundo diferentes fue en promedio de 2,46, lo que indica que los simios africanos están más estrechamente relacionados con los humanos que con los monos. Los autores consideran que el tiempo de divergencia entre los monos del Viejo Mundo y los homínidos fue hace 30 millones de años (MYA), según los datos fósiles, y se consideró que la distancia inmunológica aumentaba a un ritmo constante. Concluyeron que el tiempo de divergencia de los humanos y los simios africanos fue de aproximadamente ~5 MYA. Ese fue un resultado sorprendente. La mayoría de los científicos en ese momento pensaban que los humanos y los grandes simios divergieron mucho antes (>15 MYA).
El gorila estaba, en términos de DI, más cerca de los humanos que de los chimpancés; sin embargo, la diferencia era tan leve que la tricotomía no pudo resolverse con certeza. Estudios posteriores basados en genética molecular pudieron resolver la tricotomía: los chimpancés están filogenéticamente más cerca de los humanos que de los gorilas. Sin embargo, algunos tiempos de divergencia estimados posteriormente (utilizando métodos mucho más sofisticados en genética molecular) no difieren sustancialmente de la primera estimación de 1967, pero un artículo reciente [11] lo sitúa en 11-14 millones de años.

Los métodos actuales para determinar los tiempos de divergencia utilizan alineaciones de secuencias de ADN y relojes moleculares . Por lo general, el reloj molecular se calibra asumiendo que el orangután se separó de los simios africanos (incluidos los humanos) hace entre 12 y 16 millones de años. Algunos estudios también incluyen algunos monos del Viejo Mundo y establecen el tiempo de divergencia de ellos de los homínidos en 25-30 millones de años. Ambos puntos de calibración se basan en muy pocos datos fósiles y han sido criticados. [12]
Si se revisan estas fechas, los tiempos de divergencia estimados a partir de datos moleculares también cambiarán. Sin embargo, es poco probable que cambien los tiempos de divergencia relativos. Incluso si no podemos determinar con exactitud los tiempos de divergencia absolutos, podemos estar bastante seguros de que el tiempo de divergencia entre chimpancés y humanos es aproximadamente seis veces más corto que entre chimpancés (o humanos) y monos.
Un estudio (Takahata et al. , 1995) utilizó 15 secuencias de ADN de diferentes regiones del genoma de humanos y chimpancés y 7 secuencias de ADN de humanos, chimpancés y gorilas . [13] Determinaron que los chimpancés están más estrechamente relacionados con los humanos que los gorilas. Utilizando varios métodos estadísticos, estimaron que el tiempo de divergencia entre humanos y chimpancés era de 4,7 millones de años y el tiempo de divergencia entre gorilas y humanos (y chimpancés) era de 7,2 millones de años.
Además, estimaron que el tamaño efectivo de la población del ancestro común de los humanos y los chimpancés era de aproximadamente 100.000 individuos. Esto fue algo sorprendente, ya que se estima que el tamaño efectivo de la población humana actual es de solo aproximadamente 10.000 individuos. De ser cierto, eso significa que el linaje humano habría experimentado una inmensa disminución de su tamaño efectivo de población (y, por lo tanto, de la diversidad genética) en su evolución. (Ver la teoría de la catástrofe de Toba )

Otro estudio (Chen & Li, 2001) secuenció 53 segmentos de ADN intergénicos no repetitivos de humanos, chimpancés , gorilas y orangutanes . [8] Cuando las secuencias de ADN se concatenaron en una única secuencia larga, el árbol de unión de vecinos generado respaldó el clado Homo - Pan con un 100% de bootstrap (es decir, los humanos y los chimpancés son las especies relacionadas más cercanas de las cuatro). Cuando tres especies están bastante relacionadas entre sí (como los humanos, los chimpancés y los gorilas), los árboles obtenidos a partir de los datos de la secuencia de ADN pueden no ser congruentes con el árbol que representa la especiación (árbol de especies).
Cuanto más corto es el lapso internodal (T IN ), más comunes son los árboles genéticos incongruentes. El tamaño efectivo de la población (N e ) de la población internodal determina cuánto tiempo se conservan los linajes genéticos en la población. Un mayor tamaño efectivo de la población causa más árboles genéticos incongruentes. Por lo tanto, si se conoce el lapso internodal, se puede calcular el tamaño efectivo de la población ancestral del ancestro común de los humanos y los chimpancés.
Cuando se analizó cada segmento individualmente, 31 respaldaron el clado Homo - Pan , 10 respaldaron el clado Homo - Gorilla y 12 respaldaron el clado Pan - Gorilla . Utilizando el reloj molecular, los autores estimaron que los gorilas se separaron primero hace 6,2-8,4 millones de años y los chimpancés y los humanos se separaron entre 1,6 y 2,2 millones de años después (período internodal) hace 4,6-6,2 millones de años. El período internodal es útil para estimar el tamaño efectivo de la población ancestral del ancestro común de los humanos y los chimpancés.
Un análisis parsimonioso reveló que 24 loci apoyaban el clado Homo - Pan , 7 apoyaban el clado Homo - Gorilla , 2 apoyaban el clado Pan - Gorilla y 20 no daban ninguna resolución. Además, tomaron 35 loci de codificación de proteínas de bases de datos. De estos, 12 apoyaban el clado Homo - Pan , 3 el clado Homo - Gorilla , 4 el clado Pan - Gorilla y 16 no daban ninguna resolución. Por lo tanto, solo ~70% de los 52 loci que dieron una resolución (33 intergénicos, 19 codificadores de proteínas) apoyan el árbol de especies "correcto". A partir de la fracción de loci que no apoyaban el árbol de especies y el lapso de tiempo internodal que estimaron previamente, la población efectiva del ancestro común de humanos y chimpancés se estimó en ~52 000 a 96 000. Este valor no es tan alto como el del primer estudio (Takahata), pero aún así es mucho mayor que el tamaño de la población efectiva actual de humanos.
Un tercer estudio (Yang, 2002) utilizó el mismo conjunto de datos que Chen y Li, pero estimó la población ancestral efectiva en "solo" entre ~12.000 y 21.000, utilizando un método estadístico diferente. [14]
Los humanos y los chimpancés son idénticos en un 99,1% a nivel de codificación, con un 99,4% de similitud a nivel de no sinónimos y un 98,4% a nivel de sinónimos. [15] Las secuencias alineables dentro de los genomas de humanos y chimpancés difieren en aproximadamente 35 millones de sustituciones de un solo nucleótido. Además, aproximadamente el 3% de los genomas completos difieren en deleciones, inserciones y duplicaciones. [16]
Como la tasa de mutación es relativamente constante, aproximadamente la mitad de estos cambios se produjeron en el linaje humano. Sólo una fracción muy pequeña de esas diferencias fijas dieron lugar a los diferentes fenotipos de los humanos y los chimpancés, y encontrarlas es un gran desafío. La gran mayoría de las diferencias son neutrales y no afectan al fenotipo . [ cita requerida ]
La evolución molecular puede actuar de distintas maneras, a través de la evolución de proteínas, la pérdida de genes, la regulación diferencial de genes y la evolución del ARN. Se cree que todas ellas han desempeñado algún papel en la evolución humana.
Muchas mutaciones diferentes pueden inactivar un gen, pero pocas cambiarán su función de una manera específica. Por lo tanto, las mutaciones de inactivación estarán fácilmente disponibles para que la selección actúe sobre ellas. La pérdida de genes podría ser, por lo tanto, un mecanismo común de adaptación evolutiva (la hipótesis de "menos es más"). [17]
Se perdieron 80 genes en el linaje humano después de la separación del último ancestro común con el chimpancé. 36 de ellos eran para receptores olfativos . Los genes involucrados en la quimiorrecepción y la respuesta inmune están sobrerrepresentados. [18] Otro estudio estimó que se habían perdido 86 genes. [19]
En el linaje humano se perdió un gen de la queratina capilar de tipo I. La queratina es un componente importante del cabello. Los humanos aún tienen nueve genes de queratina capilar de tipo I funcionales, pero la pérdida de ese gen en particular puede haber causado el adelgazamiento del cabello corporal humano. Basándose en el supuesto de un reloj molecular constante, el estudio predice que la pérdida del gen ocurrió relativamente recientemente en la evolución humana, hace menos de 240 000 años, pero tanto la secuencia neandertal de Vindija como la secuencia de Denisovan de alta cobertura contienen los mismos codones de terminación prematuros que los humanos modernos y, por lo tanto, la datación debería ser mayor a 750 000 años atrás. [20]
Stedman et al. (2004) afirmaron que la pérdida del gen de miosina sarcomérica MYH16 en el linaje humano condujo a músculos masticatorios más pequeños . Estimaron que la mutación que llevó a la inactivación (una eliminación de dos pares de bases) ocurrió hace 2,4 millones de años, antes de la aparición del Homo ergaster /erectus en África. El período que siguió estuvo marcado por un fuerte aumento de la capacidad craneal , lo que promovió la especulación de que la pérdida del gen puede haber eliminado una restricción evolutiva en el tamaño del cerebro en el género Homo . [21]
Otra estimación de la pérdida del gen MYH16 es hace 5,3 millones de años, mucho antes de que apareciera el Homo . [22]
Las duplicaciones segmentarias (SD o LCR ) han desempeñado un papel en la creación de nuevos genes de primates y en la conformación de la variación genética humana.
Cuando se comparó el genoma humano con los genomas de cinco especies de primates de comparación, incluidos el chimpancé , el gorila , el orangután , el gibón y el macaco, se descubrió que hay aproximadamente 20.000 inserciones específicas de humanos que se cree que son reguladoras. Si bien la mayoría de las inserciones parecen ser neutrales en términos de aptitud, se ha identificado una pequeña cantidad en genes seleccionados positivamente que muestran asociaciones con fenotipos neuronales y algunos relacionados con fenotipos relacionados con la percepción sensorial y dental. Estos hallazgos insinúan el papel aparentemente importante de las inserciones específicas de humanos en la evolución reciente de los humanos. [23]
Las regiones aceleradas humanas son áreas del genoma que difieren entre humanos y chimpancés en mayor medida de lo que se puede explicar por la deriva genética a lo largo del tiempo desde que las dos especies compartieron un ancestro común. Estas regiones muestran signos de estar sujetas a la selección natural, lo que conduce a la evolución de rasgos distintivamente humanos. Dos ejemplos son HAR1F , que se cree que está relacionada con el desarrollo del cerebro y HAR2 (también conocido como HACNS1 ), que puede haber desempeñado un papel en el desarrollo del pulgar oponible .
También se ha planteado la hipótesis de que gran parte de la diferencia entre los humanos y los chimpancés se puede atribuir a la regulación de la expresión genética, más que a diferencias en los genes mismos. Los análisis de secuencias no codificantes conservadas , que a menudo contienen regiones reguladoras funcionales y, por lo tanto, seleccionadas positivamente, abordan esta posibilidad. [24]
Cuando se publicó el borrador de la secuencia del genoma del chimpancé común ( Pan troglodytes ) en el verano de 2005, se habían secuenciado y ensamblado 2400 millones de bases (de unos 3160 millones de bases) lo suficientemente bien como para compararlas con el genoma humano. [16] El 1,23% de esta secuencia difería en sustituciones de una sola base. De esto, se pensó que el 1,06% o menos representaba diferencias fijas entre las especies, y el resto eran sitios variantes en humanos o chimpancés. Otro tipo de diferencia, denominada indels (inserciones/deleciones), explicaba muchas menos diferencias (un 15% más), pero aportaba aproximadamente un 1,5% de secuencia única a cada genoma, ya que cada inserción o deleción puede implicar desde una base hasta millones de bases. [16]
En un artículo complementario se examinaron las duplicaciones segmentarias en los dos genomas, [25] cuya inserción y eliminación en el genoma explican gran parte de la secuencia indel. Se descubrió que un total del 2,7% de la secuencia eucromática se había duplicado de forma diferencial en uno u otro linaje.
La divergencia de secuencias tiene generalmente el siguiente patrón: humano-chimpancé < humano-gorila << humano-orangután, destacando el estrecho parentesco entre los humanos y los simios africanos. Los elementos Alu divergen rápidamente debido a su alta frecuencia de dinucleótidos CpG que mutan aproximadamente 10 veces más a menudo que el nucleótido promedio en el genoma. La tasa de mutación es mayor en la línea germinal masculina , por lo tanto, la divergencia en el cromosoma Y , que se hereda únicamente del padre, es mayor que en los autosomas . El cromosoma X se hereda con el doble de frecuencia a través de la línea germinal femenina que a través de la línea germinal masculina y, por lo tanto, muestra una divergencia de secuencia ligeramente menor. La divergencia de secuencia de la región Xq13.3 es sorprendentemente baja entre humanos y chimpancés. [26]
Las mutaciones que alteran la secuencia de aminoácidos de las proteínas (K a ) son las menos comunes. De hecho, ~29% de todas las proteínas ortólogas son idénticas entre humanos y chimpancés. La proteína típica difiere solo en dos aminoácidos. [16] Las medidas de divergencia de secuencia que se muestran en la tabla solo tienen en cuenta las diferencias sustitucionales, por ejemplo de una A ( adenina ) a una G ( guanina ). Sin embargo, las secuencias de ADN también pueden diferir por inserciones y deleciones ( indels ) de bases. Estas generalmente se eliminan de las alineaciones antes de realizar el cálculo de divergencia de secuencia.
Un grupo internacional de científicos completó un borrador de la secuencia del genoma neandertal en mayo de 2010. Los resultados indican cierta reproducción entre los humanos modernos ( Homo sapiens ) y los neandertales ( Homo neanderthalensis ) , ya que los genomas de los humanos no africanos tienen entre un 1 y un 4 % más en común con los neandertales que los genomas de los africanos subsaharianos. Los neandertales y la mayoría de los humanos modernos comparten una variante intolerante a la lactosa del gen de la lactasa que codifica una enzima que no puede descomponer la lactosa en la leche después del destete. Los humanos modernos y los neandertales también comparten la variante del gen FOXP2 asociada con el desarrollo del cerebro y con el habla en los humanos modernos, lo que indica que los neandertales pueden haber sido capaces de hablar. Los chimpancés tienen dos diferencias de aminoácidos en FOXP2 en comparación con el FOXP2 humano y neandertal. [27] [28] [29]
Se cree que el Homo sapiens surgió hace unos 300.000 años. Se dispersó por toda África y, después, hace 70.000 años, por toda Eurasia y Oceanía. Un estudio de 2009 identificó 14 "agrupaciones poblacionales ancestrales", siendo la más remota la del pueblo san del sur de África. [30] [31]
Con su rápida expansión a lo largo de diferentes zonas climáticas, y especialmente con la disponibilidad de nuevas fuentes de alimentos con la domesticación del ganado y el desarrollo de la agricultura , las poblaciones humanas han estado expuestas a importantes presiones selectivas desde su dispersión. Por ejemplo, se cree que los antepasados de los asiáticos orientales han sufrido procesos de selección de una serie de alelos, incluidas variantes de los genes EDAR , ADH1B , ABCC1 y ALDH2 .
Los tipos de ADH1B del este de Asia en particular están asociados con la domesticación del arroz y por lo tanto habrían surgido después del desarrollo del cultivo del arroz hace aproximadamente 10.000 años. [32] Varios rasgos fenotípicos característicos de los asiáticos orientales se deben a una única mutación del gen EDAR , que data de hace unos 35.000 años. [33]
En 2017 [update], la base de datos de polimorfismos de nucleótido único ( dbSNP ), que enumera los SNP y otras variantes, enumeró un total de 324 millones de variantes encontradas en genomas humanos secuenciados. [34] La diversidad de nucleótidos , la proporción promedio de nucleótidos que difieren entre dos individuos, se estima entre el 0,1% y el 0,4% para los humanos contemporáneos (en comparación con el 2% entre humanos y chimpancés). [35] [36] Esto corresponde a diferencias genómicas en unos pocos millones de sitios; el Proyecto 1000 Genomas encontró de manera similar que "un genoma [individual] típico difiere del genoma humano de referencia en 4,1 millones a 5,0 millones de sitios... afectando a 20 millones de bases de secuencia". [37]
En febrero de 2019, los científicos descubrieron evidencia, basada en estudios genéticos utilizando inteligencia artificial (IA), que sugiere la existencia de una especie de ancestro humano desconocido, no neandertal , denisovano o híbrido humano (como Denny (homínido híbrido) ), en el genoma de los humanos modernos . [38] [39]
En marzo de 2019, científicos chinos informaron haber insertado el gen MCPH1 relacionado con el cerebro humano en monos rhesus de laboratorio , lo que dio como resultado que los monos transgénicos se desempeñaran mejor y respondieran más rápido en "pruebas de memoria a corto plazo que involucraban la combinación de colores y formas", en comparación con los monos de control no transgénicos, según los investigadores. [40] [41]
En mayo de 2023, los científicos informaron, basándose en estudios genéticos, de una vía de evolución humana más compleja de lo que se creía hasta ahora. Según los estudios, los humanos evolucionaron a partir de diferentes lugares y épocas de África, en lugar de hacerlo a partir de un único lugar y período de tiempo. [42] [43]
El 31 de agosto de 2023, los investigadores informaron, basándose en estudios genéticos, que se produjo un cuello de botella en la población de ancestros humanos "hace unos 930.000 y 813.000 años... que duró unos 117.000 años y llevó a los ancestros humanos al borde de la extinción". [44] [45]
{{cite journal}}: CS1 maint: numeric names: authors list (link)