
En biología molecular , [1] [2] [3] la replicación del ADN es el proceso biológico de producir dos réplicas idénticas de ADN a partir de una molécula de ADN original . [4] La replicación del ADN ocurre en todos los organismos vivos y actúa como la parte más esencial de la herencia biológica . Esto es esencial para la división celular durante el crecimiento y la reparación de tejidos dañados, al mismo tiempo que garantiza que cada una de las nuevas células reciba su propia copia del ADN. [5] La célula posee la propiedad distintiva de la división, lo que hace que la replicación del ADN sea esencial.
El ADN está formado por una doble hélice de dos cadenas complementarias . La doble hélice describe la apariencia de un ADN bicatenario que, por tanto, está compuesto por dos cadenas lineales que corren opuestas entre sí y se retuercen para formar [6] . Durante la replicación, estas cadenas se separan. Cada cadena de la molécula de ADN original sirve entonces como plantilla para la producción de su contraparte, un proceso conocido como replicación semiconservativa . Como resultado de la replicación semiconservativa, la nueva hélice estará compuesta por una cadena de ADN original, así como por una cadena recién sintetizada [7] . Los mecanismos de corrección de errores y de verificación celular garantizan una fidelidad casi perfecta para la replicación del ADN [8] [9]
En una célula , la replicación del ADN comienza en lugares específicos, u orígenes de replicación , [10] en el genoma [11] que contiene el material genético de un organismo. [12] El desenrollado del ADN en el origen y la síntesis de nuevas hebras, acomodado por una enzima conocida como helicasa , da como resultado horquillas de replicación que crecen bidireccionalmente desde el origen. Una serie de proteínas están asociadas con la horquilla de replicación para ayudar en el inicio y la continuación de la síntesis de ADN . Más prominentemente, la ADN polimerasa sintetiza las nuevas hebras agregando nucleótidos que complementan cada hebra (plantilla). La replicación del ADN ocurre durante la etapa S de la interfase . [13]
La replicación del ADN (amplificación del ADN) también se puede realizar in vitro (artificialmente, fuera de una célula). [14] Las ADN polimerasas aisladas de células y los cebadores de ADN artificiales se pueden utilizar para iniciar la síntesis de ADN en secuencias conocidas en una molécula de ADN molde. La reacción en cadena de la polimerasa (PCR), la reacción en cadena de la ligasa (LCR) y la amplificación mediada por transcripción (TMA) son ejemplos. En marzo de 2021, los investigadores informaron evidencia que sugiere que una forma preliminar de ARN de transferencia , un componente necesario de la traducción , la síntesis biológica de nuevas proteínas de acuerdo con el código genético , podría haber sido una molécula replicadora en sí misma en el desarrollo muy temprano de la vida, o abiogénesis . [15] [16]

El ADN existe como una estructura de doble cadena, con ambas cadenas enrolladas juntas para formar la característica doble hélice . Cada cadena simple de ADN es una cadena de cuatro tipos de nucleótidos . Los nucleótidos en el ADN contienen un azúcar desoxirribosa , un fosfato y una nucleobase . Los cuatro tipos de nucleótidos corresponden a las cuatro nucleobases adenina , citosina , guanina y timina , comúnmente abreviadas como A, C, G y T. La adenina y la guanina son bases de purina [17] , mientras que la citosina y la timina son pirimidinas . Estos nucleótidos forman enlaces fosfodiéster , creando la estructura principal de fosfato-desoxirribosa de la doble hélice del ADN con las nucleobases apuntando hacia adentro (es decir, hacia la cadena opuesta). Las nucleobases se emparejan entre cadenas a través de enlaces de hidrógeno para formar pares de bases . La adenina se empareja con la timina (dos enlaces de hidrógeno) y la guanina se empareja con la citosina (tres enlaces de hidrógeno ). [18]
Las cadenas de ADN tienen una direccionalidad , y los diferentes extremos de una sola cadena se denominan "extremo 3′ (tres primos)" y "extremo 5′ (cinco primos)". Por convención, si se da la secuencia de bases de una sola cadena de ADN, el extremo izquierdo de la secuencia es el extremo 5′, mientras que el extremo derecho de la secuencia es el extremo 3′. Las cadenas de la doble hélice son antiparalelas, siendo una de 5′ a 3′, y la cadena opuesta de 3′ a 5′. Estos términos se refieren al átomo de carbono en la desoxirribosa al que se une el siguiente fosfato en la cadena. La direccionalidad tiene consecuencias en la síntesis de ADN, porque la ADN polimerasa puede sintetizar ADN en una sola dirección añadiendo nucleótidos al extremo 3′ de una cadena de ADN.
El emparejamiento de bases complementarias en el ADN (a través de enlaces de hidrógeno ) significa que la información contenida dentro de cada hebra es redundante. Los enlaces fosfodiéster (intra-hebra) son más fuertes que los enlaces de hidrógeno (entre hebras). La función real de los enlaces fosfodiéster es que en los polímeros de ADN se conecte el átomo de carbono 5' de un nucleótido con el átomo de carbono 3' de otro nucleótido, mientras que los enlaces de hidrógeno estabilizan las dobles hélices de ADN a través del eje de la hélice pero no en la dirección del eje. [19] Esto hace posible separar las hebras entre sí. Por lo tanto, los nucleótidos de una sola hebra se pueden utilizar para reconstruir nucleótidos en una hebra asociada recién sintetizada. [20]

Las ADN polimerasas son una familia de enzimas que llevan a cabo todas las formas de replicación del ADN. [22] Las ADN polimerasas en general no pueden iniciar la síntesis de nuevas hebras, sino que solo pueden extender una hebra de ADN o ARN existente emparejada con una hebra molde. Para comenzar la síntesis, se debe crear un fragmento corto de ARN, llamado cebador , y emparejarlo con la hebra molde de ADN.
La ADN polimerasa añade una nueva cadena de ADN extendiendo el extremo 3' de una cadena de nucleótidos existente, añadiendo nuevos nucleótidos que coincidan con la cadena molde, uno a la vez, mediante la creación de enlaces fosfodiéster . La energía para este proceso de polimerización del ADN proviene de la hidrólisis de los enlaces de fosfato de alta energía (fosfoanhídrido) entre los tres fosfatos unidos a cada base no incorporada . Las bases libres con sus grupos fosfato unidos se denominan nucleótidos ; en particular, las bases con tres grupos fosfato unidos se denominan trifosfatos de nucleósidos . Cuando se añade un nucleótido a una cadena de ADN en crecimiento, la formación de un enlace fosfodiéster entre el fosfato proximal del nucleótido a la cadena en crecimiento se acompaña de la hidrólisis de un enlace fosfato de alta energía con la liberación de los dos grupos fosfato distales como pirofosfato . La hidrólisis enzimática del pirofosfato resultante en fosfato inorgánico consume un segundo enlace fosfato de alta energía y hace que la reacción sea efectivamente irreversible. [Nota 1]
En general, las ADN polimerasas son muy precisas, con una tasa de error intrínseco de menos de un error por cada 10 7 nucleótidos añadidos. [23] Algunas ADN polimerasas también pueden eliminar nucleótidos del final de una cadena en desarrollo para corregir bases desapareadas. Esto se conoce como corrección de errores. Finalmente, los mecanismos de reparación de desajustes posteriores a la replicación monitorean el ADN en busca de errores, siendo capaces de distinguir los desajustes en la cadena de ADN recién sintetizada de la secuencia de la cadena original. Juntos, estos tres pasos de discriminación permiten una fidelidad de replicación de menos de un error por cada 10 9 nucleótidos añadidos. [23]
La tasa de replicación del ADN en una célula viva se midió por primera vez como la tasa de elongación del ADN del fago T4 en E. coli infectada con fagos . [24] Durante el período de aumento exponencial del ADN a 37 °C, la tasa fue de 749 nucleótidos por segundo. La tasa de mutación por par de bases por replicación durante la síntesis del ADN del fago T4 es de 1,7 por 10 8 . [25]


La replicación del ADN, como todos los procesos de polimerización biológica, se desarrolla en tres pasos coordinados y catalizados enzimáticamente: iniciación, elongación y terminación.


Para que una célula se divida , primero debe replicar su ADN. [26] La replicación del ADN es un proceso de todo o nada; una vez que comienza la replicación, se completa. Una vez que la replicación se completa, no ocurre nuevamente en el mismo ciclo celular. Esto es posible gracias a la división de iniciación del complejo de pre-replicación .
En la mitosis tardía y la fase G1 temprana , un gran complejo de proteínas iniciadoras se ensambla en el complejo de pre-replicación en puntos particulares en el ADN, conocidos como " orígenes ". [11] [10] En E. coli la proteína iniciadora primaria es el ADN A ; en levadura , este es el complejo de reconocimiento de origen . [27] Las secuencias utilizadas por las proteínas iniciadoras tienden a ser "ricas en AT" (ricas en bases de adenina y timina), porque los pares de bases AT tienen dos enlaces de hidrógeno (en lugar de los tres formados en un par CG) y, por lo tanto, son más fáciles de separar en cadenas. [28] En eucariotas, el complejo de reconocimiento de origen cataliza el ensamblaje de proteínas iniciadoras en el complejo de pre-replicación. Además, un informe reciente sugiere que el ORC de levadura en ciernes se dimeriza de una manera dependiente del ciclo celular para controlar la licencia. [29] [30] A su vez, el proceso de dimerización de ORC está mediado por un ciclo de dimerización de Noc3p dependiente del ciclo celular in vivo, y este papel de Noc3p es separable de su papel en la biogénesis de los ribosomas. Un ciclo de dimerización esencial de Noc3p media la formación del doble hexámero de ORC en la licencia de replicación ORC y Noc3p están unidos continuamente a la cromatina durante todo el ciclo celular. [31] Cdc6 y Cdt1 luego se asocian con el complejo de reconocimiento de origen unido en el origen para formar un complejo más grande necesario para cargar el complejo Mcm en el ADN. En eucariotas, el complejo Mcm es la helicasa que dividirá la hélice de ADN en las horquillas de replicación y los orígenes. El complejo Mcm es reclutado en la fase G1 tardía y cargado por el complejo ORC-Cdc6-Cdt1 en el ADN a través de la remodelación de proteínas dependiente de ATP. La carga del complejo Mcm en el ADN de origen marca la finalización de la formación del complejo de pre-replicación. [32]
Si las condiciones ambientales son las adecuadas en la fase G1 tardía, se activan los complejos G1 y G1/S ciclina - Cdk , que estimulan la expresión de genes que codifican componentes de la maquinaria sintética del ADN. La activación de G1/S-Cdk también promueve la expresión y activación de los complejos S-Cdk, que pueden desempeñar un papel en la activación de los orígenes de replicación dependiendo de la especie y el tipo de célula. El control de estas Cdk varía dependiendo del tipo de célula y la etapa de desarrollo. Esta regulación se entiende mejor en la levadura en ciernes , donde las ciclinas S Clb5 y Clb6 son las principales responsables de la replicación del ADN. [33] Los complejos Clb5,6-Cdk1 desencadenan directamente la activación de los orígenes de replicación y, por lo tanto, son necesarios durante toda la fase S para activar directamente cada origen. [32]
De manera similar, Cdc7 también es necesaria durante la fase S para activar los orígenes de replicación. Cdc7 no está activa durante todo el ciclo celular y su activación está estrictamente cronometrada para evitar el inicio prematuro de la replicación del ADN. A finales de G1, la actividad de Cdc7 aumenta abruptamente como resultado de la asociación con la subunidad reguladora DBF4 , que se une a Cdc7 directamente y promueve su actividad de proteína quinasa. Se ha descubierto que Cdc7 es un regulador limitante de la velocidad de la actividad del origen. Juntos, los G1/S-Cdks y/o S-Cdks y Cdc7 colaboran para activar directamente los orígenes de replicación, lo que lleva al inicio de la síntesis de ADN. [32]
En la fase S temprana, la activación de S-Cdk y Cdc7 conduce al ensamblaje del complejo de preiniciación, un complejo proteico masivo formado en el origen. La formación del complejo de preiniciación desplaza a Cdc6 y Cdt1 del complejo de replicación del origen, inactivando y desensamblando el complejo de pre-replicación. La carga del complejo de preiniciación en el origen activa la helicasa Mcm, lo que provoca el desenrollado de la hélice de ADN. El complejo de preiniciación también carga α-primasa y otras polimerasas de ADN en el ADN. [32]
Después de que la α-primasa sintetiza los primeros cebadores, las uniones cebador-molde interactúan con el cargador de pinzas, que carga la pinza deslizante sobre el ADN para comenzar la síntesis de ADN. Los componentes del complejo de preiniciación permanecen asociados con las horquillas de replicación a medida que se alejan del origen. [32]
La ADN polimerasa tiene actividad 5′–3′. Todos los sistemas de replicación de ADN conocidos requieren un grupo hidroxilo 3′ libre antes de que se pueda iniciar la síntesis (nota: la plantilla de ADN se lee en la dirección 3′ a 5′ mientras que una nueva cadena se sintetiza en la dirección 5′ a 3′; esto a menudo se confunde). Se reconocen cuatro mecanismos distintos para la síntesis de ADN:
Los organismos celulares utilizan la primera de estas vías, ya que es la más conocida. En este mecanismo, una vez que se separan las dos hebras, la primasa añade cebadores de ARN a las hebras molde. La hebra líder recibe un cebador de ARN mientras que la hebra rezagada recibe varios. La hebra líder se extiende continuamente desde el cebador por una ADN polimerasa con alta procesividad , mientras que la hebra rezagada se extiende de forma discontinua desde cada cebador formando fragmentos de Okazaki . La ARNasa elimina los fragmentos de ARN del cebador y una ADN polimerasa de baja procesividad distinta de la polimerasa replicativa entra para rellenar los huecos. Cuando esto se completa, se puede encontrar una única muesca en la hebra líder y varias muescas en la hebra rezagada. La ligasa trabaja para rellenar estas muescas, completando así la molécula de ADN recién replicada.
La primasa utilizada en este proceso difiere significativamente entre bacterias y arqueas / eucariotas . Las bacterias utilizan una primasa que pertenece a la superfamilia de proteínas DnaG que contiene un dominio catalítico del tipo de pliegue TOPRIM. [34] El pliegue TOPRIM contiene un núcleo α/β con cuatro hebras conservadas en una topología similar a Rossmann . Esta estructura también se encuentra en los dominios catalíticos de la topoisomerasa Ia, la topoisomerasa II, las nucleasas de la familia OLD y las proteínas de reparación del ADN relacionadas con la proteína RecR.
En cambio, la primasa que utilizan las arqueas y los eucariotas contiene una versión altamente derivada del motivo de reconocimiento de ARN (RRM). Esta primasa es estructuralmente similar a muchas ARN polimerasas dependientes de ARN virales, transcriptasas inversas, ciclasas generadoras de nucleótidos cíclicos y ADN polimerasas de las familias A/B/Y que participan en la replicación y reparación del ADN. En la replicación eucariota, la primasa forma un complejo con Pol α. [35]
Varias ADN polimerasas desempeñan diferentes funciones en el proceso de replicación del ADN. En E. coli , la ADN Pol III es la enzima polimerasa principalmente responsable de la replicación del ADN. Se ensambla en un complejo de replicación en la horquilla de replicación que exhibe una procesividad extremadamente alta, permaneciendo intacta durante todo el ciclo de replicación. Por el contrario, la ADN Pol I es la enzima responsable de reemplazar los cebadores de ARN con ADN. La ADN Pol I tiene una actividad exonucleasa de 5′ a 3′ además de su actividad polimerasa, y utiliza su actividad exonucleasa para degradar los cebadores de ARN que se encuentran delante de ella a medida que extiende la cadena de ADN detrás de ella, en un proceso llamado traducción de mellas . La Pol I es mucho menos procesiva que la Pol III porque su función principal en la replicación del ADN es crear muchas regiones cortas de ADN en lugar de unas pocas regiones muy largas.
En eucariotas , la enzima de baja procesividad, Pol α, ayuda a iniciar la replicación porque forma un complejo con la primasa. [36] En eucariotas, se cree que la síntesis de la cadena líder es realizada por Pol ε; sin embargo, esta visión ha sido cuestionada recientemente, sugiriendo un papel para Pol δ. [37] La eliminación del cebador la completa Pol δ [38] mientras que la reparación del ADN durante la replicación la completa Pol ε.
A medida que continúa la síntesis de ADN, las cadenas de ADN originales continúan desenrollándose en cada lado de la burbuja, formando una horquilla de replicación con dos puntas. En las bacterias, que tienen un único origen de replicación en su cromosoma circular, este proceso crea una " estructura theta " (que se asemeja a la letra griega theta: θ). En cambio, los eucariotas tienen cromosomas lineales más largos e inician la replicación en múltiples orígenes dentro de estos. [39]


La horquilla de replicación es una estructura que se forma dentro de la larga hélice de ADN durante la replicación del ADN. Es producida por enzimas llamadas helicasas que rompen los enlaces de hidrógeno que mantienen unidas las cadenas de ADN en una hélice. La estructura resultante tiene dos "puntas" ramificadas, cada una de ellas formada por una sola cadena de ADN. Estas dos cadenas sirven como plantilla para las cadenas líder y rezagada, que se crearán a medida que la ADN polimerasa empareje los nucleótidos complementarios con las plantillas; las plantillas pueden denominarse correctamente plantilla de la cadena líder y plantilla de la cadena rezagada.
La ADN polimerasa lee el ADN en la dirección 3' a 5', lo que significa que la nueva cadena se sintetiza en la dirección 5' a 3'. Dado que las cadenas líder y rezagada están orientadas en direcciones opuestas en la horquilla de replicación, una cuestión importante es cómo lograr la síntesis de ADN de la nueva cadena rezagada, cuya dirección de síntesis es opuesta a la dirección de la horquilla de replicación en crecimiento.
La hebra principal es la hebra de ADN nuevo que se sintetiza en la misma dirección que la horquilla de replicación en crecimiento. Este tipo de replicación del ADN es continua.
La hebra rezagada es la hebra de ADN nuevo cuya dirección de síntesis es opuesta a la dirección de la horquilla de replicación en crecimiento. Debido a su orientación, la replicación de la hebra rezagada es más complicada en comparación con la de la hebra líder. Como consecuencia, se observa que la ADN polimerasa de esta hebra "va por detrás" de la otra hebra.
La hebra rezagada se sintetiza en segmentos cortos y separados. En la plantilla de la hebra rezagada , una primasa "lee" el ADN de la plantilla e inicia la síntesis de un cebador de ARN complementario corto . Una ADN polimerasa extiende los segmentos cebados, formando fragmentos de Okazaki . Luego, los cebadores de ARN se eliminan y se reemplazan con ADN, y los fragmentos de ADN se unen mediante la ADN ligasa .

En todos los casos, la helicasa está compuesta por seis polipéptidos que envuelven una sola hebra del ADN que se está replicando. Las dos polimerasas están unidas al hexámero de la helicasa. En los eucariotas, la helicasa envuelve la hebra líder y en los procariotas, la hebra rezagada. [40]
A medida que la helicasa desenrolla el ADN en la horquilla de replicación, el ADN que se encuentra por delante se ve obligado a rotar. Este proceso da como resultado una acumulación de giros en el ADN que se encuentra por delante. [41] Esta acumulación crea una carga torsional que eventualmente detendría la horquilla de replicación. Las topoisomerasas son enzimas que rompen temporalmente las cadenas de ADN, aliviando la tensión causada por el desenrollado de las dos cadenas de la hélice de ADN; las topoisomerasas (incluida la ADN girasa ) logran esto agregando superenrollamientos negativos a la hélice de ADN. [42]
El ADN monocatenario desnudo tiende a plegarse sobre sí mismo y forma estructuras secundarias ; estas estructuras pueden interferir con el movimiento de la ADN polimerasa. Para evitar esto, las proteínas de unión monocatenarias se unen al ADN hasta que se sintetiza una segunda cadena, lo que impide la formación de estructuras secundarias. [43]
El ADN de doble cadena está enrollado alrededor de histonas que desempeñan un papel importante en la regulación de la expresión génica, por lo que el ADN replicado debe estar enrollado alrededor de las histonas en los mismos lugares que el ADN original. [44] Para garantizar esto, las chaperonas de histonas desmontan la cromatina antes de que se replique y reemplazan las histonas en el lugar correcto. Algunos pasos en este reensamblaje son algo especulativos. [45]
Las proteínas de pinza actúan como una pinza deslizante sobre el ADN, lo que permite que la ADN polimerasa se una a su plantilla y ayude en la procesividad. La cara interna de la pinza permite que el ADN pase a través de ella. Una vez que la polimerasa llega al final de la plantilla o detecta ADN de doble cadena, la pinza deslizante sufre un cambio conformacional que libera la ADN polimerasa. Las proteínas de carga de la pinza se utilizan para cargar inicialmente la pinza, reconociendo la unión entre la plantilla y los cebadores de ARN. [9] :274-5
En la horquilla de replicación, muchas enzimas de replicación se ensamblan en el ADN para formar una máquina molecular compleja llamada replisoma . A continuación, se incluye una lista de las principales enzimas de replicación del ADN que participan en el replisoma: [46]
Los experimentos in vitro con moléculas individuales (utilizando pinzas ópticas y pinzas magnéticas ) han encontrado interacciones sinérgicas entre las enzimas del replisoma ( helicasa , polimerasa y proteína de unión al ADN de cadena sencilla ) y con la horquilla de replicación del ADN, lo que mejora el desenrollado del ADN y la replicación del ADN. [14] Estos resultados conducen al desarrollo de modelos cinéticos que dan cuenta de las interacciones sinérgicas y su estabilidad. [14]

Las maquinarias de replicación consisten en factores que intervienen en la replicación del ADN y que aparecen en los ssDNA molde. Las maquinarias de replicación incluyen los primosificadores, que son enzimas de replicación; ADN polimerasa, ADN helicasas, pinzas de ADN y ADN topoisomerasas, y proteínas de replicación; por ejemplo, proteínas de unión al ADN monocatenario (SSB). En las maquinarias de replicación, estos componentes se coordinan. En la mayoría de las bacterias, todos los factores que intervienen en la replicación del ADN se encuentran en las horquillas de replicación y los complejos permanecen en las horquillas durante la replicación del ADN. Las maquinarias de replicación también se conocen como replisomas o sistemas de replicación del ADN. Estos términos son términos genéricos para las proteínas ubicadas en las horquillas de replicación. En las células eucariotas y algunas bacterianas, los replisomas no se forman.
En una figura alternativa, las fábricas de ADN son similares a los proyectores y los ADN son como películas cinematográficas que pasan constantemente por los proyectores. En el modelo de fábrica de replicación, después de que las helicasas de ADN para las cadenas principales y las cadenas rezagadas se cargan en los ADN molde, las helicasas recorren los ADN entre sí. Las helicasas permanecen asociadas durante el resto del proceso de replicación. Peter Meister et al. observaron directamente los sitios de replicación en la levadura en ciernes al monitorear las ADN polimerasas α marcadas con proteína fluorescente verde (GFP). Detectaron la replicación de ADN de pares de los loci marcados espaciados simétricamente desde un origen de replicación y encontraron que la distancia entre los pares disminuía notablemente con el tiempo. [48] Este hallazgo sugiere que el mecanismo de replicación de ADN va con las fábricas de ADN. Es decir, se cargan pares de fábricas de replicación en los orígenes de replicación y las fábricas se asocian entre sí. Además, los ADN molde se mueven hacia las fábricas, lo que provoca la extrusión de los ssADN molde y nuevos ADN. El hallazgo de Meister es la primera evidencia directa de un modelo de fábrica de replicación. Investigaciones posteriores han demostrado que las helicasas de ADN forman dímeros en muchas células eucariotas y que las maquinarias de replicación bacterianas permanecen en una única ubicación intranuclear durante la síntesis de ADN. [49]
Las fábricas de replicación desenredan las cromátidas hermanas. El desenredado es esencial para distribuir las cromátidas en las células hijas después de la replicación del ADN. Debido a que las cromátidas hermanas después de la replicación del ADN se unen entre sí mediante anillos de cohesión , existe la única posibilidad de desenredado en la replicación del ADN. La fijación de maquinarias de replicación como fábricas de replicación puede mejorar la tasa de éxito de la replicación del ADN. Si las horquillas de replicación se mueven libremente en los cromosomas, la concatenación de los núcleos se agrava e impide la segregación mitótica. [48]
Los eucariotas inician la replicación del ADN en múltiples puntos del cromosoma, por lo que las horquillas de replicación se encuentran y terminan en muchos puntos del cromosoma. Debido a que los eucariotas tienen cromosomas lineales, la replicación del ADN no puede llegar al final de los cromosomas. Debido a este problema, el ADN se pierde en cada ciclo de replicación desde el final del cromosoma. Los telómeros son regiones de ADN repetitivo cercanas a los extremos y ayudan a prevenir la pérdida de genes debido a este acortamiento. El acortamiento de los telómeros es un proceso normal en las células somáticas . Esto acorta los telómeros del cromosoma de ADN hija. Como resultado, las células solo pueden dividirse una cierta cantidad de veces antes de que la pérdida de ADN impida una mayor división. (Esto se conoce como el límite de Hayflick ). Dentro de la línea de células germinales , que pasa el ADN a la siguiente generación, la telomerasa extiende las secuencias repetitivas de la región telomérica para evitar la degradación. La telomerasa puede activarse por error en las células somáticas, lo que a veces conduce a la formación de cáncer . El aumento de la actividad de la telomerasa es una de las características del cáncer.
La terminación requiere que el avance de la horquilla de replicación del ADN se detenga o se bloquee. La terminación en un locus específico, cuando ocurre, implica la interacción entre dos componentes: (1) una secuencia del sitio de terminación en el ADN y (2) una proteína que se une a esta secuencia para detener físicamente la replicación del ADN. En varias especies bacterianas, esto se denomina proteína de unión al sitio terminal de replicación del ADN o proteína Ter .
Debido a que las bacterias tienen cromosomas circulares, la terminación de la replicación ocurre cuando las dos horquillas de replicación se encuentran en el extremo opuesto del cromosoma parental. E. coli regula este proceso mediante el uso de secuencias de terminación que, cuando están unidas por la proteína Tus , permiten que solo pase una dirección de la horquilla de replicación. Como resultado, las horquillas de replicación se ven obligadas a encontrarse siempre dentro de la región de terminación del cromosoma. [50]

En los eucariotas, la replicación del ADN se controla en el contexto del ciclo celular . A medida que la célula crece y se divide, progresa a través de etapas en el ciclo celular; la replicación del ADN tiene lugar durante la fase S (fase de síntesis). El progreso de la célula eucariota a través del ciclo está controlado por puntos de control del ciclo celular . La progresión a través de los puntos de control se controla mediante interacciones complejas entre varias proteínas, incluidas las ciclinas y las quinasas dependientes de ciclinas . [51] A diferencia de las bacterias, el ADN eucariota se replica en los confines del núcleo. [52]
El punto de control G1/S (punto de control de restricción) regula si las células eucariotas entran en el proceso de replicación del ADN y posterior división. Las células que no pasan por este punto de control permanecen en la etapa G0 y no replican su ADN.
Una vez que el ADN ha pasado por la prueba "G1/S", sólo se puede copiar una vez en cada ciclo celular. Cuando el complejo Mcm se aleja del origen, el complejo de pre-replicación se desmantela. Debido a que un nuevo complejo Mcm no se puede cargar en un origen hasta que se reactiven las subunidades de pre-replicación, un origen de replicación no se puede utilizar dos veces en el mismo ciclo celular. [32]
La activación de las S-Cdks en la fase S temprana promueve la destrucción o inhibición de componentes individuales del complejo de pre-replicación, impidiendo el reensamblaje inmediato. Las S y M-Cdks continúan bloqueando el ensamblaje del complejo de pre-replicación incluso después de que se completa la fase S, lo que garantiza que el ensamblaje no pueda ocurrir nuevamente hasta que se reduzca toda la actividad de las Cdk en la mitosis tardía. [32]
En la levadura en ciernes, la inhibición del ensamblaje es causada por la fosforilación dependiente de Cdk de los componentes del complejo de pre-replicación. Al inicio de la fase S, la fosforilación de Cdc6 por Cdk1 causa la unión de Cdc6 a la proteína ligasa de ubiquitina SCF , que causa la destrucción proteolítica de Cdc6. La fosforilación dependiente de Cdk de las proteínas Mcm promueve su exportación fuera del núcleo junto con Cdt1 durante la fase S, evitando la carga de nuevos complejos Mcm en los orígenes durante un solo ciclo celular. La fosforilación de Cdk del complejo de replicación del origen también inhibe el ensamblaje del complejo de pre-replicación. La presencia individual de cualquiera de estos tres mecanismos es suficiente para inhibir el ensamblaje del complejo de pre-replicación. Sin embargo, las mutaciones de las tres proteínas en la misma célula desencadenan la reiniciación en muchos orígenes de replicación dentro de un ciclo celular. [32] [53]
En las células animales, la proteína geminina es un inhibidor clave del ensamblaje del complejo de pre-replicación. La geminina se une a Cdt1, impidiendo su unión al complejo de reconocimiento de origen. En G1, los niveles de geminina se mantienen bajos por la APC, que ubiquitina la geminina para dirigirla hacia su degradación. Cuando se destruye la geminina, se libera Cdt1, lo que le permite funcionar en el ensamblaje del complejo de pre-replicación. Al final de G1, la APC se inactiva, lo que permite que la geminina se acumule y se una a Cdt1. [32]
La replicación de los genomas de los cloroplastos y de las mitocondrias ocurre independientemente del ciclo celular, a través del proceso de replicación del bucle D.
En las células de vertebrados, los sitios de replicación se concentran en posiciones llamadas focos de replicación . [48] Los sitios de replicación se pueden detectar mediante inmunotinción de cadenas hijas y enzimas de replicación y monitoreando los factores de replicación marcados con GFP. Mediante estos métodos se descubre que los focos de replicación de diferentes tamaños y posiciones aparecen en la fase S de la división celular y su número por núcleo es mucho menor que el número de horquillas de replicación genómica.
P. Heun et al. , [48] (2001) rastrearon focos de replicación marcados con GFP en células de levadura en gemación y revelaron que los orígenes de replicación se mueven constantemente en la fase G1 y S y la dinámica disminuye significativamente en la fase S. [48] Tradicionalmente, los sitios de replicación se fijaban en la estructura espacial de los cromosomas mediante la matriz nuclear o láminas . Los resultados de Heun negaron los conceptos tradicionales, las levaduras en gemación no tienen láminas, y respaldan que los orígenes de replicación se autoensamblan y forman focos de replicación.
Al activarse los orígenes de replicación, controlados espacial y temporalmente, se regula la formación de focos de replicación. DA Jackson et al. (1998) revelaron que los orígenes vecinos se activan simultáneamente en células de mamíferos. [48] La yuxtaposición espacial de los sitios de replicación produce la agrupación de las horquillas de replicación. La agrupación rescata las horquillas de replicación estancadas y favorece el progreso normal de las horquillas de replicación. El progreso de las horquillas de replicación se inhibe por muchos factores: colisión con proteínas o con complejos que se unen fuertemente al ADN, deficiencia de dNTP, mellas en los ADN molde, etc. Si las horquillas de replicación se atascan y el resto de las secuencias de las horquillas atascadas no se copian, entonces las hebras hijas obtienen sitios no replicados de mella-mella. Los sitios no replicados en la hebra de un progenitor mantienen unida la otra hebra, pero no las hebras hijas. Por lo tanto, las cromátidas hermanas resultantes no pueden separarse entre sí y no pueden dividirse en 2 células hijas. Cuando los orígenes vecinos se activan y una bifurcación de un origen se detiene, la bifurcación del otro origen accede en una dirección opuesta a la bifurcación detenida y duplica los sitios no replicados. Como otro mecanismo de rescate, existe la aplicación de orígenes de replicación latentes , en los que los orígenes excedentes no se activan en la replicación normal del ADN.

La mayoría de las bacterias no pasan por un ciclo celular bien definido, sino que copian continuamente su ADN; durante el crecimiento rápido, esto puede dar lugar a la ocurrencia simultánea de múltiples rondas de replicación. [54] En E. coli , la bacteria mejor caracterizada, la replicación del ADN está regulada a través de varios mecanismos, entre ellos: la hemimetilación y el secuestro de la secuencia de origen, la relación entre el trifosfato de adenosina (ATP) y el difosfato de adenosina (ADP) y los niveles de proteína DnaA. Todos estos controlan la unión de las proteínas iniciadoras a las secuencias de origen. [55]
Debido a que E. coli metila las secuencias de ADN GATC, la síntesis de ADN da como resultado secuencias hemimetiladas. Este ADN hemimetilado es reconocido por la proteína SeqA , que se une y secuestra la secuencia de origen; además, DnaA (necesaria para el inicio de la replicación) se une menos bien al ADN hemimetilado. Como resultado, se impide que los orígenes recién replicados inicien inmediatamente otra ronda de replicación de ADN. [56]
El ATP se acumula cuando la célula se encuentra en un medio rico, lo que desencadena la replicación del ADN una vez que la célula ha alcanzado un tamaño específico. El ATP compite con el ADP para unirse a DnaA, y el complejo DnaA-ATP puede iniciar la replicación. También se requiere una cierta cantidad de proteínas DnaA para la replicación del ADN: cada vez que se copia el origen, se duplica el número de sitios de unión para DnaA, lo que requiere la síntesis de más DnaA para permitir otro inicio de la replicación.
En las bacterias de crecimiento rápido, como E. coli , la replicación de cromosomas lleva más tiempo que la división celular. Las bacterias solucionan este problema iniciando una nueva ronda de replicación antes de que la anterior haya terminado. [57] La nueva ronda de replicación formará el cromosoma de la célula que nace dos generaciones después de la célula en división. Este mecanismo crea ciclos de replicación superpuestos.
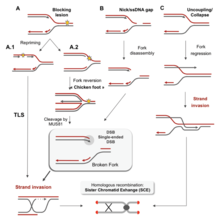
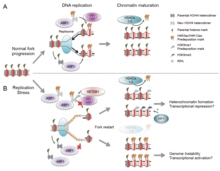
Hay muchos eventos que contribuyen al estrés de replicación, incluidos: [58]
Los investigadores suelen replicar el ADN in vitro mediante la reacción en cadena de la polimerasa (PCR). La PCR utiliza un par de cebadores para abarcar una región objetivo en el ADN molde y luego polimeriza las cadenas asociadas en cada dirección a partir de estos cebadores utilizando una ADN polimerasa termoestable . La repetición de este proceso a través de múltiples ciclos amplifica la región de ADN objetivo. Al comienzo de cada ciclo, la mezcla de plantilla y cebadores se calienta, separando la molécula recién sintetizada y la plantilla. Luego, a medida que la mezcla se enfría, ambos se convierten en plantillas para la hibridación de nuevos cebadores, y la polimerasa se extiende desde estos. Como resultado, el número de copias de la región objetivo se duplica en cada ronda, aumentando exponencialmente . [59]