La genómica es un campo interdisciplinario de la biología molecular que se centra en la estructura, función, evolución, mapeo y edición de genomas . Un genoma es el conjunto completo de ADN de un organismo , incluidos todos sus genes, así como su configuración estructural tridimensional jerárquica. [1] [2] [3] [4] A diferencia de la genética , que se refiere al estudio de genes individuales y sus funciones en la herencia, la genómica apunta a la caracterización y cuantificación colectiva de todos los genes de un organismo, sus interrelaciones e influencia en el organismo. [5] Los genes pueden dirigir la producción de proteínas con la ayuda de enzimas y moléculas mensajeras. A su vez, las proteínas forman estructuras corporales como órganos y tejidos, así como controlan reacciones químicas y transportan señales entre células. La genómica también implica la secuenciación y el análisis de genomas mediante el uso de secuenciación de ADN de alto rendimiento y bioinformática para ensamblar y analizar la función y la estructura de genomas completos. [6] [7] Los avances en genómica han desencadenado una revolución en la investigación basada en el descubrimiento y la biología de sistemas para facilitar la comprensión incluso de los sistemas biológicos más complejos, como el cerebro. [8]
El campo también incluye estudios de fenómenos intragenómicos (dentro del genoma) como la epistasis (efecto de un gen sobre otro), la pleiotropía (un gen que afecta a más de un rasgo), la heterosis (vigor híbrido) y otras interacciones entre loci y alelos dentro del genoma. [9]
Del griego ΓΕΝ [10] gen , "gen" (gamma, epsilon, nu, epsilon) que significa "llegar a ser, crear, creación, nacimiento", y variantes posteriores: genealogía, génesis, genética, genic, genomere, genotipo, género, etc. Mientras que la palabra genoma (del alemán Genom , atribuida a Hans Winkler ) se utilizaba en inglés ya en 1926, [11] el término genómica fue acuñado por Tom Roderick, un genetista del Laboratorio Jackson ( Bar Harbor, Maine ), tomando unas cervezas con Jim Womack, Tom Shows y Stephen O'Brien en una reunión celebrada en Maryland sobre el mapeo del genoma humano en 1986. [12] Primero como nombre para una nueva revista y luego como una disciplina científica completamente nueva. [13]
Tras la confirmación de la estructura helicoidal del ADN por parte de Rosalind Franklin , la publicación de la estructura del ADN por parte de James D. Watson y Francis Crick en 1953 y la publicación de la secuencia de aminoácidos de la insulina por parte de Fred Sanger en 1955, la secuenciación de ácidos nucleicos se convirtió en un objetivo importante de los primeros biólogos moleculares . [14] En 1964, Robert W. Holley y sus colegas publicaron la primera secuencia de ácidos nucleicos jamás determinada, la secuencia de ribonucleótidos del ARN de transferencia de alanina . [15] [16] Ampliando este trabajo, Marshall Nirenberg y Philip Leder revelaron la naturaleza triplete del código genético y pudieron determinar las secuencias de 54 de los 64 codones en sus experimentos. [17] En 1972, Walter Fiers y su equipo del Laboratorio de Biología Molecular de la Universidad de Gante ( Gante , Bélgica ) fueron los primeros en determinar la secuencia de un gen: el gen de la proteína de la cubierta del bacteriófago MS2 . [18] El grupo de Fiers amplió su trabajo sobre la proteína de cubierta MS2, determinando la secuencia de nucleótidos completa del ARN del bacteriófago MS2 (cuyo genoma codifica sólo cuatro genes en 3569 pares de bases [pb]) y del virus simio 40 en 1976 y 1978, respectivamente. [19] [20]
Además de su trabajo seminal sobre la secuencia de aminoácidos de la insulina, Frederick Sanger y sus colegas desempeñaron un papel clave en el desarrollo de técnicas de secuenciación de ADN que permitieron el establecimiento de proyectos integrales de secuenciación del genoma. [9] En 1975, él y Alan Coulson publicaron un procedimiento de secuenciación utilizando ADN polimerasa con nucleótidos radiomarcados que llamó la técnica Plus and Minus . [21] [22] Esto involucraba dos métodos estrechamente relacionados que generaban oligonucleótidos cortos con extremos 3' definidos. Estos podían fraccionarse por electroforesis en un gel de poliacrilamida (llamada electroforesis en gel de poliacrilamida) y visualizarse mediante autorradiografía. El procedimiento podía secuenciar hasta 80 nucleótidos de una sola vez y fue una gran mejora, pero todavía era muy laborioso. Sin embargo, en 1977 su grupo pudo secuenciar la mayoría de los 5.386 nucleótidos del bacteriófago monocatenario φX174 , completando el primer genoma basado en ADN completamente secuenciado. [23] El refinamiento del método Plus y Minus resultó en la terminación de cadena, o método Sanger (ver abajo), que formó la base de las técnicas de secuenciación de ADN, mapeo genómico, almacenamiento de datos y análisis bioinformático más ampliamente utilizadas en el siguiente cuarto de siglo de investigación. [24] [25] En el mismo año, Walter Gilbert y Allan Maxam de la Universidad de Harvard desarrollaron independientemente el método Maxam-Gilbert (también conocido como el método químico ) de secuenciación de ADN, que implica la escisión preferencial del ADN en bases conocidas, un método menos eficiente. [26] [27] Por su trabajo pionero en la secuenciación de ácidos nucleicos, Gilbert y Sanger compartieron la mitad del Premio Nobel de Química de 1980 con Paul Berg ( ADN recombinante ).
La llegada de estas tecnologías dio lugar a una rápida intensificación del alcance y la velocidad de finalización de los proyectos de secuenciación genómica . La primera secuencia completa del genoma de un orgánulo eucariota , la mitocondria humana (16.568 pb, aproximadamente 16,6 kb [kilobase]), se informó en 1981, [28] y los primeros genomas de cloroplastos siguieron en 1986. [29] [30] En 1992, se secuenció el primer cromosoma eucariota , el cromosoma III de la levadura cervecera Saccharomyces cerevisiae (315 kb). [31] El primer organismo de vida libre en ser secuenciado fue el de Haemophilus influenzae (1,8 Mb [megabase]) en 1995. [32] Al año siguiente, un consorcio de investigadores de laboratorios de América del Norte , Europa y Japón anunció la finalización de la primera secuencia completa del genoma de un eucariota, S. cerevisiae (12,1 Mb), y desde entonces los genomas han seguido siendo secuenciados a un ritmo exponencialmente creciente. [33] A octubre de 2011 [update], las secuencias completas están disponibles para: 2.719 virus , 1.115 arqueas y bacterias , y 36 eucariotas , de los cuales aproximadamente la mitad son hongos . [34] [35]

La mayoría de los microorganismos cuyos genomas han sido completamente secuenciados son patógenos problemáticos , como Haemophilus influenzae , lo que ha resultado en un sesgo pronunciado en su distribución filogenética en comparación con la amplitud de la diversidad microbiana. [36] [37] De las otras especies secuenciadas, la mayoría fueron elegidas porque eran organismos modelo bien estudiados o prometían convertirse en buenos modelos. La levadura ( Saccharomyces cerevisiae ) ha sido durante mucho tiempo un organismo modelo importante para la célula eucariota , mientras que la mosca de la fruta Drosophila melanogaster ha sido una herramienta muy importante (notablemente en la genética premolecular temprana ). El gusano Caenorhabditis elegans es un modelo simple a menudo utilizado para organismos multicelulares . El pez cebra Brachydanio rerio se utiliza para muchos estudios de desarrollo a nivel molecular, y la planta Arabidopsis thaliana es un organismo modelo para plantas con flores. El pez globo japonés ( Takifugu rubripes ) y el pez globo verde moteado ( Tetraodon nigroviridis ) son interesantes debido a sus genomas pequeños y compactos, que contienen muy poco ADN no codificante en comparación con la mayoría de las especies. [38] [39] Los mamíferos perro ( Canis familiaris ), [40] rata parda ( Rattus norvegicus ), ratón ( Mus musculus ) y chimpancé ( Pan troglodytes ) son todos animales modelo importantes en la investigación médica. [27]
A principios de 2001, el Proyecto Genoma Humano completó un borrador del genoma humano , lo que generó mucha fanfarria. [41] Este proyecto, completado en 2003, secuenció todo el genoma de una persona específica, y en 2007 esta secuencia se declaró "terminada" (menos de un error en 20.000 bases y todos los cromosomas ensamblados). [41] En los años transcurridos desde entonces, se han secuenciado los genomas de muchos otros individuos, en parte bajo los auspicios del Proyecto 1000 Genomas , que anunció la secuenciación de 1.092 genomas en octubre de 2012. [42] La finalización de este proyecto fue posible gracias al desarrollo de tecnologías de secuenciación mucho más eficientes y requirió el compromiso de importantes recursos bioinformáticos de una gran colaboración internacional. [43] El análisis continuo de los datos genómicos humanos tiene profundas repercusiones políticas y sociales para las sociedades humanas. [44]

El neologismo en inglés ómica se refiere informalmente a un campo de estudio en biología que termina en -ómica , como la genómica, la proteómica o la metabolómica . El sufijo relacionado -oma se utiliza para referirse a los objetos de estudio de dichos campos, como el genoma , el proteoma o el metaboloma ( lipidoma ), respectivamente. El sufijo -oma , tal como se utiliza en biología molecular, se refiere a una totalidad de algún tipo; de manera similar, ómica ha llegado a referirse en general al estudio de grandes conjuntos de datos biológicos completos. Si bien el crecimiento en el uso del término ha llevado a algunos científicos ( Jonathan Eisen , entre otros [45] ) a afirmar que se ha sobrevendido, [46] refleja el cambio de orientación hacia el análisis cuantitativo del surtido completo o casi completo de todos los constituyentes de un sistema. [47] En el estudio de las simbiosis , por ejemplo, los investigadores que antes estaban limitados al estudio de un único producto genético ahora pueden comparar simultáneamente el complemento total de varios tipos de moléculas biológicas. [48] [49]
Una vez seleccionado un organismo, los proyectos genómicos implican tres componentes: la secuenciación del ADN, el ensamblaje de esa secuencia para crear una representación del cromosoma original y la anotación y análisis de esa representación. [9]
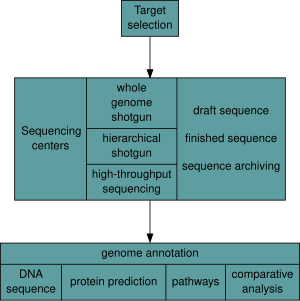
Históricamente, la secuenciación se hacía en centros de secuenciación , instalaciones centralizadas (que van desde grandes instituciones independientes como el Joint Genome Institute , que secuencia docenas de terabases al año, hasta instalaciones centrales de biología molecular locales) que contienen laboratorios de investigación con la costosa instrumentación y el soporte técnico necesarios. Sin embargo, a medida que la tecnología de secuenciación continúa mejorando, una nueva generación de secuenciadores de sobremesa eficaces y de respuesta rápida se ha puesto al alcance del laboratorio académico promedio. [50] [51] En general, los enfoques de secuenciación del genoma se dividen en dos grandes categorías: secuenciación shotgun y secuenciación de alto rendimiento (o de próxima generación ). [9]

La secuenciación por escopeta es un método de secuenciación diseñado para el análisis de secuencias de ADN de más de 1000 pares de bases, hasta cromosomas enteros inclusive. [52] Se denomina así por analogía con el patrón de disparo cuasialeatorio y de rápida expansión de una escopeta . Dado que la secuenciación por electroforesis en gel solo se puede utilizar para secuencias bastante cortas (de 100 a 1000 pares de bases), las secuencias de ADN más largas se deben dividir en pequeños segmentos aleatorios que luego se secuencian para obtener lecturas . Se obtienen múltiples lecturas superpuestas para el ADN objetivo realizando varias rondas de esta fragmentación y secuenciación. Luego, los programas de computadora utilizan los extremos superpuestos de diferentes lecturas para ensamblarlas en una secuencia continua. [52] [53] La secuenciación por escopeta es un proceso de muestreo aleatorio, que requiere un sobremuestreo para garantizar que un nucleótido dado esté representado en la secuencia reconstruida; el número promedio de lecturas por las que se sobremuestrea un genoma se conoce como cobertura . [54]
Durante gran parte de su historia, la tecnología subyacente a la secuenciación shotgun fue el método clásico de terminación de cadena o " método Sanger ", que se basa en la incorporación selectiva de didesoxinucleótidos de terminación de cadena por la ADN polimerasa durante la replicación de ADN in vitro . [23] [55] Recientemente, la secuenciación shotgun ha sido suplantada por métodos de secuenciación de alto rendimiento , especialmente para análisis genómicos automatizados a gran escala . Sin embargo, el método Sanger sigue siendo ampliamente utilizado, principalmente para proyectos de menor escala y para obtener lecturas de secuencias de ADN contiguas especialmente largas (>500 nucleótidos). [56] Los métodos de terminación de cadena requieren una plantilla de ADN monocatenario, un cebador de ADN , una ADN polimerasa , desoxinucleósidotrifosfatos normales (dNTP) y nucleótidos modificados (didesoxiNTP) que terminan la elongación de la cadena de ADN. Estos nucleótidos que terminan la cadena carecen de un grupo 3'- OH necesario para la formación de un enlace fosfodiéster entre dos nucleótidos, lo que hace que la ADN polimerasa detenga la extensión del ADN cuando se incorpora un ddNTP. Los ddNTP pueden marcarse radiactivamente o con fluorescencia para su detección en secuenciadores de ADN . [9] Por lo general, estas máquinas pueden secuenciar hasta 96 muestras de ADN en un solo lote (sesión) en hasta 48 ejecuciones al día. [57]
La gran demanda de secuenciación de bajo costo ha impulsado el desarrollo de tecnologías de secuenciación de alto rendimiento que paralelizan el proceso de secuenciación, produciendo miles o millones de secuencias a la vez. [58] [59] La secuenciación de alto rendimiento tiene como objetivo reducir el costo de la secuenciación de ADN más allá de lo que es posible con los métodos estándar de terminación por colorante. En la secuenciación de ultra alto rendimiento, se pueden ejecutar hasta 500.000 operaciones de secuenciación por síntesis en paralelo. [60] [61]

El método de secuenciación por tinte Illumina se basa en terminadores de tinte reversibles y fue desarrollado en 1996 en el Instituto de Investigación Biomédica de Ginebra por Pascal Mayer y Laurent Farinelli. [62] En este método, las moléculas de ADN y los cebadores se adhieren primero a un portaobjetos y se amplifican con polimerasa para que se formen colonias clonales locales, inicialmente denominadas "colonias de ADN". Para determinar la secuencia, se añaden cuatro tipos de bases de terminación reversibles (bases RT) y se eliminan los nucleótidos no incorporados. A diferencia de la pirosecuenciación, las cadenas de ADN se extienden un nucleótido a la vez y la adquisición de imágenes se puede realizar en un momento diferido, lo que permite capturar conjuntos muy grandes de colonias de ADN mediante imágenes secuenciales tomadas con una sola cámara. La disociación de la reacción enzimática y la captura de imágenes permite un rendimiento óptimo y una capacidad de secuenciación teóricamente ilimitada; con una configuración óptima, el rendimiento final del instrumento depende únicamente de la tasa de conversión A/D de la cámara. La cámara toma imágenes de los nucleótidos marcados con fluorescencia , luego el tinte junto con el bloqueador terminal 3' se elimina químicamente del ADN, lo que permite el siguiente ciclo. [63]
Un enfoque alternativo, la secuenciación de semiconductores iónicos, se basa en la química estándar de replicación del ADN. Esta tecnología mide la liberación de un ion de hidrógeno cada vez que se incorpora una base. Un micropocillo que contiene ADN molde se inunda con un solo nucleótido ; si el nucleótido es complementario a la cadena molde, se incorporará y se liberará un ion de hidrógeno. Esta liberación activa un sensor de iones ISFET . Si hay un homopolímero presente en la secuencia molde, se incorporarán múltiples nucleótidos en un solo ciclo de inundación y la señal eléctrica detectada será proporcionalmente mayor. [64]
El ensamblaje de secuencias se refiere a la alineación y fusión de fragmentos de una secuencia de ADN mucho más larga para reconstruir la secuencia original. [9] Esto es necesario ya que la tecnología actual de secuenciación de ADN no puede leer genomas completos como una secuencia continua, sino que lee pequeños fragmentos de entre 20 y 1000 bases, dependiendo de la tecnología utilizada. Las tecnologías de secuenciación de tercera generación como PacBio u Oxford Nanopore generan rutinariamente lecturas de secuenciación de 10 a 100 kb de longitud; sin embargo, tienen una alta tasa de error de aproximadamente el 1 por ciento. [65] [66] Normalmente, los fragmentos cortos, llamados lecturas, resultan de la secuenciación shotgun de ADN genómico o transcripciones de genes ( EST ). [9]
El ensamblaje se puede categorizar ampliamente en dos enfoques: ensamblaje de novo , para genomas que no son similares a ninguno secuenciado en el pasado, y ensamblaje comparativo, que utiliza la secuencia existente de un organismo estrechamente relacionado como referencia durante el ensamblaje. [54] En relación con el ensamblaje comparativo, el ensamblaje de novo es computacionalmente difícil ( NP-hard ), lo que lo hace menos favorable para las tecnologías NGS de lectura corta. Dentro del paradigma de ensamblaje de novo hay dos estrategias principales para el ensamblaje, estrategias de ruta euleriana y estrategias de consenso de diseño de superposición (OLC). Las estrategias OLC en última instancia intentan crear una ruta hamiltoniana a través de un gráfico de superposición, que es un problema NP-hard. Las estrategias de ruta euleriana son computacionalmente más manejables porque intentan encontrar una ruta euleriana a través de un gráfico de DeBruijn. [54]
Los genomas terminados se definen como aquellos que tienen una única secuencia contigua sin ambigüedades que representan cada replicón . [67]
El ensamblaje de la secuencia de ADN por sí solo tiene poco valor sin un análisis adicional. [9] La anotación del genoma es el proceso de adjuntar información biológica a las secuencias y consta de tres pasos principales: [68]
Las herramientas de anotación automática intentan realizar estos pasos in silico , a diferencia de la anotación manual (también conocida como curación) que involucra experiencia humana y una posible verificación experimental. [69] Idealmente, estos enfoques coexisten y se complementan entre sí en el mismo proceso de anotación (ver también a continuación).
Tradicionalmente, el nivel básico de anotación es el uso de BLAST para encontrar similitudes y luego anotar genomas basados en homólogos. [9] Más recientemente, se agrega información adicional a la plataforma de anotación. La información adicional permite a los anotadores manuales deconvolucionar discrepancias entre genes que reciben la misma anotación. Algunas bases de datos utilizan información de contexto del genoma, puntajes de similitud, datos experimentales e integraciones de otros recursos para proporcionar anotaciones del genoma a través de su enfoque de subsistemas. Otras bases de datos (por ejemplo, Ensembl ) se basan tanto en fuentes de datos curadas como en una variedad de herramientas de software en su canalización de anotación automatizada del genoma. [70] La anotación estructural consiste en la identificación de elementos genómicos, principalmente ORFs y su localización, o estructura genética. La anotación funcional consiste en adjuntar información biológica a elementos genómicos.
La necesidad de reproducibilidad y gestión eficiente de la gran cantidad de datos asociados a los proyectos genómicos significa que los procesos computacionales tienen aplicaciones importantes en la genómica. [71]
La genómica funcional es un campo de la biología molecular que intenta hacer uso de la gran cantidad de datos producidos por proyectos genómicos (como proyectos de secuenciación genómica ) para describir las funciones e interacciones de los genes (y las proteínas ). La genómica funcional se centra en los aspectos dinámicos, como la transcripción génica , la traducción y las interacciones proteína-proteína , en contraposición a los aspectos estáticos de la información genómica, como la secuencia o las estructuras del ADN. La genómica funcional intenta responder preguntas sobre la función del ADN a nivel de genes, transcripciones de ARN y productos proteicos. Una característica clave de los estudios de genómica funcional es su enfoque de todo el genoma para estas preguntas, que generalmente implica métodos de alto rendimiento en lugar de un enfoque más tradicional "gen por gen".
Una rama importante de la genómica sigue ocupándose de la secuenciación de los genomas de diversos organismos, pero el conocimiento de los genomas completos ha creado la posibilidad de que se desarrolle el campo de la genómica funcional , que se ocupa principalmente de los patrones de expresión genética en diversas condiciones. Las herramientas más importantes en este campo son los microarrays y la bioinformática .

La genómica estructural busca describir la estructura tridimensional de cada proteína codificada por un genoma dado . [72] [73] Este enfoque basado en el genoma permite un método de alto rendimiento para la determinación de la estructura mediante una combinación de enfoques experimentales y de modelado . La principal diferencia entre la genómica estructural y la predicción estructural tradicional es que la genómica estructural intenta determinar la estructura de cada proteína codificada por el genoma, en lugar de centrarse en una proteína en particular. Con secuencias de genoma completo disponibles, la predicción de la estructura se puede hacer más rápidamente a través de una combinación de enfoques experimentales y de modelado, especialmente porque la disponibilidad de grandes cantidades de genomas secuenciados y estructuras de proteínas resueltas previamente permite a los científicos modelar la estructura de las proteínas en las estructuras de homólogos resueltos previamente. La genómica estructural implica adoptar una gran cantidad de enfoques para la determinación de la estructura, incluidos métodos experimentales que utilizan secuencias genómicas o enfoques basados en modelado basados en la homología de secuencia o estructural con una proteína de estructura conocida o basados en principios químicos y físicos para una proteína sin homología con ninguna estructura conocida. A diferencia de la biología estructural tradicional , la determinación de la estructura de una proteína mediante un esfuerzo de genómica estructural a menudo (pero no siempre) se produce antes de que se conozca nada sobre la función de la proteína. Esto plantea nuevos desafíos en la bioinformática estructural , es decir, la determinación de la función de la proteína a partir de su estructura 3D . [74]
La epigenómica es el estudio del conjunto completo de modificaciones epigenéticas en el material genético de una célula, conocido como epigenoma . [75] Las modificaciones epigenéticas son modificaciones reversibles en el ADN o las histonas de una célula que afectan la expresión génica sin alterar la secuencia de ADN (Russell 2010 p. 475). Dos de las modificaciones epigenéticas más caracterizadas son la metilación del ADN y la modificación de las histonas . [76] Las modificaciones epigenéticas desempeñan un papel importante en la expresión y regulación génica, y están implicadas en numerosos procesos celulares como la diferenciación/desarrollo [77] y la tumorigénesis . [75] El estudio de la epigenética a nivel global ha sido posible solo recientemente gracias a la adaptación de ensayos genómicos de alto rendimiento. [78]
La metagenómica es el estudio de los metagenomas , material genético recuperado directamente de muestras ambientales . Este amplio campo también puede denominarse genómica ambiental, ecogenómica o genómica de comunidades. Mientras que la microbiología tradicional y la secuenciación del genoma microbiano se basan en cultivos clonales cultivados , la secuenciación temprana de genes ambientales clonó genes específicos (a menudo el gen ARNr 16S ) para producir un perfil de diversidad en una muestra natural. Este trabajo reveló que la gran mayoría de la biodiversidad microbiana se había pasado por alto con los métodos basados en cultivos . [79] Estudios recientes utilizan la secuenciación de Sanger "shotgun" o la pirosecuenciación masiva paralela para obtener muestras en gran medida imparciales de todos los genes de todos los miembros de las comunidades muestreadas. [80] Debido a su poder para revelar la diversidad previamente oculta de la vida microscópica, la metagenómica ofrece una lente poderosa para ver el mundo microbiano que tiene el potencial de revolucionar la comprensión de todo el mundo viviente. [81] [82]
Los bacteriófagos han desempeñado y siguen desempeñando un papel clave en la genética bacteriana y la biología molecular . Históricamente, se utilizaron para definir la estructura y la regulación de los genes . Además, el primer genoma en secuenciarse fue un bacteriófago . Sin embargo, la investigación sobre bacteriófagos no lideró la revolución genómica, que está claramente dominada por la genómica bacteriana. Solo muy recientemente se ha hecho prominente el estudio de los genomas de bacteriófagos, lo que permite a los investigadores comprender los mecanismos subyacentes a la evolución de los fagos . Las secuencias del genoma de los bacteriófagos se pueden obtener mediante la secuenciación directa de bacteriófagos aislados, pero también se pueden derivar como parte de genomas microbianos. El análisis de genomas bacterianos ha demostrado que una cantidad sustancial de ADN microbiano consiste en secuencias de profagos y elementos similares a profagos. [83] Una minería de datos detallada de estas secuencias ofrece información sobre el papel de los profagos en la conformación del genoma bacteriano: en general, este método verificó muchos grupos de bacteriófagos conocidos, lo que lo convierte en una herramienta útil para predecir las relaciones de los profagos a partir de genomas bacterianos. [84] [85]
En la actualidad, hay 24 cianobacterias para las que se dispone de una secuencia genómica completa. Quince de estas cianobacterias proceden del entorno marino. Se trata de seis cepas de Prochlorococcus , siete cepas de Synechococcus marino , Trichodesmium erythraeum IMS101 y Crocosphaera watsonii WH8501. Varios estudios han demostrado cómo estas secuencias podrían utilizarse con mucho éxito para inferir importantes características ecológicas y fisiológicas de las cianobacterias marinas. Sin embargo, hay muchos más proyectos genómicos en curso, entre los que se encuentran otros aislados de Prochlorococcus y Synechococcus marino, Acaryochloris y Prochloron , las cianobacterias filamentosas fijadoras de N 2 Nodularia spumigena , Lyngbya aestuarii y Lyngbya majuscula , así como bacteriófagos que infectan a las cianobacterias marinas. Por tanto, el creciente conjunto de información genómica también puede aprovecharse de una forma más general para abordar problemas globales aplicando un enfoque comparativo. Algunos ejemplos nuevos y apasionantes de progreso en este campo son la identificación de genes para ARN reguladores, conocimientos sobre el origen evolutivo de la fotosíntesis o la estimación de la contribución de la transferencia horizontal de genes a los genomas que se han analizado. [86]

La genómica ha proporcionado aplicaciones en muchos campos, incluida la medicina , la biotecnología , la antropología y otras ciencias sociales . [44]
Las tecnologías genómicas de próxima generación permiten a los médicos e investigadores biomédicos aumentar drásticamente la cantidad de datos genómicos recopilados en grandes poblaciones de estudio. [87] Cuando se combina con nuevos enfoques informáticos que integran muchos tipos de datos con datos genómicos en la investigación de enfermedades, esto permite a los investigadores comprender mejor las bases genéticas de la respuesta a los medicamentos y la enfermedad. [88] [89] Los primeros esfuerzos para aplicar el genoma a la medicina incluyeron los de un equipo de Stanford dirigido por Euan Ashley , quien desarrolló las primeras herramientas para la interpretación médica de un genoma humano. [90] [91] [92] El programa de investigación Genomes2People en el Brigham and Women's Hospital , Broad Institute y Harvard Medical School se estableció en 2012 para realizar investigaciones empíricas en la traducción de la genómica en salud. El Brigham and Women's Hospital abrió una Clínica de Genómica Preventiva en agosto de 2019, y el Massachusetts General Hospital lo siguió un mes después. [93] [94] El programa de investigación All of Us tiene como objetivo recopilar datos de secuencias del genoma de 1 millón de participantes para convertirse en un componente crítico de la plataforma de investigación de medicina de precisión. [95]
El crecimiento del conocimiento genómico ha permitido aplicaciones cada vez más sofisticadas de la biología sintética . [96] En 2010, investigadores del Instituto J. Craig Venter anunciaron la creación de una especie parcialmente sintética de bacteria , Mycoplasma laboratorium , derivada del genoma de Mycoplasma genitalium . [97]
La genómica de poblaciones se ha desarrollado como un campo popular de investigación, donde los métodos de secuenciación genómica se utilizan para realizar comparaciones a gran escala de secuencias de ADN entre poblaciones, más allá de los límites de los marcadores genéticos como los productos de PCR de corto alcance o los microsatélites utilizados tradicionalmente en la genética de poblaciones . La genómica de poblaciones estudia los efectos de todo el genoma para mejorar nuestra comprensión de la microevolución, de modo que podamos aprender lahistoria filogenética y la demografía de una población. [98] Los métodos genómicos de poblaciones se utilizan para muchos campos diferentes, incluidos la biología evolutiva , la ecología , la biogeografía , la biología de la conservación y la gestión pesquera . De manera similar, la genómica del paisaje se ha desarrollado a partir de la genética del paisaje para utilizar métodos genómicos para identificar relaciones entre patrones de variación ambiental y genética.
Los conservacionistas pueden utilizar la información recopilada mediante la secuenciación genómica para evaluar mejor los factores genéticos clave para la conservación de las especies, como la diversidad genética de una población o si un individuo es heterocigoto para un trastorno genético hereditario recesivo. [99] Al utilizar datos genómicos para evaluar los efectos de los procesos evolutivos y detectar patrones de variación en una población determinada, los conservacionistas pueden formular planes para ayudar a una especie determinada sin dejar tantas variables desconocidas como las que no se abordan con los enfoques genéticos estándar . [100]
{{cite book}}: CS1 maint: DOI inactive as of September 2024 (link)libro electrónico ISBN 978-94-007-5561-1 ISSN 1559-0836 ISSN electrónico 1868-0402