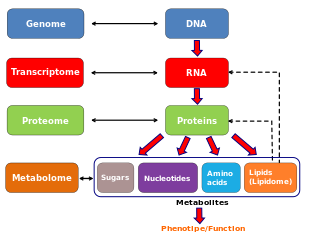
El metaboloma se refiere al conjunto completo de sustancias químicas de moléculas pequeñas que se encuentran dentro de una muestra biológica. [1] La muestra biológica puede ser una célula , un orgánulo celular , un órgano , un tejido , un extracto de tejido, un biofluido o un organismo completo. Las sustancias químicas de moléculas pequeñas que se encuentran en un metaboloma determinado pueden incluir tanto metabolitos endógenos que son producidos naturalmente por un organismo (como aminoácidos , ácidos orgánicos , ácidos nucleicos , ácidos grasos , aminas , azúcares , vitaminas , cofactores , pigmentos , antibióticos , etc.) como sustancias químicas exógenas (como medicamentos, contaminantes ambientales , aditivos alimentarios , toxinas y otros xenobióticos ) que no son producidos naturalmente por un organismo. [2] [3]
En otras palabras, existe un metaboloma endógeno y un metaboloma exógeno. El metaboloma endógeno puede subdividirse aún más para incluir un metaboloma "primario" y uno "secundario" (particularmente cuando se hace referencia a metabolomas de plantas o microbios). Un metabolito primario está directamente involucrado en el crecimiento, desarrollo y reproducción normales. Un metabolito secundario no está directamente involucrado en esos procesos, pero generalmente tiene una función ecológica importante. Los metabolitos secundarios pueden incluir pigmentos, antibióticos o productos de desecho derivados de xenobióticos parcialmente metabolizados. El estudio del metaboloma se llama metabolómica .
La palabra metaboloma parece ser una combinación de las palabras "metabolito" y " cromosoma ". Fue construida para implicar que los metabolitos son codificados indirectamente por los genes o actúan sobre los genes y productos génicos. El término "metaboloma" se utilizó por primera vez en 1998 [1] [4] y probablemente fue acuñado para coincidir con los términos biológicos existentes que se refieren al conjunto completo de genes (el genoma ), el conjunto completo de proteínas (el proteoma ) y el conjunto completo de transcripciones (el transcriptoma ). El primer libro sobre metabolómica se publicó en 2003. [5] La primera revista dedicada a la metabolómica (titulada simplemente "Metabolomics") se lanzó en 2005 y actualmente está editada por el profesor Roy Goodacre . Algunos de los primeros artículos más significativos sobre el análisis del metaboloma se enumeran en las referencias a continuación. [6] [7] [8] [9]
El metaboloma refleja la interacción entre el genoma de un organismo y su entorno. Como resultado, el metaboloma de un organismo puede servir como una excelente sonda de su fenotipo (es decir, el producto de su genotipo y su entorno). Los metabolitos se pueden medir (identificar, cuantificar o clasificar) utilizando una serie de tecnologías diferentes, incluyendo la espectroscopia de RMN y la espectrometría de masas . [10] La mayoría de los métodos de espectrometría de masas (MS) deben estar acoplados a varias formas de cromatografía líquida (LC), cromatografía de gases (GC) o electroforesis capilar (CE) para facilitar la separación de compuestos. Cada método suele ser capaz de identificar o caracterizar entre 50 y 5000 metabolitos diferentes o "características" de metabolitos a la vez, dependiendo del instrumento o protocolo que se utilice. Actualmente no es posible analizar toda la gama de metabolitos con un solo método analítico.
La espectroscopia de resonancia magnética nuclear (RMN) es una técnica de química analítica que mide la absorción de la radiación de radiofrecuencia de núcleos específicos cuando las moléculas que contienen esos núcleos se colocan en campos magnéticos fuertes . La frecuencia (es decir, el desplazamiento químico ) a la que absorbe un átomo o núcleo determinado depende en gran medida del entorno químico (enlace, estructura química, vecinos más cercanos, disolvente) de ese átomo en una molécula determinada. Los patrones de absorción de RMN producen picos de "resonancia" a diferentes frecuencias o diferentes desplazamientos químicos; esta colección de picos se denomina espectro de RMN . Debido a que cada compuesto químico tiene una estructura química diferente, cada compuesto tendrá un espectro de RMN único (o casi único). Como resultado, la RMN es particularmente útil para la caracterización, identificación y cuantificación de moléculas pequeñas, como los metabolitos. El uso generalizado de la RMN para estudios metabólicos "clásicos", junto con su capacidad excepcional para manejar mezclas complejas de metabolitos, es probablemente la razón por la que la RMN fue una de las primeras tecnologías en ser ampliamente adoptadas para mediciones rutinarias del metaboloma. Como técnica analítica, la RMN no es destructiva, no tiene sesgos, es fácilmente cuantificable, requiere poca o ninguna separación, permite la identificación de compuestos nuevos y no necesita derivatización química. La RMN es particularmente adecuada para detectar compuestos que son menos manejables para el análisis LC-MS , como azúcares, aminas o líquidos volátiles o análisis GC-MS , como moléculas grandes (>500 Da) o compuestos relativamente no reactivos. La RMN no es una técnica muy sensible con un límite inferior de detección de aproximadamente 5 μM. Por lo general, se pueden identificar entre 50 y 150 compuestos mediante estudios metabolómicos basados en RMN.
La espectrometría de masas es una técnica analítica que mide la relación masa-carga de las moléculas. Las moléculas o fragmentos moleculares se cargan o ionizan típicamente rociándolos a través de un campo cargado ( ionización por electrospray ), bombardeándolos con electrones desde un filamento caliente ( ionización electrónica ) o disparándolos con un láser cuando se colocan en placas especialmente recubiertas (ionización por desorción láser asistida por matriz). Las moléculas cargadas se impulsan a través del espacio utilizando electrodos o imanes y se miden su velocidad, tasa de curvatura u otras características físicas para determinar su relación masa-carga. A partir de estos datos se puede determinar la masa de la molécula original. Una mayor fragmentación de la molécula a través de colisiones controladas con moléculas de gas o con electrones puede ayudar a determinar la estructura de las moléculas. También se pueden utilizar mediciones de masa muy precisas para determinar las fórmulas elementales o la composición elemental de los compuestos. La mayoría de las formas de espectrometría de masas requieren algún tipo de separación mediante cromatografía líquida o cromatografía de gases . Este paso de separación es necesario para simplificar los espectros de masas resultantes y permitir una identificación más precisa de los compuestos. Algunos métodos de espectrometría de masas también requieren que las moléculas se deriven o modifiquen químicamente para que sean más adecuadas para la separación cromatográfica (esto es particularmente cierto para GC-MS). Como técnica analítica, MS es un método muy sensible que requiere muy poca muestra (<1 ng de material o <10 μL de un biofluido) y puede generar señales para miles de metabolitos a partir de una sola muestra. Los instrumentos MS también se pueden configurar para análisis de metabolomas de muy alto rendimiento (cientos a miles de muestras al día). La cuantificación de metabolitos y la caracterización de nuevas estructuras de compuestos es más difícil por MS que por RMN. LC-MS es particularmente adecuada para detectar moléculas hidrófobas ( lípidos , ácidos grasos) y péptidos, mientras que GC-MS es mejor para detectar moléculas pequeñas (<500 Da) y compuestos altamente volátiles ( ésteres , aminas, cetonas , alcanos , tioles ).
A diferencia del genoma o incluso del proteoma , el metaboloma es una entidad altamente dinámica que puede cambiar drásticamente en un período de apenas segundos o minutos. Como resultado, existe un creciente interés en medir los metabolitos durante múltiples períodos de tiempo o en intervalos de tiempo cortos utilizando versiones modificadas de la metabolómica basada en RMN o MS.
Como el metaboloma de un organismo está definido en gran medida por su genoma, las distintas especies tendrán metabolomas diferentes. De hecho, el hecho de que el metaboloma de un tomate sea diferente del de una manzana es la razón por la que estas dos frutas tienen un sabor tan diferente. Además, los distintos tejidos, órganos y biofluidos asociados a esos órganos y tejidos también pueden tener metabolomas claramente diferentes. El hecho de que los distintos organismos y tejidos y biofluidos tengan metabolomas tan diferentes ha llevado al desarrollo de una serie de bases de datos de metabolomas específicas de cada organismo y de cada biofluido. Algunas de las bases de datos de metabolomas más conocidas incluyen la Base de Datos del Metaboloma Humano o HMDB, [11] la Base de Datos del Metaboloma de Levadura o YMDB, [12] la Base de Datos del Metaboloma de E. coli o ECMDB, [13] la base de datos del metaboloma de Arabidopsis o AraCyc [14] así como la Base de Datos del Metaboloma de Orina, [15] la Base de Datos del Metaboloma del Líquido Cefalorraquídeo (LCR) [16] y la Base de Datos del Metaboloma del Suero . [17] Las últimas tres bases de datos son específicas para los biofluidos humanos. También existen varias bases de datos de metabolitos generales muy populares, incluyendo KEGG , [18] MetaboLights, [19] la Base de Datos del Metaboloma de Golm , [20] MetaCyc , [21] LipidMaps [22] y Metlin . [23] Las bases de datos de metabolomas se pueden distinguir de las bases de datos de metabolitos en que las bases de datos de metabolitos contienen datos de metabolitos ligeramente anotados o sinópticos de múltiples organismos, mientras que las bases de datos de metabolomas contienen datos químicos, de vías, espectrales y de concentración de metabolitos muy detallados y ampliamente referenciados para organismos específicos.
La base de datos del metaboloma humano (HMDB) es una base de datos de libre acceso que contiene información detallada sobre más de 40.000 metabolitos que ya se han identificado o que es probable que se encuentren en el cuerpo humano. La HMDB contiene tres tipos de información:
Los datos químicos incluyen más de 40.000 estructuras de metabolitos con descripciones detalladas, clasificaciones químicas extensas, información de síntesis y propiedades químicas observadas/calculadas. También contiene casi 10.000 espectros de RMN , GC-MS y LC/MS medidos experimentalmente de más de 1.100 metabolitos diferentes. La información clínica incluye datos sobre más de 10.000 concentraciones de metabolitos en biofluidos, información sobre la concentración de metabolitos en más de 600 enfermedades humanas diferentes y datos de vías para más de 200 errores innatos del metabolismo diferentes. La información bioquímica incluye casi 6.000 secuencias de proteínas (y ADN) y más de 5.000 reacciones bioquímicas vinculadas a estas entradas de metabolitos. La HMDB admite una amplia variedad de consultas en línea, incluidas búsquedas de texto, búsquedas de estructura química, búsquedas de similitud de secuencias y búsquedas de similitud espectral. Esto lo hace particularmente útil para los investigadores metabolómicos que intentan identificar o comprender los metabolitos en estudios metabolómicos clínicos. La primera versión de la HMDB se publicó el 1 de enero de 2007 y fue compilada por científicos de la Universidad de Alberta y la Universidad de Calgary . En ese momento, informaron datos sobre 2.500 metabolitos, 1.200 medicamentos y 3.500 componentes de alimentos. Desde entonces, estos científicos han ampliado enormemente la colección. La versión 3.5 de la HMDB contiene más de 16.000 metabolitos endógenos, más de 1.500 medicamentos y más de 22.000 constituyentes de alimentos o metabolitos de alimentos. [24]
Los científicos de la Universidad de Alberta han estado caracterizando sistemáticamente metabolomas específicos de biofluidos, incluyendo el metaboloma sérico, [17] el metaboloma urinario, [15] el metaboloma del líquido cefalorraquídeo (LCR) [16] y el metaboloma de la saliva. Estos esfuerzos han implicado tanto análisis metabolómico experimental (que implica ensayos de RMN, GC-MS, ICP-MS , LC-MS y HPLC ) como una extensa exploración bibliográfica. Según sus datos, el metaboloma sérico humano contiene al menos 4.200 compuestos diferentes (incluidos muchos lípidos), el metaboloma urinario humano contiene al menos 3.000 compuestos diferentes (incluidos cientos de volátiles y metabolitos microbianos intestinales), el metaboloma del LCR humano contiene casi 500 compuestos diferentes, mientras que el metaboloma de la saliva humana contiene aproximadamente 400 metabolitos diferentes, incluidos muchos productos bacterianos.
La base de datos del metaboloma de la levadura es una base de datos en línea de libre acceso que contiene más de 2000 metabolitos de moléculas pequeñas que se encuentran en la Saccharomyces cerevisiae ( levadura de panadería ) o que esta produce. La YMDB contiene dos tipos de información:
La información química de YMDB incluye 2027 estructuras de metabolitos con descripciones detalladas de metabolitos, clasificaciones químicas extensas, información de síntesis y propiedades químicas observadas/calculadas. También contiene casi 4000 espectros de RMN, GC-MS y LC/MS obtenidos de más de 500 metabolitos diferentes. La información bioquímica de YMDB incluye más de 1100 secuencias de proteínas (y ADN) y más de 900 reacciones bioquímicas. YMDB admite una amplia variedad de consultas, incluidas búsquedas de texto, búsquedas de estructura química, búsquedas de similitud de secuencias y búsquedas de similitud espectral. Esto lo hace particularmente útil para investigadores metabolómicos que estudian la levadura como organismo modelo o que buscan optimizar la producción de bebidas fermentadas (vino, cerveza).
La ionización por electrospray secundaria ( SESI-HRMS) es una técnica analítica no invasiva que nos permite monitorear las actividades metabólicas de la levadura. SESI-HRMS ha encontrado alrededor de 300 metabolitos en el proceso de fermentación de la levadura, lo que sugiere que una gran cantidad de metabolitos de glucosa no se encuentran reportados en la literatura. [25]
La base de datos del metaboloma de E. coli es una base de datos en línea de libre acceso que contiene más de 2700 metabolitos de moléculas pequeñas que se encuentran en Escherichia coli (cepa de E. coli K12, MG1655) o que son producidos por ella. La base de datos del metaboloma de E. coli contiene dos tipos de información:
La información química incluye más de 2.700 estructuras de metabolitos con descripciones detalladas de metabolitos, clasificaciones químicas extensas, información de síntesis y propiedades químicas observadas/calculadas. También contiene casi 5.000 espectros de RMN, GC-MS y LC-MS de más de 600 metabolitos diferentes. La información bioquímica incluye más de 1.600 secuencias de proteínas (y ADN) y más de 3.100 reacciones bioquímicas vinculadas a estas entradas de metabolitos. El ECMDB admite muchos tipos diferentes de consultas en línea, incluidas búsquedas de texto, búsquedas de estructura química, búsquedas de similitud de secuencias y búsquedas de similitud espectral. Esto lo hace particularmente útil para investigadores metabolómicos que estudian E. coli como organismo modelo.
La ionización por electrospray secundaria (SESI-MS) puede discriminar entre once cepas de E. Coli gracias al perfil de compuestos orgánicos volátiles. [26]
En 2021 se publicó en línea el primer atlas del metaboloma cerebral del cerebro del ratón (y de un animal (un mamífero) en diferentes etapas de la vida). Los datos se diferencian por regiones cerebrales y los cambios metabólicos podrían "mapear con atlas cerebrales de genes y proteínas existentes". [27] [28]
La microbiota intestinal humana contribuye a la etiología del cáncer colorrectal a través de su metaboloma. [29] En particular, la conversión de ácidos biliares primarios en ácidos biliares secundarios como consecuencia del metabolismo bacteriano en el colon promueve la carcinogénesis . [29]