
Hi-C es una técnica genómica y epigenómica de alto rendimiento para capturar la conformación de la cromatina (3C) . [1] En general, Hi-C se considera un derivado de una serie de tecnologías de captura de conformación cromosómica , que incluyen, entre otras, 3C (captura de conformación cromosómica), 4C (captura de conformación cromosómica en chip/captura de conformación cromosómica circular) y 5C (captura de conformación cromosómica con copia de carbón). [1] [2] [3] [4] Hi-C detecta de manera integral las interacciones de la cromatina en todo el genoma en el núcleo celular mediante la combinación de enfoques de 3C y secuenciación de próxima generación (NGS) y se ha considerado como un salto cualitativo en el desarrollo de la tecnología C (tecnologías basadas en captura de conformación cromosómica) y el comienzo de la genómica 3D. [2] [3] [4]
Similar a la técnica clásica 3C, Hi-C mide la frecuencia (como promedio sobre una población celular) en la que dos fragmentos de ADN se asocian físicamente en el espacio 3D, vinculando la estructura cromosómica directamente a la secuencia genómica. [4] El procedimiento general de Hi-C implica primero la reticulación del material de cromatina utilizando formaldehído . [3] [4] Luego, la cromatina se solubiliza y fragmenta, y los loci interactuantes se vuelven a ligar para crear una biblioteca genómica de moléculas de ADN quimérico . [4] La abundancia relativa de estas quimeras, o productos de ligadura, está correlacionada con la probabilidad de que los respectivos fragmentos de cromatina interactúen en el espacio 3D en toda la población celular. [4] Mientras que 3C se centra en el análisis de un conjunto de loci genómicos predeterminados para ofrecer investigaciones de “uno contra algunos” de la conformación de las regiones cromosómicas de interés, Hi-C permite la elaboración de perfiles de interacción de “todos contra todos” al etiquetar toda la cromatina fragmentada con un nucleótido biotinilado antes de la ligadura. [3] [4] Como resultado, las uniones de ligadura marcadas con biotina se pueden purificar de manera más eficiente mediante perlas magnéticas recubiertas de estreptavidina , y los datos de interacción de la cromatina se pueden obtener mediante secuenciación directa de la biblioteca Hi-C. [3] [4]
Los análisis de datos de Hi-C no solo revelan la estructura genómica general de los cromosomas de mamíferos , sino que también ofrecen información sobre las propiedades biofísicas de la cromatina, así como contactos más específicos y de largo alcance entre elementos genómicos distantes (por ejemplo, entre genes y elementos reguladores ), [4] [5] [6] incluyendo cómo estos cambian con el tiempo en respuesta a estímulos. [7] En los últimos años, Hi-C ha encontrado su aplicación en una amplia variedad de campos biológicos, incluido el crecimiento y la división celular , la regulación de la transcripción , la determinación del destino , el desarrollo, las enfermedades autoinmunes y la evolución del genoma . [7] [5] [6] Al combinar los datos de Hi-C con otros conjuntos de datos, como mapas de todo el genoma de modificaciones de la cromatina y perfiles de expresión genética, también se pueden delinear los roles funcionales de la conformación de la cromatina en la regulación y estabilidad del genoma. [4]
En sus inicios, Hi-C era una tecnología de baja resolución y alto ruido que solo era capaz de describir regiones de interacción de cromatina dentro de un tamaño de bin de 1 millón de pares de bases (Mb). [1] La biblioteca Hi-C también requería varios días para construirse, [4] [8] y los conjuntos de datos en sí mismos eran bajos tanto en rendimiento como en reproducibilidad. [9] Sin embargo, los datos Hi-C ofrecían nuevos conocimientos sobre la conformación de la cromatina, así como sobre las arquitecturas nucleares y genómicas, y estas perspectivas motivaron a los científicos a realizar esfuerzos para modificar la técnica durante la última década.
Entre 2012 y 2015, se han realizado varias modificaciones al protocolo Hi-C, con digestión con 4 cortadores [10] o profundidad de secuenciación más profunda adaptada para obtener una mayor resolución. [8] [9] [11] El uso de endonucleasas de restricción que cortan con mayor frecuencia, o DNaseI y nucleasas microcócicas también aumentaron significativamente la resolución del método. [12] Más recientemente (2017), Belaghzal et al. describieron un protocolo Hi-C 2.0 que pudo lograr una resolución de kilobase (kb). [12] La adaptación clave al protocolo base fue la eliminación del paso de solubilización de SDS después de la digestión para preservar la estructura nuclear y evitar la ligadura aleatoria entre la cromatina fragmentada mediante ligadura dentro de los núcleos intactos, que formaron la base del Hi-C in situ. [12] En 2021, Lafontaine et al. describieron Hi-C 3.0, con una resolución más alta lograda al mejorar la reticulación con formaldehído seguido de glutarato de disuccinimidilo (DSG). [13] Mientras que el formaldehído captura los grupos amino e imino tanto de las proteínas como del ADN, los ésteres NHS en DSG reaccionan con aminas primarias en las proteínas y pueden capturar interacciones amina-amina. [13] Estas actualizaciones del protocolo base permitieron a los científicos observar estructuras conformacionales más detalladas, como el compartimento cromosómico y los dominios de asociación topológica (TAD), así como características conformacionales de alta resolución, como los bucles de ADN. [12] [13]
Hasta la fecha, ya han surgido diversos derivados de Hi-C, incluidos Hi-C in situ, Hi-C bajo, Hi-C SAFE y Micro-C, con características distintivas relacionadas con diferentes aspectos del Hi-C estándar, pero el principio básico sigue siendo el mismo.
El esquema del flujo de trabajo clásico de Hi-C es el siguiente: las células se reticulan con formaldehído; la cromatina se digiere con una enzima de restricción que genera un saliente 5' ; el saliente 5' se llena con bases biotiniladas y el ADN de extremos romos resultante se liga. [1] Los productos de ligadura, con biotina en la unión, se seleccionan para usar estreptavidina y se procesan posteriormente para preparar una biblioteca lista para los esfuerzos de secuenciación posteriores. [1]
Las interacciones por pares que Hi-C puede capturar en todo el genoma son inmensas, por lo que es importante analizar un tamaño de muestra adecuadamente grande para capturar interacciones únicas que solo se pueden observar en una minoría de la población general. [4] Para obtener una biblioteca de alta complejidad de productos de ligación que garantice una alta resolución y profundidad de los datos, se requiere una muestra de 20 a 25 millones de células como entrada para Hi-C. [3] [4] Las muestras humanas primarias, que pueden estar disponibles solo en un menor número de células, podrían usarse para la preparación estándar de la biblioteca Hi-C con tan solo 1 a 5 millones de células. [4] Sin embargo, el uso de una entrada de células tan baja puede estar asociado con una baja complejidad de la biblioteca, lo que da como resultado un alto porcentaje de lecturas duplicadas durante la preparación de la biblioteca. [4]
El Hi-C estándar proporciona datos sobre interacciones por pares con una resolución de 1 a 10 Mb, requiere una gran profundidad de secuenciación y el protocolo tarda alrededor de 7 días en completarse. [3] [4] [14]
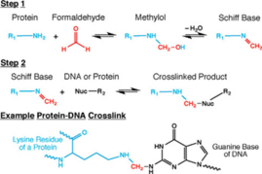
Las membranas celulares y nucleares son altamente permeables al formaldehído. [4] [15] [16] La reticulación con formaldehído se emplea con frecuencia para la detección y cuantificación de interacciones ADN-proteína y proteína-proteína. [15] De interés en el contexto de Hi-C, y todos los métodos basados en 3C, es la capacidad del formaldehído para capturar interacciones cromosómicas cis entre segmentos distales de cromatina. [1] [4] [15] [16] Lo hace formando enlaces covalentes entre segmentos de cromatina espacialmente adyacentes. El formaldehído puede reaccionar con macromoléculas en dos pasos: primero reacciona con un grupo nucleofílico en una base de ADN, por ejemplo, y forma un aducto de metilol, que luego se convierte en una base de Schiff . [15] En el segundo paso, la base de Schiff, que puede descomponerse rápidamente, forma un puente de metileno con otro grupo funcional en otra molécula. [15] También puede hacer este puente de metileno con una pequeña molécula en solución como la glicina , que se usa en exceso para apagar el formaldehído en Hi-C. [1] [4] [15] [16] Los extintores normalmente pueden ejercer un efecto sobre el formaldehído desde fuera de la célula. [15] Una característica clave de esta reacción de reticulación de formaldehído de dos pasos es que todas las reacciones son reversibles, lo que es vital para la captura de cromatina. [1] [4] [15] [16]
La reticulación es un paso fundamental del flujo de trabajo de captura de cromatina, ya que la lectura funcional de la técnica es la frecuencia con la que dos regiones genómicas se reticulan entre sí. [4] Por lo tanto, la estandarización de este paso es importante y para eso, se deben considerar las posibles fuentes de variación. [4] La presencia de suero, que contiene una alta concentración de proteína, en los medios de cultivo puede disminuir la concentración efectiva de formaldehído disponible para la reticulación de la cromatina, al secuestrarlo en los medios de cultivo. [4] Por lo tanto, en los casos en que se utiliza suero en el cultivo, debe eliminarse para el paso de reticulación. [4] La naturaleza de las células, es decir, si están en suspensión o son adherentes, también es una consideración pertinente para el paso de reticulación. [4] Las células adherentes se unen a las superficies con la ayuda de mecanismos moleculares de los citoesqueletos . [4] Se ha demostrado que existe un vínculo entre la morfología nuclear y celular mantenida por el citoesqueleto que, si se altera, puede afectar negativamente a la organización nuclear global. [4] Por lo tanto, las células adherentes deben reticularse mientras aún están unidas a su superficie de cultivo. [4]
Las células se lisan en hielo con un tampón hipotónico frío que contiene cloruro de sodio , Tris-HCl a pH 8,0 y detergente no iónico IGEPAL CA-630 , suplementado con inhibidores de proteasa . [4] [16] Los inhibidores de proteasa y la incubación en hielo ayudan a preservar la integridad de los complejos de cromatina reticulada de las proteasas endógenas. [4] [16] El paso de lisis ayuda a liberar el material nucleico de las células. [1] [4] [16]
Después de la lisis celular, la cromatina se solubiliza con SDS diluido para eliminar las proteínas que no se han reticulado y para abrir la cromatina y hacerla más accesible para la posterior digestión mediada por endonucleasas de restricción. [4] Si la incubación con SDS excede los 10 minutos recomendados, los enlaces cruzados de formaldehído se pueden revertir y, por lo tanto, la incubación con SDS debe ser seguida inmediatamente por una incubación en hielo. [4] Se utiliza un detergente no iónico llamado Triton X-100 para apagar el SDS con el fin de evitar la desnaturalización de la enzima en el siguiente paso. [4]
Cualquier enzima de restricción que genere un saliente 5', como HindIII , se puede utilizar para digerir la cromatina ahora accesible durante la noche. [4] [16] Este saliente 5' proporciona la plantilla requerida por el fragmento Klenow de la ADN polimerasa I para agregar CTP o ATP biotinilado a los extremos digeridos de la cromatina. [4] [16] Este paso permite la selección de productos de ligadura Hi-C para la preparación de la biblioteca. [4] [16]
Se realiza una ligadura por dilución en fragmentos de ADN que aún están reticulados entre sí para favorecer la ligadura intramolecular de fragmentos dentro del mismo complejo de cromatina en lugar de eventos de ligadura entre fragmentos a través de diferentes complejos. [4] [16] Dado que este paso de ligadura ocurre entre fragmentos de ADN de extremos romos (ya que los extremos pegajosos se han rellenado con bases marcadas con biotina), se permite que la reacción continúe hasta 4 horas para compensar su ineficiencia inherente. [16] Como resultado de la ligadura de proximidad, los sitios terminales HindIII se pierden y se genera un sitio NheI. [1]
Los productos de ligación marcados con biotina se pueden purificar utilizando extracción de ADN con fenol-cloroformo . [4] [16] [17] Para eliminar cualquier fragmento con extremos marcados con biotina que no se hayan ligado, se utiliza la ADN polimerasa T4 con actividad exonucleasa 3' a 5' para eliminar nucleótidos de los extremos de dichos fragmentos. [4] [16] [18] Este paso asegura que ninguno de estos fragmentos no ligados se seleccione para la preparación de la biblioteca. [4] [16] La reacción se detiene con EDTA y el ADN se purifica una vez más utilizando extracción de ADN con fenol-cloroformo. [4] [16]
El tamaño ideal de los fragmentos de ADN para la biblioteca de secuenciación depende de la plataforma de secuenciación que se utilizará. [4] [16] El ADN se puede cortar primero en fragmentos de alrededor de 300 a 500 pb de longitud mediante sonicación . [4] [16] [17] Los fragmentos de este tamaño son adecuados para la secuenciación de alto rendimiento. [4] [16] [17] Después de la sonicación, los fragmentos se pueden seleccionar por tamaño utilizando perlas AMPure XP de Beckman Coulter para obtener productos de ligadura con una distribución de tamaño entre 150 y 300 pb. [4] [17] Esta es la ventana de tamaño de fragmento óptima para la formación de grupos HiSeq. [4] [17]
El cizallamiento del ADN provoca roturas asimétricas del ADN y debe repararse antes de la extracción con biotina y la ligadura del adaptador de secuenciación. [4] [16] Esto se logra utilizando una combinación de enzimas que rellenan los salientes 5' y añaden grupos fosfato 5' y adenilato a los extremos 3' de los fragmentos para permitir la ligadura de los adaptadores de secuenciación. [4] [16]
Usando un exceso de perlas de estreptavidina, como la solución de perlas de estreptavidina My-One C1 de Dynabeads , los productos de ligadura Hi-C biotinilados se pueden extraer y enriquecer para. [4] [16] La ligadura de los adaptadores de extremos emparejados de Illumina se realiza mientras los fragmentos de ADN están unidos a las perlas de estreptavidina. [4] [16] [17] La adsorción a las perlas aumenta la eficiencia de la ligadura de estos fragmentos de ADN de extremos romos a los adaptadores, ya que disminuye su movilidad. [4] [16] [17]
Una vez completada la ligadura de los adaptadores, se realiza la amplificación por PCR de la biblioteca. [4] [16] El paso de PCR puede introducir una gran cantidad de duplicados en una muestra de producto de ligadura Hi-C de baja complejidad como resultado de la sobreamplificación. [4] [16] Esto da como resultado que se capturen muy pocas interacciones y, a menudo, esto se debe a que el tamaño de la muestra de entrada tenía una baja cantidad de células. [4] [16] Es importante titular la cantidad de ciclos necesarios para obtener al menos 50 ng de ADN de la biblioteca Hi-C para la secuenciación. [4] [16] Cuanto menor sea el número de ciclos, mejor para que no haya artefactos de PCR (como amplicones fuera del objetivo, no especificidad, etc.). [4] [16] El rango ideal de ciclos de PCR es de 9 a 15 y es más ideal agrupar múltiples reacciones de PCR para obtener suficiente ADN para la secuenciación, que aumentar la cantidad de ciclos para una reacción de PCR. [4] [16] Los productos de PCR se purifican nuevamente utilizando perlas AMPure para eliminar los dímeros de cebadores y luego se cuantifican antes de secuenciarlos. [4] [16] Las regiones de cromatina que interactúan entre sí se identifican luego mediante secuenciación de extremos emparejados de los productos biotinilados y ligados. [4] [16]
Cualquier plataforma que permita que los fragmentos ligados se secuencien a través de la unión NheI ( Roche 454) o mediante lecturas de extremos emparejados o emparejados ( plataformas Illumina GA y HiSeq ) sería adecuada para Hi-C. [4] Antes de la secuenciación de alto rendimiento, se debe verificar la calidad de la biblioteca utilizando la secuenciación de Sanger , en la que la lectura de secuenciación larga se leerá a través de la unión de biotina. [4] Las lecturas de treinta y seis o 50 pb son suficientes para identificar la mayoría de los pares de interacción de cromatina utilizando la secuenciación de extremos emparejados de Illumina. [4] Dado que el tamaño promedio de los fragmentos en la biblioteca es de 250 pb, se ha descubierto que las lecturas de extremos emparejados de 50 pb son óptimas para la secuenciación de la biblioteca Hi-C. [4]
Existen varios puntos de presión a lo largo del flujo de trabajo de preparación de muestras de Hi-C que están bien documentados y reportados. [4] [16] El ADN en varias etapas se puede ejecutar en geles de agarosa al 0,8% para analizar la distribución del tamaño de los fragmentos. [4] [16] Esto es particularmente importante después del corte de los pasos de selección de tamaño. [4] [16] La degradación del ADN también se puede monitorear como manchas que aparecen como resultado de productos de bajo peso molecular en geles. [4] [16] La degradación puede ocurrir debido a no agregar suficientes inhibidores de proteasa durante la lisis, actividad de nucleasa endógena o degradación térmica debido a una formación de hielo incorrecta. [4] [16] Se pueden realizar reacciones de PCR 3C para probar la formación de productos de ligadura de proximidad. [4] [16]
El Hi-C estándar tiene un alto costo de entrada de número de células, requiere una secuenciación profunda, genera datos de baja resolución y sufre la formación de moléculas redundantes que contribuyen a las bibliotecas de baja complejidad cuando el número de células es bajo. [4] [16] [17] Para combatir estos problemas con el fin de poder aplicar esta técnica en contextos donde el número de células es un factor limitante, por ejemplo, con el trabajo primario con células humanas, se han desarrollado varias variantes de Hi-C desde la primera conceptualización de Hi-C. [3]
Las cuatro clases principales bajo las cuales se incluyen las variantes de Hi-C son: ligadura por dilución, ligadura in situ, célula única y sistemas de mejora de bajo ruido. [3] La Hi-C estándar es un tipo de ligadura por dilución y otras ligaduras por dilución incluyen DNase Hi-C y Capture Hi-C. [3] A diferencia de la Hi-C estándar y Capture, la Hi-C de DNase requiere solo de 2 a 5 millones de células como entrada, utiliza DNaseI para la fragmentación de la cromatina y emplea una ligadura de proximidad por dilución en gel. [3] [19] [20] Se ha demostrado que el uso de DNaseI mejora en gran medida la eficiencia y la resolución de Hi-C. [3] [19] Capture Hi-C es una técnica de ensayo de todo el genoma para observar las interacciones de la cromatina de loci específicos utilizando una captura basada en hibridación de regiones genómicas específicas. [20] Fue desarrollado por primera vez por Mifsud et al. para mapear contactos promotores de largo alcance en células humanas mediante la generación de una biblioteca de cebos de ARN biotinilados que apuntaba a 21.841 regiones promotoras. [20] Estas variantes, además de otras (descritas a continuación), representan modificaciones a la técnica fundamental del Hi-C estándar y abordan y alivian una o más limitaciones del método original.
In situ Hi-C combina Hi-C estándar con ensayo de ligadura nuclear, es decir, ligadura de proximidad realizada en núcleos intactos. [14] [21] El protocolo es similar al Hi-C estándar en términos del esquema de flujo de trabajo básico pero difiere en otros aspectos. [14] In situ Hi-C requiere de 2 a 5 millones de células en comparación con los 20 a 25 millones ideales requeridos para Hi-C estándar y requiere solo 3 días para completar el protocolo versus 7 días para Hi-C estándar. [14] Además, la ligadura de proximidad no se lleva a cabo en solución como en Hi-C estándar, disminuyendo la frecuencia de contactos y ligaduras aleatorios, biológicamente irrelevantes, como lo indica la menor frecuencia de contactos de ADN mitocondrial y nuclear en ADN biotinilado capturado. [14] Esto se logra dejando los núcleos intactos para el paso de ligadura. [14] Las células todavía se lisan con un tampón que contiene Tris-HCl a pH 8,0, cloruro de sodio y el detergente IGEPAL CA630 antes de la ligadura, pero en lugar de homogeneizar el lisado celular, los núcleos celulares se sedimentan después de la lisis inicial para degradar la membrana celular. [14] Una vez completada la ligadura de proximidad, los núcleos celulares se incuban durante al menos 1,5 horas a 68 grados Celsius para permeabilizar la membrana nuclear y liberar su contenido nuclear. [14]
La resolución que se puede lograr con Hi-C in situ puede ser de hasta 950 a 1000 pb en comparación con la resolución de 1 a 10 Mb de Hi-C estándar y la resolución de 100 kb de DNase Hi-C. [3] [4] [14] [19] Mientras que Hi-C estándar hace uso de un cortador de 6 pb como HindIII para el paso de digestión de restricción, Hi-C in situ utiliza un cortador de 4 pb como MboI o su isoesquizómero DpnII (que no es sensible a la metilación de CpG ) para aumentar la eficiencia y la resolución (ya que los sitios de restricción de MboI y DpnII ocurren con mayor frecuencia en el genoma). [3] [4] [14] Los datos entre réplicas para Hi-C in situ son consistentes y altamente reproducibles, con muy poco ruido de fondo y demostrando interacciones claras de la cromatina. [3] [14] Sin embargo, es posible que algunas de las interacciones capturadas no sean interacciones intermoleculares precisas, ya que el núcleo está densamente lleno de proteínas y ADN, por lo que realizar ligaduras de proximidad en núcleos intactos puede generar interacciones confusas que solo pueden formarse debido a la naturaleza del empaquetamiento nuclear y no tanto a interacciones cromosómicas únicas con impacto funcional celular. [3] [14] También requiere una profundidad de secuenciación extremadamente alta de alrededor de 5 mil millones de lecturas de extremos emparejados por muestra para lograr la resolución de los datos descrita por Rao et al. [3] [14] [22] Existen varias técnicas que han adaptado el concepto de Hi-C in situ, incluidas Sis Hi-C, OCEAN-C y captura in situ Hi-C. [3] A continuación se describen dos de las técnicas basadas en Hi-C in situ más destacadas. [3]
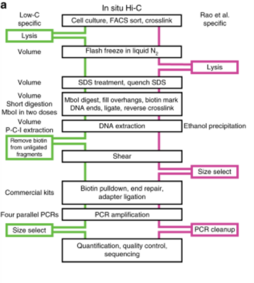
Low-C es un protocolo Hi-C in situ adaptado para su uso en números bajos de células, lo que es particularmente útil en contextos donde el número de células es un agente limitante, por ejemplo, en cultivos primarios de células humanas. [23] Este método hace uso de cambios menores, incluidos los volúmenes y concentraciones utilizados y el tiempo y orden de ciertos pasos experimentales para permitir la generación de bibliotecas Hi-C de alta calidad a partir de números de células tan bajos como 1000 células. [23] A pesar del potencial de generar datos utilizables y de alta resolución con tan solo 1000 células, Diaz et al. aún recomiendan usar al menos 1 a 2 millones de células si es posible, o si no un mínimo de 500 K células. [23] La calidad de la biblioteca se evaluó primero en la plataforma Illumina MiSeq (lecturas de extremos emparejados de 2x84 np) y una vez que pasó los criterios de control de calidad (incluidos los duplicados de PCR bajos), la biblioteca se secuenció en Illumina NextSeq (lecturas de extremos emparejados de 2x80 pb). [23] En general, esta técnica evita el problema de requerir una gran cantidad de células para Hi-C y la alta profundidad de secuenciación requerida para obtener datos de alta resolución, pero solo puede lograr resoluciones de hasta 5 kb y puede no ser siempre reproducible debido a la naturaleza variable de los tamaños de muestra utilizados y los datos generados a partir de ellos. [23]
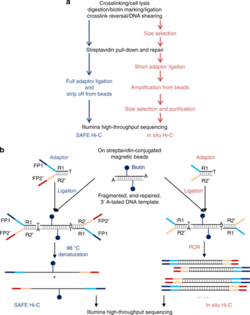
SAFE Hi-C, o Hi-C simplificado, rápido y económicamente eficiente, genera suficientes fragmentos ligados sin amplificación para una secuenciación de alto rendimiento. [17] Los datos in situ de Hi-C que se han publicado indican que la amplificación (en el paso de PCR para la preparación de la biblioteca) introduce un sesgo de amplificación dependiente de la distancia, lo que da como resultado una mayor relación ruido-señal frente a la distancia genómica. [17] SAFE Hi-C se utilizó con éxito para generar una biblioteca de ligadura Hi-C in situ sin amplificación a partir de tan solo 250 mil células K562 . [17] Los fragmentos de ligadura tienen entre 200 y 500 pb de longitud, con un promedio de aproximadamente 370 pb. [17] Todas las bibliotecas de productos de ligadura se secuenciaron utilizando la plataforma Illumina HiSeq (lecturas de extremos emparejados de 2x150 pb). [17] Aunque SAFE Hi-C se puede utilizar para una entrada de células tan baja como 250 mil, Niu et al. Se recomienda utilizar de 1 a 2 millones de células. [17] Las muestras producen suficientes ligantes para ser secuenciados en una cuarta parte de un carril. [17] Se ha demostrado que SAFE Hi-C aumenta la complejidad de la biblioteca debido a la eliminación de duplicados de PCR que reducen el porcentaje general de lecturas pareadas únicas. [17] En general, SAFE Hi-C preserva la integridad de las interacciones cromosómicas al mismo tiempo que reduce la necesidad de tener una alta profundidad de secuenciación y ahorra costos generales y mano de obra. [17]
Micro-C es una versión de Hi-C que incluye un paso de digestión con nucleasa microcócica (MNasa) para observar las interacciones entre pares de nucleosomas , lo que permite la resolución de estructuras TAD subgenómicas a escala de 1 a 100 nucleosomas. [24] [25] Primero se desarrolló para su uso en levadura y se demostró que conservaba los datos estructurales obtenidos de un Hi-C estándar pero con una mayor relación señal-ruido. [24] [25] Cuando se usó con células madre embrionarias humanas y fibroblastos , se obtuvieron de 2.6 a 4.5 mil millones de lecturas mapeadas de forma única por muestra. [24] [25] Hsieh et al. analizaron 2.64 mil millones de lecturas de células madre embrionarias de ratón y demostraron que había un mayor poder para detectar interacciones de corto alcance. [24] [25] [26]

Hi-C también se ha adaptado para su uso con células individuales, pero estas técnicas requieren altos niveles de experiencia para su realización y están plagadas de problemas como baja calidad de datos, cobertura y resolución. [3]
Los productos de ligación de ADN quimérico generados por Hi-C representan interacciones de cromatina por pares o contactos físicos 3D dentro del núcleo, [1] [2] [3] [4] y pueden analizarse mediante una variedad de enfoques posteriores. Brevemente, los datos de secuenciación profunda se utilizan para construir mapas imparciales de interacción de cromatina en todo el genoma. [3] [4] [27] [28] [29] [30] Luego, se pueden emplear varios métodos diferentes para analizar estos mapas para identificar patrones estructurales cromosómicos y sus interpretaciones biológicas. Muchos de estos enfoques de análisis de datos también se aplican a la secuenciación 3C u otros datos equivalentes.
Los datos Hi-C producidos por secuenciación profunda tienen la forma de un archivo FASTQ tradicional , y las lecturas se pueden alinear con el genoma de interés utilizando un software de alineación de secuencias (por ejemplo, Bowtie , [31] bwa, [9] [32] etc.). [27] [28] Debido a que los productos de ligadura Hi-C pueden abarcar cientos de megabases y pueden unir loci en diferentes cromosomas, [3] [4] [27] [28] la alineación de lecturas Hi-C a menudo es quimérica en el sentido de que diferentes partes de una lectura pueden alinearse con loci distantes, posiblemente en diferentes orientaciones. Los alineadores de lectura larga (por ejemplo, minimap2 [33] ) a menudo admiten la alineación quimérica y se pueden aplicar directamente a datos Hi-C de lectura larga. La alineación Hi-C de lectura corta es más desafiante.
En particular, Hi-C genera uniones de ligadura de tamaños variables, pero no se mide la posición exacta del sitio de ligadura. [3] [4] [27] Para evitar este problema, se utiliza el mapeo iterativo [27] para evitar la búsqueda del sitio de unión antes de poder dividir las lecturas en dos y mapearlas por separado para identificar los pares de interacción. La idea detrás del mapeo iterativo es mapear una secuencia lo más corta posible para asegurar la identificación única de los pares de interacción antes de llegar al sitio de unión. [27] [28] Como resultado, las lecturas de 25 pb de longitud que comienzan desde el extremo 5' se mapean al genoma primero, y las lecturas que no se mapean de manera única a un solo loci se extienden por 5 pb adicionales y luego se vuelven a mapear. [27] Este proceso se repite hasta que todas las lecturas se mapean de manera única, o hasta que las lecturas se extienden a su totalidad. [27] [28] Solo se mantienen las lecturas de extremos pareados con cada lado mapeado de manera única a un solo loci genómico. [28] Se descartan todas las demás lecturas de extremos emparejados.
En muchos procesos bioinformáticos se implementan varias variaciones de técnicas de mapeo de lecturas, como ICE, [34] HiC-Pro, [35] HIPPIE, [36] HiCUP, [37] y TADbit, [38] para mapear dos porciones de una lectura de extremo emparejado por separado, en el caso de que las dos porciones coincidan con posiciones genómicas distintas, abordando así el desafío donde las lecturas abarcan las uniones de ligadura. [28]
Con una mayor longitud de lectura, los procesos más recientes (por ejemplo, Juicer [39] y el portal de datos 4D-Nucleosome [40] ) a menudo alinean lecturas cortas de Hi-C con un algoritmo de alineación capaz de realizar alineamiento quimérico, como bwa-mem, [41] chromap [42] y dragmap. Este procedimiento llama a la alineación una vez y es más simple que el mapeo iterativo.
A cada una de las lecturas mapeadas se le asigna una única ubicación de alineación genómica de acuerdo con su posición mapeada 5' en el genoma. [27] Para cada par de lecturas, se asigna una ubicación a solo uno de los fragmentos de restricción , por lo tanto, debe caer en proximidad cercana a un sitio de restricción y a menos de la longitud máxima de la molécula. [27] [28] Las lecturas mapeadas a más de la longitud máxima de la molécula de los sitios de restricción más cercanos son el resultado de la rotura física de la cromatina o de actividades de nucleasas no canónicas. [27] Debido a que estas lecturas también instruyen información sobre las interacciones de la cromatina, no se descartan, pero se debe realizar un filtrado apropiado después de asignar ubicaciones genómicas para eliminar el ruido técnico en el conjunto de datos. [27] [28] [29] [30]
Dependiendo de si el par de lecturas cae dentro del mismo fragmento de restricción o de diferentes, se aplican diferentes criterios de filtrado. Si las lecturas emparejadas se asignan al mismo fragmento de restricción, es probable que representen extremos colgantes no ligados o fragmentos circularizados que no son informativos y, por lo tanto, se eliminan del conjunto de datos. [27] [28] Estas lecturas también podrían representar artefactos de PCR, fragmentos de cromatina no digeridos o, simplemente, lecturas con baja calidad de alineación. [8] [28] Cualquiera sea su origen, las lecturas asignadas al mismo fragmento se consideran "señales espurias" [28] y, por lo general, se descartan antes del procesamiento posterior.
Las lecturas emparejadas restantes asignadas a fragmentos de restricción distintos también se filtran para descartar productos de PCR idénticos/redundantes, y esto se logra eliminando las lecturas que comparten exactamente la misma secuencia o posiciones de alineación 5'. [27] También se podrían aplicar niveles adicionales de filtrado para adaptarse al propósito experimental. Por ejemplo, los posibles sitios de restricción no digeridos podrían filtrarse específicamente, en lugar de identificarse pasivamente, eliminando las lecturas asignadas a la misma cadena cromosómica con una pequeña distancia (definida por el usuario, basada en la experiencia) entre ellos. [27]

Basándose en sus coordenadas de punto medio, los fragmentos de restricción Hi-C se agrupan en intervalos genómicos fijos, con tamaños de bin que van desde 40 kb a 1 Mb. [27] La razón detrás de este enfoque es que al reducir la complejidad de los datos y disminuir el número de interacciones candidatas a nivel de genoma por bin, los bins genómicos permiten la construcción de señales más robustas y menos ruidosas, en forma de frecuencias de contacto, a expensas de la resolución (aunque la longitud del fragmento de restricción sigue siendo el límite físico definitivo para la resolución de Hi-C). [27] [28] Las interacciones de bin a bin se agregan simplemente tomando la suma, aunque también se han desarrollado métodos más centrados e informativos a lo largo de los años para mejorar aún más la señal. [27] Uno de estos métodos descrito por Rao et al. tiene como objetivo ampliar el límite del tamaño de bin a bins cada vez más pequeños, hasta tener eventualmente > 80% de bins cubiertos por 1000 lecturas cada uno, lo que aumentó significativamente la resolución de los resultados del análisis final. [14]
El filtrado a nivel de bin, al igual que el filtrado a nivel de fragmento, también se lleva a cabo para eliminar artefactos experimentales de los datos obtenidos. Los bins con alto ruido y bajas señales se eliminan, ya que generalmente representan contenidos genómicos altamente repetitivos alrededor de los telómeros y centrómeros . [27] Esto se hace comparando las sumas de bins individuales con la suma de todos los bins y eliminando el 1% inferior de bins, o utilizando la varianza como una medida de ruido. [27] Los bins de baja cobertura, o bins tres desviaciones estándar por debajo del centro de una distribución log-normal (que se ajusta al número total de contactos por bin genómico), se eliminan utilizando el filtro MAD-max (máxima desviación absoluta mediana permitida). [43] [44] Después de la clasificación, los datos Hi-C se almacenarán en un formato de matriz simétrica. [27] [28] [29] [30]
Más recientemente, se han propuesto muchos enfoques para predeterminar el tamaño óptimo de bin para diferentes experimentos Hi-C. Li et al. en 2018 describieron deDoc, un método en el que el tamaño de bin se selecciona como aquel en el que la entropía estructural de la matriz Hi-C alcanza un mínimo estable. [45] QuASAR, por otro lado, ofrece un poco más de evaluación de la calidad y compara las puntuaciones de las réplicas de las muestras (dado que las réplicas se incluyen de hecho para el propósito experimental) para encontrar la resolución máxima utilizable. [46] Algunas publicaciones [8] [47] también intentaron puntuar las frecuencias de interacción a nivel de fragmento único, donde se puede lograr una cobertura mayor incluso con un menor número de lecturas. HiCPlus, [48] una herramienta desarrollada por Zhang et al. en 2018, puede imputar matrices Hi-C similares a las originales utilizando solo 1/16 de las lecturas originales. [48]
El balanceo se refiere al proceso de corrección de sesgo de los datos Hi-C obtenidos, y puede ser explícito o implícito. [27] [28] Los métodos de balanceo explícito requieren las definiciones explícitas de sesgos que se sabe que están asociados con las lecturas Hi-C (o cualquier técnica de secuenciación de alto rendimiento en general), incluida la capacidad de mapeo de lectura, el contenido de GC , así como la longitud de fragmentos individuales. [27] [28] Primero se calcula un factor de corrección para cada uno de los sesgos considerados, seguido de cada una de sus combinaciones, y luego se aplica a los recuentos de lectura por bin genómico. [27] [28]
Sin embargo, algunos sesgos pueden provenir de un origen desconocido, en cuyo caso se utiliza en su lugar un enfoque de equilibrio implícito. El equilibrio implícito se basa en el supuesto de que cada locus genómico debe tener "igual visibilidad", lo que sugiere que la señal de interacción en cada locus genómico en los datos Hi-C debe sumar la misma cantidad total. [28] Un enfoque llamado corrección iterativa utiliza el algoritmo de equilibrio Sinkhorn-Knopp [49] e intenta equilibrar la matriz simétrica utilizando el supuesto mencionado anteriormente (igualando la suma de todas y cada una de las filas y columnas de la matriz). [27] [28] [49] El algoritmo alterna iterativamente entre dos pasos: 1) dividir cada fila por su media, y 2) dividir cada columna por su media, que se garantiza que convergen al final y no dejan filas o columnas obviamente altas en la matriz de interacción. [27] [49] También existen otros métodos computacionales para normalizar los sesgos inherentes a los datos Hi-C, incluyendo la normalización de componentes secuenciales (SCN), [50] el enfoque de balanceo de matriz de Knight-Ruiz, [14] [51] y la normalización de descomposición de vectores propios (ICE). [34] Al final, tanto los métodos de corrección de sesgo explícitos como los implícitos producen resultados comparables. [27]
Con una matriz de interacción agrupada a nivel del genoma, es posible identificar e interpretar biológicamente los patrones de interacción comunes observados en genomas de mamíferos, mientras que los patrones más raros y menos frecuentemente observados, como los cromosomas circulares y la agrupación de centrómeros, pueden requerir métodos adicionales especialmente diseñados para su identificación.
Las interacciones cis / trans son uno de los dos patrones de interacción más fuertes observados en los mapas Hi-C. [27] No son específicas de locus y, por lo tanto, se consideran un patrón a nivel de genoma. [27] Por lo general, se observa una frecuencia de interacción más alta, en promedio, para pares de loci que residen en el mismo cromosoma (en cis) que para pares de loci que residen en cromosomas diferentes (en trans). [27] En las matrices de interacción Hi-C, las interacciones cis/trans aparecen como bloques cuadrados centrados a lo largo de una diagonal, que coinciden con los cromosomas individuales al mismo tiempo. [27] Debido a que este patrón es relativamente consistente en diferentes especies y tipos de células, se puede utilizar para evaluar la calidad de los datos. Un experimento más ruidoso, debido a la ligadura de fondo aleatoria o cualquier factor desconocido, dará como resultado una relación de interacción cis a trans más baja (ya que se espera que el ruido afecte tanto a las interacciones cis como a las trans en una medida similar), y los experimentos de alta calidad suelen tener una relación de interacción cis/trans entre 40 y 60 para el genoma humano. [27]
Este patrón se refiere a la disminución dependiente de la distancia de las frecuencias de interacción a nivel del genoma, y representa el segundo de los dos patrones de interacción Hi-C más fuertes. [27] A medida que las frecuencias de interacción entre los loci que interactúan en cis disminuyen (como resultado de una mayor distancia entre ellos), se puede observar una disminución gradual de la frecuencia de interacción alejándose de la diagonal en la matriz de interacción. [27]
Existen varios modelos de polímeros [52] [53] para caracterizar estadísticamente las propiedades de pares de loci separados por una distancia dada, pero la clasificación discreta y el ajuste de funciones continuas son dos formas comunes de analizar las frecuencias de interacción dependientes de la distancia entre puntos de datos. [27] Primero, las frecuencias de interacción se pueden clasificar en función de su distancia genómica, luego se ajusta una función continua a los datos utilizando información del promedio de cada clasificación. [27] La función de decaimiento resultante se grafica en un gráfico logarítmico-logarítmico para que se pueda usar una línea lineal para representar los decaimientos de la ley de potencia predichos por los modelos de polímeros. [52] [53] Sin embargo, a menudo un modelo de polímero simple no será suficiente para representar completamente las frecuencias de interacción dependientes de la distancia, en cuyo punto pueden resultar funciones de decaimiento más complicadas, lo que puede afectar la reproducibilidad de los datos debido a la presencia de patrones específicos del locus en lugar de patrones de todo el genoma observados en la matriz Hi-C (que no son tomados en cuenta por los modelos de polímeros). [27] [52] [53]
El patrón específico de locus más fuerte que se encuentra en los mapas Hi-C son los compartimentos de cromatina, [1] que toman la forma de un patrón de cuadros o de “tablero de ajedrez” en la matriz de interacción, con bloques alternados que varían entre 1 y 10 Mb de tamaño (lo que los hace fáciles de extraer incluso en experimentos con un muestreo muy bajo) en el genoma humano. [27] [28] [30] Este patrón se puede encontrar tanto en frecuencias altas como bajas. Debido a que los cromosomas constan de dos tipos de regiones genómicas que se alternan a lo largo de la longitud de los cromosomas individuales, las frecuencias de interacción entre dos regiones del mismo tipo y las frecuencias de interacción entre dos regiones de diferentes tipos pueden ser bastante diferentes. [27] [28]
La definición de los compartimentos de cromatina activa (A) e inactiva (B) se basa en el análisis de componentes principales , establecido por primera vez por Lieberman-Aiden et al. en 2009. [1] [27] [28] [30] Su enfoque calculó la correlación de la matriz Hi-C de la relación de la señal observada frente a la esperada (obtenida de una matriz de contacto normalizada por distancia), y utilizó el signo del primer vector propio para denotar las partes positivas y negativas del gráfico resultante como compartimentos A y B, respectivamente. [1] [27] [28] [30] Muchos estudios genómicos han indicado que los compartimentos de cromatina están correlacionados con los estados de la cromatina, como la densidad genética , la accesibilidad del ADN, el contenido de GC, el tiempo de replicación y las marcas de histonas . [1] [27] [28 ] [30] Por lo tanto, los compartimentos de tipo A se definen más específicamente para representar las regiones densas en genes de la eucromatina , mientras que los compartimentos de tipo B representan regiones heterocromáticas con menos actividades genéticas. [27] [28] [30] En general, los compartimentos de cromatina ofrecen información sobre los principios generales de organización del genoma de interés.
Durante la última década se han desarrollado cada vez más herramientas bioinformáticas capaces de realizar llamadas de compartimentos, entre las que se incluyen HOMER, [54] HiTC R, [35] y CscoreTool. [55] Aunque cada una tiene sus propias diferencias y optimizaciones realizadas en el enfoque original de 2009, sus protocolos básicos aún se basan en el análisis de componentes principales.
Los TAD son estructuras sub-Mb que pueden albergar características reguladoras de genes, como interacciones promotor - potenciador locales. [27] De manera más general, los TAD se consideran una propiedad emergente de los mecanismos biológicos subyacentes, lo que define a los TAD como extrusiones de bucle, compartimentación o cualquier patrón genómico dinámico en lugar de una característica estructural estática del genoma. [56] Por lo tanto, los TAD representan microambientes reguladores y generalmente aparecen en un mapa Hi-C como bloques de regiones altamente autointeractuantes en las que las frecuencias de interacción dentro de la región son significativamente más altas que las frecuencias de interacción entre dos regiones adyacentes. [27] [28] [30] En las matrices de interacción Hi-C, los TAD son bloques cuadrados de frecuencias de interacción elevadas centradas a lo largo de la diagonal. [27] Sin embargo, esta es simplemente una descripción simplificada, e identificar el patrón real requiere mucho más procesamiento y estimación estadística.
Un enfoque para identificar los TAD fue descrito por Dixon et al., [9] donde primero calcularon (dentro de un rango genómico) la diferencia entre las interacciones promedio aguas arriba y las interacciones promedio aguas abajo de cada bin en la matriz. [9] Esta diferencia luego se transformó en una estadística de chi-cuadrado basada en el Modelo Oculto de Markov , y cualquier cambio brusco en este valor de chi-cuadrado, llamado índice de direccionalidad, definirá los límites de los TAD. [9] [27] Alternativamente, uno podría simplemente tomar la relación entre las interacciones promedio aguas arriba y aguas abajo para definir los límites de los TAD, como lo hicieron Naumova et al. [57]
Otro enfoque es calcular las frecuencias de interacción promedio que cruzan cada bin, nuevamente dentro de un rango genómico predeterminado. [27] [28] [58] El valor resultante se conoce como puntaje de aislamiento y puede considerarse como el promedio de un cuadrado que se desliza a lo largo de la diagonal de la matriz (Crane et al.). [58] Se espera que este valor sea menor en los límites de TAD; por lo tanto, se pueden usar técnicas estadísticas estándar para encontrar mínimos locales (límites) y definir regiones entre límites consecutivos como TAD. [27] [28] [58]
Sin embargo, como se reconoce cada vez más hoy en día, los TAD representan una serie jerárquica de estructuras que no se pueden caracterizar completamente mediante puntuaciones unidimensionales dadas por los métodos anteriores. [28] La mayor resolución disponible en los conjuntos de datos más nuevos ahora puede abordar explícitamente los TAD con enfoques de análisis multiescala. Como lo introdujo por primera vez Armatus, [59] se pueden identificar dominios específicos de resolución y se puede calcular un conjunto de consenso de dominios conservados en todas las resoluciones, [28] [59] lo que transforma el problema de la llamada de TAD en la optimización de funciones de puntuación basadas en sus densidades de interacción local. [59] También se desarrollan variaciones de este enfoque con diferentes funciones objetivo, como Lavaburst, [60] MrTADFinder, [61] 3DNetMod, [62] y Matryoshka, [63] para lograr un mejor rendimiento computacional en conjuntos de datos de mayor resolución.
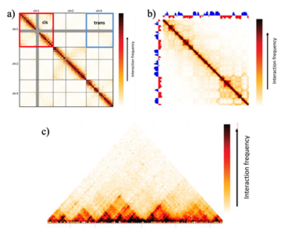
Biológicamente, las interacciones reguladoras suelen ocurrir a una escala mucho menor que los TAD, y dos elementos genómicos pueden activar/inhibir la expresión de un gen dentro de una distancia tan pequeña como 1 kb. [27] Por lo tanto, las interacciones puntuales son importantes para interpretar los mapas Hi-C, y se espera que aparezcan como enriquecimientos locales en la probabilidad de contacto. [27] [28] Sin embargo, las metodologías actuales para la identificación de interacciones puntuales son todas implícitas en la naturaleza, en el sentido de que no instruyen cómo debería lucir una interacción puntual. [27] [28] En cambio, las mutaciones puntuales se identifican como valores atípicos con frecuencias de interacción más altas de lo esperado dentro de la matriz Hi-C, dado que el modelo de fondo consta solo de las señales más fuertes, como las funciones de decaimiento de la distancia. [27] [28] El modelo de fondo se puede estimar y construir utilizando distribuciones de señales locales y enfoques globales (es decir, a nivel de cromosoma/genoma). [28] Muchos de los paquetes de bioinformática mencionados anteriormente incorporan algoritmos para identificar interacciones puntuales. En resumen, se calcula la importancia de la interacción individual por pares y los valores atípicos significativamente altos se corrigen mediante múltiples pruebas antes de que se los reconozca como interacciones puntuales verdaderamente informativas. [27] Es útil complementar las interacciones puntuales identificadas con evidencia adicional, como el análisis de los puntajes de enriquecimiento y las réplicas biológicas, para indicar que estas interacciones son realmente de importancia biológica. [27]
Hi-C puede revelar cambios en la conformación de la cromatina durante la división celular. En la interfase , las cromatinas son generalmente sueltas y vivaces para que pueda tener lugar la regulación de la transcripción y otras actividades reguladoras. [64] Al entrar en mitosis y división celular, las cromatinas se pliegan de forma compacta en cromosomas cilíndricos densos. [64] En los últimos cinco años, el desarrollo de Hi-C de una sola célula ha permitido la representación de todo el paisaje estructural 3D de las cromatinas/cromosomas a lo largo del ciclo celular , y muchos estudios han descubierto que estos dominios genómicos identificados permanecen sin cambios en la interfase y se borran mediante mecanismos de silenciamiento cuando la célula entra en mitosis. [65] [66] Cuando se completa la división mitótica y la célula vuelve a entrar en la interfase, se observa que las estructuras 3D de la cromatina se restablecen y se restaura la regulación de la transcripción. [65]
Se ha sospechado que la diferenciación de células madre embrionarias (ESC) y células madre pluripotentes inducidas (iPSC) en varios linajes de células maduras está acompañada de cambios globales en las estructuras cromosómicas y, en consecuencia, en la dinámica de interacción para permitir la regulación de la activación/silenciamiento transcripcional. [3] Se puede utilizar Hi-C estándar para investigar esta pregunta de investigación.
En 2015, Dixon et al. [11] aplicaron Hi-C estándar para capturar la dinámica 3D global en células madre embrionarias humanas durante su diferenciación en células high five . Debido a la capacidad de Hi-C para representar interacciones dinámicas en TAD relacionadas con la diferenciación, los investigadores descubrieron aumentos en la cantidad de sitios DHS, capacidad de unión de CTCF , modificaciones activas de histonas y expresiones de genes diana dentro de estos TAD de interés, y encontraron una participación significativa de los principales factores de pluripotencia como OCT4 , NANOG y SOX2 en la red de interacción durante la reprogramación de células somáticas . [11] Desde entonces, Hi-C ha sido reconocido como uno de los métodos estándar para investigar las actividades reguladoras de la transcripción y ha confirmado que la arquitectura cromosómica está estrechamente relacionada con el destino celular. [11] [67]
El crecimiento y desarrollo somático de los mamíferos comienza con la fertilización del espermatozoide y el ovocito , seguido por la etapa de cigoto , la etapa de 2 células, 4 células y 8 células, la etapa de blastocisto y finalmente la etapa de embrión . [68] Hi-C hizo posible explorar la arquitectura genómica integral durante el crecimiento y el desarrollo, ya que tanto sis-Hi-C [69] como in situ Hi-C [70] han informado que los TAD y los compartimentos A y B genómicos no están obviamente presentes y parecen estar menos bien estructurados en las células de ovocito. [69] [70] Estas características estructurales de la cromatina solo se establecen gradualmente desde frecuencias más débiles hasta puntos de datos más limpios y frecuentes después de la fertilización, a medida que progresan las etapas de desarrollo. [69] [70]
A medida que los datos sobre las estructuras del genoma 3D se vuelven cada vez más frecuentes en los últimos años, Hi-C comienza a usarse como un medio para rastrear características/cambios estructurales evolutivos. Los polimorfismos de nucleótido único (SNP) genómicos y los TAD generalmente se conservan entre especies, [71] junto con el factor CTCF en la evolución del dominio de la cromatina. [72] Sin embargo, las técnicas Hi-C han revelado que otros factores experimentan evoluciones estructurales en la arquitectura 3D. Estos incluyen la similitud de frecuencia de uso de codones (CUFS), [73] la corregulación de genes parálogos, [74] y los módulos ortólogos que coevolucionan espacialmente (SCOM). [75] Para la evolución de dominios a gran escala, las translocaciones cromosómicas , las regiones sinténicas, así como las regiones de reordenamiento genómico, se conservaron relativamente. [2] [67] [72] [76] [77] Estos hallazgos implican que las tecnologías Hi-C son capaces de proporcionar un punto de vista alternativo en el árbol de la vida eucariota . [3]
Varios estudios han empleado el uso de Hi-C para describir y estudiar la arquitectura de la cromatina en diferentes cánceres y su impacto en la patogénesis de la enfermedad. Kloetgen et al. utilizaron Hi-C in situ para estudiar la leucemia linfoblástica aguda de células T (T-ALL) y encontraron un evento de fusión TAD que eliminó un sitio de aislamiento CTCF, lo que permitió que el promotor del oncogén MYC interactuara directamente con un superpotenciador distal . [78] Fang et al. también han demostrado cómo hay ganancia o pérdida específica de aislamiento de cromatina de T-ALL, lo que altera la fuerza de la arquitectura TAD del genoma, utilizando Hi-C in situ. [79] Low-C se ha utilizado para mapear la estructura de la cromatina de células B primarias de un paciente con linfoma difuso de células B grandes y se utilizó para encontrar una alta variación estructural cromosómica entre el paciente y las células B sanas. [23] En general, la aplicación de Hi-C y sus variantes en la investigación del cáncer proporciona una visión única de los fundamentos moleculares de los factores impulsores de la anomalía celular. [23] [78] [79] Puede ayudar a explicar fenómenos biológicos (alta expresión de MYC en LLA-T) y ayudar al desarrollo de fármacos dirigidos a mecanismos exclusivos de las células cancerosas. [23] [78] [79]
{{cite book}}: |journal=ignorado ( ayuda ){{cite journal}}: Requiere citar revista |journal=( ayuda )