Las proteínas de hierro-azufre son proteínas caracterizadas por la presencia de cúmulos de hierro-azufre que contienen centros de di-, tri- y tetrahierro unidos a sulfuro en estados de oxidación variables . Los cúmulos de hierro-azufre se encuentran en una variedad de metaloproteínas , como las ferredoxinas , así como en la NADH deshidrogenasa , las hidrogenasas , la coenzima Q-citocromo c reductasa , la succinato-coenzima Q reductasa y la nitrogenasa . [1] Los cúmulos de hierro-azufre son más conocidos por su papel en las reacciones de oxidación-reducción del transporte de electrones en mitocondrias y cloroplastos . Tanto el complejo I como el complejo II de la fosforilación oxidativa tienen múltiples cúmulos de Fe–S. Tienen muchas otras funciones, incluida la catálisis , como lo ilustra la aconitasa , la generación de radicales, como lo ilustran las enzimas dependientes de SAM , y como donantes de azufre en la biosíntesis de ácido lipoico y biotina . Además, algunas proteínas de Fe–S regulan la expresión genética. Las proteínas Fe–S son vulnerables al ataque del óxido nítrico biogénico , que forma complejos de hierro dinitrosílico . En la mayoría de las proteínas Fe–S, los ligandos terminales del Fe son tiolatos , pero existen excepciones. [2]
La prevalencia de estas proteínas en las vías metabólicas de la mayoría de los organismos conduce a teorías de que los compuestos de hierro y azufre tuvieron un papel importante en el origen de la vida en la teoría del mundo hierro-azufre .
En algunos casos, los grupos Fe–S son inactivos en términos redox, pero se propone que tienen funciones estructurales. Algunos ejemplos son la endonucleasa III y MutY. [3] [4]
En casi todas las proteínas Fe–S, los centros Fe son tetraédricos y los ligandos terminales son centros de azufre tiolato de residuos de cisteinilo. Los grupos sulfuro tienen coordinación bi o tricoordinada. Los tres tipos distintos de grupos Fe–S con estas características son los más comunes.
Las proteínas de hierro y azufre participan en varios procesos biológicos de transporte de electrones, como la fotosíntesis y la respiración celular, que requieren una transferencia rápida de electrones para satisfacer las necesidades energéticas o bioquímicas del organismo. Para cumplir con sus diversas funciones biológicas, las proteínas de hierro y azufre efectúan transferencias rápidas de electrones y abarcan todo el rango de potenciales redox fisiológicos, desde -600 mV hasta +460 mV.
Los enlaces Fe 3+ -SR tienen una covalencia inusualmente alta, lo cual es de esperar. [ ¿según quién? ] Al comparar la covalencia de Fe 3+ con la covalencia de Fe 2+ , Fe 3+ tiene casi el doble de covalencia de Fe 2+ (20% a 38,4%). [5] Fe 3+ también está mucho más estabilizado que Fe 2+ . Los iones duros como Fe 3+ normalmente tienen una covalencia baja debido al desajuste de energía del orbital molecular desocupado más bajo del metal con el orbital molecular ocupado más alto del ligando .
Las moléculas de agua externas ubicadas cerca del sitio activo de hierro-azufre reducen la covalencia; esto se puede demostrar mediante experimentos de liofilización en los que se elimina el agua de la proteína. Esta reducción se debe a que el agua externa forma enlaces de hidrógeno con la cisteína S, lo que disminuye la donación de electrones de pares solitarios de esta última al Fe 3+/2+ al retirar los electrones S. [5] Dado que la covalencia estabiliza más al Fe 3+ que al Fe 2+ , el Fe 3+ se desestabiliza más por los enlaces de hidrógeno HOH-S.
Las energías orbitales 3d de Fe 3+ siguen el esquema de enlace "invertido" que fortuitamente tiene los orbitales d de Fe 3+ estrechamente emparejados en energía con los orbitales 3p del azufre, lo que da una alta covalencia en el orbital molecular de enlace resultante. [3] Esta alta covalencia reduce la energía de reorganización de la esfera interna [3] y, en última instancia, contribuye a una rápida transferencia de electrones.

El sistema polimetálico más simple, el grupo [Fe 2 S 2 ], está constituido por dos iones de hierro unidos por dos iones de sulfuro y coordinados por cuatro ligandos cisteinilo (en las ferredoxinas Fe 2 S 2 ) o por dos cisteínas y dos histidinas (en las proteínas de Rieske ). Las proteínas oxidadas contienen dos iones Fe 3+ , mientras que las proteínas reducidas contienen un ion Fe 3+ y un ion Fe 2+ . Estas especies existen en dos estados de oxidación, (Fe III ) 2 y Fe III Fe II . El dominio de azufre de hierro de CDGSH también está asociado con grupos 2Fe-2S.

Las proteínas de Rieske contienen grupos de Fe–S que se coordinan como una estructura 2Fe–2S y se pueden encontrar en el complejo III del citocromo bc1 unido a la membrana en las mitocondrias de eucariotas y bacterias. También son parte de las proteínas del cloroplasto, como el complejo del citocromo b 6 f en los organismos fotosintéticos. Estos organismos fotosintéticos incluyen plantas, algas verdes y cianobacterias , el precursor bacteriano de los cloroplastos. Ambos son parte de la cadena de transporte de electrones de sus respectivos organismos, que es un paso crucial en la recolección de energía para muchos organismos. [6]
Un motivo común presenta cuatro iones de hierro y cuatro iones de sulfuro colocados en los vértices de un grupo de tipo cubano . Los centros de Fe suelen estar coordinados además por ligandos de cisteinilo. Las proteínas de transferencia de electrones [Fe 4 S 4 ] ( ferredoxinas [Fe 4 S 4 ] ) pueden subdividirse en ferredoxinas de bajo potencial (tipo bacteriano) y de alto potencial (HiPIP) . Las ferredoxinas de bajo y alto potencial están relacionadas por el siguiente esquema redox:
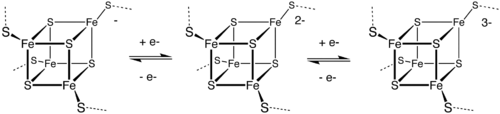
En HiPIP, el grupo oscila entre [2Fe 3+ , 2Fe 2+ ] (Fe 4 S 4 2+ ) y [3Fe 3+ , Fe 2+ ] (Fe 4 S 4 3+ ). Los potenciales para este par redox varían de 0,4 a 0,1 V. En las ferredoxinas bacterianas, el par de estados de oxidación son [Fe 3+ , 3Fe 2+ ] (Fe 4 S 4 + ) y [2Fe 3+ , 2Fe 2+ ] (Fe 4 S 4 2+ ). Los potenciales para este par redox varían de −0,3 a −0,7 V. Las dos familias de grupos 4Fe–4S comparten el estado de oxidación Fe 4 S 4 2+ . La diferencia en los pares redox se atribuye al grado de enlace de hidrógeno, que modifica fuertemente la basicidad de los ligandos de tiolato de cisteinilo. [ cita requerida ] Otro par redox, que es aún más reductor que las ferredoxinas bacterianas, está implicado en la nitrogenasa .
Algunos grupos 4Fe–4S se unen a sustratos y, por lo tanto, se clasifican como cofactores enzimáticos. En la aconitasa , el grupo Fe–S se une al aconitato en el único centro Fe que carece de un ligando tiolato. El grupo no experimenta redox, pero sirve como catalizador de ácido de Lewis para convertir el citrato en isocitrato . En las enzimas SAM radicales , el grupo se une y reduce la S-adenosilmetionina para generar un radical, que participa en muchas biosíntesis. [7]

El segundo cubano mostrado aquí con pares de valencia mixtos (2 Fe3+ y 2 Fe2+), tiene una mayor estabilidad gracias a la comunicación covalente y una fuerte deslocalización covalente del electrón “extra” del Fe2+ reducido que resulta en un acoplamiento ferromagnético completo.
También se sabe que las proteínas contienen centros [Fe 3 S 4 ], que presentan un hierro menos que los núcleos [Fe 4 S 4 ] más comunes. Tres iones de sulfuro unen dos iones de hierro cada uno, mientras que el cuarto sulfuro une tres iones de hierro. Sus estados de oxidación formales pueden variar de [Fe 3 S 4 ] + (forma todo Fe 3+ ) a [Fe 3 S 4 ] 2− (forma todo Fe 2+ ). En varias proteínas de hierro-azufre, el grupo [Fe 4 S 4 ] se puede convertir reversiblemente por oxidación y pérdida de un ion de hierro en un grupo [Fe 3 S 4 ]. Por ejemplo, la forma inactiva de la aconitasa posee un [Fe 3 S 4 ] y se activa mediante la adición de Fe 2+ y reductor.
Los ejemplos incluyen los sitios activos de varias enzimas:

La biosíntesis de los grupos Fe–S ha sido bien estudiada. [15] [16] [17] La biogénesis de los grupos de hierro y azufre se ha estudiado más extensamente en las bacterias E. coli y A. vinelandii y la levadura S. cerevisiae . Hasta ahora se han identificado al menos tres sistemas biosintéticos diferentes, a saber, los sistemas nif, suf e isc, que se identificaron por primera vez en bacterias. El sistema nif es responsable de los grupos en la enzima nitrogenasa. Los sistemas suf e isc son más generales.
El sistema isc de la levadura es el mejor descrito. Varias proteínas constituyen la maquinaria biosintética a través de la vía isc. El proceso ocurre en dos pasos principales: (1) el grupo Fe/S se ensambla en una proteína de andamiaje seguido de (2) la transferencia del grupo preformado a las proteínas receptoras. El primer paso de este proceso ocurre en el citoplasma de los organismos procariotas o en las mitocondrias de los organismos eucariotas . En los organismos superiores, los grupos son transportados fuera de la mitocondria para ser incorporados a las enzimas extramitocondriales. Estos organismos también poseen un conjunto de proteínas involucradas en los procesos de transporte e incorporación de grupos Fe/S que no son homólogas a las proteínas que se encuentran en los sistemas procariotas.
Holm y sus colaboradores fueron los primeros en informar sobre análogos sintéticos de los grupos Fe–S que se producen en la naturaleza . [ 18 ] El tratamiento de sales de hierro con una mezcla de tiolatos y sulfuro produce derivados como ( Et4N ) 2Fe4S4 (SCH2Ph ) 4 ] . [19] [ 20 ]
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace ){{cite book}}: CS1 maint: varios nombres: lista de autores ( enlace )