La secuenciación del ADN es el proceso de determinar la secuencia del ácido nucleico : el orden de los nucleótidos en el ADN . Incluye cualquier método o tecnología que se utilice para determinar el orden de las cuatro bases: adenina , guanina , citosina y timina . La llegada de métodos rápidos de secuenciación de ADN ha acelerado enormemente la investigación y los descubrimientos biológicos y médicos. [1] [2]
El conocimiento de las secuencias de ADN se ha vuelto indispensable para la investigación biológica básica, los proyectos genográficos de ADN y en numerosos campos aplicados como el diagnóstico médico , la biotecnología , la biología forense , la virología y la sistemática biológica . La comparación de secuencias de ADN sanas y mutadas puede diagnosticar diferentes enfermedades, incluidos varios cánceres, [3] caracterizar el repertorio de anticuerpos, [4] y puede usarse para guiar el tratamiento del paciente. [5] Tener una forma rápida de secuenciar el ADN permite administrar una atención médica más rápida e individualizada, y identificar y catalogar más organismos. [4]
La rápida velocidad de secuenciación lograda con la tecnología moderna de secuenciación de ADN ha sido fundamental para la secuenciación de secuencias completas de ADN, o genomas , de numerosos tipos y especies de vida, incluido el genoma humano y otras secuencias completas de ADN de muchos animales, plantas y microbios. especies.

Las primeras secuencias de ADN fueron obtenidas a principios de los años 1970 por investigadores académicos utilizando laboriosos métodos basados en la cromatografía bidimensional . Tras el desarrollo de métodos de secuenciación basados en fluorescencia con un secuenciador de ADN , [6] la secuenciación de ADN se ha vuelto más fácil y mucho más rápida. [7] [8]
La secuenciación de ADN se puede utilizar para determinar la secuencia de genes individuales , regiones genéticas más grandes (es decir, grupos de genes u operones ), cromosomas completos o genomas completos de cualquier organismo. La secuenciación de ADN es también la forma más eficaz de secuenciar indirectamente ARN o proteínas (a través de sus marcos de lectura abiertos ). De hecho, la secuenciación de ADN se ha convertido en una tecnología clave en muchas áreas de la biología y otras ciencias como la medicina, la ciencia forense y la antropología .
La secuenciación se utiliza en biología molecular para estudiar los genomas y las proteínas que codifican. La información obtenida mediante secuenciación permite a los investigadores identificar cambios en genes y ADN no codificante (incluidas secuencias reguladoras), asociaciones con enfermedades y fenotipos, e identificar posibles objetivos farmacológicos.
Dado que el ADN es una macromolécula informativa en términos de transmisión de una generación a otra, la secuenciación del ADN se utiliza en biología evolutiva para estudiar cómo se relacionan los diferentes organismos y cómo evolucionaron. En febrero de 2021, los científicos informaron, por primera vez, de la secuenciación de ADN de restos de animales , en este caso un mamut , de más de un millón de años, el ADN más antiguo secuenciado hasta la fecha. [9] [10]
El campo de la metagenómica implica la identificación de organismos presentes en una masa de agua, aguas residuales , suciedad, desechos filtrados del aire o muestras de hisopos de organismos. Saber qué organismos están presentes en un entorno particular es fundamental para la investigación en ecología , epidemiología , microbiología y otros campos. La secuenciación permite a los investigadores determinar qué tipos de microbios pueden estar presentes en un microbioma , por ejemplo.
Como la mayoría de los virus son demasiado pequeños para ser vistos con un microscopio óptico, la secuenciación es una de las principales herramientas en virología para identificar y estudiar el virus. [11] Los genomas virales pueden estar basados en ADN o ARN. Los virus de ARN requieren más tiempo para la secuenciación del genoma, ya que se degradan más rápido en muestras clínicas. [12] La secuenciación tradicional de Sanger y la secuenciación de próxima generación se utilizan para secuenciar virus en la investigación básica y clínica, así como para el diagnóstico de infecciones virales emergentes, la epidemiología molecular de patógenos virales y las pruebas de resistencia a los medicamentos. Hay más de 2,3 millones de secuencias virales únicas en GenBank . [11] Recientemente, NGS ha superado al Sanger tradicional como el enfoque más popular para generar genomas virales. [11]
Durante el brote de influenza aviar de 1990, la secuenciación viral determinó que el subtipo de influenza se originó a través del recombinamiento entre codornices y aves de corral. Esto llevó a que en Hong Kong se aprobara una legislación que prohibía la venta conjunta de codornices y aves de corral vivas en el mercado. La secuenciación viral también se puede utilizar para estimar cuándo comenzó un brote viral mediante una técnica de reloj molecular . [12]
Los técnicos médicos pueden secuenciar genes (o, teóricamente, genomas completos) de pacientes para determinar si existe riesgo de enfermedades genéticas. Esta es una forma de prueba genética , aunque es posible que algunas pruebas genéticas no impliquen la secuenciación del ADN.
La secuenciación del ADN también se utiliza cada vez más para diagnosticar y tratar enfermedades raras. A medida que se identifican más y más genes que causan enfermedades genéticas raras, los diagnósticos moleculares para los pacientes se vuelven más comunes. La secuenciación de ADN permite a los médicos identificar enfermedades genéticas, mejorar el manejo de las enfermedades, brindar asesoramiento reproductivo y terapias más efectivas. [13]
Además, la secuenciación de ADN puede ser útil para determinar una bacteria específica, para permitir tratamientos con antibióticos más precisos , reduciendo así el riesgo de crear resistencia a los antimicrobianos en las poblaciones de bacterias. [14] [15] [16] [17] [18] [19]
La secuenciación de ADN se puede utilizar junto con métodos de elaboración de perfiles de ADN para la identificación forense [20] y pruebas de paternidad . Las pruebas de ADN han evolucionado enormemente en las últimas décadas para, en última instancia, vincular una huella de ADN con lo que se está investigando. Los patrones de ADN en las huellas dactilares, la saliva, los folículos pilosos, etc. separan de manera única a cada organismo vivo de otro. La prueba de ADN es una técnica que puede detectar genomas específicos en una cadena de ADN para producir un patrón único e individualizado.
La estructura canónica del ADN tiene cuatro bases: timina (T), adenina (A), citosina (C) y guanina (G). La secuenciación de ADN es la determinación del orden físico de estas bases en una molécula de ADN. Sin embargo, hay muchas otras bases que pueden estar presentes en una molécula. En algunos virus (específicamente, bacteriófagos ), la citosina puede ser reemplazada por hidroximetilo o hidroximetilglucosacitosina. [21] En el ADN de los mamíferos, se pueden encontrar bases variantes con grupos metilo o fosfosulfato. [22] [23] Dependiendo de la técnica de secuenciación, una modificación particular, por ejemplo, el 5mC ( 5 metil citosina ) común en humanos, puede detectarse o no. [24]
En casi todos los organismos, el ADN se sintetiza in vivo utilizando únicamente las 4 bases canónicas; La modificación que se produce después de la replicación crea otras bases como 5 metil C. Sin embargo, algunos bacteriófagos pueden incorporar una base no estándar directamente. [25]
Además de las modificaciones, el ADN está bajo constante ataque de agentes ambientales como los rayos UV y los radicales de oxígeno. En la actualidad, la mayoría de los métodos de secuenciación de ADN no detectan la presencia de dichas bases dañadas, aunque PacBio ha publicado en este https://www.pacb.com/publications/direct-detection-and-sequencing-of-damged- bases-adn/
El ácido desoxirribonucleico ( ADN ) fue descubierto y aislado por primera vez por Friedrich Miescher en 1869, pero permaneció poco estudiado durante muchas décadas porque se pensaba que las proteínas, más que el ADN, eran las que mantenían el modelo genético de la vida. Esta situación cambió después de 1944 como resultado de algunos experimentos realizados por Oswald Avery , Colin MacLeod y Maclyn McCarty que demostraron que el ADN purificado podía transformar una cepa de bacterias en otra. Esta fue la primera vez que se demostró que el ADN era capaz de transformar las propiedades de las células.
En 1953, James Watson y Francis Crick propusieron su modelo de ADN de doble hélice , basado en estructuras cristalizadas de rayos X que estaba estudiando Rosalind Franklin . Según el modelo, el ADN se compone de dos cadenas de nucleótidos enrollados entre sí, unidos por enlaces de hidrógeno y que van en direcciones opuestas. Cada cadena está compuesta por cuatro nucleótidos complementarios: adenina (A), citosina (C), guanina (G) y timina (T), con una A en una cadena siempre emparejada con una T en la otra, y una C siempre emparejada con G. Propusieron que dicha estructura permitía utilizar cada hebra para reconstruir la otra, una idea central para la transmisión de información hereditaria entre generaciones. [26]

Las bases para la secuenciación de proteínas fueron establecidas por primera vez por el trabajo de Frederick Sanger , quien en 1955 había completado la secuencia de todos los aminoácidos de la insulina , una pequeña proteína secretada por el páncreas. Esto proporcionó la primera evidencia concluyente de que las proteínas eran entidades químicas con un patrón molecular específico y no una mezcla aleatoria de material suspendido en un fluido. El éxito de Sanger en la secuenciación de la insulina estimuló a los cristalógrafos de rayos X, incluidos Watson y Crick, que ya estaban tratando de comprender cómo el ADN dirigía la formación de proteínas dentro de una célula. Poco después de asistir a una serie de conferencias impartidas por Frederick Sanger en octubre de 1954, Crick comenzó a desarrollar una teoría que sostenía que la disposición de los nucleótidos en el ADN determinaba la secuencia de aminoácidos en las proteínas, lo que a su vez ayudaba a determinar la función de una proteína. Publicó esta teoría en 1958. [27]
La secuenciación de ARN fue una de las primeras formas de secuenciación de nucleótidos. El principal hito de la secuenciación de ARN es la secuencia del primer gen completo y el genoma completo del bacteriófago MS2 , identificado y publicado por Walter Fiers y sus colaboradores en la Universidad de Gante ( Gante , Bélgica ), en 1972 [28] y 1976. [29] Los métodos tradicionales de secuenciación de ARN requieren la creación de una molécula de ADNc que debe secuenciarse. [30]
El primer método para determinar secuencias de ADN implicó una estrategia de extensión de cebadores de ubicación específica establecida por Ray Wu en la Universidad de Cornell en 1970. [31] Para secuenciar se utilizó catálisis de ADN polimerasa y marcaje de nucleótidos específico, los cuales ocupan un lugar destacado en los esquemas de secuenciación actuales. los extremos cohesivos del ADN del fago lambda. [32] [33] [34] Entre 1970 y 1973, Wu, R Padmanabhan y sus colegas demostraron que este método se puede emplear para determinar cualquier secuencia de ADN utilizando cebadores sintéticos específicos de ubicación. [35] [36] [8] Frederick Sanger luego adoptó esta estrategia de extensión de cebadores para desarrollar métodos de secuenciación de ADN más rápidos en el Centro MRC , Cambridge , Reino Unido y publicó un método para la "secuenciación de ADN con inhibidores de terminación de cadena" en 1977. [37] Walter Gilbert y Allan Maxam en Harvard también desarrollaron métodos de secuenciación, incluido uno para la "secuenciación del ADN por degradación química". [38] [39] En 1973, Gilbert y Maxam informaron de la secuencia de 24 pares de bases utilizando un método conocido como análisis de punto errante. [40] Los avances en la secuenciación se vieron favorecidos por el desarrollo simultáneo de la tecnología del ADN recombinante , que permitió aislar muestras de ADN de fuentes distintas a los virus.

El primer genoma de ADN completo que se secuenció fue el del bacteriófago φX174 en 1977. [41] Los científicos del Consejo de Investigación Médica descifraron la secuencia completa de ADN del virus de Epstein-Barr en 1984 y descubrieron que contenía 172.282 nucleótidos. La finalización de la secuencia marcó un punto de inflexión significativo en la secuenciación del ADN porque se logró sin conocimiento previo del perfil genético del virus. [42] [8]
A principios de la década de 1980, Herbert Pohl y sus colaboradores desarrollaron un método no radiactivo para transferir las moléculas de ADN de mezclas de reacción de secuenciación a una matriz inmovilizadora durante la electroforesis . [43] [44] Seguido de la comercialización del secuenciador de ADN "Direct-Blotting-Electrophoresis-System GATC 1500" de GATC Biotech , que se utilizó intensivamente en el marco del programa de secuenciación del genoma de la UE, se obtuvo la secuencia completa de ADN del Levadura Saccharomyces cerevisiae cromosoma II. [45] El laboratorio de Leroy E. Hood en el Instituto de Tecnología de California anunció la primera máquina de secuenciación de ADN semiautomática en 1986. [46] A esto le siguió la comercialización por parte de Applied Biosystems de la primera máquina de secuenciación totalmente automatizada, la ABI 370. , en 1987 y por Génesis 2000 de Dupont [47] que utilizó una nueva técnica de etiquetado fluorescente que permitía identificar los cuatro didesoxinucleótidos en un solo carril. En 1990, los Institutos Nacionales de Salud (NIH) de Estados Unidos habían iniciado ensayos de secuenciación a gran escala de Mycoplasma capricolum , Escherichia coli , Caenorhabditis elegans y Saccharomyces cerevisiae a un costo de 0,75 dólares por base. Mientras tanto, en el laboratorio de Craig Venter comenzó la secuenciación de secuencias de ADNc humano llamadas etiquetas de secuencia expresadas , un intento de capturar la fracción codificante del genoma humano . [48] En 1995, Venter, Hamilton Smith y sus colegas del Instituto de Investigación Genómica (TIGR) publicaron el primer genoma completo de un organismo de vida libre, la bacteria Haemophilus influenzae . El cromosoma circular contiene 1.830.137 bases y su publicación en la revista Science [49] marcó el primer uso publicado de la secuenciación escopeta del genoma completo, eliminando la necesidad de esfuerzos iniciales de mapeo.
En 2001, se habían utilizado métodos de secuenciación rápida para producir un borrador de secuencia del genoma humano. [50] [51]

Se desarrollaron varios métodos nuevos para la secuenciación de ADN entre mediados y finales de la década de 1990 y se implementaron en secuenciadores de ADN comerciales en el año 2000. En conjunto, se denominaron métodos de secuenciación (NGS) de "próxima generación" o "segunda generación", para distinguirlos. a partir de los métodos anteriores, incluida la secuenciación de Sanger . A diferencia de la primera generación de secuenciación, la tecnología NGS se caracteriza típicamente por ser altamente escalable, lo que permite secuenciar todo el genoma de una vez. Por lo general, esto se logra fragmentando el genoma en pedazos pequeños, tomando muestras aleatorias de un fragmento y secuenciandolo usando una de una variedad de tecnologías, como las que se describen a continuación. Un genoma completo es posible porque se secuencian múltiples fragmentos a la vez (lo que le da el nombre de secuenciación "masivamente paralela") en un proceso automatizado.
La tecnología NGS ha empoderado enormemente a los investigadores para buscar ideas sobre la salud, a los antropólogos para investigar los orígenes humanos y está catalizando el movimiento de la " Medicina Personalizada ". Sin embargo, también ha abierto la puerta a más margen de error. Existen muchas herramientas de software para llevar a cabo el análisis computacional de datos NGS, a menudo compilados en plataformas en línea como CSI NGS Portal, cada una con su propio algoritmo. Incluso los parámetros dentro de un paquete de software pueden cambiar el resultado del análisis. Además, las grandes cantidades de datos producidos por la secuenciación del ADN también han requerido el desarrollo de nuevos métodos y programas para el análisis de secuencias. Se han intentado varios esfuerzos para desarrollar estándares en el campo NGS para abordar estos desafíos, la mayoría de los cuales han sido esfuerzos a pequeña escala surgidos de laboratorios individuales. Más recientemente, un gran esfuerzo organizado y financiado por la FDA culminó en el estándar BioCompute .
El 26 de octubre de 1990, Roger Tsien , Pepi Ross, Margaret Fahnestock y Allan J. Johnston presentaron una patente que describe la secuenciación por pasos ("base por base") con bloqueadores 3' removibles en matrices de ADN (transferencias y moléculas de ADN individuales). [53] En 1996, Pål Nyrén y su alumno Mostafa Ronaghi en el Real Instituto de Tecnología de Estocolmo publicaron su método de pirosecuenciación . [54]
El 1 de abril de 1997, Pascal Mayer y Laurent Farinelli presentaron patentes a la Organización Mundial de la Propiedad Intelectual que describen la secuenciación de colonias de ADN. [55] La preparación de muestras de ADN y los métodos de disposición aleatoria en cadena de la polimerasa de superficie (PCR) descritos en esta patente, junto con el método de secuenciación "base por base" de Roger Tsien et al., ahora se implementan en Illumina . Secuenciadores del genoma Hi-Seq.
En 1998, Phil Green y Brent Ewing de la Universidad de Washington describieron su puntuación de calidad phred para el análisis de datos de secuenciadores, [56] una técnica de análisis histórica que obtuvo una adopción generalizada y que sigue siendo la métrica más común para evaluar la precisión de una secuenciación. plataforma. [57]
Lynx Therapeutics publicó y comercializó la secuenciación masiva de firmas paralelas (MPSS) en 2000. Este método incorporó una tecnología de secuenciación basada en cuentas paralelizada, mediada por adaptador/ligación y sirvió como el primer método de secuenciación de "próxima generación" disponible comercialmente, aunque no Los secuenciadores de ADN se vendieron a laboratorios independientes. [58]
Allan Maxam y Walter Gilbert publicaron un método de secuenciación del ADN en 1977 basado en la modificación química del ADN y la posterior escisión en bases específicas. [38] También conocido como secuenciación química, este método permitió utilizar muestras purificadas de ADN bicatenario sin clonación adicional. El uso de etiquetado radiactivo en este método y su complejidad técnica desalentaron su uso extensivo después de que se realizaron mejoras en los métodos de Sanger.
La secuenciación de Maxam-Gilbert requiere un marcaje radiactivo en un extremo 5' del ADN y la purificación del fragmento de ADN que se va a secuenciar. Luego, el tratamiento químico genera roturas en una pequeña proporción de una o dos de las cuatro bases de nucleótidos en cada una de las cuatro reacciones (G, A+G, C, C+T). La concentración de los químicos modificadores se controla para introducir en promedio una modificación por molécula de ADN. De este modo se genera una serie de fragmentos marcados, desde el extremo radiomarcado hasta el primer sitio "cortado" de cada molécula. Los fragmentos de las cuatro reacciones se someten a electroforesis uno al lado del otro en geles desnaturalizantes de acrilamida para la separación por tamaños. Para visualizar los fragmentos, el gel se expone a una película de rayos X para realizar una autorradiografía, lo que produce una serie de bandas oscuras, cada una de las cuales corresponde a un fragmento de ADN radiomarcado, a partir del cual se puede inferir la secuencia. [38]
Este método está prácticamente obsoleto a partir de 2023. [59]
El método de terminación de cadena desarrollado por Frederick Sanger y sus compañeros de trabajo en 1977 pronto se convirtió en el método elegido, debido a su relativa facilidad y confiabilidad. [37] [60] Cuando se inventó, el método terminador de cadena utilizaba menos sustancias químicas tóxicas y menores cantidades de radiactividad que el método de Maxam y Gilbert. Debido a su relativa facilidad, el método Sanger pronto se automatizó y fue el método utilizado en la primera generación de secuenciadores de ADN .
La secuenciación de Sanger es el método que prevaleció desde los años 1980 hasta mediados de los años 2000. Durante ese período, se lograron grandes avances en la técnica, como el etiquetado fluorescente, la electroforesis capilar y la automatización general. Estos avances permitieron una secuenciación mucho más eficiente, lo que generó costos más bajos. El método Sanger, en forma de producción en masa, es la tecnología que produjo el primer genoma humano en 2001, marcando el comienzo de la era de la genómica . Sin embargo, más adelante en la década, llegaron al mercado enfoques radicalmente diferentes, lo que redujo el costo por genoma de 100 millones de dólares en 2001 a 10 000 dólares en 2011. [61]
El objetivo de la secuenciación secuencial por síntesis (SBS) es determinar la secuenciación de una muestra de ADN mediante la detección de la incorporación de un nucleótido por una ADN polimerasa . Se utiliza una polimerasa diseñada para sintetizar una copia de una sola cadena de ADN y se monitorea la incorporación de cada nucleótido. El principio de secuenciación en tiempo real por síntesis se describió por primera vez en 1993 [62] y se publicaron mejoras algunos años después. [63] Las partes clave son muy similares para todas las realizaciones de SBS e incluyen (1) amplificación de ADN (para mejorar la señal posterior) y unir el ADN que se va a secuenciar a un soporte sólido, (2) generación de ADN monocatenario en el soporte sólido, (3) incorporación de nucleótidos usando una polimerasa diseñada y (4) detección en tiempo real de la incorporación de nucleótidos. Se repiten los pasos 3-4 y la secuencia se ensambla a partir de las señales obtenidas en el paso 4. Este principio de La secuenciación por síntesis en tiempo real se ha utilizado para casi todos los instrumentos de secuenciación paralela masiva , incluidos 454 , PacBio , IonTorrent , Illumina y MGI .
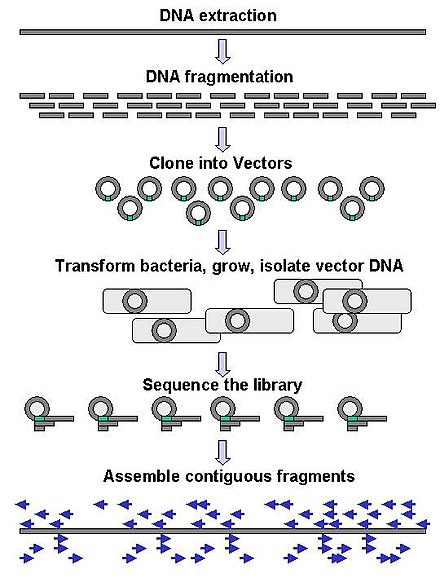
La secuenciación a gran escala a menudo tiene como objetivo secuenciar piezas de ADN muy largas, como cromosomas completos , aunque la secuenciación a gran escala también se puede utilizar para generar cantidades muy grandes de secuencias cortas, como las que se encuentran en la presentación en fagos . Para objetivos más largos, como los cromosomas, los enfoques comunes consisten en cortar (con enzimas de restricción ) o cortar (con fuerzas mecánicas) grandes fragmentos de ADN en fragmentos de ADN más cortos. El ADN fragmentado puede luego clonarse en un vector de ADN y amplificarse en un huésped bacteriano como Escherichia coli . Los fragmentos cortos de ADN purificados de colonias bacterianas individuales se secuencian individualmente y se ensamblan electrónicamente en una secuencia larga y contigua. Los estudios han demostrado que agregar un paso de selección de tamaño para recolectar fragmentos de ADN de tamaño uniforme puede mejorar la eficiencia de la secuenciación y la precisión del ensamblaje del genoma. En estos estudios, el dimensionamiento automatizado ha demostrado ser más reproducible y preciso que el dimensionamiento manual del gel. [64] [65] [66]
El término " secuenciación de novo " se refiere específicamente a métodos utilizados para determinar la secuencia de ADN sin una secuencia conocida previamente. De novo se traduce del latín como "desde el principio". Los huecos en la secuencia ensamblada pueden llenarse mediante el recorrido del cebador . Las diferentes estrategias tienen diferentes compensaciones en cuanto a velocidad y precisión; Los métodos de escopeta se utilizan a menudo para secuenciar genomas grandes, pero su ensamblaje es complejo y difícil, particularmente porque las repeticiones de secuencias a menudo causan brechas en el ensamblaje del genoma.
La mayoría de los enfoques de secuenciación utilizan un paso de clonación in vitro para amplificar moléculas de ADN individuales, porque sus métodos de detección molecular no son lo suficientemente sensibles para la secuenciación de una sola molécula. La PCR en emulsión [67] aísla moléculas de ADN individuales junto con perlas recubiertas con cebador en gotitas acuosas dentro de una fase oleosa. Luego, una reacción en cadena de la polimerasa (PCR) recubre cada perla con copias clonales de la molécula de ADN seguida de una inmovilización para su posterior secuenciación. La PCR en emulsión se utiliza en los métodos desarrollados por Marguilis et al. (comercializado por 454 Life Sciences ), Shendure y Porreca et al. (también conocida como " secuenciación polony ") y secuenciación SOLiD , (desarrollada por Agencourt , más tarde Applied Biosystems , ahora Life Technologies ). [68] [69] [70] La PCR en emulsión también se utiliza en las plataformas GemCode y Chromium desarrolladas por 10x Genomics . [71]
La secuenciación de escopeta es un método de secuenciación diseñado para el análisis de secuencias de ADN de más de 1000 pares de bases, hasta cromosomas completos inclusive. Este método requiere que el ADN objetivo se rompa en fragmentos aleatorios. Después de secuenciar fragmentos individuales utilizando el método de terminación de cadena , las secuencias se pueden volver a ensamblar en función de sus regiones superpuestas. [72]

La secuenciación de alto rendimiento, que incluye métodos de secuenciación de "lectura corta" de próxima generación y de "lectura larga" de tercera generación, [nt 1] se aplica a la secuenciación del exoma , la secuenciación del genoma, la resecuenciación del genoma, la elaboración de perfiles del transcriptoma ( RNA-Seq ), Interacciones ADN-proteína ( secuenciación ChIP ) y caracterización del epigenoma . [73]
La gran demanda de secuenciación de bajo costo ha impulsado el desarrollo de tecnologías de secuenciación de alto rendimiento que paralelizan el proceso de secuenciación y producen miles o millones de secuencias simultáneamente. [74] [75] [76] Las tecnologías de secuenciación de alto rendimiento tienen como objetivo reducir el costo de la secuenciación de ADN más allá de lo que es posible con los métodos estándar de terminación de colorante. [77] En la secuenciación de rendimiento ultraalto se pueden ejecutar en paralelo hasta 500.000 operaciones de secuenciación por síntesis. [78] [79] [80] Estas tecnologías permitieron secuenciar un genoma humano completo en tan solo un día. [81] A partir de 2019 [actualizar], los líderes corporativos en el desarrollo de productos de secuenciación de alto rendimiento incluyeron Illumina , Qiagen y ThermoFisher Scientific . [81]
La secuenciación SMRT se basa en el enfoque de secuenciación por síntesis. El ADN se sintetiza en guías de ondas de modo cero (ZMW), pequeños contenedores parecidos a pozos con las herramientas de captura ubicadas en el fondo del pozo. La secuenciación se realiza con el uso de polimerasa no modificada (unida al fondo del ZMW) y nucleótidos marcados con fluorescencia que fluyen libremente en la solución. Los pocillos están construidos de manera que sólo se detecta la fluorescencia que se produce en el fondo del pocillo. La etiqueta fluorescente se desprende del nucleótido tras su incorporación a la cadena de ADN, dejando una cadena de ADN sin modificar. Según Pacific Biosciences (PacBio), desarrollador de la tecnología SMRT, esta metodología permite detectar modificaciones de nucleótidos (como la metilación de citosina). Esto sucede mediante la observación de la cinética de la polimerasa. Este enfoque permite lecturas de 20.000 nucleótidos o más, con longitudes de lectura promedio de 5 kilobases. [88] [98] En 2015, Pacific Biosciences anunció el lanzamiento de un nuevo instrumento de secuenciación llamado Sequel System, con 1 millón de ZMW en comparación con 150 000 ZMW en el instrumento PacBio RS II. [99] [100] La secuenciación SMRT se conoce como secuenciación de " tercera generación " o de "lectura larga".
El ADN que pasa a través del nanoporo cambia su corriente iónica. Este cambio depende de la forma, el tamaño y la longitud de la secuencia de ADN. Cada tipo de nucleótido bloquea el flujo de iones a través del poro durante un período de tiempo diferente. El método no requiere nucleótidos modificados y se realiza en tiempo real. La secuenciación de nanoporos se conoce como secuenciación de " tercera generación " o de "lectura larga", junto con la secuenciación SMRT.
Las primeras investigaciones industriales sobre este método se basaron en una técnica llamada "secuenciación de exonucleasas", donde la lectura de señales eléctricas se producía cuando los nucleótidos pasaban por los poros de alfa (α) -hemolisina unidos covalentemente con ciclodextrina . [101] Sin embargo, el método comercial posterior, la 'secuenciación de hebras', secuenció bases de ADN en una hebra intacta.
Dos áreas principales de secuenciación de nanoporos en desarrollo son la secuenciación de nanoporos en estado sólido y la secuenciación de nanoporos basada en proteínas. La secuenciación de nanoporos de proteínas utiliza complejos de proteínas de membrana como la α-hemolisina, MspA ( Mycobacterium smegmatis Porin A) o CssG, que se muestran muy prometedores dada su capacidad para distinguir entre nucleótidos individuales y grupos. [102] Por el contrario, la secuenciación de nanoporos en estado sólido utiliza materiales sintéticos como nitruro de silicio y óxido de aluminio y se prefiere por su capacidad mecánica superior y su estabilidad térmica y química. [103] El método de fabricación es esencial para este tipo de secuenciación dado que la matriz de nanoporos puede contener cientos de poros con diámetros inferiores a ocho nanómetros. [102]
El concepto se originó a partir de la idea de que las moléculas de ADN o ARN monocatenarias pueden ser impulsadas electroforéticamente en una secuencia lineal estricta a través de un poro biológico que puede tener menos de ocho nanómetros, y pueden detectarse dado que las moléculas liberan una corriente iónica mientras se mueven a través del poro. El poro contiene una región de detección capaz de reconocer diferentes bases, generando cada base varias señales específicas en el tiempo correspondientes a la secuencia de bases a medida que cruzan el poro, que luego se evalúan. [103] El control preciso sobre el transporte de ADN a través del poro es crucial para el éxito. Se han utilizado varias enzimas, como exonucleasas y polimerasas, para moderar este proceso colocándolas cerca de la entrada del poro. [104]
La primera de las tecnologías de secuenciación de alto rendimiento, la secuenciación masiva de firmas paralelas (o MPSS), se desarrolló en la década de 1990 en Lynx Therapeutics, una empresa fundada en 1992 por Sydney Brenner y Sam Eletr . MPSS era un método basado en cuentas que utilizaba un enfoque complejo de ligación de adaptadores seguido de decodificación del adaptador, leyendo la secuencia en incrementos de cuatro nucleótidos. Este método lo hizo susceptible a sesgos específicos de secuencia o pérdida de secuencias específicas. Debido a que la tecnología era tan compleja, Lynx Therapeutics solo realizó MPSS "internamente" y no se vendieron máquinas de secuenciación de ADN a laboratorios independientes. Lynx Therapeutics se fusionó con Solexa (luego adquirida por Illumina ) en 2004, lo que llevó al desarrollo de la secuenciación por síntesis, un enfoque más simple adquirido de Manteia Predictive Medicine , que dejó obsoleto el MPSS. Sin embargo, las propiedades esenciales de la salida MPSS eran típicas de tipos de datos posteriores de alto rendimiento, incluidos cientos de miles de secuencias cortas de ADN. En el caso de MPSS, estos se utilizaron normalmente para secuenciar ADNc para medir los niveles de expresión génica . [58]
El método de secuenciación polony , desarrollado en el laboratorio de George M. Church en Harvard, fue uno de los primeros sistemas de secuenciación de alto rendimiento y se utilizó para secuenciar un genoma completo de E. coli en 2005. [105] Combinaba un método de secuenciación emparejado in vitro. biblioteca de etiquetas con PCR en emulsión, un microscopio automatizado y química de secuenciación basada en ligación para secuenciar un genoma de E. coli con una precisión de >99,9999% y un costo de aproximadamente 1/9 del de la secuenciación de Sanger. [105] La tecnología obtuvo la licencia de Agencourt Biosciences, posteriormente se dividió en Agencourt Personal Genomics y, finalmente, se incorporó a la plataforma SOLiD de Applied Biosystems . Posteriormente, Applied Biosystems fue adquirida por Life Technologies , ahora parte de Thermo Fisher Scientific .
454 Life Sciences , que desde entonces ha sido adquirida por Roche Diagnostics, desarrolló una versión paralelizada de pirosecuenciación . El método amplifica el ADN dentro de gotas de agua en una solución de aceite (PCR en emulsión), y cada gota contiene una única plantilla de ADN unida a una única perla recubierta con un cebador que luego forma una colonia clonal. La máquina de secuenciación contiene muchos pocillos de volumen de picolitros , cada uno de los cuales contiene una sola perla y enzimas de secuenciación. La pirosecuenciación utiliza luciferasa para generar luz para la detección de los nucleótidos individuales agregados al ADN naciente, y los datos combinados se utilizan para generar lecturas de secuencia . [68] Esta tecnología proporciona una longitud de lectura intermedia y un precio por base en comparación con la secuenciación Sanger en un extremo y Solexa y SOLiD en el otro. [77]
Solexa , ahora parte de Illumina , fue fundada por Shankar Balasubramanian y David Klenerman en 1998 y desarrolló un método de secuenciación basado en tecnología de terminadores de tinte reversibles y polimerasas diseñadas. [106] El concepto de química terminada reversible fue inventado por Bruno Canard y Simon Sarfati en el Instituto Pasteur de París. [107] [108] Fue desarrollado internamente en Solexa por aquellos nombrados en las patentes relevantes. En 2004, Solexa adquirió la empresa Manteia Predictive Medicine para obtener una tecnología de secuenciación paralela masiva inventada en 1997 por Pascal Mayer y Laurent Farinelli. [55] Se basa en "grupos de ADN" o "colonias de ADN", que implica la amplificación clonal del ADN en una superficie. La tecnología del clúster fue adquirida conjuntamente con Lynx Therapeutics de California. Solexa Ltd. posteriormente se fusionó con Lynx para formar Solexa Inc.


En este método, las moléculas de ADN y los cebadores se unen primero a un portaobjetos o a una celda de flujo y se amplifican con polimerasa para que se formen colonias de ADN clonal locales, que más tarde se denominarán "grupos de ADN". Para determinar la secuencia, se añaden cuatro tipos de bases terminadoras reversibles (bases RT) y los nucleótidos no incorporados se eliminan por lavado. Una cámara toma imágenes de los nucleótidos marcados con fluorescencia . Luego, el tinte, junto con el bloqueador terminal 3', se elimina químicamente del ADN, lo que permite que comience el siguiente ciclo. A diferencia de la pirosecuenciación, las cadenas de ADN se extienden un nucleótido a la vez y la adquisición de imágenes se puede realizar en un momento retrasado, lo que permite capturar conjuntos muy grandes de colonias de ADN mediante imágenes secuenciales tomadas con una sola cámara.

Desacoplar la reacción enzimática y la captura de imágenes permite un rendimiento óptimo y una capacidad de secuenciación teóricamente ilimitada. Con una configuración óptima, el rendimiento final alcanzable del instrumento viene dictado únicamente por la tasa de conversión de analógico a digital de la cámara, multiplicada por el número de cámaras y dividida por el número de píxeles por colonia de ADN necesarios para visualizarlas de forma óptima (aproximadamente 10 píxeles/colonia). En 2012, con cámaras funcionando a velocidades de conversión A/D de más de 10 MHz y sistemas ópticos, fluídicos y enzimáticos disponibles, el rendimiento puede ser múltiplos de 1 millón de nucleótidos/segundo, lo que corresponde aproximadamente a 1 equivalente de genoma humano con una cobertura de 1x por hora por instrumento. y 1 genoma humano resecuenciado (a aproximadamente 30x) por día por instrumento (equipado con una sola cámara). [109]
Este método es una modificación mejorada de la tecnología combinatoria de ligadura de anclaje de sonda (cPAL) descrita por Complete Genomics [110] , que desde 2013 pasó a formar parte de la empresa china de genómica BGI . [111] Las dos empresas han perfeccionado la tecnología para permitir lecturas más largas longitudes, reducciones del tiempo de reacción y tiempos más rápidos para obtener resultados. Además, los datos ahora se generan como lecturas completas contiguas en el formato de archivo FASTQ estándar y se pueden usar tal cual en la mayoría de los procesos de análisis bioinformáticos basados en lecturas cortas. [112] [ cita necesaria ]
Las dos tecnologías que forman la base de esta tecnología de secuenciación de alto rendimiento son las nanobolas de ADN (DNB) y las matrices estampadas para la unión de nanobolas a una superficie sólida. [110] Las nanobolas de ADN se forman simplemente desnaturalizando bibliotecas bicatenarias ligadas a adaptadores y ligando la cadena delantera solo a un oligonucleótido férula para formar un círculo de ADN monocatenario. Se producen copias fieles de los círculos que contienen el inserto de ADN utilizando Rolling Circle Amplification, que genera aproximadamente entre 300 y 500 copias. La larga hebra de ADNss se pliega sobre sí misma para producir una estructura de nanobola tridimensional de aproximadamente 220 nm de diámetro. La creación de DNB reemplaza la necesidad de generar copias de PCR de la biblioteca en la celda de flujo y, como tal, puede eliminar grandes proporciones de lecturas duplicadas, ligaduras de adaptador-adaptador y errores inducidos por la PCR. [112] [ cita necesaria ]

La matriz estampada de puntos cargados positivamente se fabrica mediante fotolitografía y técnicas de grabado, seguidas de modificación química para generar una celda de flujo de secuenciación. Cada punto de la celda de flujo tiene aproximadamente 250 nm de diámetro, está separado por 700 nm (de centro a centro) y permite conectar fácilmente un único DNB con carga negativa a la celda de flujo y, por lo tanto, reducir la acumulación excesiva o insuficiente en la celda de flujo. [110] [ cita necesaria ]
Luego, la secuenciación se realiza mediante la adición de una sonda oligonucleotídica que se une en combinación a sitios específicos dentro del DNB. La sonda actúa como un ancla que luego permite que uno de los cuatro nucleótidos etiquetados e inactivados de forma reversible se una después de fluir a través de la celda de flujo. Los nucleótidos libres se eliminan antes de la excitación láser de las etiquetas adheridas, luego emiten fluorescencia y la señal es capturada por cámaras que se convierten en una salida digital para la llamada de bases. La base adjunta tiene su terminador y etiqueta escindidos químicamente al finalizar el ciclo. El ciclo se repite con otro flujo de nucleótidos libres marcados a través de la celda de flujo para permitir que el siguiente nucleótido se una y capture su señal. Este proceso se completa varias veces (generalmente de 50 a 300 veces) para determinar la secuencia del fragmento de ADN insertado a una velocidad de aproximadamente 40 millones de nucleótidos por segundo a partir de 2018. [ cita necesaria ]


La tecnología SOLiD de Applied Biosystems (ahora una marca de Life Technologies ) emplea secuenciación por ligación . Aquí, se marca un conjunto de todos los oligonucleótidos posibles de una longitud fija según la posición secuenciada. Los oligonucleótidos se hibridan y ligan; la ligación preferencial por la ADN ligasa para emparejar secuencias da como resultado una señal informativa del nucleótido en esa posición. Cada base de la plantilla se secuencia dos veces y los datos resultantes se decodifican según el esquema de codificación de 2 bases utilizado en este método. Antes de la secuenciación, el ADN se amplifica mediante PCR en emulsión. Las perlas resultantes, cada una de las cuales contiene copias individuales de la misma molécula de ADN, se depositan en un portaobjetos de vidrio. [113] El resultado son secuencias de cantidades y longitudes comparables a la secuenciación de Illumina. [77] Se ha informado que este método de secuenciación por ligadura tiene algunos problemas al secuenciar secuencias palindrómicas. [96]
Ion Torrent Systems Inc. (ahora propiedad de Life Technologies ) desarrolló un sistema basado en el uso de química de secuenciación estándar, pero con un novedoso sistema de detección basado en semiconductores. Este método de secuenciación se basa en la detección de iones de hidrógeno que se liberan durante la polimerización del ADN , a diferencia de los métodos ópticos utilizados en otros sistemas de secuenciación. Un micropocillo que contiene una cadena de ADN molde que se va a secuenciar se inunda con un único tipo de nucleótido . Si el nucleótido introducido es complementario al nucleótido molde principal, se incorpora a la cadena complementaria en crecimiento. Esto provoca la liberación de un ion de hidrógeno que activa un sensor de iones hipersensible, lo que indica que se ha producido una reacción. Si hay repeticiones de homopolímero presentes en la secuencia plantilla, se incorporarán múltiples nucleótidos en un solo ciclo. Esto conduce a una cantidad correspondiente de hidrógenos liberados y a una señal electrónica proporcionalmente mayor. [114]

La secuenciación de nanobolas de ADN es un tipo de tecnología de secuenciación de alto rendimiento que se utiliza para determinar la secuencia genómica completa de un organismo. La empresa Complete Genomics utiliza esta tecnología para secuenciar muestras enviadas por investigadores independientes. El método utiliza la replicación de círculos rodantes para amplificar pequeños fragmentos de ADN genómico en nanobolas de ADN. A continuación se utiliza la secuenciación no encadenada por ligación para determinar la secuencia de nucleótidos. [115] Este método de secuenciación de ADN permite secuenciar una gran cantidad de nanobolas de ADN por ejecución y con bajos costos de reactivos en comparación con otras plataformas de secuenciación de alto rendimiento. [116] Sin embargo, solo se determinan secuencias cortas de ADN a partir de cada nanobola de ADN, lo que dificulta el mapeo de las lecturas cortas a un genoma de referencia . [115]
La secuenciación con heliscopio es un método de secuenciación de una sola molécula desarrollado por Helicos Biosciences . Utiliza fragmentos de ADN con adaptadores de cola poli-A añadidos que se unen a la superficie de la celda de flujo. Los siguientes pasos implican la secuenciación basada en extensión con lavados cíclicos de la celda de flujo con nucleótidos marcados con fluorescencia (un tipo de nucleótido a la vez, como con el método Sanger). Las lecturas las realiza el secuenciador Heliscope. [117] [118] Las lecturas son cortas, con un promedio de 35 pb. [119] Lo que hizo que esta tecnología fuera especialmente novedosa fue que fue la primera de su clase en secuenciar ADN no amplificado, evitando así cualquier error de lectura asociado con los pasos de amplificación. [120] En 2009 se secuenció un genoma humano utilizando el Heliscopio, sin embargo, en 2012 la empresa quebró. [121]
Hay dos sistemas de microfluidos principales que se utilizan para secuenciar el ADN; Microfluidos basados en gotas y microfluidos digitales . Los dispositivos de microfluidos resuelven muchas de las limitaciones actuales de las matrices de secuenciación actuales.
Abate et al. estudió el uso de dispositivos de microfluidos basados en gotas para la secuenciación de ADN. [4] Estos dispositivos tienen la capacidad de formar y procesar gotas del tamaño de un picolitro a una velocidad de miles por segundo. Los dispositivos se crearon a partir de polidimetilsiloxano (PDMS) y utilizaron ensayos FRET de transferencia de energía por resonancia de Forster para leer las secuencias de ADN contenidas en las gotitas. Cada posición en la matriz se probó para una secuencia específica de 15 bases. [4]
Justo y col. utilizó dispositivos de microfluidos digitales para estudiar la pirosecuenciación del ADN . [122] Las ventajas importantes incluyen la portabilidad del dispositivo, el volumen de reactivo, la velocidad de análisis, la capacidad de fabricación en masa y el alto rendimiento. Este estudio proporcionó una prueba de concepto que muestra que los dispositivos digitales se pueden utilizar para la pirosecuenciación; El estudio incluyó el uso de síntesis, que implica la extensión de las enzimas y la adición de nucleótidos marcados. [122]
Boles et al. También estudió la pirosecuenciación en dispositivos de microfluidos digitales. [123] Utilizaron un dispositivo de electrohumectación para crear, mezclar y dividir gotas. La secuenciación utiliza un protocolo de tres enzimas y plantillas de ADN ancladas con perlas magnéticas. El dispositivo se probó utilizando dos protocolos y obtuvo una precisión del 100 % según los niveles de pirogramos sin procesar. Las ventajas de estos dispositivos de microfluidos digitales incluyen tamaño, costo y niveles alcanzables de integración funcional. [123]
La investigación de secuenciación de ADN, que utiliza microfluidos, también tiene la capacidad de aplicarse a la secuenciación de ARN , utilizando técnicas de microfluidos de gotas similares, como el método inDrops. [124] Esto muestra que muchas de estas técnicas de secuenciación de ADN podrán aplicarse más y usarse para comprender más sobre los genomas y transcriptomas.
Los métodos de secuenciación de ADN actualmente en desarrollo incluyen la lectura de la secuencia a medida que una cadena de ADN transita a través de nanoporos (un método que ahora es comercial pero las generaciones posteriores, como los nanoporos de estado sólido, aún están en desarrollo), [125] [126] y técnicas basadas en microscopía. , como la microscopía de fuerza atómica o la microscopía electrónica de transmisión que se utilizan para identificar las posiciones de nucleótidos individuales dentro de fragmentos largos de ADN (>5000 pb) mediante el marcaje de nucleótidos con elementos más pesados (p. ej., halógenos) para detección y registro visual. [127] [128] Las tecnologías de tercera generación tienen como objetivo aumentar el rendimiento y disminuir el tiempo para obtener resultados y el costo eliminando la necesidad de reactivos excesivos y aprovechando la procesividad de la ADN polimerasa. [129]
Otro enfoque utiliza mediciones de las corrientes eléctricas de túnel a través del ADN monocatenario a medida que se mueve a través de un canal. Dependiendo de su estructura electrónica, cada base afecta la corriente de túnel de manera diferente, [130] permitiendo la diferenciación entre diferentes bases. [131]
El uso de corrientes de túnel tiene el potencial de secuenciar órdenes de magnitud más rápido que los métodos de corriente iónica y ya se ha logrado la secuenciación de varios oligómeros de ADN y microARN. [132]
La secuenciación por hibridación es un método no enzimático que utiliza un microarray de ADN . Un único conjunto de ADN cuya secuencia se va a determinar se marca con fluorescencia y se hibrida con una matriz que contiene secuencias conocidas. Las fuertes señales de hibridación procedentes de un punto determinado de la matriz identifican su secuencia en el ADN que se está secuenciando. [133]
Este método de secuenciación utiliza características de unión de una biblioteca de moléculas cortas de ADN monocatenario (oligonucleótidos), también llamadas sondas de ADN, para reconstruir una secuencia de ADN objetivo. Los híbridos no específicos se eliminan mediante lavado y se eluye el ADN diana. [134] Los híbridos se reorganizan de manera que se pueda reconstruir la secuencia de ADN. El beneficio de este tipo de secuenciación es su capacidad para capturar una gran cantidad de objetivos con una cobertura homogénea. [135] Generalmente se requiere una gran cantidad de productos químicos y ADN inicial. Sin embargo, con la llegada de la hibridación basada en soluciones, se necesitan muchos menos equipos y productos químicos. [134]
Se puede utilizar espectrometría de masas para determinar secuencias de ADN. La espectrometría de masas de tiempo de vuelo por ionización y desorción láser asistida por matriz, o MALDI-TOF MS , se ha investigado específicamente como un método alternativo a la electroforesis en gel para visualizar fragmentos de ADN. Con este método, los fragmentos de ADN generados por reacciones de secuenciación de terminación de cadena se comparan por masa en lugar de por tamaño. La masa de cada nucleótido es diferente de los demás y esta diferencia es detectable mediante espectrometría de masas. Las mutaciones de un solo nucleótido en un fragmento se pueden detectar más fácilmente con la EM que mediante electroforesis en gel únicamente. MALDI-TOF MS puede detectar más fácilmente diferencias entre fragmentos de ARN, por lo que los investigadores pueden secuenciar indirectamente el ADN con métodos basados en MS convirtiéndolo primero en ARN. [136]
La mayor resolución de los fragmentos de ADN permitida por los métodos basados en MS es de especial interés para los investigadores en ciencias forenses, ya que es posible que deseen encontrar polimorfismos de un solo nucleótido en muestras de ADN humano para identificar individuos. Estas muestras pueden estar muy degradadas, por lo que los investigadores forenses suelen preferir el ADN mitocondrial por su mayor estabilidad y sus aplicaciones para estudios de linaje. Se han utilizado métodos de secuenciación basados en MS para comparar las secuencias de ADN mitocondrial humano de muestras de una base de datos de la Oficina Federal de Investigaciones [137] y de huesos encontrados en fosas comunes de soldados de la Primera Guerra Mundial. [138]
Los métodos tempranos de terminación de cadena y TOF MS demostraron longitudes de lectura de hasta 100 pares de bases. [139] Los investigadores no han podido superar este tamaño de lectura promedio; Al igual que la secuenciación de terminación de cadena por sí sola, la secuenciación de ADN basada en MS puede no ser adecuada para grandes proyectos de secuenciación de novo . Aun así, un estudio reciente utilizó lecturas de secuencia corta y espectroscopia de masas para comparar polimorfismos de un solo nucleótido en cepas patógenas de Streptococcus . [140]
En la secuenciación de microfluidos de Sanger, toda la amplificación por termociclado de los fragmentos de ADN, así como su separación por electroforesis, se realiza en una sola oblea de vidrio (aproximadamente 10 cm de diámetro), lo que reduce el uso de reactivos y el costo. [141] En algunos casos, los investigadores han demostrado que pueden aumentar el rendimiento de la secuenciación convencional mediante el uso de microchips. [142] Aún será necesario realizar investigaciones para que este uso de la tecnología sea eficaz.
Este enfoque visualiza directamente la secuencia de moléculas de ADN mediante microscopía electrónica. La primera identificación de pares de bases de ADN dentro de moléculas de ADN intactas mediante la incorporación enzimática de bases modificadas, que contienen átomos de mayor número atómico, visualización directa e identificación de bases marcadas individualmente dentro de una molécula de ADN sintética de 3272 pares de bases y un genoma viral de 7249 pares de bases. ha sido demostrado. [143]
Este método se basa en el uso de ARN polimerasa (RNAP), que está unida a una perla de poliestireno . Un extremo del ADN que se va a secuenciar se une a otra perla y ambas perlas se colocan en trampas ópticas. El movimiento de RNAP durante la transcripción acerca las perlas y su distancia relativa cambia, lo que luego se puede registrar con una resolución de un solo nucleótido. La secuencia se deduce basándose en las cuatro lecturas con concentraciones reducidas de cada uno de los cuatro tipos de nucleótidos, de manera similar al método de Sanger. [144] Se realiza una comparación entre regiones y la información de secuencia se deduce comparando las regiones de secuencia conocidas con las regiones de secuencia desconocida. [145]
Se ha desarrollado un método para analizar conjuntos completos de interacciones de proteínas utilizando una combinación de pirosecuenciación 454 y un método de visualización de ARNm de virus in vitro . Específicamente, este método une covalentemente proteínas de interés a los ARNm que las codifican y luego detecta los fragmentos de ARNm mediante PCR de transcripción inversa . A continuación, el ARNm puede amplificarse y secuenciarse. El método combinado se tituló IVV-HiTSeq y se puede realizar en condiciones libres de células, aunque sus resultados pueden no ser representativos de las condiciones in vivo . [146]
Si bien hay muchas formas diferentes de secuenciar el ADN, sólo unas pocas dominan el mercado actual. Según esto, Illumina representa aproximadamente el 80% del mercado en 2022; el resto del mercado está en manos de unos pocos actores (PacBio, Oxford, 454, MGI) [147]
El éxito de cualquier protocolo de secuenciación de ADN depende de la extracción y preparación de la muestra de ADN o ARN a partir del material biológico de interés.
Después de la extracción de ADN o ARN, es posible que las muestras requieran una preparación adicional según el método de secuenciación. Para la secuenciación Sanger, se requieren procedimientos de clonación o PCR antes de la secuenciación. En el caso de los métodos de secuenciación de próxima generación, se requiere la preparación de la biblioteca antes del procesamiento. [149] La evaluación de la calidad y cantidad de ácidos nucleicos después de la extracción y después de la preparación de la biblioteca identifica muestras degradadas, fragmentadas y de baja pureza y produce datos de secuenciación de alta calidad. [150]
La naturaleza de alto rendimiento de las tecnologías actuales de secuenciación de ADN/ARN ha planteado un desafío para la ampliación del método de preparación de muestras. Se están utilizando varios instrumentos de manipulación de líquidos para la preparación de un mayor número de muestras con un tiempo práctico total menor:

En octubre de 2006, la Fundación X Prize estableció una iniciativa para promover el desarrollo de tecnologías de secuenciación completa del genoma , llamada Premio Archon X , con la intención de otorgar 10 millones de dólares al "primer equipo que pueda construir un dispositivo y utilizarlo para secuenciar 100 genomas humanos". en 10 días o menos, con una precisión de no más de un error por cada 100.000 bases secuenciadas, con secuencias que cubran con precisión al menos el 98% del genoma y con un costo recurrente de no más de 10.000 dólares (EE.UU.) por genoma". [151]
Cada año, el Instituto Nacional de Investigación del Genoma Humano , o NHGRI, promueve subvenciones para nuevas investigaciones y desarrollos en genómica . Las subvenciones de 2010 y los candidatos de 2011 incluyen trabajo continuo en metodologías de secuenciación de microfluidos, polonia y bases pesadas. [152]
Las tecnologías de secuenciación que se describen aquí producen datos sin procesar que deben ensamblarse en secuencias más largas, como genomas completos ( ensamblaje de secuencias ). Hay muchos desafíos computacionales para lograr esto, como la evaluación de los datos de secuencia sin procesar que se realiza mediante programas y algoritmos como Phred y Phrap . Otros desafíos tienen que ver con secuencias repetitivas que a menudo impiden ensamblajes completos del genoma porque ocurren en muchos lugares del genoma. Como consecuencia, es posible que muchas secuencias no se asignen a cromosomas concretos . La producción de datos de secuencia sin procesar es sólo el comienzo de su análisis bioinformático detallado. [153] Sin embargo, se desarrollaron nuevos métodos para secuenciar y corregir errores de secuenciación. [154]
A veces, las lecturas sin procesar producidas por el secuenciador son correctas y precisas sólo en una fracción de su longitud. El uso de la lectura completa puede introducir artefactos en los análisis posteriores, como el ensamblaje del genoma, la llamada de SNP o la estimación de la expresión genética. Se han introducido dos clases de programas de recorte, basados en clases de algoritmos basados en ventanas o de suma acumulada. [155] Esta es una lista parcial de los algoritmos de recorte disponibles actualmente, especificando la clase de algoritmo a la que pertenecen:
La genética humana ha sido incluida dentro del campo de la bioética desde principios de la década de 1970 [162] y el crecimiento en el uso de la secuenciación de ADN (particularmente la secuenciación de alto rendimiento) ha introducido una serie de cuestiones éticas. Una cuestión clave es la propiedad del ADN de un individuo y los datos producidos cuando se secuencia ese ADN. [163] Con respecto a la molécula de ADN en sí, el principal caso legal sobre este tema, Moore v. Regents de la Universidad de California (1990) dictaminó que los individuos no tienen derechos de propiedad sobre las células desechadas ni ningún beneficio obtenido utilizando estas células (por ejemplo, como línea celular patentada ). Sin embargo, las personas tienen derecho a obtener un consentimiento informado con respecto a la extracción y el uso de células. En cuanto a los datos producidos mediante la secuenciación del ADN, Moore no otorga al individuo ningún derecho sobre la información derivada de su ADN. [163]
A medida que la secuenciación del ADN se generaliza, el almacenamiento, la seguridad y el intercambio de datos genómicos también se han vuelto más importantes. [163] [164] Por ejemplo, una preocupación es que las aseguradoras puedan utilizar los datos genómicos de un individuo para modificar su cotización, dependiendo de la salud futura percibida del individuo en función de su ADN. [164] [165] En mayo de 2008, se firmó en los Estados Unidos la Ley de No Discriminación por Información Genética (GINA), que prohíbe la discriminación basada en información genética con respecto al seguro médico y el empleo. [166] [167] En 2012, la Comisión Presidencial de EE. UU. para el Estudio de Cuestiones Bioéticas informó que la legislación de privacidad existente para los datos de secuenciación de ADN, como GINA y la Ley de Responsabilidad y Portabilidad del Seguro Médico, eran insuficientes, y señaló que los datos de secuenciación del genoma completo eran insuficientes. particularmente sensible, ya que podría usarse para identificar no solo a la persona a partir de la cual se crearon los datos, sino también a sus familiares. [168] [169]
En la mayor parte de los Estados Unidos, el ADN "abandonado", como el que se encuentra en un sello o sobre lamido, en una taza de café, en un cigarrillo, en un chicle, en la basura doméstica o en el cabello caído en una acera pública, puede recolectarse legalmente. y secuenciados por cualquier persona, incluida la policía, investigadores privados, opositores políticos o personas involucradas en disputas de paternidad. En 2013, once estados tienen leyes que pueden interpretarse para prohibir el "robo de ADN". [170]
También han surgido cuestiones éticas por el uso cada vez mayor de pruebas de detección de variaciones genéticas, tanto en recién nacidos como en adultos, por parte de empresas como 23andMe . [171] [172] Se ha afirmado que la detección de variaciones genéticas puede ser dañina y aumentar la ansiedad en personas que tienen un mayor riesgo de enfermedad. [173] Por ejemplo, en un caso señalado en Time , los médicos que examinaban a un bebé enfermo en busca de variantes genéticas optaron por no informar a los padres sobre una variante no relacionada vinculada con la demencia debido al daño que causaría a los padres. [174] Sin embargo, un estudio de 2011 en The New England Journal of Medicine ha demostrado que las personas sometidas a un perfil de riesgo de enfermedad no mostraron mayores niveles de ansiedad. [173] Además, el desarrollo de tecnologías de secuenciación de próxima generación, como la secuenciación basada en nanoporos, también ha planteado más preocupaciones éticas. [175]
Las tecnologías de secuenciación de próxima generación (NGS) han revolucionado la investigación genómica. (frase inicial del artículo)
{{cite press release}}: |last=tiene nombre genérico ( ayuda )